通过转录组测序分析识别糖尿病患者冠状动脉粥样硬化的新途径
2019-04-25陈丁源
陈丁源
(青海省心脑血管病专科医院 急诊科,青海 西宁 810012)
心外膜脂肪组织(epicardial adipose tissus,EAT)是一种内脏脂肪组织,与冠状动脉和心肌紧密相邻,可分泌大量脂肪因子。EAT 被认为在冠状动脉疾病(coronary artery disease,CAD)的发生、发展中发挥作用[1]。EAT 可通过巨噬细胞活化、氧化应激,以及先天性炎症反应等参与CAD[2]。有研究表明,在脂肪细胞功能异常、内脏肥胖症以及糖尿病相关的冠状动脉粥样硬化间存在相互作用[3-4]。尽管如此,糖尿病对CAD 患者EAT 转录组的影响尚未得到很好的阐释。此外,之前的研究大多使用微阵列技术,而EAT的转录组测序(RNA-seq)分析在该研究为首次应用。本 研 究 对8 例CAD 和2 型 糖 尿 病(type 2 diabetes,T2DM)的受试者的EAT 进行RNA-Seq 分析,鉴定有差异表达基因,并通过实验验证氧化应激途径下有差异表达的基因。
1 资料与方法
1.1 样本收集
选取2015年6月—2016年1月青海省心脑血管病专科医院8 例接受心胸外科手术(冠状动脉旁路移植术或心脏瓣膜置换术)CAD 患者。冠状动脉粥样硬化性心脏病(以下简称冠心病)患者纳入标准[5]:①经冠状动脉造影检查确诊,符合WHO 制订的冠心病诊断指标;②血清TC>5.2 mmol/L 或LDL-C>3.1 mmol/L; ③按NYHA 心功能分级Ⅱ、Ⅳ级;④常见症状有胸闷、胸痛、心悸、乏力等。该8 例CAD 患者根据有无T2DM 将患者分为糖尿病组(D 组,n=5)和非糖尿病组(ND 组,n=3)。T2DM 患者纳入标准根据1999年世界卫生组织(WHO)诊断标准[6]:①糖尿病症状,空腹血糖≥7.0 mmol/L 或餐后2 h 血糖≥11.1 mmol/L; ②无糖尿病症状,空腹血糖≥7.0 mmol/L 或餐后2 h 血糖≥11.1 mmol/L,重复再测1 次,仍达以上值者;③无糖尿病症状,空腹血糖≥7.0 mmol/L 或餐后2 h 血糖≥11.1 mmol/L,糖耐量实验2 h 血糖≥ 11.1 mmol/L 者中,符合任意2 条标准或符合同一标准2 次者诊断为糖尿病。此研究由青海省心脑血管病专科医院医学伦理委员会批准,患者知情并签署知情同意书。本研究收治的T2DM 患者有糖尿病史,目前使用抗糖尿病药物(口服或注射用药)且血红蛋白A1c(HbA1c)≥6%。D 组5 例患者。男性2 例,女性3 例;年龄60 ~75 岁,平均(69.6± 5.81)岁,符合T2DM 条件;ND 组3 例患者。男性2例,女性1 例;年龄51 ~66 岁,平均(58.0±7.55)岁,既往有糖尿病或糖尿病前期病史,糖化血红蛋白≤ 5.7%且未服用抗糖尿病药物。心脏病专家根据临床病史,术前冠状动脉造影和其他常规检查确定CAD是否存在。发现60 ~75 岁的6 例患者具有CAD,而51 ~57 岁的2 例患者无CAD。基本资料见表1。对每位受试患者进行术前空腹抽血,检测其血糖、血红蛋白A1c(HbA1c)、丙氨酸氨基转移酶(ALT)和天冬氨酸转氨酶(AST)。ALT 被当作胰岛素抵抗的替代指标[7]。在患者右冠状动脉近端部分附近获得EAT样本,其深度至心包的内脏层。获得组织样本储存于液氮中直至提取RNA 进行测序处理。
1.2 脂肪RNA 提取
对收集的脂肪组织提取RNA。在2 ml TRI 试剂缓冲液中分离大约200 μg 脂肪组织。将样品在室温下温育5 min,并在4℃下12 377 r/min 离心10 min。除去脂肪单层,并将上清液收集到2 个1.5 ml 微量离心管中,每管中加入200 μl 氯仿(CHCl3)。通过涡旋混合后,将样品在4℃下12 377 r/min 离心30 min。收集上层液相并与200 μl 氯仿混合,然后在4℃、12 377 r/min 离心30 min。然后将富含RNA 的上层相与1.5 倍体积的100%乙醇混合。将混合物装载到RNeasy 离心柱(德国凯杰公司)中。多次洗涤后,用60 μl H2O 洗脱RNA。使用生物分析仪测量RNA 的质量和数量。所有样本的RNA 完整性(RIN)都在8 以上。

表1 患者基本信息
续表1
1.3 测序数据处理
本转录组测序由上海致禾生物公司实施,将提取的RNA 在HiSeq 2000 仪器进行测序,最终获得原始测序数据。利用TopHat 软件,将序列比对到人类参考基因组中。采用百万外显子的碱基片段(fragments per kilo bases per million reads,FPKM)评估基因表达水平。应用Cufflinks 对实验组以及对照组的组织的比对文件进行分析,挖掘出错误发现率<0.05 有差异表达的基因。
1.4 逆转录聚合酶链反应
根据相同的纳入标准,经过数据挖掘后,将后选的基因在扩大样本量中进行表达验证,扩大样本为另外单独收集的20 对,包括20 个糖尿病患者和20 个非糖尿病患者的EAT 样本。并按照qScript Flex cDNA试剂盒(美国安诺伦公司)说明书的步骤进行逆转录。使用具有10 μl 反应物的Perfe CTA SYBR Green Fast Mix ROX(美国安诺伦公司)在ABI 7900(美国生命技术科技公司)进行RT-PCR。使用ABI 7900 HT 软件分析转录物扩增结果,并且使用2-ΔΔCt方法将所有值归一化为SDHA 的水平。
1.5 基因富集分析
使用EnrichR[8]对识别的差异表达基因进行富集分析。具体分析类型有基因本体论(GO)富集,信号通路(KEGG)分析。
1.6 Western blotting 检测FRA1 和PTX2 表达
BCA 蛋白定量试剂盒、Western 制胶试剂盒均购自上海碧云天生物技术有限公司。BCA 法测蛋白质浓度,取50 μg 蛋白质用10%的分离胶和浓缩胶进行电泳分离,电转移至PVDF 膜。5%脱脂牛奶室温摇床封闭1 h,孵育1 ∶2 000 比例FRA1 和PTX3 一抗4℃摇床过夜。次日,回收一抗,1×TBST 洗膜8 min/次,3 次,室温孵育1 ∶5 000 比例二抗1 h,再次用1×TBST 洗膜8 min/次,3 次,ECL 发光试剂曝光。
1.7 统计学方法
数据分析采用R 语言2.39 软件。计量资料以均数±标准差(±s)表示,比较采用t检验,P<0.05为差异有统计学意义。
2 结果
2.1 D 与ND 组患者EAT 的RNA-seq 分析
通过D 组与ND 组EAT 的RNA-Seq 数据识别到592 个差异表达基因,其中433 个基因在糖尿病EAT中表现为上调,159 个基因表现为下调。见图1。
2.2 D 组EAT 中上调的基因
图1 ND 组和N 组差异表达基因聚类图
在D 组EAT 中高表达基因是ARC(Fold Change,FC=80),与调控细胞骨架相关蛋白有关。根据差异表达的分析结果,11 个高表达基因其参与的功能作用各不相同(见表2)。随后,将识别到的所有上调基因进行富集分析,挖掘其潜在的功能,结果显示D组EAT 中上调的基因与炎症反应密切相关,包括炎症反应途径、细胞因子产生及白细胞迁移途径。KEGG通路分析进一步证实,上调基因主要参与炎症相关通路,如cytokine-cytokine 相互作用、TNF、NF-κB,JAK-STAT。值得注意的是,在D 组EAT 中发现有20 个上调基因富集于AGE-RAGE 信号途径,该途径的重要配体包括S100A8 和S100A9,其FC 分别为4.79 和4.90。此外,本研究结果识别26 个脂肪因子和脂肪因子受体(如TNF 家族成员TNFRSF12A、TNFRSF13C 和趋化因子受体CXCR1 和CXCR2 等)在D 组EAT 中表现为高表达(见表3)。大部分脂肪因子如CSF3、IL-1β、IL-6 及CCL2 均表现为促炎性。使用qRT-PCR 进一步验证由RNA-Seq 识别一些具有差异表达的基因,该基因包括CSF3、EMP1、FOSL1、GOS2、IL-6、MT2A和THSB1。qRT-PCR 的结果证实该基因在D 组EAT 中为高表达。见图2。
2.3 D 组EAT 中下调的基因
在D 组EAT 中下调的基因包括KRT19、RSP01、PTX3、KLK11 和DNAJC22等。该基因可能参与CAD和糖尿病性心脏病的发生、发展。
2.4 在D 组EAT 中氧化应激标志物的应用
为探究EAT 中的RNA 表达变化是否由高血糖引起的氧化应激增加所致,本研究分析以前研究报道过的在氧化应激作用下表达有改变的基因。结果发现,15 个基因在D 组和ND 组的EAT 中均有表达,且其 中8 个 基 因(FRA1、PTX3、CDKN1A、GDF15、TIRAPP、RGS16、PLK3 和ETS2)的差异有统计学意义。
2.5 蛋白表达
根据基因表达的FDR 显著性,选择与氧化应激途径有关并且有差异表达的2 个基因(FRA1和PTX3)(见图3),通过免疫印迹在EAT 中证实相关基因编码蛋白的表达情况。结果表明,在另外的20对D 组 和ND 组的EAT 中FRA1(FOSL1 蛋 白)的Western blotting 结果为增加;PTX3 的Western blotting结果为降低(见图4)。该蛋白表达结果与RNA-Seq结果一致。
表2 各基因的表达变化 (±s)
表2 各基因的表达变化 (±s)
基因名称 参与的功能表达量 两组基因表达值的倍数变化 t 值 P 值ND 组 D 组ARC 骨架信号 0.291±0.002 25.242±3.021 87.312±23.123 34.211 0.000 LINC01348 未富集 0.262±0.001 15.991±2.232 60.711±18.342 45.212 0.000 LIPG 脂蛋白代谢 0.263±0.002 10.591±7.521 40.573±14.231 23.233 0.000 PTX3 免疫信号 2.562±0.013 97.712±14.321 38.231±13.234 24.232 0.000 CSF3 免疫信号 1.053±0.023 28.171±5.342 26.792±12.123 24.112 0.000 LBP 免疫信号 1.362±0.021 35.241±6.342 25.884±13.245 23.783 0.000 FOSL1 细胞增殖 1.562±0.032 37.191±6.311 23.912±13.245 22.324 0.000 SLC7A5 新陈代谢 1.212±0.012 22.432±8.234 18.543±9.234 21.231 0.000 MMP-19 细胞外基质重塑 8.923±2.043 162.072±34.321 18.171±9.073 20.123 0.000 SLC16A10 营养运输 0.682±0.022 11.251±3.211 16.472±8.421 18.231 0.000 TNFRSF12A 内皮细胞 6.253±2.021 100.471±23.251 16.083±7.782 17.234 0.000
表3 各脂肪因子及其受体的差异表达 (±s)
表3 各脂肪因子及其受体的差异表达 (±s)
基因名称 参与的功能表达量 两组基因表达 值的倍数变化 t 值 P 值ND 组 D 组CSF3 免疫信号 1.051±0.021 28.172±9.241 26.792±13.123 20.123 0.000 LBP 免疫信号 1.361±0.021 35.241±13.244 25.882±12.345 21.244 0.000 CXCL8 免疫信号 6.471±1.213 104.112±23.133 16.112±9.282 18.342 0.000 IL1RN 免疫信号 1.481±0.011 21.623±5.213 14.592±8.081 16.231 0.000 INHBA 免疫信号 0.711±0.002 11.332±2.234 15.982±7.625 15.232 0.000 IL-6 免疫信号 25.811±5.114 301.742±33.123 11.692±6.861 13.232 0.000 FGF9 免疫信号 3.432±0.123 0.453±0.002 0.132±0.023 3.234 0.000 LIF 免疫信号 4.692±0.421 69.062±23.12 12.593±5.982 11.234 0.000 STC1 免疫信号 3.292±0.231 35.423±7.234 10.772±5.982 10.232 0.000 PROK2 免疫信号 0.792±0.001 7.512±2.121 9.532±5.091 8.232 0.000 CXCL3 免疫信号 1.432±0.012 17.282±6.234 12.131±6.012 9.234 0.000 OSM 免疫信号 2.092±0.012 17.682±5.829 8.483±5.051 8.234 0.000 OPN 免疫信号 6.522±1.231 60.762±20.231 9.722±4.811 8.234 0.000 CHI3L1 免疫信号 5.751±1.012 42.921±13.232 7.462±4.013 7.231 0.000 IL-1β 免疫信号 4.141±0.781 30.123±10.234 7.283±3.872 6.232 0.000 CCL2 免疫信号 85.812±18.122 671.532±78.231 7.832±3.641 6.231 0.000 AREG 免疫信号 4.321±0.781 31.741±10.232 7.392±3.342 5.811 0.000 TNC 免疫信号 7.292±1.231 43.812±15.232 6.012±3.021 5.552 0.000 CXCL2 免疫信号 26.253±8.213 152.232±34.234 5.821±2.671 5.134 0.000 PLAU 免疫信号 9.813±1.245 53.182±16.232 5.422±2.512 5.012 0.000 SEMA4A 免疫信号 18.935±6.431 94.612±20.121 5.012±2.341 4.812 0.000 ALPL 免疫信号 6.462±2.123 32.672±15.231 5.053±2.221 4.712 0.000 CCL19 免疫信号 18.161±6.123 79.332±17.231 4.373±2.123 4.521 0.000 SEMA7A 免疫信号 1.152±0.123 5.212±1.263 4.552±2.412 4.411 0.000 BMP-4 免疫信号 15.792±6.331 4.633±1.234 0.293±0.003 2.123 0.000 VEGFA 免疫信号 31.112±9.123 112.711±34.234 3.623±1.341 4.012 0.000 SEMA6B 免疫信号 6.273±1.248 22.132±6.21 3.534±1.347 3.712 0.000 ECGF1 免疫信号 24.892±9.193 77.943±23.112 3.133±1.623 3.012 0.000 FSTL3 免疫信号 21.543±9.124 48.943±32.344 2.274±1.221 2.813 0.000 SLIT2 免疫信号 18.262±6.731 10.142±2.124 0.563±0.032 1.123 0.000
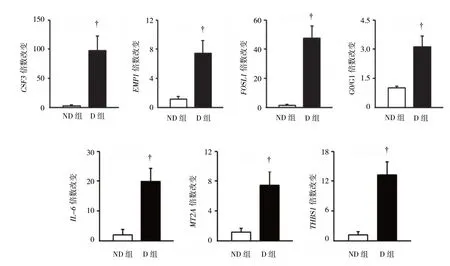
图2 qRT-PCR 验证基因表达结果 (±s)
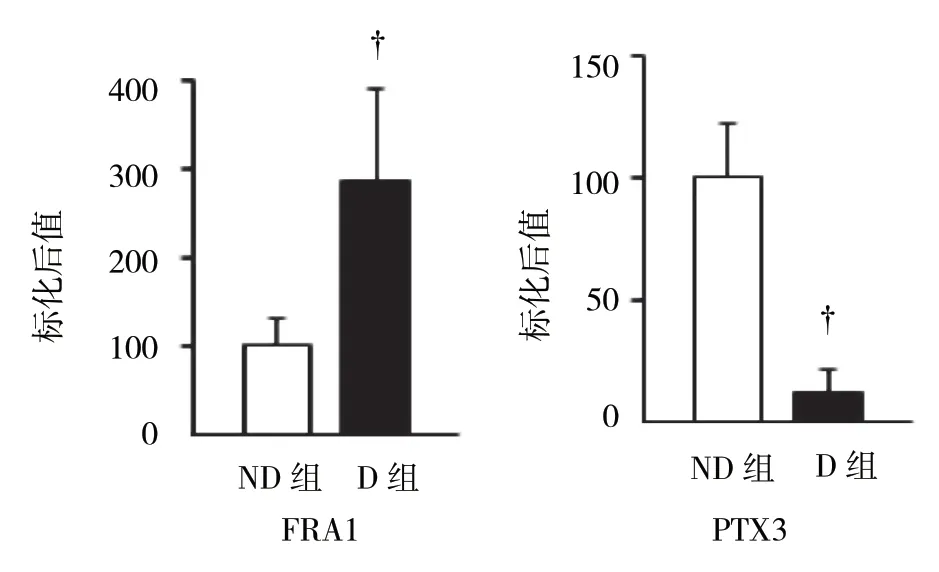
图3 FRA1 和PTX3 凝胶图像处理系统定量检测结果 (±s)
3 讨论
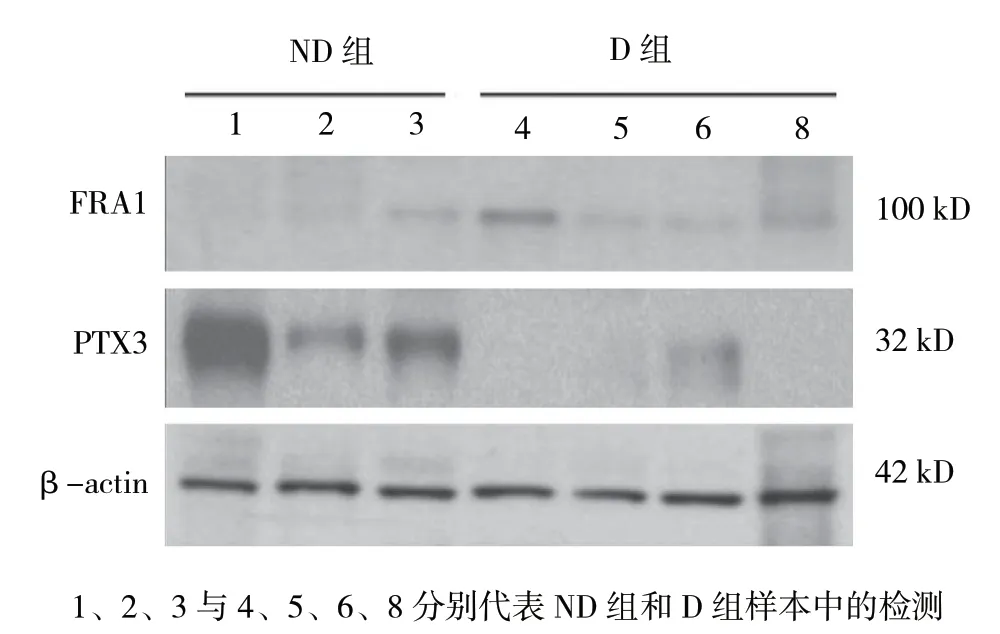
图4 FRA1 和PTX2 Western blotting 结果
本研究发现,糖尿病EAT 的差异表达基因高度富集于炎症反应。这支持以前研究的数据,证实EAT 中炎症反应引起的转录组水平差异[9-11]。在糖尿病EAT中上调的基因包括不同的炎症反应途径,例如细胞因子产生、白细胞迁移、细胞因子-细胞因子相互作用,以及TNF、NF-κB、JAK-STAT 和AGE-RAGE信号传导等。值得注意的是,在糖尿病EAT 中,20 个上调基因属于AGE-RAGE 信号传导,目前该信号途径在加速糖尿病患者冠状动脉粥样硬化过程中的促炎和促纤维化通路的激活中发挥关键作用[12]。RAGE 是结合AGE 和其他炎症细胞因子的多配体受体。激活的AGE-RAGE 途径可能引起会糖尿病EAT 患者的局部和慢性炎症反应。在EAT 中RAGE 的表达增加与炎症、脂肪细胞肥大、脂联素降低以及胰岛素信号受损相关[13]。有人提出CAD 患者中EAT 代谢紊乱可能与RAGE 过度表达有关[13]。高血糖激活的AGERAGE 途径可以上调转录因子的表达。该转录因子的失衡和上调导致许多基因的差异转录。在糖尿病EAT中显著上调的转录因子为NF-κB 家族和FOS 家族成员。值得注意的是,本研究发现,NF-κB 途径中的14 个基因在糖尿病EAT 中表现为上调,表明NF-κB信号通路在糖尿病EAT 中被激活。糖尿病中的AGERAGE 通路可首先激活NF-κB 信号。因此,NF-κB家族转录因子可进一步调节EAT 中靶向基因的表达。总之,本研究发现,NF-κB 信号的激活可能是引起糖尿病EAT 中基因表达变化的原因,从而进一步促进CAD 的发病。
促炎脂肪因子TNF、ILs 和趋化因子受体(如CXCR1 和CXCR2)在糖尿病EAT 中表现为高表达,这与以前的研究结果一致[2,14]。心外膜炎症分子的旁分泌可能引起代谢和炎症环境的改变从而促进动脉粥样硬化形成。细胞增殖和斑块形成增加了动脉壁厚度,动脉粥样硬化形成过程中使旁分泌扩散更困难。本研究结果表明许多基因在糖尿病EAT 的炎症反应中被激活。
糖尿病EAT 的转录组变化可能与高血糖引起的氧化应激有关。本研究发现,FRA1、PTX3、CDKN1A、GDF15、TIRAPP、RGS16、PLK3和ETS2的 差 异 表达表明氧化应激对糖尿病EAT 样本的影响。特别是CDKN1A 和GDF15 是p53 信号通路的成员,该信号通路先前已被证明与氧化应激和心血管疾病有关[15]。后续研究根据是否有差异表达,筛选出2 个基因(FRA1和PTX3)进行实验验证,与测序结果一致。
本研究首次发现,在糖尿病EAT 中高度富集的FOSL1 蛋白编码基因,是一种参与细胞增殖、分化和转化的调节因子。FOSL1 与Toll 样受体(TLRs)信号通路有关。该发现可能具有重要意义,因为EAT 内导致动脉粥样硬化的炎症过程也可能涉及先天免疫炎症反应。Toll 样受体TLR 刺激炎症介质的转录从而引起先天性免疫反应。TLRs 的激活将NF-κB 易位至胞核内,从而启动IL-1β,IL-6,TNF-α 和抵抗素的转录、释放[16]。
另一个新的糖尿病EAT 下调基因为PTX3,它是先天性和适应性免疫应答的关键组成部分。该发现与EAT 在参与动脉粥样硬化发展和进展的先天性炎症反应中的作用一致。PTX3 在动脉粥样硬化中的作用存在争议,因为最近的观察表明,PTX3 实际上可能具有心血管保护功能[17]。PTX3 在糖尿病EAT 中的下调可能反映对严重CAD 的保护性反应。适应性或者不适应性免疫应答的机制可能导致EAT 心脏保护基因的上调或下调[9]。
本研究存在一些局限性。首先,用于RNA-Seq的样本数量较少,需要大样本量来证明结果的可靠性。然而,本研究通过独立队列对鉴定的重要基因进行RT-PCR 和Western blotting 的验证,结果与本研究的RNA-Seq 一致。其次,尽管本研究分析一些脂肪细胞因子参与的分子/细胞功能和信号途径,但仍可能存在被忽略的其他功能和信号途径。最后,本研究缺少对鉴定基因的功能验证。
综上所述,本研究为T2DM 和CAD 受试者的EAT 转录组测序分析提供新的研究方向。糖尿病EAT转录组主要富集参与炎症,免疫应答和内皮损伤的基因。本研究数据显示,EAT炎症基因表达可能是由于上调的细胞因子,主要是NF-κB 和FOS 的作用,主要由AGE-RAGE 信号传导激活。此外,发现糖尿病EAT 的转录组变化与高血糖引起的氧化应激关系。本研究的数据结果可能描述糖尿病患者导致冠状动脉粥样硬化的新途径,为理解糖尿病冠心病的病理生理学作用的新假说提供了理论依据。
