枯草芽孢杆菌MX-6产纳豆激酶特性分析
2019-04-04满丽莉向殿军
满丽莉,向殿军
1(内蒙古民族大学 生命科学学院,内蒙古 通辽,028042)2(内蒙古民族大学 农学院,内蒙古 通辽,028042)
血栓形成是由大量纤维蛋白和血小板异常凝聚引起的,与心肌梗死、脑梗塞和中风等疾病的发生密切相关,是一种严重威胁人类健康的疾病[1-2]。纳豆在发酵过程中纳豆菌会产生一种碱性丝氨酸蛋白酶—纳豆激酶,该酶最早于1987年由日本的须见洋行等发现,其在体内体外均为一种有效的溶栓酶,它不仅直接降解纤维蛋白,而且可促进细胞释放组织纤溶酶原激活物,降解纤维蛋白酶,纳豆激酶是纳豆、豆豉等发酵食品的主要功能成分,作为溶栓药物的应用备受关注[3-4]。与临床溶栓药物(尿激酶和链激酶等)相比,纳豆激酶具有安全、低成本、易口服、易吸收、不易导致出血倾向、并发症少等优点,是一种理想的绿色溶栓药物,具有广阔的开发和应用前景[5-6]。纳豆激酶除具有溶解血栓作用外,还具有抑制血小板凝固、降血压、改善血流等多种生理功能,因此,纳豆激酶相关产品(如纳豆激酶药品、纳豆激酶保健品及功能性食品等)的开发引起了世界范围内的广泛关注[7-8]。
纳豆激酶高产菌株的来源较为广泛,如纳豆、豆豉、中国豆酱、韩国的Chungkook-Jang和Doen-Jang等[4],菌株筛选是研究纳豆激酶特性、纯化、应用等的基础,对纳豆激酶特性的深入研究有利于保障人民的身体健康,具有重要的经济价值和社会效益。
本研究以市售纳豆、豆豉及自然发酵纳豆为供试样品,筛选溶栓酶高产菌株,通过个体形态、菌落形态及16S rDNA基因同源性比对确定菌株属种,应用聚合酶链式反应、多序列比对、系统进化树构建、聚丙烯酰胺凝胶电泳、发酵曲线测定进一步研究纳豆激酶相关特性,为更好地实现纳豆激酶的开发及应用提供一定的理论依据和方法参考。
1 材料与方法
1.1 材料
1.1.1 供试样品及模式菌株
供试样品为市售的商品化纳豆、豆豉及自然发酵纳豆。模式菌株,枯草芽孢杆菌1.108 6购自中科院北京微生物研究所。
1.1.2 试剂与培养基
凝血酶,购自中国药品生物制品检定所;纤维蛋白原和琼脂糖,购自美国Sigma公司;细菌基因组提取试剂盒,购自北京天根生化科技有限公司;Marker、Taq聚合酶等,购自宝生物工程(上海)有限公司;其他药品均为国产分析纯。
酪蛋白高盐平板(g/L):酵母提取物5,酪蛋白10,NaCl 40,琼脂20,pH 7.2~7.4,121 ℃灭菌20 min。
种子培养基(g/L):蛋白胨10,牛肉膏3,NaCl 5,pH 7.2~7.4,121 ℃灭菌20 min。
发酵培养基(g/L):葡萄糖20,蛋白胨20,NaCl 5,pH 7.2~7.4,121 ℃灭菌20 min。
1.1.3 仪器与设备
MJ-54A/MJ-78A型STIK灭菌锅,北京天赐科仪商贸有限公司;Microfuge16型离心机,美国贝克曼公司;WS-600型恒温振荡培养箱,德国Wiggens公司;110-801型三量ip67防水原点数显卡尺不锈钢零点电子游标尺,东莞市景有模具五金有限公司;9700 PCR仪,美国Applied Biosystems公司;DYY-10C型电泳仪及DYCZ-28A型电泳槽,北京市六一仪器厂;1708195型凝胶成像系统,伯乐(Bio-Rad)公司。
1.2 方法
1.2.1 培养及发酵方法
培养方法:挑取菌株接种于装有50 mL种子培养基的250 mL锥形瓶中,37 ℃、180 r/min条件下振荡培养12 h(OD600=1.5~1.6)。
发酵方法:将菌液按5%接种于装有50 mL发酵培养基的250 mL锥形瓶中,37 ℃、180 r/min条件下振荡培养72 h后,发酵液以5 000×g离心10 min后,取10 μL上清液用于纳豆激酶活性的测定。
1.2.2 纳豆激酶活力的测定
纳豆激酶活力的测定采用纤维蛋白平板法[9]。溶圈直径采用电子游标卡尺测定。
1.2.3 纳豆激酶高产菌株的筛选及鉴定
1.2.3.1 菌种的分离及筛选
将6~8粒供试样品放入无菌试管中,经10 mL无菌水洗涤分装于离心管中,5 000×g离心10 min,收集菌体,用无菌生理盐水悬起,95 ℃水浴5 min,将菌液进行10倍的梯度稀释,取10-3、10-4、10-5分别吸取100 μL菌液涂布于酪蛋白高盐平板,37 ℃培养36 h,电子游标卡尺测定透明圈大小,在每个培养皿中选择透明圈/菌落比值大的菌落挑菌,经反复纯化后保藏。挑取菌株按1.2.1方法培养发酵测定纳豆激酶活性,溶圈直径最大的菌株用于进一步研究。
1.2.3.2 纳豆激酶高产菌株的鉴定
通过菌落形态、菌体形态、生理生化鉴定和16S rDNA 基因序列测定进行菌株的鉴定。比较分离菌株及模式菌株在固体种子培养基上的菌落形态、革兰氏染色后的菌体形态及生理生化鉴定结果,根据《常见细菌系统鉴定手册》及《伯杰氏细菌鉴定手册(第八版)》进行菌株的初步鉴定。
应用细菌基因组提取试剂盒进行菌体DNA的提取,通过PCR扩增16S rDNA基因序列进一步鉴定菌株。根据GenBank中已公布的枯草芽孢杆菌的16S rDNA 基因序列(序列号分别为AY219900.1、EF423598.1、JN399219.1、EF528288.1、DQ416796.1、JX402129.1、JQ435698.1、AB188212.1、HM030757.1)设计引物,上游引物(MX-6-16S rDNA-F):5′-AGAGKTTGAWYMTGGCTCAGGA CG-3′,下游引物(MX-6-16S rDNA-R):5′-ARGGAGGTGATCCAGCCRCACCTT-3′。PCR扩增产物通过10 g/L琼脂糖凝胶电泳(100 V)检测,送上海生工生物工程技术有限公司进行序列测定。利用NCBI中的Blast(http://www.ncbi.nlm.nih.gov/BLAST),将所测定的序列与数据库中的序列进行比对。
1.2.4 纳豆激酶编码基因aprN的克隆
根据GenBank中已公布的纳豆激酶编码基因aprN序列设计引物,上游引物aprN-F(5′-GTATGAAAATAGTTATTTCGAGTCTC-3′)和下游引物aprN-R(5′-TCCGGTGCTTGTGAAG ATTTTCAGA-3′)用于克隆枯草芽孢杆菌MX-6中的aprN基因。PCR反应程序为:94 ℃预变性5 min;94 ℃、30 s,52 ℃、30 s,72 ℃、1 min,30次循环;72 ℃延伸10 min。PCR扩增产物通过10 g/L琼脂糖凝胶电泳(100 V)检测,送上海生工生物工程技术有限公司进行序列测定。利用NCBI中的Blast(http://www.ncbi.nlm.nih.gov/BLAST)将所测定的序列与数据库中的序列进行比对,系统进化树用MEGA 6.0构建,氨基酸多序列比对利用Clustal X进行,蛋白质的分子质量用ExPASy的ProtParam tool在线程序预测。
1.2.5 纳豆激酶的分子质量测定
枯草芽孢杆菌MX-6采用1.2.1方法获得纳豆激酶发酵上清液。上清液经过硫酸铵沉淀、DEAE-Sepharose Fast Flow及CM-Sepharose Fast Flow层析和Sephadex G-75凝胶过滤实现纳豆激酶的纯化。纯化的纳豆激酶的相对分子质量通过SDS-PAGE测定[10]。
1.2.6 枯草芽孢杆菌MX-6产纳豆激酶的发酵曲线
枯草芽孢杆菌MX-6采用1.2.1方法培养,将菌液按体积分数5%接种于装有50 mL发酵培养基的250 mL锥形瓶中,37 ℃,180 r/min条件下振荡培养102 h后,每隔6 h取样,发酵液经5 000×g离心10 min 后,取10 μL上清液用于纳豆激酶活性的测定。
1.2.7 数据分析
纳豆激酶活性的测定数据均重复3次,应用SPSS 17.0软件计算标准差。
2 结果与分析
2.1 菌株的筛选与鉴定
2.1.1 菌株的分离及筛选
应用酪蛋白高盐平板从商品化的纳豆、豆豉及自然发酵纳豆中分离出60株产蛋白酶菌株,应用纤维蛋白平板法筛选出4株纤溶酶活性明显较高的菌株(P<0.01),分别命名为菌株MX-1、MX-3、MX-5、MX-6,其中分离于豆豉中的菌株MX-6纤溶酶活性最高,溶圈直径达到(21.60±0.48)mm,将菌株MX-6应用于进一步的研究。
2.1.2 菌落形态及菌体形态鉴定
与模式菌株-枯草芽孢杆菌1.108 6相比,不同点在于菌株MX-6的菌落直径较大,菌落形状为不规则圆形,边缘呈现波状,中央凸起,有褶皱,孢子位于端生或偏端生(表1)。
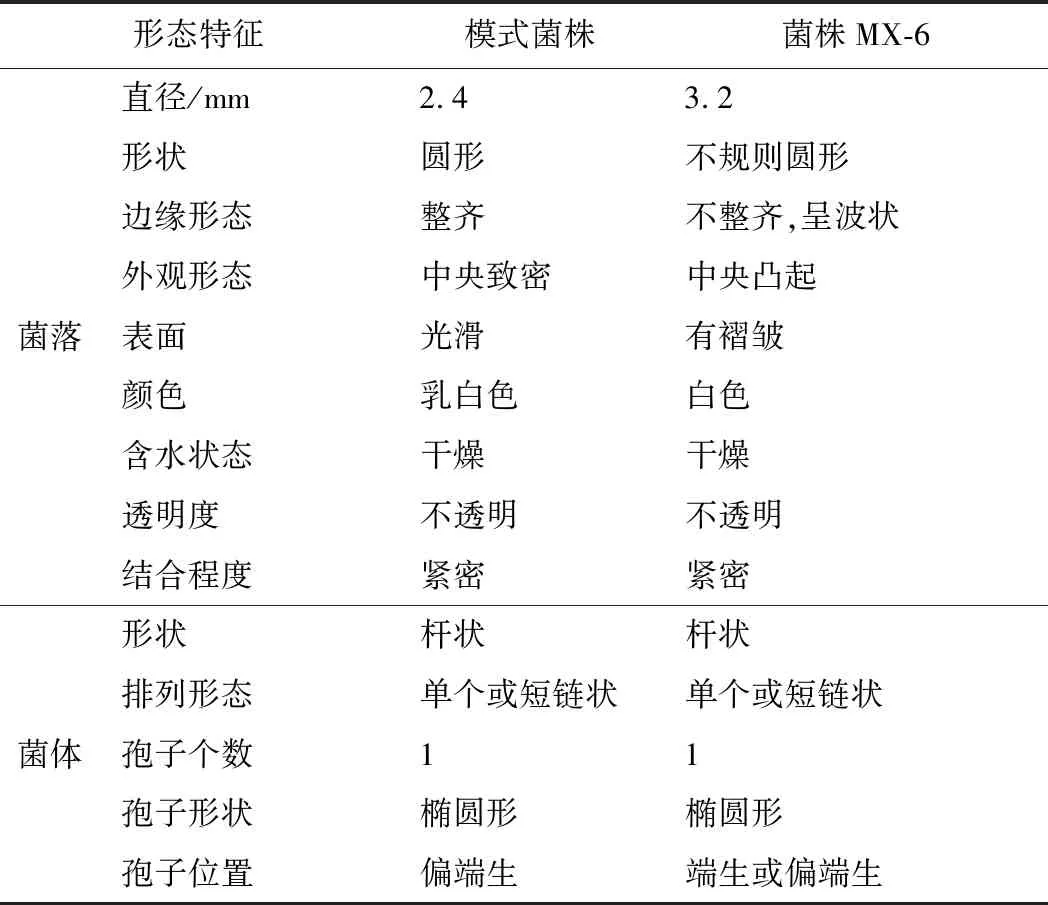
表1 菌落形态及菌体形态鉴定结果Table 1 Identification results of colony morphology and bacterial morphology
2.1.3 生理生化鉴定
通过革兰氏染色阳性、过氧化氢酶实验阳性及其他生理生化鉴定结果显示,菌株MX-6符合枯草芽孢杆菌属的典型特征。与模式菌株相比较,不同点在于菌株MX-6的D-甘露醇实验为阴性,其他生理生化鉴定结果均与模式菌株相同(表2),且都具有较好的耐盐性和耐热性,此结果可说明菌株MX-6属于枯草芽孢杆菌属。

表2 生理生化鉴定结果Table 2 The results of physiological and biochemical identification
注:“+”或“-”分别表示生化反应(或革兰氏染色)为阳性或阴性。
2.1.4 菌株的16S rDNA基因序列分析
以菌株MX-6的总基因组DNA为模板,应用简并引物MX-6-16S rDNA-F和MX-6-16S rDNA-R扩增16S rDNA基因片段,获得的序列片段长度为1 542 bp(图1),其序列与GenBank中已公布的枯草芽胞杆菌IAM 12118(NR112116.1)和枯草芽孢杆菌168(NR102783.1)的16S rDNA基因序列同源性高达99%以上,进一步证明菌株MX-6为枯草芽孢杆菌,其16S rDNA基因序列已提交到GenBank,序列号为KY575151。

M-DNA Marker Ⅲ;1-16S rDNA的PCR产物图1 琼脂糖凝胶电泳鉴定菌株MX-6的16S rDNA基因Fig.1 Identification of16S rDNA gene in strain MX-6 by agarose gel electrophoresis
2.2 纳豆激酶编码基因aprN的克隆及序列分析
以枯草芽孢杆菌MX-6的总基因组DNA为模板,根据GenBank中已公布的纳豆激酶编码基因aprN序列设计引物,PCR产物通过琼脂糖凝胶电泳进行检测,以DNA Marker Ⅲ为对照,在1 473 bp处获得1条特异性条带(图2),测序结果与GenBank中枯草芽胞杆菌29R(CP017763.1)、枯草芽孢杆菌KH2(CP018184.1)及纳豆芽孢杆菌CGMCC 2108(CP014471.1)的aprN基因序列的同源性高达99%,表明克隆到的aprN基因序列的正确性。将克隆测序得到的aprN基因序列提交至GenBank数据库,获得序列号为KY576911。枯草芽孢杆菌MX-6的aprN基因序列长度为1 473 bp,开放阅读框为1 152 bp,起始密码子为GTG,终止序列包括3个终止密码子(TAA、TAG、TAA)。

M-DNA Marker Ⅲ;1-aprN的PCR产物图2 琼脂糖凝胶电泳鉴定枯草芽孢杆菌MX-6的aprN基因Fig.2 Identification of aprN gene in Bacillus subtilis MX-6 by agarose gel electrophoresis
与枯草芽孢杆菌LSSE-22所产纳豆激酶的结构相一致[11],由推断获得的381个氨基酸组成的阅读框架包括29个氨基酸组成的信号肽、77个氨基酸组成的前导肽及275个氨基酸组成的成熟肽。预测分子质量为27 712.72 u。推断的氨基酸序列与已公布枯草芽孢杆菌纳豆激酶序列(AA065246.1)的同源性高达99%,结果表明,枯草芽孢杆菌MX-6所产的纤溶酶为纳豆激酶。氨基酸多序列比对结果显示,枯草芽孢杆菌MX-6的溶栓酶序列与已知的溶栓酶序列具有高度保守性,与枯草芽胞杆菌的纳豆激酶序列(WP 014479360.1)仅相差1个氨基酸残基(图3)。由系统进化树可知(图4),枯草芽孢杆菌MX-6的纤溶酶与枯草芽孢杆菌纳豆激酶NAT(WP 014479360.1)、枯草芽孢杆菌纳豆激酶前体(AA065246.1)、枯草芽孢杆菌纳豆激酶E(WP 009966941.1)同在一个进化分支上,可进一步确定枯草芽孢杆菌MX-6所产的纤溶酶为纳豆激酶。
2.3 纳豆激酶的相对分子质量测定
发酵上清液经40%~60%硫酸铵分段沉淀、DEAE-Sepharose Fast Flow及CM-Sepharose Fast Flow层析及Sephadex G-75凝胶过滤获得纯化纳豆激酶,纯度提高53.4倍,回收率达到11.7%,纯化纳豆激酶可用于进行SDS-PAGE。SDS-PAGE结果显示在约28 ku获得单一条带(图5),与预测的相对分子质量27 712.72 u相吻合。相关研究显示,微生物纤溶酶的相对分子质量在14~52 ku,枯草芽孢杆菌MX-6所产纳豆激酶的相对分子质量在此范围内[12],其相对分子质量测定结果(约28 ku)与DABBAGH等的研究结果(27.7 ku)相一致[7],而与LU等(118 ku和49 ku)[13]、KOTB等(18.2 ku)[14]和ZHANG等(约31 ku)[9]的研究结果不同,溶栓酶相对分子质量的差异可能是由于菌株属种不同所造成。

图3 枯草芽孢杆菌MX-6的纤溶酶序列与已知的纤溶酶序列的多序列比对结果Fig.3 Comparison results of plasminogen sequence of Bacillus subtilis MX-6 with known plasminogen sequence注:MX6-枯草芽孢杆菌MX-6的纤溶酶;Bs-枯草芽孢杆菌纳豆激酶NAT(WP 014479360.1);Bm-莫海威芽孢杆菌肽酶S8(WP 010333625.1);Bh-耐盐短杆菌肽酶S8(WP 044156525.1);Bv-死谷芽孢杆菌肽酶S8(WP 010329279.1);Bl-地衣芽孢杆菌酶AprE 3-17(ACU32756.1);Bn-纳卡穆雷芽孢杆菌肽酶(WP 061521470.1)。

图4 枯草芽孢杆菌MX-6的纤溶酶与同源纤溶酶构建的系统进化树Fig.4 Phylogenetic tree of plasminogen in Bacillus subtilis MX-6 and homologous plasminogens

1-粗酶液;2-纯化纳豆激酶图5 粗酶液及纯化纳豆激酶的SDS-PAGE结果Fig.5 SDS-PAGE result of crude enzyme and purified nattokinase
2.4 枯草芽孢杆菌MX-6产纳豆激酶的发酵曲线
由图6可知,在0~12 h枯草芽孢杆菌MX-6所产纳豆激酶的合成量较低,在12~72 h,纳豆激酶的合成量快速增加,72 h达到最高合成量,溶圈直径达到21.60 mm。在72~102 h纳豆激酶的合成量呈现下降趋势。

图6 枯草芽孢杆菌MX-6所产纳豆激酶的发酵曲线Fig.6 Fermentation curve of nattokinase produced by Bacillus subtilis MX-6
3 结论
由于16S rDNA基因在菌株进化过程中具有高度保守性,结合PCR技术可建立基于16S rDNA序列来确定其生物分类的方法,此法具有快捷、方便、准确等优势,可有效地分析不同菌株种内差异,是细菌分类研究中最有效的方法之一[15]。本研究结合菌落形态、菌体形态、生理生化鉴定及16S rDNA基因同源性比对结果准确鉴定了菌株MX-6为枯草芽孢杆菌。
目前,具有溶栓活性的蛋白酶除了纳豆激酶之外,还有许多其他的酶类如subtilisin DJ-4、subtilisin DFE、纤维蛋白分解酶BSF1等,由于纳豆激酶编码基因aprN具有高度保守性[11],为了进一步确定枯草芽胞杆菌MX-6所产溶栓酶是否为纳豆激酶,根据GenBank中已公布的纳豆激酶编码基因aprN序列设计引物,应用PCR技术克隆获得一条特异性条带,结合测序、多序列比对及系统进化树结果确定枯草芽胞杆菌MX-6所产的溶栓酶为纳豆激酶。SDS-PAGE获得纳豆激酶的分子质量约28 ku,与预测的分子质量277 12.72 u相吻合,表明检测结果的准确性。本研究可为纳豆激酶的工业化应用奠定一定的理论与技术参考。
