养殖废水处理系统中微生物菌群结构及动态变化
2019-02-27刘兰英吴妙鸿陈丽华叶美锋林代炎宋永康
黄 薇,刘兰英,吴妙鸿,陈丽华,吕 新,叶美锋,林代炎,宋永康*
养殖废水处理系统中微生物菌群结构及动态变化
黄 薇1,刘兰英1,吴妙鸿1,陈丽华1,吕 新1,叶美锋2,林代炎2,宋永康1*
(1.福建省农业科学院农业质量标准与检测技术研究所,福建 福州 350003;2.福建省农业科学院农业工程技术研究所,福建 福州 350003)
采用高通量测序技术对养猪场废水处理系统的细菌群落组成进行分析,同时对细菌群落、污水水质和抗生素含量相关性进行分析.研究结果表明,污水各处理单元中的菌群结构存在显著差异,且细菌菌群结构随处理进程逐步改变,其中集污池与厌氧反应器的细菌菌群优势门类为Firmicutes、Bacteroidetes和Proteobacteria,曝气池的细菌菌群优势门类变为Firmicutes、Verrucomicrobia、Bacteroidetes、Proteobacteria和Candidatus Saccharibacteria,氧化塘的细菌菌群优势门类为Firmicutes、Proteobacteria、Chloroflexi、Euryarchaeota和Bacteroidetes.相关性分析结果表明,污水中的细菌群落与水质环境因子之间存在显著相关(<0.05).其中,种属与COD、TN、NH4+-N呈显著正相关,与TN、NH4+-N呈显著负相关,与磺胺甲恶唑、磺胺间甲氧嘧啶、强力霉素、土霉素呈极显著正相关,与恩诺沙星、四环素呈显著正相关,与环丙沙星呈显著正相关,与氧氟沙星呈显著正相关.
养猪废水;细菌群落;水质;抗生素
近年来,随着我国畜禽养殖业的迅猛发展,其污染排放问题也日趋严重[1].畜禽养殖污水不仅富含化学需氧量(COD)、总氮(TN)、氨氮(NH4+-N)、总磷(TP)等常规污染物,还含有大量的抗生素和各种病原菌,对公共健康和生态安全构成严重威胁[2].研发经济高效的畜禽养殖废水处理工艺已成为养殖企业和环保行业的研究热点,且已开发出包括UASB-SBR、ABR-MABR、AMBR-MABR等在内的多种处理工艺[3-7].然而,目前的畜禽养殖废水处理工艺开发主要从工程角度进行设计,鲜有考虑微生物群落结构和功能及其与环境因素的响应规律[8].
在污水处理系统中,微生物是污染物去除的主体,系统的处理效果、稳定运行和出水安全性与其中的微生物群落结构及其功能紧密相关[8-9].Fang等[10]和Liu[11]等的研究结果均显示特定的微生物菌群能够促进废水处理系统中某些复杂大分子有机化合物的降解.因此,分析养殖废水处理系统各单元细菌群落组成、明确占主导地位的细菌群落以及细菌群落与污染物浓度之间的关联性,对优化废水处理系统菌群结构,提高水质净化效率具有重要作用[12].但目前对养殖废水处理过程中微生物群落结构及其丰度变化的研究仍少见报道.
传统细菌培养法在养殖废水微生物研究中的应用较多[13].然而由于细菌在自然条件的生长情况与培养基不同,以及自然界中大多数微生物不可培养,使得传统的分离培养方法很难对处理系统中各单元的优势种群及其差异进行识别[14].近几年,随着高通量测序及其分析技术的发展,国内外学者通过高通量技术在土壤、肠道微生物、极端环境等多种复杂介质中微生物群落的研究都取得了较好的进展[15-17].因此,本文拟采用高通量测序技术对某稳定运行养猪废水处理系统中的微生物群落结构进行分析,以期明确各处理单元中起主导作用的细菌及其在整个处理过程中的动态变化;同时通过相关性分析对环境因子和微生物群落的变化进行分析,探索对污染物质降解起重要作用的功能微生物,以期发现微生物群落变化的主要推动力.
1 材料与方法
1.1 养殖废水处理工艺流程
选择福建省三明市一家典型的规模化生猪养殖场为研究对象,该养猪场的生猪养殖规模约5000头,废水排放量为80m3/d.养猪场废水处理工艺流程见图1,养殖废水通过管道进入机械粗格栅拦截废水中的大颗粒物质及纤维状污染物,出水用振动筛挤压分离机进行固液分离,分离后的液体进入集污池继续沉淀废水中的部分细小颗粒,集污池中的污水经管道进入厌氧反应器中进行厌氧发酵,排除的沼液通过管道依次进入一级、二级、三级曝气池,经曝气处理后的污水进入氧化塘进行深度净化,氧化塘中种植穗花狐尾藻()以及养殖胡子鲶(),最终出水达到《畜禽养殖业污染物排放标准》(GB 18596-2001)[18],用于猪舍冲洗和周边苗木、蔬菜灌溉.
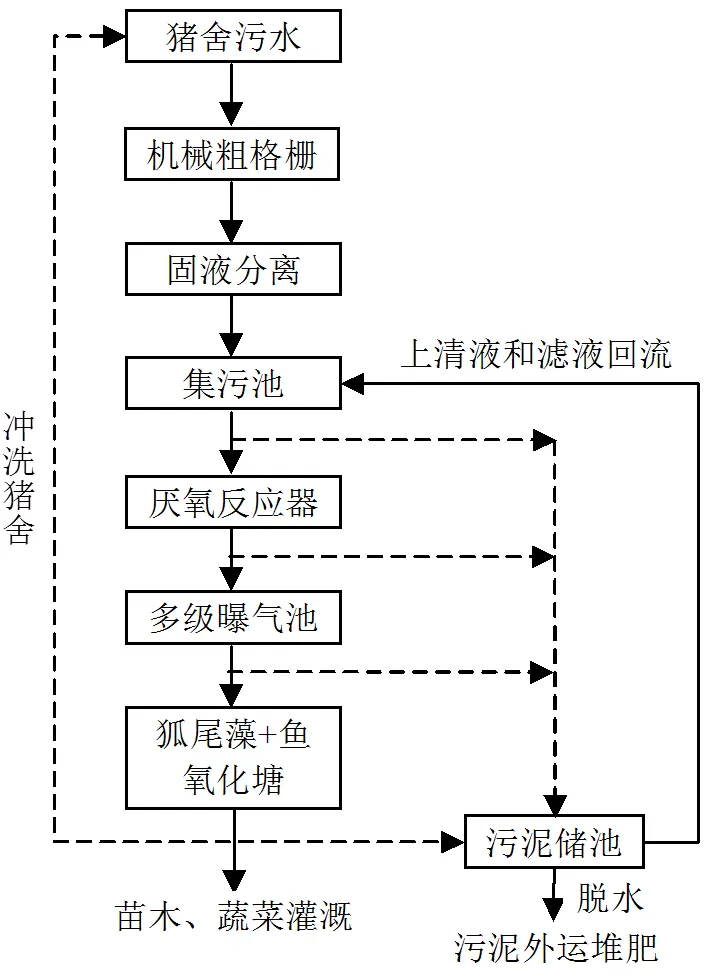
图1 养猪废水处理工艺流程示意
1.2 水样采集
依据处理系统的运行情况,于2018年1月19日进行1次系统地样品采集,共采集12个点,其中在集污池、厌氧反应器、三级曝气池、氧化塘各取3个,采样点布置在处理单元的出水口或出水口一侧,样品保存方法参考文献[19],在0~4℃条件下保存运输,48h内完成污水水质和抗生素的检测分析.
1.3 微生物菌群分析
1.3.1 DNA提取、PCR扩增和高通量测序 收集的水样通过0.45µm微孔滤膜进行过滤,取滤膜采用PowerWater DNA Isolation kit(MOBio, CarIsbad, CA, USA)进行提取,提取过程参考操作说明书进行.提取得到的DNA样品浓度和质量采用Qubit 2.0 Fluorometer(Invitrogen, Carlsbad, CA, USA)检测,将符合试验要求的DNA样品送生工生物工程(上海)股份有限公司扩增,PCR扩增采用16S rRNA基因V3~V4区的通用引物341F(5′-CCCTACACGCT- CTTCCGATCTG (barcode) CCTACGGGNGGCWG- CAG-3′)和805R(5′-GACTGGAGTTCCTTGGCA- CCCGAGAATTCCA (barcode) GACTACHVGGGT- ATCTAATCC-3′).PCR产物使用QIAquick PCR Purification kit(Qiagen, Düsseldorf, Germany)进行纯化.纯化后的PCR产物采用Illumina MiSeq测序平台进行高通量测序,原始数据提交NCBI’s Sequence Read Archive(SRA)数据库保存,序列号SRP151692.
1.3.2 高通量数据处理 Illumina MiSeq测序数据首先使用FLASH软件[20]进行拼接,之后运用cutadapt软件[21]裁切序列中的正反向引物,使用Prinseq软件[22]质控去除score<20的低质量序列,然后使用Usearch软件[23]去除嵌合体以及按97%序列相似度划分分类操作单元(OTUs).利用Ribosomal Database Project (RDP) classifier将OUT代表序列与数据库对比进行物种注释(置信度阈值默认为0.8以上),得到每条序列从门到属各个水平的分类信息,基于分类学信息,可以在各个分类水平上进行群落结构的统计分析[24].利用Mothur软件统计每个生物样本的Good’s coverage、ACE、Chao1、Shannon、Simpson等alpha多样性指数[25].使用QIIME软件计算weighted UniFrac距离,用于beta多样性的评估分析[26].
1.4 水质与抗生素含量检测分析
COD采用HJ/T 399-2007(快速消解分光光度法)[27]测定,TN采用HJ636-2012(碱性过硫酸钾消解紫外分光光度法)[28]测定,NH4+-N采用HJ 535-2009 (纳氏试剂分光光度法)[29].污水中抗生素含量的测定采用超高效液相色谱/串联质谱法(HPLC-MS),测定的抗生素有:磺胺甲恶唑(SMZ)、磺胺间甲氧嘧啶(SMM)、环丙沙星(CIP)、恩诺沙星(ENR)、氧氟沙星(OFLX)、强力霉素(DOX)、四环素(TET)、土霉素(OTC)[30].
1.5 数据统计分析
运用EXCEL软件统计平均数和标准方差.采用SPSS软件对污水水质、抗生素含量、差异物种、多样性指数等进行单因素方差分析(LSD法)和Spearman相关性分析.应用R软件统计细菌群落数据并绘制柱状图、热图、进化树、PCoA图,同时运行相似度分析(Anosim)对样品组间差异显著性进行检测以及对环境因素和细菌群落结构进行冗余分析(RDA)、Pearson Correlation相关性分析[31-32].通过LEfSe软件线性判别分析找出对样品划分产生显著性影响的差异物种[33].
2 结果与讨论
2.1 废水水质及抗生素含量动态变化
养殖污水净化处理过程中,水质动态变化见表1.从表中可知,养殖废水有机物浓度较高,集污池中污水COD浓度高达5180mg/L,经过厌氧反应器后,COD浓度急剧降低,降至1246mg/L,去除率达到75.95%,而曝气池和氧化塘对COD的贡献率仅为15.41%和1.68%,最终COD出水浓度为361mg/L.这与已有的多数研究结果基本一致[2-5],养殖废水中的有机物被厌氧微生物分解、代谢、消化,去除率可达到65%~80%;但出水后的沼液具有低COD/N的特性,其有机质不易被微生物利用[34].在脱氮方面,养殖废水中的TN含量主要以NH4+-N为主,TN和NH4+-N在厌氧反应器中的去除量不高,而在曝气池和氧化塘中被大量去除,去除率分别为75.75%和83.83%,出水TN浓度为137.67mg/L,NH4+-N浓度为63.67mg/L.有的研究认为曝气使自养型硝化细菌处于优势,可以稳定高效地转化NH4+-N为NO3--N[1].

表1 各处理单元出水水质分析(mg/L)
注:数值为平均值±标准差(=3),同行不同字母表示处理单元间差异显著(<0.05),LSD法.
在抗生素去除方面,本研究共检测了2种磺胺类、3种氟喹诺酮类以及3种四环素类,这几种抗生素在猪场中被普遍使用且检出率较高[35-37].抗生素浓度在各处理单元的变化情况具体见表2.总体上看,除TET外,其他7种抗生素都具有明显的去除效果,去除率达到86.91%~100%.从表中可知,厌氧反应器对污水中CIP、TET没有明显的消减作用,对ENR的消减作用达到89.76%,但SMZ、SMM、OFX、DOX、OTC等均表现出负去除现象.分析认为,原因可能是固体颗粒中吸附的抗生素解吸到水相中[1,36].曝气池处理后,多种抗生素浓度大幅降低,SMZ、SMM、CIP、DOX、OTC浓度均显著(<0.05)小于厌氧反应器中浓度,其中CIP浓度已经低于仪器检测下限.曝气池处理后的出水经过包含穗花狐尾藻和胡子鲶的氧化塘,各种抗生素浓度均得到进一步降低,SMZ和SMM浓度低于仪器检测下限,ENR和OFX浓度仅为0.01µg/L和0.17µg/L.此外,养殖污水中四环素类抗生素TET、DOX、OTC经处理系统处理后,出水中的浓度仍较高,分别为4.80µg/L、5.77µg/L和2.30µg/L,这与多项研究结果大体相似[38-39],表明处理系统对四环素类抗生素的降解能力较小.这可能与四环素类抗生素化学结构有关,其降解机制复杂、半衰期长,甚至在特定情况下降解产物还会转化为母体化合物;且四环素类抗生素是疏水性有机物,与其他抗生素相比,更容易被吸附在固体颗粒上而受到保护[40].

表2 各处理单元出水抗生素含量分析(µg/L)
注: -为末检验,数值为平均值±标准差(=3),同行不同字母表示处理单元间差异显著(<0.05),LSD法.
2.2 污水细菌群落结构特征与动态变化
如表3所示,12个污水样品获得的有效16S rRNA序列数为42537~69256条,共计608526条.基于97%的序列相似性,共获得8508个OTUs,平均每个样品为2263个OTUs.OUT覆盖率(Good’s coverage)显示97%以上的微生物被捕捉到,表明本研究获取的数据量能够很好地反映特定样品的细菌多样性情况.Alpha多样性分析结果表明(表3),不同处理单元出水中的微生物群落的丰富度指数(ACE和Chaol)和多样性指数(Shannon和Simpson)都存在一定的差异,上游处理单元的细菌物种数和群落多样性要比下游处理单元高,厌氧反应器中的细菌物种数和群落多样性要比有氧处理单元高.一般来说,细菌物种数和群落多样性越丰富,污染物的去除效果越好[41].
利用RDP classifier对97%相似度水平的OTU代表序列进行物种分类注释,共得到31个门水平的细菌和一些无法归类的细菌(图2).从图2中可见,集污池中的污水优势菌为Firmicutes、Bacteroidetes和Proteobacteria,分别占全部细菌总数的41.47%、30.52%和24.07%;Lamendella等[42]研究显示猪肠道微生物的主要类群为Firmicutes和Bacteroidetes,表明生猪养殖废水中的微生物主要以肠道微生物为主.在厌氧反应器中,优势群体的种类没有改变,但数量明显发生了变化,Firmicutes(62.99%)显著(<0.05)高于集污池,而Bacteroidetes(23.32%)和Proteobacteria (5.15%)显著(<0.05)低于集污池;在曝气池中,优势细菌变为Firmicutes(35.33%)、Verrucomicrobia (12.94%)、Bacteroidetes(12.40%)、Proteobacteria (12.09%)和Candidatus Saccharibacteria(8.28%),且各种群含量与厌氧反应器均存在显著差异(<0.05);进入氧化塘后,优势群体的种类为Firmicutes (37.39%)、Proteobacteria(12.09%)、Chloroflexi (7.84%)、Euryarchaeota(6.12%)和Bacteroidetes (5.63%),除Firmicutes和Euryarchaeota,其他种群含量与曝气池均存在显著差异(<0.05);说明不同的污水处理单元,细菌的优势菌群具有明显差异.

表3 Illumina测序数据统计分析
注:数值为平均值±标准差(=3),同行不同字母表示处理单元间差异显著(<0.05),LSD法.

图2 门水平下不同处理单元污水中细菌群落结构组成

图3 属水平下不同处理单元污水中细菌群落结构组成
通过高通量测序,在整个生猪养殖污水处理过程中共检测到548个属的细菌,对属水平的细菌群落数量在前50的属进行热图分析,结果见图3.从样品间的属水平的聚类关系可以看出,污水中细菌种群有高达20.30%~38.40%的物种未分类,且其种类及数量随着处理进程逐步发生改变.在集污池中,优势细菌属为(10.81%)、(9.25%)和(7.25%);进入厌氧反应器中,优势细菌属发生了明显的变化,变为(9.80%)和(7.93%);在曝气池中,含量显著降低(<0.05),和大幅增长(<0.05),优势菌属为(7.80%)、(7.70%)、(7.50%),进入氧化塘后,、和显著增长(<0.05),优势菌属为(9.09%)、(7.33%)、(6.11%)和(5.43%).
2.3 组间群落结果差异分析

图4 生猪养殖污水净化系统各处理单元细菌群落结构比较
a为聚类分析;b为PCoA
为比较污水各处理单元样品中细菌群落的差异性,根据各样本物种丰度计算Beta多样性距离矩阵,计算样本间距离的方法为weighted Unifrac,样本间的距离越大,说明样本间的细菌群落差异越大.基于距离矩阵进行聚类分析可以发现(图4a),细菌群落结构的变化是随着处理进程逐步发生改变的;基于距离矩阵进行PCoA发现(图4b),前3个主坐标可以代表90%的变量信息,并且各样品细菌群落随处理进程的变化过程主要是在PCoA1轴方向发生.曝气池和氧化塘样品的距离较近,表明这2个处理单元的微生物组成相似.

图5 生猪养殖污水净化系统各处理单元细菌群落结构相似性分析
Anosim分析结果可以看出各处理单元污水样品组间差异大于组内差异(=1),并且具有显著的统计学差异(=0.002<0.05),表明生猪养殖污水的各处理单元的群落结构发生了明显的变化(图5).进一步通过LEfSe判别分析估算各物种类群对差异效果影响的大小,筛选出对样本分类有显著影响作用的物种,结果见图6.从图中可以看出,集污池、厌氧反应器、曝气池和氧化塘对组间基因或功能差异有显著影响的物种类群分别为45,17,29以及53类.其中集污池与其他处理单元存在显著差异的菌群主要包括Proteobacteria中的Gammaproteobacteria和Bacteroidales等,研究表明Proteobacteria和Bacteroidetes均为畜禽养殖污水中的优势菌群, Proteobacteria主要负责有机和营养物的去除, Bacteroidetes在污水中的作用仅次于Proteobacteria[43-44].厌氧反应器中存在显著差异的门类为Firmicutes和Spirochaetes,Ducey等[45]研究表明,包含产酸菌Clostridiales、Clostridium、Acidobacteria以及产甲烷菌的Firmicutes在厌氧处理阶段起着关键作用.Verrucomicrobia是曝气池显著高于其他处理阶段的菌群门类,其是高效活性污泥中常见门类,主要用于对有机物的降解[46-47].在氧化塘中检测到众多与其他处理单元存在显著差异的菌群,主要隶属于门Proteobacteria、Planctomycetes、Euryarchaeota、Deinococcus- Thermus、Chloroflexi和Actinobacteria,这些差异菌群的存在可能与穗花狐尾藻根系微生物和胡子鲶肠道微生物有一定的关系.

图6 生猪养殖污水净化系统各处理单元细菌物种类群LEfSe分析环形树状
不同颜色表示不同分组,不同颜色的节点表示在该颜色所代表的分组中起重要作用的微生物群,黄色节点表示在不同分组中没有起到重要作用的微生物类群,图例为组间在丰度上存在显著差异的物种名称(<0.05)
2.4 细菌群落与水质环境因子之间的关系
水质环境因子影响着微生物的微生态生存环境,微生物的生长代谢反过来影响着水质中化学物质的存在形态和浓度[48-49].在属分类上对菌群(相对丰度>2%)进行Pearson Correlation相关性分析,结果如表4所示.
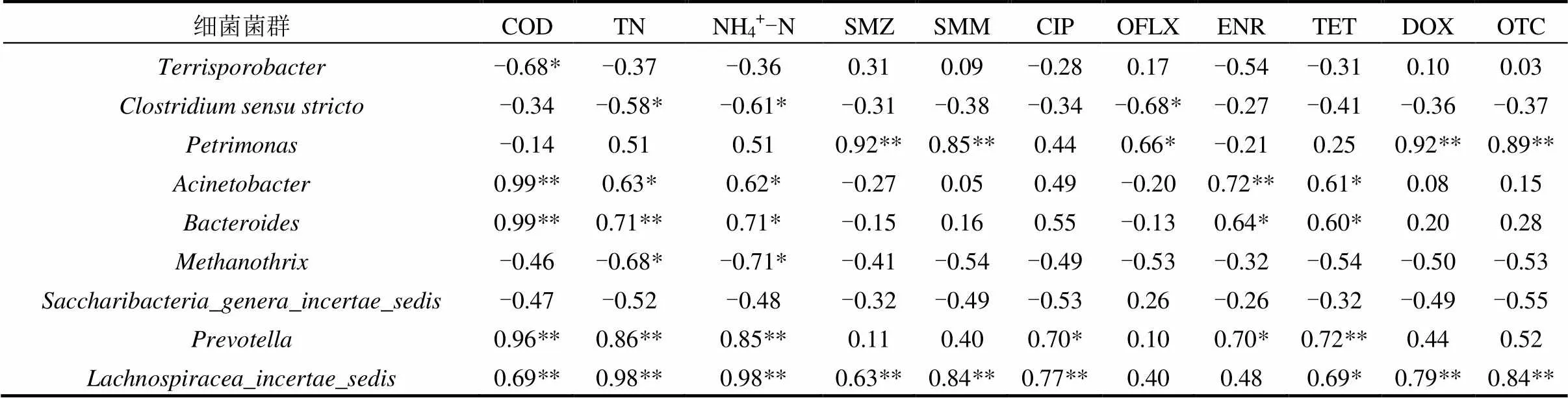
表4 属分类水平上细菌菌群与水质环境因子的Pearson Correlation分析(相对丰度>2%)
注:*表示显著相关(<0.05),**表示极显著相关(<0.01).
与COD、TN、NH4+-N呈显著正相关,与TN、NH4+-N呈显著负相关.在抗生素方面,磺胺类SMZ、SMM和四环素类DOX、OTC与菌群的相关性一致,均与呈极显著正相关;氟喹诺酮类ENR和四环素类TET与菌群的相关性相似,与呈显著正相关;氟喹诺酮类CIP与呈显著正相关,而OFLX与菌群呈显著正相关.
由上述分析结果可知,不同的外部环境因子对于生猪养殖净化系统中的微生物群落组成均有着显著的影响.因此,对于生猪养殖污水的处理,创造合理的生存条件,促进微生物特别是功能菌的增殖,最大程度发挥降解有机物和脱除氮的功能,可能是提高其处理效果的有效途径[2].
3 结论
3.1 本研究的生猪养殖污水净化处理系统中具有极其丰富的生物多样性,共检测得到31个门、548个属的细菌.其中,集污池的优势种群为Firmicutes (41.47%)、Bacteroidetes(30.52%)和Proteobacteria (24.07%);厌氧反应器优势群体为Firmicutes (62.99%)、Bacteroidetes(23.32%)和Proteobacteria (5.15%);在曝气池中,优势细菌种群变为Firmicutes (35.33%)、Verrucomicrobia(12.94%)、Bacteroidetes (12.40%)、Proteobacteria(12.09%)和Candidatus Saccharibacteria(8.28%);进入氧化塘后,优势群体的种类为Firmicutes(37.39%)、Proteobacteria(12.09%)、Chloroflexi(7.84%)、Euryarchaeota(6.12%)和Bacteroidetes(5.63%).
3.2 生猪养殖污水各处理单元中的细菌群落结构随处理进程逐步发生改变,并且存在显著的统计学差异(<0.05).集污池、厌氧反应器、曝气池和氧化塘对各处理单元细菌基因或功能差异有显著影响的物种类群分别为45,17,29以及53类.其中集污池存在显著差异的菌群为Proteobacteria和Bacteroidales,厌氧反应器中存在显著差异的门类为Firmicutes和Spirochaetes,曝气池显著差异的门类为Verrucomicrobia,而氧化塘中检测到众多与其他处理单元存在显著差异的菌群,隶属于Proteobacteria、Planctomycetes、Euryarchaeota、Deinococcus- Thermus、Chloroflexi和Actinobacteria.
3.3 本研究的生猪养殖污水中的细菌群落与水质环境因子之间存在显著相关(<0.05).其中, Acinetobacter、Bacteroides、Prevotella、Lachnospiracea_incertae_sedis与COD、TN、NH4+-N呈显著正相关,Clostridium sensu strict、Methanothrix与TN、NH4+-N呈显著负相关.在抗生素方面,SMZ、SMM、DOX、OTC与Petrimonas、Lachnospiracea_ incertae_sedis、Clostridium XlVa、Tissierella呈极显著正相关,ENR、TET与Acinetobacter、Bacteroides、Prevotella呈显著正相关,CIP与Prevotella、Lachnospiracea_incertae_sedis呈显著正相关,OFLX与菌群Clostridium sensu stricto、Petrimonas呈显著正相关.
[1] 庄榆佳,高阳俊,邓玉君,等.微生物固化曝气技术对养殖废水的深度处理 [J]. 环境化学, 2015,34(7):1356-1362. Zhuang Yu-jia, Gao Yang-jun, Deng Yu-jun, et al.Advanced treatment of swine wastewater by the immobilized-microorganism and aeration technology [J]. Environmental Chemistry, 2015,34(7):1356-1362.
[2] 翟一帆,袁青彬,胡 南.养猪废水处理系统微生物群落结构变化及影响因素研究 [J]. 水资源保护, 2018,34(1):88-94. Zhai Yi-fan, Yuan Qing-bin, Hu Nan. Study on structure variation and influencing factors of microbial community in swine wastewater treatment system [J]. Water Resources Protection, 2018,34(1):88-94.
[3] 申童童,袁林江,温丹丹,等.侧流污泥返送到缺氧池对A2/O系统效能影响研究[J]. 中国环境科学, 2018,38(9):3335-3342. Shen Tong-tong, Yuan Lin-jiang, Wen Dan-dan, et al. Effect of side- stream sludge returning to anoxic tank on A2/Osystem effectiveness [J]. China Environmental Science, 2018,38(9):3335-3342.
[4] 杜 龑,周北海,袁蓉芳,等.UASB-SBR工艺处理规模化畜禽养殖废水 [J]. 环境工程学报, 2018,12(2):497-504. Du Yan, Zhou Bei-hai, Yuan Rong-fang, et al. Treatment of large- scale livestock wastewater by UASB-SBR process [J]. Chinese Journalof Environmental Engineering, 2018,12(2):497-504.
[5] 支 尧,张光生,钱 凯,等.生物吸附/MBR/硫铁自养反硝化组合工艺优化研究[J]. 中国环境科学, 2018,38(6):2097-2104. Zhi Yao, Zhang Guang-sheng, Qian Kai, et al. Research on optimal operation by a combined biological adsorption-MBR-Sulfur/Iron autotrophic denitrification process [J]. China Environmental Science, 2018,38(6):2097-2104.
[6] 陈 晴,王毅力,赵 丽,等.ABR-MABR耦合工艺处理畜禽养殖废水的同步启动[J]. 环境科学研究, 2017,30(2):298-305. Chen Qing, Wang Yi-li, Zhao Li, et al. Synchronous Start-Up of Anaerobic Baffled Reactor-Membrane Biofilm Bioreactor (ABR- MABR) Coupling Process Treating Synthetic Livestock and Poultry Wastewater [J]. Research of Environmental Sciences, 2017,30(2): 298-305.
[7] 赵 丽,王毅力.AMBR-MABR耦合工艺处理模拟畜禽养殖废水的启动和运行[J]. 环境工程学报, 2017,11(11):5799-5809. Zhao Li, Wang Yi-li. Start-up and operation of anaerobic migrating blanket reactor-membrane biofilm bioreactor (AMBR-MABR) coupling process treating synthetic livestock wastewater [J]. Chinese Journal of Environmental Engineering, 2017,11(11):5799-5809.
[8] Cydzikkwiatkowska A, Zielińska M. Bacterial communities in full-scale wastewater treatment systems [J]. World Journal of Microbiology & Biotechnology, 2016,32(4):66.
[9] 王维奇,王秀杰,李 军,等.包埋厌氧氨氧化的脱氮特性及其微生物群落结构[J]. 中国环境科学, 2018,38(9):3343-3350. Wang Wei-qi, Wang Xiu-jie, Li Jun, et al. Nitrogen removal characteristics and microbial community structure analysis of entrapped anaerobic ammonium oxidizingbacteria [J]. China Environmental Science, 2018,38(9):3343-3350.
[10] Fang F, Han H, Zhao Q, et al. Bioaugmentation of biological contact oxidation reactor (BCOR) with phenol-degrading bacteria for coal gasification wastewater (CGW) treatment [J]. Bioresource Technology, 2013,150:314-320.
[11] Liu X, Chen Y, Zhang X, et al. Aerobic granulation strategy for bioaugmentation of a sequencing batch reactor (SBR) treating high strength pyridine wastewater [J]. Journal of Hazardous Materials, 2015,295:153-160.
[12] Chen Y, Lan S, Wang L, et al. A review: Driving factors and regulation strategies of microbial community structure and dynamics in wastewater treatment systems [J]. Chemosphere, 2017,174:173-182.
[13] Renbao L, Cheng M P, Wu M C, et al. Use of metagenomic approaches to isolate lipolytic genes from activated sludge [J]. Bioresource Technology, 2010,101(21):8323-8329.
[14] Garciaarmisen T, Anzil A, Cornelis P, et al. Identification of antimicrobial resistant bacteria in rivers: insights into the cultivation bias [J]. Water Research, 2013,47(14):4938-4947.
[15] Li Y, Adams J, Shi Y, et al. Distinct Soil Microbial Communities in habitats of differing soil water balance on the Tibetan Plateau [J]. Scientific Reports, 2017,7:46407.
[16] Kallenbach C M, Frey S D, Grandy A S. Direct evidence for microbial-derived soil organic matter formation and its ecophysiological controls [J]. Nature Communications, 2016,7:13630.
[17] Barton W, Penney N C, Cronin O, et al. The microbiome of professional athletes differs from that of more sedentary subjects in composition and particularly at the functional metabolic level [J]. Gut, 2018,67(4):625-633.
[18] GB 18596-2001 畜禽养殖业污染物排放标准 [S]. GB 18596-2001 Discharge standard of pollutants forlivestock and poultry breeding [S].
[19] GB 12999-1991 水质采样样品的保存和管理技术规定 [S]. GB 12999-1991 Water quality sampling-Technical regulation of the preservation and handling of samples [S].
[20] Magoä T, Salzberg S L. FLASH: fast length adjustment of short reads to improve genome assemblies [J]. Bioinformatics, 2011,27(21): 2957-2963.
[21] Martin M. Cutadapt removes adapter sequences from high-throughput sequencing reads [J]. Embnet Journal, 2011,17(1):10-12.
[22] Schmieder R, Edwards R. Quality control and preprocessing of metagenomic datasets [J]. Bioinformatics, 2011,27(6):863-864.
[23] Edgar R C. Search and clustering orders of magnitude faster than BLAST [J]. Bioinformatics, 2010,26(19):2460-2461.
[24] Ye L, Shao M F, Zhang T, et al. Analysis of the bacterial community in a laboratory-scale nitrification reactor and a wastewater treatment plant by 454-pyrosequencing [J]. Water Research, 2011,45(15):4390- 4398.
[25] Schloss P D, Westcott S L, Ryabin T, et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities [J]. Applied & Environmental Microbiology, 2009,75(23):7537-7541.
[26] Caporaso J G, Kuczynski J, Stombaugh J, et al. QIIME allows analysis ofhigh-throughput communitysequencingdata [J]. NatMethods, 2010, 7(5):335-336.
[27] HJ/T 399-2007 水质化学需氧量的测定快速消解分光光度法. HJ/T 399-2007 Water quality-Determination of the chemical oxygen demand-Fast digestion-Spectrophotometric method.
[28] HJ636-2012 水质总氮的测定碱性过硫酸钾消解紫外分光光度法. HJ636-2012 Water quality-Determination of total nitrogen-Alkaline potassium persulfate digestion UV spectrophotometric method.
[29] HJ/T 535-2009 水质氨氮的测定纳氏试剂分光光度法. HJ/T 535-2009 Waterquality-Determinationofammonianitrogen- Nessler’sreagentspectrophotometry.
[30] 郭欣妍,王 娜,郝利君,等.超高效液相色谱/串联质谱法同时测定水、土壤及粪便中25种抗生素 [J]. 分析化学, 2015,(1):13-20. Guo Xin-Yan, Wang Na, Hao Li-Jun, et al. Simultaneous detection of 25kinds of veterinary antibiotics in soil, manure and water samples using liquid chromatography_tandem mass spectrometry [J]. Chinese journal of analytical chemistry, 2015,(1):13-20.
[31] Oksanen J B F, Kindt R, Legendre P, et al. Vegan: community ecology R package, v2.0-10 [Z]. https://CRAN.R-project.org/package=vegan/ 2013-12-12.
[32] Warnes G R, Bolker B, Bonebakker L, et al. gplots: Various R programming tools for plotting data.https://CRAN.R-project.org/ package=gplots /2016-03-30.
[33] Segata N, Izard J, Waldron L, et al. Metagenomic biomarker discovery and explanation [J]. Genome Biology, 2011,12(6):R60.
[34] 朱映红.微生物强化猪场沼液脱氮工艺研究 [D]. 武汉:华中农业大学, 2015. Zhu Ying-hong. Research on high-perform ancenitrogen removalofpiggerybiogaseffluentbymicrobialbio-augmentation [D]. Wuhan: Huazhong Agricultural University, 2015.
[35] 刘元望,李兆君,冯 瑶,等.微生物降解抗生素的研究进展[J]. 农业环境科学学报, 2016,35(2):212-224.Liu Yuan-wang, Li Zhao-jun, Feng Yao, et al. Research progress in microbial degradation of antibiotics [J]. Journal of Agro-Environment Science, 2016,35(2):212-224.
[36] 靳红梅,黄红英,管永祥,等.规模化猪场废水处理过程中四环素类和磺胺类抗生素的降解特征[J]. 生态与农村环境学报, 2016,32(6): 978-985.Jin Hong-mei, Huang Hong-ying, Guan Yong-xiang, et al. Characteristics of degradation tetracyclines and sulfonamides during wastewater treating processes in an in-tensive swine farm [J]. Journal of Ecology and Rural Environment, 2016,32(6):978-985.
[37] Wang N, Guo X, Xu J, et al. Pollution characteristics and environmental risk assessment of typical veterinary antibiotics in livestock farms in Southeastern China [J]. Journal ofenvironmental science and health. Part B, 2014,49(7):468-479.
[38] Prado N, Ochoa J, Amrane A. Biodegradation and biosorption of tetracycline and tylosin antibiotics in activated sludge system [J]. Process Biochemistry. 2009,44(11):1302-1306.
[39] Kim S E P, Jensen J N, et al. Removal of antibiotics in wastewater: Effect of hydraulic and solid retention times on the fate of tetracycline in the activated sludge process [J]. Environmental Science & Technology, 2005,39(15):5816-5823.
[40] 杨晓芳,杨 涛,王 莹,等.四环素类抗生素污染现状及其环境行为研究进展[J]. 环境工程, 2014,32(2):123-127. Yang Xiao-fang, Yang Tao, Wang Ying, et al. Research progress in progress in pollution status and environmental behavior of tetracycline antibiotics [J]. Environmental Engineering, 2014,32(2):123-127.
[41] 刘志伟,周美修,宋俊玲,等.复合垂直流人工湿地污染物去除特征及微生物群落多样性分析[J]. 环境工程, 2014,32(2):38-42. Liu Zhi-wei, Zhou Mei-xiu, Song Jun-ling, et al. Pollutants removal characteristics and analysis of microbial communitu diversity in integrated vertical-flow constructed wetland [J]. Environmental Engineering, 2014,32(2):38-42.
[42] Lamendella R, Domingo J W, Ghosh S, et al. Comparative fecal metagenomics unveils unique functional capacity of the swine gut [J]. Bmc Microbiology, 2011,11(1):103.
[43] Wagner M, Loy A. Bacterial community composition and function in sewage treatment systems [J]. Current Opinion in Biotechnology, 2002, 13(3):218-227.
[44] Hu M, Wang X, Wen X, et al. Microbial community structures in different wastewater treatment plants as revealed by 454- pyrosequencing analysis [J]. Bioresource Technology, 2012,117:72– 79.
[45] Ducey T F, Hunt P G. Microbial community analysis of swine wastewater anaerobic lagoons by next-generation DNA sequencing [J]. Anaerobe, 2013,21(6):50-57.
[46] Wang X, Hu M, Xia Y, et al. Pyrosequencing analysis of bacterial diversity in 14wastewater treatment systems in China [J]. Bioresource Technology, 2012,78(19):7042-7047.
[47] Yu K, Zhang T. Metagenomic and metatranscriptomicanalysis of microbial community structure and gene expression of activated sludge [J]. PLoS One, 2012,7(5):e38183.
[48] Cydzik-Kwiatkowska A, Zielińska M. Bacterial communities in full-scale wastewater treatment systems [J]. World J Microbiology & Biotechnology, 2016,32(4):66.
[49] Looft T J T, Allen H K, Bayles D O, et al. In-feed antibiotic effects on the swine intestinal microbiome [J]. Proceedings of the National Academy of Sciences of the United States of America, 2012,109(5): 1691-1696.
Microbial community structure and dynamics in swine wastewater treatment system.
HUANG Wei1, LIU Lan-ying1, WU Miao-hong1, CHEN Li-hua1, LÜ Xin1, YE Mei-feng2, LIN Dai-yan2, SONG Yong-kang1*
(1.Institute of Agricultural Quality Standards and Testing Technology Research, Fujian Academy of Agricultural Sciences, Fuzhou 350003, China;2.Institute of Agricultural Engineering and Technology, Fujian Academy of Agricultural Sciences, Fuzhou 350003, China)., 2019,39(2):839~848
To elucidate the bacterial community dynamics in swine wastewater treatment system, the bacterial community characteristics in different treatment units of swine wastewater was analyzed by Illumina Miseq high-throughput sequencing technologies, and the correlation between bacterial community and water quality or antibiotics was investigated. The results demonstrated that microbial community structure varies greatly in different wastewater treatment processes. The dominant bacteria in fouling pool and anaerobic reator were Firmicutes, Bacteroidetes and Proteobacteria, the dominant bacteria in aeration tan were Firmicutes, Verrucomicrobia, Bacteroidetes, Proteobacteria and Candidatus Saccharibacteria, while in oxidation pond, the dominant bacteria were Firmicutes, Proteobacteria, Chloroflexi, Euryarchaeota and Bacteroidetes. The microbial comminity structure in wasterwater treatment system was significantly correlated with both water quality and antibiotics (<0.05).,,,correlated closely with COD、TN、NH4+-N.correlated closely with TN、NH4+-N.,,,extremely significant positively correlated with sulfamethoxazole, sulfamethoxazole, doxycycline, oxytetracycline.,,significant positively correlated with enrofloxacin and tetracycline.,positively correlated with ciprofloxacin.,positively correlated ofloxacin.
swine wastewater;bacterial community;water quality;antibiotics
X172
A
1000-6923(2019)02-0839-10
黄 薇(1986-),女,江西抚州人,助理研究员,硕士,主要从事环境微生物组学方面研究.发表论文40余篇.
2018-07-03
福建省公益类科研院所专项(2015R1025-8,2018R1018-1);福建省农科院全产业链科技示范项目(kjFW19)
* 责任作者, 研究员, songlibby@sina.com
