大豆赖氨酸合成关键酶基因的克隆与定位分析
2019-01-09陈庆山邱红梅蒋洪蔚李灿东唐敬仙
于 妍, 姜 威, 陈庆山, 邱红梅, 管 宇, 蒋洪蔚, 李灿东, 唐敬仙
(1.长春科技学院,吉林长春 130600; 2.东北农业大学农学院,黑龙江哈尔滨 150030; 3.吉林省农业科学院大豆研究所,吉林长春 130033;4.黑河出入境检验检疫局综合技术中心,黑龙江黑河 164300; 5.黑龙江省农业科学院佳木斯分院,黑龙江佳木斯 154007)
大豆(Glycinemax)是重要的油料作物、经济作物,植物性蛋白质的重要来源之一便是大豆蛋白质,其营养价值较高。大豆中人体必需的氨基酸含量丰富,其中赖氨酸含量远远高于谷物。由于赖氨酸能够促进人体充分吸收和利用其他营养物质,一旦缺乏将直接或间接影响机体的生长和发育,所以进一步提高大豆中的赖氨酸含量十分必要。在植物赖氨酸合成途径中,二氢吡啶、二羧二氢吡啶、二羧酸先后在2,3-二氢吡啶二羧酸还原酶(DHDPR)、二氨基庚二酸异构酶(DAPE)、N-α-酰基-L,L-二氨基庚二酸脱酰酶(ADPD)的催化下生成赖氨酸[1]。表达序列标签(expressed sequence tag,简称EST)已经成为人类克隆不同物种特异性表达基因以及寻找未知基因的重要手段[2-3]。截至目前,已有多项研究通过电子克隆手段获得了许多人类功能基因[4-6]。但是由于目标物种序列信息有限,所以在植物基因克隆研究中,大部分试验方法仍局限于原有的、常规的思路。但是,随着植物EST数据库的逐步完善,其信息量快速增加,借助生物信息学技术来电子克隆基因势必成为基因克隆的一个方便快捷、有效的途径。另外,在分子标记研究领域,目前的研究多是将标记定位在连锁群上的,真正的基因定位鲜见报道。如果以基因自身的序列为依据,进而获得连锁群的定位,这样可以实现基因更加准确精细的定位。
本研究利用电子克隆手段,获得了大豆赖氨酸合成3个关键酶基因的全长cDNA序列,同时以已构建的大豆基因组遗传图和物理图的整合图谱为基础,完成这些基因的定位,并通过cDNA和gDNA序列的比对,获得所有基因的结构信息,从而为后续基因功能的研究及分子辅助育种打下良好基础。
1 材料与方法
1.1 序列信息下载及生物软件
本研究从美国国立生物技术信息中心(National Center for Biotechnology Information,简称NCBI)网站上分别下载了不同外源物种赖氨酸合成3个关键酶基因信息(表1),并用phytozome网站(http://www.phytozome.net/soybean.php)进行在线比对。从phytozome网站(http://soybase.org/SequenceIntro.php)下载获得标记图谱BARCSOYSSR Potential SSR Database。从NCBI的FTP网站(ftp://ftp.ncbi.nih.gov/blast/executables/release/2.2.16/)下载用于本地比对的blast2.2.16软件包。利用DNAMan软件进行EST片段比对,并筛选序列进行拼接。

表1 外源物种赖氨酸合成关键酶基因信息
1.2 试验植物
先将大豆品种东农42的种子在自来水中浸泡过夜,再将吸胀后的种子种在花盆中,于光照培养箱中培养,待长出3出复叶后,将幼嫩的叶片取下用作试验材料。
1.3 电子克隆
本研究依据不同物种间存在相同基因核苷酸序列同源性较高的特点,借助已构建的本地化大豆EST数据库,将已获得的不同物种的赖氨酸合成关键酶基因序列利用Blast软件与其进行比对,从比对出的数条EST序列中选取多条同源性高,且能完全覆盖整个基因cDNA片段的EST序列才可进行后续拼接工作。利用DNAMan 5.0软件的Multiple sequence alignment功能将目的基因与其同源序列进行联配分析,之后运用Sequence Assembly工具对所选EST序列进行进一步拼接组装完成电子延伸,进而获得全长基因序列。
1.4 引物设计
以拼接预测的基因序列为模版,利用DNAMan软件设计引物,引物序列见表2。引物委托生工生物工程(上海)股份有限公司合成。

表2 赖氨酸合成关键酶基因引物信息
1.5 大豆叶片总RNA提取及第一链cDNA的合成
依据TRIzol说明书提供的方法进行大豆叶片总RNA的提取。参照M-MLV使用说明书反转录合成cDNA的第一链,直接用作模板用于PCR扩增。
1.6 基因的克隆及测序
以东农42大豆品种cDNA作为模板,利用已设计的大豆DAPD、DAPE和DHDP3个酶基因特异引物,借助PCR扩增全长基因片段。反应条件如下:94 ℃ 5 min;94 ℃ 35 s,60 ℃ 35 s,72 ℃ 2 min,28个循环;72 ℃ 7 min。待反应结束后,分别取10 μL PCR产物于10 g/L琼脂糖凝胶中进行电泳检测。回收目的片段并连接到pGEM T-easy载体上,转化大肠杆菌(Escherichiacoli) DH5α感受态细胞,在氨苄青霉素(Amp)抗性X-gal/IPTG LB培养基平板上将白色克隆挑出,并送交生工生物工程(上海)股份有限公司进行测序。
1.7 基因的电子定位
将已获得的3个大豆赖氨酸合成关键酶基因序列与本地数据库进行比对,将获得的序列与从phytozome网站下载的数据库再一次进行比对分析,由此便可准确地将基因定位在遗传图谱上。结合Song等已构建的图谱[7],进而确定这些基因定位的连锁群以及距基因较近的两侧标记信息。
1.8 基因结构分析
根据比对分析结果,进一步确定基因的内含子与外显子数量,从而获得基因的精细结构。
2 结果与分析
2.1 大豆赖氨酸合成关键酶基因的电子克隆
2.1.1 基因的电子预测 本试验将获得的不同外源基因序列与已构建的大豆本地化EST数据库进行比对,从比对结果中选取多条同源性高的序列,并进行拼接,进而获得全长基因,从而预测出目的基因的全长序列。经比对、拼接,大豆赖氨酸合成3个关键酶基因的全长序列预测信息见表3。

表3 大豆赖氨酸合成关键酶基因的预测信息
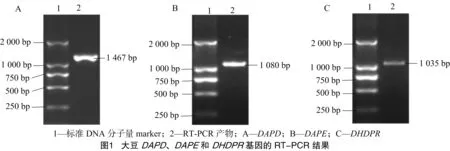
2.1.2 基因的RT-PCR鉴定与测序结果分析 以大豆品种东农42叶片总RNA反转录的cDNA为模板,通过RT-PCR扩增,获得大豆DAPD、DAPE和DHDPR3个关键酶基因PCR产物,经10 g/L琼脂糖凝胶电泳检测,结果显示泳道均出现单一条带,DAPD、DAPE、DHDPR3个关键酶基因大小分别约为1 460、1 080、1 030 bp,全部与预测的基因长度相符(图1)。将3个PCR产物分别与pGEM-T easy载体连接,进而获得对应的重组质粒。最后将重组质粒进行PCR扩增,经电泳检测,证实结果均与预测基因长度相符。
借助DNAMan软件,将大豆赖氨酸合成DAPD、DAPE和DHDPR3个关键酶基因重组质粒的测序结果与之前的预测结果进行比对分析,结果显示,在核苷酸水平上,所获得的3个基因cDNA序列与预测序列的同源性分别为99.66%、99.17%、95.94%。利用EBI→InterProScan分别对DAPD、DAPE和DHDPR3个酶基因编码的蛋白保守结构域进行预测。由表4可以看出,DAPD、DAPE、DHDPR3个关键酶基因含有的保守结构域分别具有2,6-二氨基庚二酸脱羧酶、二氨基庚二酸异构酶和2,3-二氢吡啶二羧酸还原酶功能。通过以上分析,进一步验证了通过电子克隆获得的这3个全长基因序列是正确的。
2.2 大豆赖氨酸合成关键酶基因的电子定位与结构分析
2.2.1 基因的物理图定位 借助大豆基因组数据库,将已获得的基因序列片段与之进行比对,进而完成物理图定位分析。如果与基因组进行比对时出现多个片段同时匹配的情况,是由于该基因具有内含子。表5中显示了基因以及与基因组匹

表4 大豆赖氨酸合成关键酶基因编码蛋白保守结构域信息

表5 大豆赖氨酸合成关键酶基因的物理图定位
配区段的信息,此外还可以看出,E值均趋近于0,平均匹配率均在99%以上。这些数据足以说明,这些基因与基因组序列的匹配是真实且可信的。
2.2.2 基因的遗传图定位 根据赖氨酸合成3个关键酶基因与基因组匹配区段,同时参考标记图谱BARCSOYSSR Potential SSR Database,DAPD、DAPE和DHDPR3个基因分别定位在L、N、J 3个连锁群上,获得了基因的两侧标记以及定位区间。由表6、图2可以看出,DAPD基因被定位在L连锁群Super40的314 760~319 421之间,且BARC-014655-01607-BARC-028787-06014是分别位于此区间两侧最近的标记,距离是1.1 cM;DAPE基因被定位在N连锁群Super62的2 825 711~2 829 438之间,且BARC-024329-04849-BARC-028205-05792是分别位于此区间两侧最近的标记,距离是0.6 cM;DHDPR基因被定位在J连锁群Super154的1 521 652~1 524 809之间,BARC-022453-04332-BARC-014459-01381是分别位于此区间两侧最近的标记,且定位于连锁群的同一位置,定位距离是3个基因中最近的。

表6 大豆赖氨酸合成关键酶基因遗传图基因定位
2.2.3 基因结构分析 通过对大豆赖氨酸合成3个关键酶基因进行cDNA和gDNA序列的比对,进一步确定各个基因长度以及外显子、内含子的数量。表7结果显示,DAPD、DAPE、DHDPR3个酶基因cDNA长度分别为1 467、1 080、1 035 bp;gDNA的长度分别为4 662、3 728、3 158 bp;外显子数量分别为8、9、8个;内含子的数量分别为7、8、7个。

表7 赖氨酸合成关键酶基因结构比对信息
3 讨论与结论
3.1 大豆赖氨酸合成关键酶基因的电子克隆
目前,电子克隆方法已经被广泛应用于很多领域,例如在人[8]、甘蓝[9]和牛[10]的研究中均有报道。但大部分研究仅局限于利用外源物种核苷酸或氨基酸序列与GenBank网站上提供的EST数据库进行比对,且由于EST数据库中的信息包含多个物种,所以造成信息量过大,因而比对起来必然存在以下问题:(1)时效性差;(2)会将许多不需要的物种信息同时比对出来,所以不仅须要比对多次,另外还须对结果进行细致筛选,为序列拼接带来不必要的麻烦。本研究在成功构建大豆EST数据库的基础上进行本地比对,这样针对性更强,结果更准确。由此可见,以本地化EST数据库为基础进行基因电子克隆,既准确又快捷。
3.2 大豆赖氨酸合成关键酶基因的电子定位
目前,针对大豆脂肪酸含量[11]、种子油分和蛋白[12]、大豆花叶病毒抗性[13]及胞囊线虫抗性[14]等性状的基因定位均有报道,但关于基因方面的研究并不多见。本研究借助已有的大豆基因信息,构建了本地化EST数据库。同时利用生物学软件,完成了基因准确、快速的定位。而传统的分子标记,需要一定的群体以及大量人力、物力,既耗时又费力。基因电子定位是借助生物信息学相关软件并建立在测序基础之上的另一种用于基因定位的手段,其获得的基因两侧标记与基因位点之间的物理距离非常近,可为今后基于基因分子标记的开发奠定良好基础,从而真正完成基因准确精细的定位。
由本试验结果还可以看出,通过基因的电子定位,不仅能够更加快速、准确地相互找到对应标记,同时还能获得相邻序列的信息,这样就可以挖掘更多未知的功能性基因,进而完成QTL到QTG更深入的转换,同时也很好地将挖掘基因与遗传作图二者密切地联系在一起。除此之外,电子定位还可将基因cDNA序列和gDNA序列进行比对,同时对精细的基因结构,即内含子与外显子信息进行分析,为后续进一步研究基因提供了必要条件。目前基于基因序列的研究大部分仍然局限于cDNA水平, 却忽视了基于gDNA水平上基因结构的分析,本研究对这方面内容进行了有效的补充。

