洛阳、吉林生态区谷子抗倒伏性的全基因组关联分析
2018-12-29贾小平全建章王永芳董志平袁玺垒李剑峰
贾小平,张 博,全建章,王永芳,董志平,袁玺垒,李剑峰
(1.河南科技大学 农学院,河南 洛阳 471023;2.河北省农林科学院 谷子研究所/国家谷子改良中心,河北 石家庄 050035)
倒伏是农作物的茎秆从相对的直立形态发生不可逆错位的现象[1],作物的倒伏将会对群体的结构和茎秆的输导能力造成一定程度的破坏,并且能够显著地降低光合效率和光合作用产物向穗部运输的能力,最终造成作物大量减产[2]。谷子作为一种重要的杂粮作物,具有光合效率高、耐旱耐瘠、营养均衡等特点,近些年广受人们的喜爱,然而,由于茎秆偏软、种植密度较大,在遭遇风雨气候等因素时,谷子容易发生倒伏,对产量和商品价值造成极大的影响[3]。造成谷子倒伏的因素有外因与内因两个方面:内因是指品种的区别,不同谷子品种间抗倒伏性存在巨大的差异;外因则主要包含具有灾害性的气候(例如暴雨、冰雹、大风等)、施肥情况、病虫害、栽培密度[4]。施肥和栽培密度配合可以一定程度上改善水稻茎秆理化特性,有效提高抗倒伏性[5]。通过分析施肥对谷子抗倒伏性和产量的影响,发现不同品种对施肥处理的反应不同,有些品种抗倒伏能力提高而有些却降低,这主要取决于品种对肥料的需求与否[6]。也有研究发现矮化处理后能有效防止谷子倒伏[7]。
目前有关谷子抗倒伏性评价的研究较多,如袁立新[2]研究发现,谷子的株型与茎秆的倒伏性状关系十分密切,茎部性状中的株高、节间长度和穗部性状中的穗长、穗柄长、穗弯曲度与谷子的抗倒伏性呈现出显著负相关,而茎部性状茎粗和谷子根量则与谷子的抗倒伏性呈现出正相关。研究结果表明,株型相关性状的选择对于选育出抗倒伏性强的谷子品种具有重要意义;袁志华等[8]建立的力学模型可用来评估谷子的抗倒伏能力;刘艳丽等[9]应用倒伏系数研究表明,倒伏性状对于谷子的产量、品质和机械化收获等有着明显的影响,同时确定了对谷子倒伏性有着重要影响的一些关键性状指标;贾小平等[10]进行了不同谷子品种(系)的抗倒性分析,发现谷子的倒数第2茎节倒伏指数对谷子植株的倒伏性起到关键作用。
当前对谷子倒伏性的研究主要集中在形态学特征、理化特性方面,有关抗倒伏性的QTL定位等分子水平的研究尚未见报道,在模式作物水稻中相关研究报道较多[11-13]。近些年谷子基因组学研究取得了重大进展,全基因组测序工作已经分别由中国和美国完成[14-15],而基于重测序的全基因组关联分析方法已经广泛应用于植物QTL定位研究[16-17]。Jia等[18]首次对916份谷子材料进行了重测序,开发了250多万个SNP标记,并用其中80万个SNP标记构建了谷子基因组单倍型图谱,对47个性状进行了全基因组关联分析,为开展谷子全基因组关联分析研究提供了借鉴。本研究选择98份来自国内外不同地区的谷子材料,在洛阳、吉林两地调查了这98份谷子材料的倒伏率,并对这些谷子材料进行基因组重测序,开发大量的SNP标记,进行突变位点与抗倒伏性的全基因组关联分析,发掘与抗倒伏性显著相关的SNP位点,筛选抗倒伏性候选基因,为进一步定位、克隆控制谷子抗倒伏性的基因奠定基础。
1 材料与方法
1.1 试验材料及种植方法
本研究所用的98份谷子材料由中国农业科学院作物科学研究所(国家作物遗传育种改良中心)提供(表1),98份谷子材料于2016年5月中旬分别播种于河南科技大学试验田(洛阳)、吉林市农业科学院试验田(吉林),株距5 cm,行距40 cm,每品种种植两行,行长2 m。
1.2 谷子抗倒伏性的测定
在谷子成熟期调查倒伏率,每个品种除去行两端各2株,统计每一品种的总株数,并仔细调查出每个品种的倒伏株数,倒伏的标准为茎秆与地面的倾角小于45°,计算倒伏率。
1.3 谷子基因组DNA的提取及重测序
在洛阳试验田当98份谷子材料长到3叶期时,采集幼嫩叶片保存于液氮中,随后用2×CTAB法提取基因组DNA。质量合格的DNA送上海美吉生物工程有限公司进行基因组文库构建,用Illumina HiSeqTM进行测序。
1.4 突变位点检测和注释
运用BWA软件将质控后的测序片段与谷子参考基因组序列进行比对,利用GATK进行SNP检测。对于检测到的SNP位点,用Samtools提供的vcfutils工具对其过滤。测序深度大于等于3,即支持该变异的测序片段数目要大于等于3,质量值Qual>50.0。然后利用ANNOVAR软件对由多个基因组检测出的基因变异进行功能注释。
1.5 群体结构分析及连锁不平衡分析
选择均匀分布于谷子9条染色体上的27 000个SNP标记,用STRUCTURE Version 1.8软件对98份谷子材料进行群体结构分析,首先假设98份谷子材料的分群数(K值)为7,进行聚类,根据CV error(cross validation error)最低点对应的K值来确定最佳分群数。用VCFtools软件计算标记间连锁不平衡系数(r2),进行连锁不平衡分析,r2越接近1,代表连锁关系越强。
1.6 抗倒伏性的关联分析及候选基因预测
利用Tassel5.0软件进行关联分析,采用混合线性模型(mixed linear model,MLM),同时对群体结构和个体亲缘关系进行校正,以减少计算误差。以群体结构得到的Q值和TASSEL软件计算各材料之间的K系数矩阵作为协变量,以P Marker值来衡量关联标记的显著性。用Manhattan图来显示关联分析结果,在检测到与抗倒伏性显著相关的标记位点两侧200 kb范围内寻找候选基因,并在GO数据库对候选基因进行功能注释。
2 结果与分析
2.1 洛阳、吉林两地谷子倒伏率调查
在洛阳地区,谷子材料倒伏率变异范围在0~42.5%,倒伏率为0的品种最多,占有效调查品种数目的50%以上,其次为倒伏率在10%以内的品种,约占有效调查品种数的39%,倒伏率在15%以上的品种最少,约占有效调查品种数的5%。在吉林地区,谷子材料倒伏率变异范围在0~30%,与洛阳调查结果相似,倒伏率为0的品种最多,也达到有效调查品种数的50%以上,其次是倒伏率在10%~15%的品种,占有效调查品种数的29%,倒伏率在15%以上的品种最少,约占有效调查品种数的14%(图1)。总体来看,洛阳、吉林两地谷子倒伏率分布规律表现一定的一致性。
2.2 基于重测序谷子SNP标记的开发
通过对98份谷子材料进行基因组重测序检测到9 740 706个SNP,经过滤获得4 482 208个高质量的SNP标记,这些SNP标记中有4 037 579个位于基因间隔区,约占SNP总数的90%;其次为分布在内含子区域、转录起始位点的上游区域以及基因下游1 kb区域的SNP位点,其相应数目为122 698、102 355和81 070;剩下的SNP主要分布于外显子区、5′非翻译区、3′非翻译区以及非编码RNA的外显子、内含子区域(表1)。

图1 洛阳(A)、吉林(B)两地倒伏率分布情况Fig.1 Distribution of lodging percentage in Luoyang(A) and Jilin (B)

表1 重测序开发的SNP位点统计
2.3 98份谷子品种群体结构分析
在SNP标记开发的基础上,通过STRUCTURE软件分析98个样品的群体结构,分别假设98个样品的分群数,即K值为1~7,进行聚类分析。根据CV error最低点对应的K值来确定最佳分群数为3(图2-A)。以各品种的遗传成分值Q>0.5作为标准进行品种归类:亚群1包括了44个品种,占总品种的44.9%,主要源自河南、河北、吉林、甘肃,还包括部分黑龙江、辽宁、陕西、山东、内蒙古、四川、山西的品种,2个北京品系和2个日本引进品系、1个美国引进品系;亚群2包括了10个品种,占总品种的10.2%,主要源自国内,包括陕西、新疆、宁夏、湖南、吉林、甘肃、河南各地区的品种;亚群3包括了38个品种,占总品种的38.8%,主要源自河北、河南、山西,还包括了黑龙江、辽宁、吉林、山东、新疆、宁夏、甘肃、内蒙古的部分品种,以及朝鲜、美国、日本、南非各一个引进品系。混合型亚群包括了6个品种,占总品种的6.1%,主要源自黑龙江,还包括了河南、吉林、河北,以及一个法国引进品系。98份材料的群体结构较简单,有效减弱了群体结构对关联分析所造成的干扰(图2-B)。
2.4 连锁不平衡分析
通过计算覆盖谷子全基因组的4 482 208个SNP标记间的连锁不平衡系数(r2)来分析谷子群体全基因组的LD水平,而常用r2衰退到一半时所对应的距离作为连锁不平衡的衰减的数值。由图3可知,伴随着SNP间距的不断增加,谷子基因组的r2在逐渐下降,r2衰退到一半时LD-decay长度为47.5 kb,说明谷子基因组平均LD衰减距离是47.5 kb,因此可以在47.5 kb范围对应的LD区域内扫描抗倒伏性相关候选基因。
2.5 谷子抗倒伏性的全基因组关联分析
在洛阳生态区,检测到与倒伏率存在显著关联的SNP位点有764个(P<10-4),这些位点分布在谷子所有9个染色体上,其中8号染色体分布的SNP位点最多,为221个;其次为9号染色体,达到141个;3号、5号染色体分布的SNP位点数也达到104、102个;6号染色体分布的SNP数目最少,为15个。在吉林生态区,检测到24个SNP位点与倒伏率显著关联(P<10-4),这些位点分布在谷子1~8号染色体上,其中5号、8号染色体分布的SNP位点最多,均为6个;其次为2号染色体和1号染色体,分布的SNP位点数分别为4个和3个;其余染色体分布的SNP数目为1~2个(图4)。
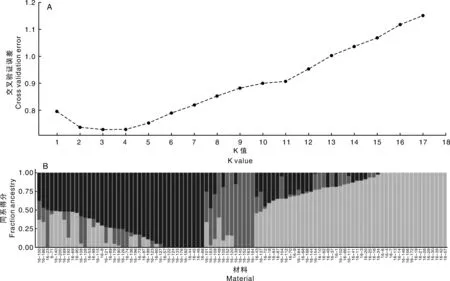
A,K值分布曲线;B,98份谷子材料群体结构。A,K value distribution curve; B,Population structure of 98 foxtail millet materials.图2 九十八份谷子材料群体结构分析Fig.2 Population structure analysis of 98 foxtail millet materials

横坐标代表SNP 的间距,纵坐标代表r2值。r2衰退到一半时对应的SNP 间距即为LD-decay。The abscissa represented the spacing of SNP; the ordinate represented the r2 value.The corresponding SNP spacing was LD-decay when r2 fell to half.图3 LD衰减分布图Fig.3 LD decay distribution diagram
在关联SNP位点两侧区域200 kb的范围搜索候选基因,洛阳生态区在谷子1~9号染色体上均检测到候选基因,其中激酶类基因分布在谷子的1~8号染色体上,且数量最多,包括LRR受体类丝氨酸/苏氨酸蛋白激酶、L-型受体激酶、亮氨酸重复受体类丝氨酸/苏氨酸蛋白激酶、蛋白激酶G11A、细胞壁关联受体激酶等,抗病蛋白数量也较多,分布在除1号、4号、5号、6号染色体之外的其他5条染色体上。此外,三角状五肽蛋白、糖类分解、转移、运输相关的蛋白,如糖转运蛋白、蔗糖运输蛋白、1,4-α-葡聚糖分支酶、EGF结构域特异性O-连接N-乙酰葡糖胺转移酶、葡萄糖醛酸转移酶、葡聚糖内切-1,3-β-葡糖苷酶、β-1,4-甘露糖基糖蛋白4-β-N-乙酰葡糖胺基转移酶、多聚半乳糖醛酸酶、UDP糖基转移酶也在各染色体广泛分布,关联到数量较少的基因包括GA合成途径相关酶类、E3泛素-蛋白连接酶、液泡蛋白分选相关蛋白、含有NAC结构域的蛋白质及细胞色素P450相关蛋白,此外还关联到一些微管蛋白、肌动蛋白、甲基转移酶及生长素合成途径的一些酶相关基因。吉林生态区在谷子1~8号染色体上均关联到抗倒伏性相关候选基因,但是数量远少于洛阳生态区,主要集中在5号、6号、8号染色体上。吉林关联到的候选基因同样是激酶类最多,包括丝氨酸/苏氨酸蛋白激酶、受体类蛋白激酶、LRR受体类丝氨酸/苏氨酸蛋白激酶和细胞壁关联受体。此外关联到的候选基因还有UDP糖基转移酶、细胞色素P450相关、甲基转移酶、驱动蛋白、几丁质酶类蛋白质等少数基因。
洛阳、吉林两地都关联到较多的激酶类基因,特别是6号染色体上两地都关联到同一个候选基因——LRR受体类丝氨酸/苏氨酸蛋白激酶基因(LOC101776261)。此外,在5号、6号、8号染色体上还关联到一些位点不同但功能接近的蛋白激酶基因,如5号染色体上洛阳关联到基因(LOC101754749)和吉林关联到的基因(LOC101756923)均为受体类蛋白激酶,6号、8号染色体上两地均关联到一些细胞壁相关的受体激酶基因(LOC105914550、LOC101786429、LOC105913474、LOC101766879),还有一个8号染色体上的激酶基因(LOC101760794),在洛阳地区检测到的也是LRR受体类丝氨酸/苏氨酸蛋白激酶基因,与6号染色体洛阳、吉林两地共同检测到的激酶基因类似。还有一些其他类型的基因同样虽然在两地位于不同的染色体上,但是功能很接近,如甲基转移酶LOC101766453和LOC101771920、UDP糖基转移酶LOC101764556和LOC101753540等(表2)。

图4 洛阳(A)、吉林(B)关联分析结果的曼哈顿图Fig.4 Manhattan map of correlation analysis results in Luoyang (A) and Jilin (B)

表2 洛阳、吉林关联到的抗倒伏性候选基因

续表2

续表2

续表2
3 讨论
倒伏是影响作物产量和品质的重要因素,定位控制倒伏性的基因位点可以揭示抗倒伏性的遗传机制,为利用分子育种技术培育抗倒伏作物新品种奠定基础。目前有关抗倒伏性的QTL定位、基因克隆方面的研究在水稻中报道较多。研究表明,控制水稻株型、茎秆性状、细胞壁性状的相关基因和抗倒伏性关系密切,如和株高相关的赤霉素合成途径基因SD1突变可以导致植株矮化,提高抗倒伏能力[19];DEP1为控制水稻直立穗型的主要基因,具有直立穗型的水稻品种抗倒伏性明显增加[20]。与水稻茎秆物理特性相关的基因SMOS1编码一个转录因子,通过生长素依赖形式调节细胞伸长,该基因突变能增加秆壁厚度,提高抗倒伏性[21];控制水稻茎秆硅含量的硅转运蛋白基因突变会导致抗倒伏性、抗病性降低[22]。与水稻次生细胞壁相关的基因与抗倒伏性密切相关,如对香豆酸辅酶A连接酶是木质素合成的关键酶,编码该酶的基因表达量下调会导致植株变矮,茎秆强度变低[23]。
本研究通过全基因组关联分析,在洛阳、吉林两地均检测到较多的蛋白激酶基因,特别是在谷子6号染色体,洛阳、吉林都检测到同一个LRR受体类丝氨酸/苏氨酸蛋白激酶基因(LOC101776261),在6号、8号染色体还检测到一些细胞壁关联的类受体激酶基因(LOC105914550、LOC101786429、LOC105913474、LOC101766879)。目前报道的类受体蛋白激酶多数集中在植物抗病防御反应,有关在抗倒伏性方面的报道极少[24]。近期,华中农业大学王石平教授领导的团队发现水稻抗白叶枯病主效基因Xa4编码一个细胞壁相关的激酶,该激酶通过促进纤维素的合成增强了细胞壁的强度,不仅防御了白叶枯病菌的侵染,而且增加了茎秆机械强度,提高了水稻抗倒伏性[25]。据此推测,本研究关联到的LRR受体类丝氨酸/苏氨酸蛋白激酶基因、细胞壁相关的受体激酶基因可能与谷子抗倒伏性有密切关系。此外,一些虽然在洛阳、吉林两地检测到的位点不同但功能接近的基因,如细胞色素P450相关蛋白、UDP糖基转移酶基因、驱动蛋白基因、甲基转移酶基因也可能与抗倒伏性有一定关系,这些基因仍需要进一步进行功能验证。
