传统自然发酵黏豆包面团微生物菌群结构分析
2018-10-08赵烜影苑秀娟董艳如
赵烜影,苑秀娟,郭 鸰,*,董艳如,孙 琦
(1.东北农业大学食品学院,乳品科学教育部重点实验室,黑龙江 哈尔滨 150030;2.安达市畜牧兽医局,黑龙江 安达 151400)
发酵食品是指以微生物发酵为基础而制作的一类食品的总称,常见的发酵食品按原料来源可以分为发酵豆类制品、发酵谷类制品、发酵肉制品、发酵蔬菜类制品、发酵乳制品以及发酵茶制品[1]。在中国谷类发酵食品是最常见的主食,主要有馒头、醪糟、面包、醋、酒、发酵米粉、面酱及发面饼类等[2]。馒头、面包、包子、发面饼和黏豆包等需要将粉碎的面粉与其他成分(液体)揉捏形成面团作为发酵的基础,因此酵母和乳酸菌发酵的面粉与水的混合物构建了一个生态系统[3-6]。世界范围内的典型面团生态系统有[7]:埃及Balady-Soltani酸面团,发酵微生物主要是乳杆菌(约占63%~64%);印度南部Idli酸面团,其优势菌是肠系膜明串珠菌,其次为粪肠球菌,这种面团与传统以酵母菌起发的面团不同,主要是由肠系膜明串珠菌起发产生;中欧式面团Lindneri酸面团中的代表微生物为从啤酒中分离得到的短乳杆菌;美国旧金山地区的San Francisco面团中产酸主要微生物是异型发酵乳杆菌;伊朗Sangak酸面团里产酸发酵的主要微生物是约占微生物数量77%以上的肠系膜明串珠菌、短乳杆菌和植物乳杆菌。在稳定的面团中,通常含有特定的酵母菌和乳酸菌。多项相关研究报道指出,单一面团在特定时间内一般只含有一种或两种酵母菌,其中矮小假丝酵母(Candida humilis)、库德里阿兹威氏毕赤酵母(Pichia kudriavzevii)和酿酒酵母(Saccharomyces cerevisiae)的出现较为频繁[8]。Desiye等[9]应用传统纯培养技术对埃塞俄比亚主食之一煎饼Enjera中的乳酸菌和酵母菌进行了分离、鉴定和识别,在34 种样品中得到107 株乳酸菌和68 株酵母菌。
变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)是一种不依赖于纯培养的分离技术[10],而且DGGE技术相较于琼脂糖电泳和聚丙烯酰氨凝胶电泳分辨率高[11],分析微生物群落可以获取其高分辨率图谱并对图谱的信息进行生物学上的合理解释[12]。因其快速、灵敏、分析全面等特点已被广泛应用于微生物生态学的研究中[13-14]。近年来,聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)技术已经直接应用于食品领域[15-16]。乌日娜等[17]利用PCR-DGGE技术成功研究了酸汤子面团中的微生物菌群多样性。Ercolini等[18]应用此技术纯化天然乳清培养基的DNA片段,从中鉴定出德氏乳杆菌和乳球菌。Yoshikawa等[19]用活菌计数和DGGE分别监测了小麦和酸面团中菌群的动态变化。
黏豆包又称黄豆包或豆包,由大黄米或江米与玉米按一定比例混合经自然发酵后,包裹红豆馅蒸制而成的冷冻类面制食品。具体做法是把大黄米或江米泡12 h,适当晾干后磨面,将黄米或江米与玉米适度混合,冷水和面,进行发酵(一般1~3 d),待有酸味飘出,用手揉面,此步决定黏豆包制作的成功与否,及口感与色泽。将红豆制馅,用揉好的面团将豆馅包入,蒸制20 min即可。将这种易于储藏、食材易得、营养丰富的满族特色美食推广开来,丰富多样化饮食餐谱,对黏豆包加工进行升级与产业化具有重要意义。
目前国内对传统黏豆包的研究较少,仅有李琦等[20]对采用低温长时间发酵的自然发酵糯玉米黏豆包中优势乳杆菌进行了纯培养分离鉴定的研究,然而应用分子生物学方法对其产品的研究鲜见详细报道。其中黏豆包发酵面团的微生物菌群结构是影响产品功能特性的重要因素之一[21-24],并且微生物菌群组成是发酵条件受限的关键节点,同时也是黏豆包工业化生产受到阻碍的主因。本实验采用传统黏豆包原料大黄米和江米,通过高温过夜发酵,并利用16S rDNA V3区和26S rDNA D1区通用引物进行PCR-DGGE指纹技术分析,对传统自然发酵黏豆包面团中微生物菌群组成进行快速、深入的探索,以期为实现黏豆包产品的工业化、规模化、自动化生产提供理论依据。
1 材料与方法
1.1 材料与试剂
随机抽取2012年11月份东北地区黑龙江省安达市不同家庭的传统自然发酵黏豆包发酵终点面团样品5 种,其中3 种样品为大黄米黏豆包发酵面团,标记为P1、P2、P3;另外2 种样品为江米黏豆包发酵面团,标记为G1、G2;样品P4为同一时间段实验室条件下模拟制备的大黄米黏豆包发酵面团,无菌操作取样并收集于已灭菌的收集盒中,保存于-20 ℃。
2×Taq PCR Master Mix(含染料)双丙烯酰胺、过硫酸铵、F338-G、引物 北京博奥拓达科技有限公司。
1.2 仪器与设备
PL203型电子分析天平 梅特勒-托利多仪器有限公司;TCL-16C离心机 上海安亭科学仪器厂;ABI-9700型PCR扩增仪 美国ABI公司;DYY-10C型电泳仪 北京市六一仪器厂;UVP凝胶呈相系统、DGGE仪 美国Bio-Rad公司;紫外-可见透射反射仪 上海精科实业有限公司。
1.3 方法
1.3.1 黏豆包发酵面团的收集
实验室条件下,模拟传统黏豆包自然发酵面团的制备流程如下:

原料取用市售大黄米与玉米面,大黄米用清水先淘洗以淘净沙子,开水浸洗,浸泡大约10 h后,晾至大半干后用粉碎机磨成面。设定玉米面-大黄米面按质量比1∶3混合均匀,再用热水和面,掺水量约是面量的80%。先用少量开水烫面,再加温水混合到面团黏合而不黏手为合适。和好的面置42 ℃温箱内发酵12 h,取出翻新揉面揣平,再继续醒发2 h。
1.3.2 黏豆包发酵面团中微生物总DNA提取
按1∶9(g/mL)的比例将面团样品与灭菌的0.9%食盐水振荡混匀,参照十六烷基三甲基溴化铵(hexadecyl trimethyl ammonium bromide,CTAB)-溶菌酶法[24]对样品微生物总DNA提取。将得到DNA沉淀自然风干,然后加入40 μL TE缓冲液(含10 μg/mL的RNaseA)溶解,37 ℃保温2 h后,-20 ℃冰箱保存。
1.3.3 PCR扩增
细菌16S rDNA V3区采用表1中的通用引物对338f-GC和518r进行扩增[22-23]。PCR体系50 μL:4 μL(约50 ng)模板DNA,1 μL 338f-GC1,1 μL 518r,25 μL 2×Taq PCR Master Mix(含染料),去离子水补足总体系为50 μL。反应参数:94 ℃预变性2 min,94 ℃变性1 min,65~56 ℃(-1 ℃/ 2 个循环)退火1 min,72 ℃延伸1 min,20 个循环;94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸1 min,10 个循环;72 ℃延伸7 min,4 ℃保存。对所得的PCR产物用1.5%的琼脂糖凝胶电泳进行检测,对符合要求的PCR产物进行后续实验。
酵母菌26S rDNA D1区采用表1中的通用引物对NL1-GC2和LS2进行扩增[25-26]。PCR体系50 μL:4 μL模板DNA,1 μL的NL1-GC2,1 μL的LS2,25 μL的2×Taq PCR Master Mix(含染料)用去离子水稀释至50 μL。反应参数:95 ℃预变性2 min,95 ℃变性1 min,52 ℃退火1 min,72 ℃延伸1 min,35 个循环;72 ℃延伸7 min,4 ℃保存。对所得的PCR产物用1.5%的琼脂糖凝胶电泳进行检测,对符合要求的PCR产物进行后续实验。
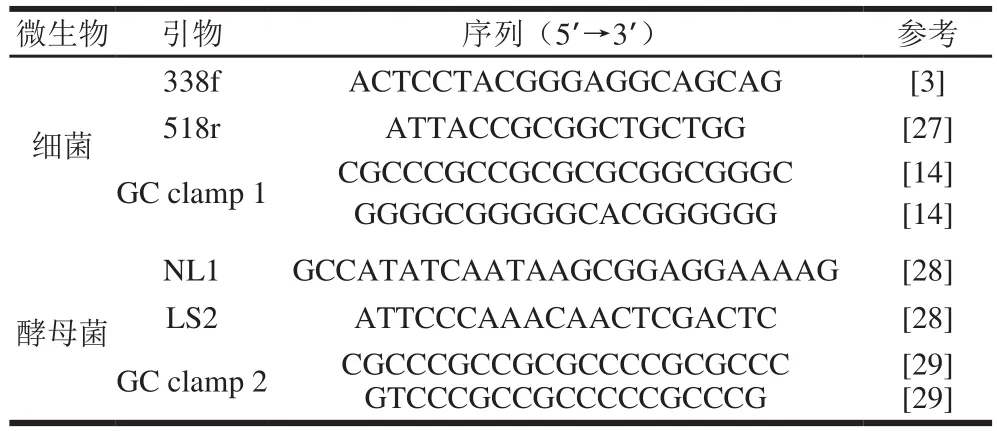
表1 PCR引物和GC夹序列Table 1 PCR primers and GC clamps used in this study
1.3.4 黏豆包发酵面团16S rDNA V3区和26S rDNA D1区DGGE分析
电泳采用The DCodeTM Universal Mutation Detection System系统,基因片段大小在200 bp左右,选用8%的DGGE变性凝胶,16S rDNA V3区变性梯度为35%~60%,26S rDNA D1区变性凝胶梯度为35%~70%。电泳条件:上样量为40 μL,1×TAE电泳缓冲液,60 ℃(DNA的最适变性温度),电压130 V,电泳时间8 h。电泳结束后,胶样用GeneFinder核酸染料染色30 min。UVP凝胶成像系统拍照,并用Quantity One软件进行相应分析。所得到的DGGE图谱中,在紫外割胶仪中用无菌手术刀将清晰、亮度较高且分离较明显的优势带切下,装入无菌的EP管中。将切好的胶块用100 μL去离子无菌水清洗2~3 次,然后再加入50 μL去离子无菌水将胶块捣碎,4 ℃过夜后取其上清液作为模板,16S rDNA V3区去掉“GC夹”的338f和518r引物对其进行PCR扩增,26S rDNA D1区采用去掉“GC夹”的NL1和LS2引物对对DNA样本进行PCR扩增,PCR体系及条件同1.3.3节。最后,将PCR产物送苏州金唯智测序公司进行基因测序,测序结果对比GenBank数据库进行BLAST分析。
2 结果与分析
2.1 16S rDNA V3区和26S rDNA D1区的扩增结果
2.1.1 黏豆包发酵面团细菌16S rDNA V3区扩增结果
采用细菌16S rDNA V3区通用引物(表1),以提取获得的6 种样品的总DNA为模板,按照1.3.3节的体系及相应的降落式PCR程序进行扩增,通过1.5%琼脂糖凝胶电泳分析扩增产物,得到图谱见图1,Marker I标记显示其DNA片段对应大小约为200 bp,而且获得的条带完整一致,无明显杂带,可认定为目标条带。
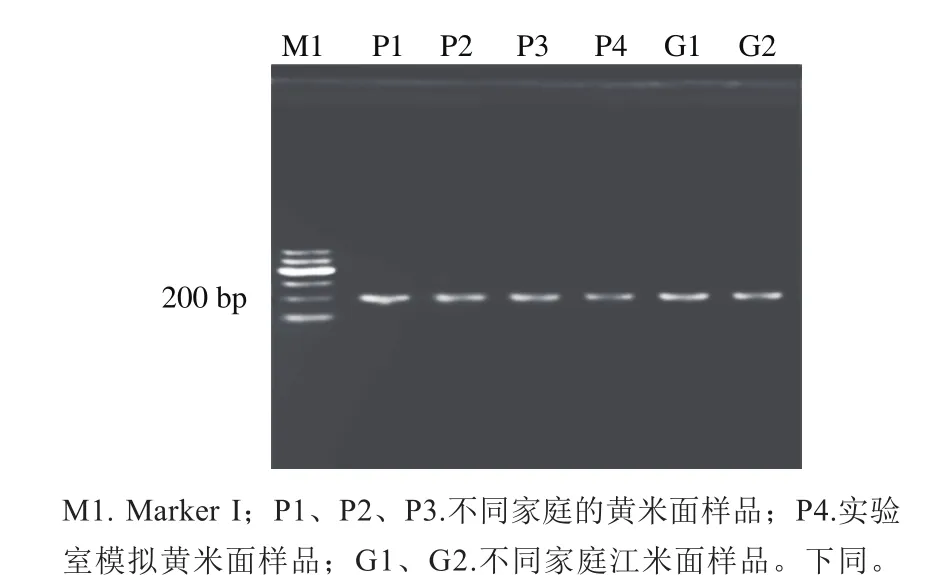
图1 不同黏豆包样品16S rDNA V3区PCR琼脂糖凝胶电泳图Fig. 1 PCR fingerprints of V3 region of bacterial 16S rDRNA genes from sticky bean stun samples
2.1.2 黏豆包发酵面团酵母菌26S rDNA D1区扩增结果
采用酵母菌26S rDNA D1区通用引物(表1),对提取获得的6 种样品的总DNA按照1.3.3节的体系及相应的PCR程序进行扩增,使用1.5%琼脂糖凝胶电泳分析,得到图谱见图2,D2000标记显示其DNA片段大小约为250 bp,获得的条带清晰均一,无明显杂带,因此可认定为目标条带。
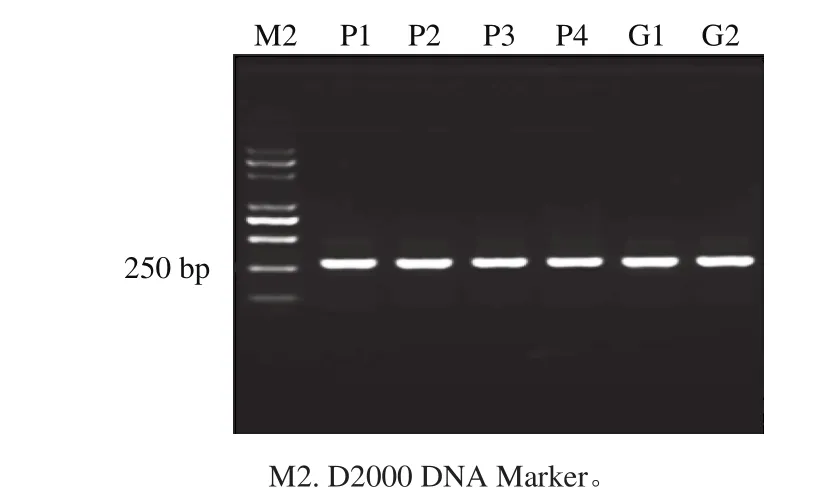
图2 26S rDNA V3区PCR琼脂糖凝胶电泳图Fig. 2 PCR fingerprints of D1 region of 26S rDNA genes of yeasts from different sticky bean bun samples
2.2 黏豆包发酵面团中菌群组成DGGE指纹图谱分析
2.2.1 黏豆包发酵面团细菌DGGE指纹图谱分析

表2 细菌和酵母菌DGGE条带测序结果Table 2 Sequencing results of the bands selected from the bacteria and yeast DGGE profiles
DGGE图谱条带数目可以直观地反映黏豆包发酵面团样品中细菌组成的多样性。表2显示,黏豆包面团6 种样品的细菌DGGE图谱总共发现7 个特异性扩增条带,其条带数量和细菌分布情况各不相同,说明黏豆包面团样品中细菌分布有着较大的差异性。样品G1、G2和P4中条带数目与其他3 种样品相比较多,说明采用江米为原料制作黏豆包的样品中细菌的种类比较丰富;P1的条带数目最少,说明原材料的选择与制作人的技巧及环境对细菌多样性有一定影响。对7 个特征条带继续回收和测序,测序结果进行BLAST同源性比对,结果如表2所示。
黏豆包面团6 个样品中共检测出5 种细菌,他们分别属于4 个菌属。食窦魏斯氏菌和肠膜明串珠菌在6 个样品中均检测出,且在多个样品中条带明亮,更宽,说明二者无论是大黄米或江米为原料面团中都存在很多。融合魏斯氏菌仅在大黄米和玉米混合的黏豆包面团样品中检出,说明为大黄米发酵面团的特有菌属。此外,乳杆菌属中的罗氏乳杆菌、乳酸乳球菌也在黏豆包面团中检出。由此推断,食窦魏斯氏菌和肠膜明串珠菌为黏豆包发酵面团样品中所占比例最高,属于黏豆包的优势细菌菌群;且不同谷物为原料制作的黏豆包面团可能存在独有的特有菌属。
从条带分布结合鉴定结果可知,即使同一地区采集的相似原料加工的黏豆包样品中细菌结构也存在差异,这一发现与同为东北满族谷物发酵食品“酸汤子”的细菌结构分析结果一致。乌日娜等[17]发现辽宁丹东地区3 个不同人家的酸汤子中细菌菌群分布差异明显,植物乳杆菌和类肠膜魏斯氏菌为酸汤子样品的优势细菌菌群。李晓红[30]运用传统分离鉴定方法,从自然发酵小麦面粉面团中发现植物乳杆菌和嗜热链球菌为优势菌。而对5 份中国传统小麦面粉发酵面团的高通量测序结果显示明串珠菌属、乳酸杆菌和魏斯氏菌属是其中的优势菌属[31]。以江米为原料的黏豆包发酵面团中还鉴定到了作为国际公认益生菌的罗氏乳杆菌,该菌曾在一种发酵食品Ting中分离得到,早在2003年中国卫生部即批准它为可用于人类健康的保健产品,具有很高的理论研究和生产应用价值[32-33]。

图3 不同黏豆包样品的细菌DGGE指纹图谱Fig. 3 DGGE profiles of V3 fragments of bacterial 16S rDNA genes from sticky bean bun samples
2.2.2 黏豆包发酵面团中酵母菌DGGE指纹图谱分析
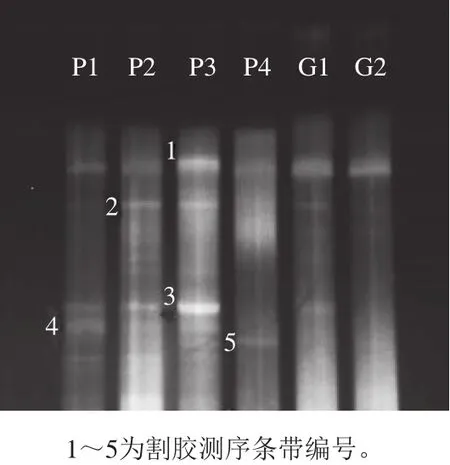
图4 不同黏豆包样品的酵母菌DGGE指纹图谱Fig. 4 DGGE profiles of D1 fragments of 26S rDNA genes of yeasts from sticky bean bun samples
黏豆包发酵面团样品中酵母菌PCR鉴定后所得对应的DGGE图谱及其各条带基因测序后在NCBI中GenBank数据库BLAST比对后的结果分别见图4和表2。结果显示,酵母菌DGGE图谱中获得5 个优势条带,每个样品的扩增条带呈现出较大的差异性,得到的条带数量、酵母分布情况以及清晰度较细菌相比,都有较大下降。其中发现的诞沫假丝酵母在6 个样品中均有条带显现,传统手工家庭制作的黏豆包面团中都发现了卡利比克毕赤酵母,普兰久浩酵母中以大黄米和玉米为原料发酵的黏豆包面团中出现的条带明亮而清晰。此外在实验室模拟加工的黏豆包面团里出现了一种不能培养的酵母菌。
由此推测,黏豆包面团中的优势酵母菌属为诞沫假丝酵母。不同原料发酵的黏豆包存在各自的特征条带,暗示可能存在机会发酵菌种,专一利用特有的酵母进行发酵,本研究中的卡利比克毕赤酵母就可能属于其中之一。本研究还鉴定出的普兰久浩酵母属于耐冷酵母,黏豆包的加工多为冬天,而普兰久浩酵母能高产乳糖酶,东北极冷条件下也保有很高活性。以玉米为原料发酵制成的酸汤子中的优势真菌也有普兰久浩酵母。将普兰久浩酵母的产低温乳糖酶分离用于食品加工领域具有较高的应用价值[34]。陶东娅等[35]研究黑龙江牡丹江市东宁县以大黄米-玉米质量比3∶1的黏豆包酸面团的真菌菌群发现米根霉、热带假丝酵母、酿酒酵母和异常毕赤酵母为优势菌属;同时也发现不同地域黏豆包面团中细菌的多样性比真菌的丰富。
3 结 论
本研究对黑龙江省传统自然发酵黏豆包面团微生物菌群组成进行探究,发现菌群结构与面团的原料组成有关,揭示了可能在黏豆包发酵过程中起关键作用的菌群,了解东北地区利用传统方法自然发酵的黏豆包中微生物的种类。结果发现黏豆包面团中细菌较酵母相比种类更为多样,明串珠菌属、魏斯氏菌和假丝酵母为优势菌群,乳杆菌属为优势细菌菌属。今后可以通过纯培养从黏豆包中获得相关菌株研究其功能特性,以期找出黏豆包发酵的具体功能菌,进一步为改良黏豆包品质,实现黏豆包工业化、规模化、自动化提供理论依据。
