抗生素胁迫暂养下菲律宾蛤仔黏附污泥的耐药絮凝活性菌研究
2018-08-19崔霞,穆军
崔 霞,穆 军
(浙江海洋大学海洋科学与技术学院,浙江舟山 316022)
絮凝剂作为一种常见的水处理试剂,广泛用于不同的工业领域,按其分子组成可分为无机絮凝剂、合成有机高分子絮凝剂和天然高分子絮凝剂[1-2]。菲律宾蛤仔黏附污泥作为一种已被报道的新型天然生物絮凝剂,具备絮凝、脱色、重金属去除等典型微生物絮凝剂活性,且能自然沉淀、适合淡水和海水处理[3-6]。菲律宾蛤仔黏附污泥是一种具有复杂生态关系的活性生物污泥,周海军等[7]在探究其絮凝活性产生机制过程中,选取了影响菲律宾蛤仔生态环境的生物因素,包括抑制细菌(加青霉素和链霉素),抑制真菌(加灰黄霉素和5-氟胞嘧啶),无菌(加青霉素,链霉素,灰黄霉素和5-氟胞嘧啶)等,以及不加泥沙栖息地的非生物影响因素,以正常养殖条件下的菲律宾蛤仔作为对照组,通过高岭土悬浊液絮凝活性测定方法对比各影响因素下的菲律宾蛤仔黏附污泥絮凝性能后发现,细菌在黏附污泥絮凝机制中起到重要作用,泥沙栖息环境是黏附污泥具有絮凝活性的必要非生物因素。值得注意的是,在加入青霉素,链霉素等抗生素的蛤仔暂养系统中,收集的菲律宾蛤仔黏附污泥仍具有一定的絮凝活性,将该条件下的黏附污泥进行微生物分离筛选后,获得了一批耐药型絮凝活性菌,但目前针对耐药絮凝活性菌的微生物多样性及其物种组成还尚未进行探讨。
本文利用Illumina高通量测序方法研究了抑制细菌条件下的菲律宾蛤仔黏附污泥中耐药型絮凝活性菌的微生物组成,同时,对多数菌落形态相似的代表性耐药絮凝活性菌进行形态学与16S rDNA鉴定,旨在探讨菲律宾蛤仔抗生素胁迫暂养下黏附污泥的耐药絮凝活性菌的物种组成,目前,这是首个关于菲律宾蛤仔粘附污泥中耐药型絮凝活性菌微生物组成的研究。
1 材料与方法
1.1 耐药絮凝活性菌的来源
选取加入青霉素,链霉素等抗生素养殖系统中的菲律宾蛤仔,收集其所吐黏附污泥,通过与无菌水混合进行梯度稀释,在含有抗生素的固体平板培养基上涂布分离,培养基成分为:葡萄糖20 g,酵母膏0.5 g,尿素0.5 g,硫酸铵0.2 g,硫酸镁0.2 g,磷酸二氢钾2.0 g,磷酸氢二钾5.0 g,青霉素0.06 g,链霉素0.08 g,琼脂20 g,溶解于1 L的陈化海水中,pH值7.2~7.4,培养基在115°C条件下灭菌30 min。涂布后的培养基在25°C下培养7 d,随机挑选单菌落并进行划线分离后,获得50株纯化的耐药细菌。将纯化的耐药细菌分别接种到液体培养基中(与固体培养基成分相同,不含琼脂),25°C、180 r/min条件下培养5 d,培养后的发酵液上清液通过高岭土悬浊液来测定其絮凝活性,最终筛选得到50株具有30%左右絮凝活性的耐药细菌[7]。
1.2 耐药絮凝活性菌的DNA提取
使用TaKaRa全基因组提取试剂盒DNA Extraction Kit Ver.3.0进行耐药细菌DNA的提取和纯化。
(1)菌体裂解:将50株耐药絮凝活性菌混合发酵培养,取细菌发酵液到离心管中,12 000 r/min条件下离心2 min,弃掉上清液,加入500 μL Buffer BS缓冲液得到细胞悬浮液,再加入50 μL Lysozyme溶菌酶充分混合,在37°C水浴中加热60 min;将加热后的混合液在12 000 r/min条件下离心5 min,丢弃上清液;向离心后的物质中加入180 μL Buffer GL缓冲液,20 μL Proteinase K蛋白酶和10 μL Proteinase A蛋白酶,充分混匀后置于56°C水浴中加热10 min;最后向溶液中加入200 μL Buffer GB缓冲液和200 μL无水乙醇,充分混合。
(2)DNA的提取与纯化:将步骤(1)中的溶液转移到旋转柱,再插入收集管中,12 000 r/min条件下离心2 min,弃掉下部过滤液;取500 μL Buffer WA缓冲液加入旋转柱中,12 000 r/min条件下离心1 min,弃掉下部过滤液;再取700μLBufferWB缓冲液沿管壁加入旋转柱中,以清除沾附于管壁上的盐份,12 000 r/min条件下离心1 min,再次丢弃过滤液。重复加入700 μL的Buffer WB缓冲液进行离心过滤。将上述步骤操作后的旋转柱放入新的1.5 mL试管中,在膜的中心处加入50~200 μL的无菌蒸馏水,室温静置5 min,12 000 r/min条件下离心2 min洗脱DNA。将提取的基因组DNA通过1%的凝胶电泳进行检测。
1.3 PCR扩增和Illumina Miseq测序
通常对于细菌多样性分析,选用16S rDNA的V3-V4区域用于扩增构建,PCR扩增使用常见引物343F(5'-TACGGRAGGCAGCAG-3')和798R(5'-AGGGTATCTAATCCT-3')。取适量的DNA样品于离心管中,使用无菌水稀释样品至1 ng/μL,以稀释后的基因组DNA为模板,根据测序区域的选择,使用带Barcode的特异引物,KAPA公司的HiFi Hot Start Ready Mix高保真酶进行PCR,确保扩增效率和准确性。取稀释后的DNA(50 ng)作为聚合反应的模板,反应中包含1 μL正向和反向引物,15 μL的2xTaq Master Mix溶液(Vazyme,南京)和13 μL的ddH2O。PCR反应条件为:94 °C 预变性5 min,90 °C 变性 30 s,60 °C退火30 s,72°C延伸30 s,变性到延伸反应需25~35个循环,最后反应在72°C修复延伸7 min,并在4°C条件下终止反应。
PCR产物使用1%的凝胶电泳检测,检测后使用AMPure XP beads(Agencourt AMPure XP kit)进行磁珠纯化,取1 μL纯化过的一轮产物进行浓度检测,将检测后的PCR产物作为模板,进行二轮PCR扩增,再次使用电泳检测,检测后进行二次磁珠纯化。纯化后的PCR产物通过Qubit dsDNA检测试剂盒(Life Technologies)进行定量,再用Agilent生物分析仪2100(美国)进行检测。根据PCR产物浓度进行等量混样,并进行上机测序。
1.4 测序原始数据处理
将PCR产物通过Illumina Miseq测序平台进行测序,将得到的双端序列通过Trimmomatic(Version 0.35)软件[8]和FLASH(Version 1.2.11)软件[9]进行去杂和优质序列拼接;生成的序列嵌合体使用UCHIME(Version 4.2)软件[10]进行去除,作为下一步分类学单元聚类的优质序列。将上述预处理生成的优质序列采用CD-HIT方法[11]进行OTU(Operational Taxonomic Units)挑选聚类,采用Naive Bayesian分类算法[12]将97%相似水平的代表序列归为一个分类单元OTU,并挑选OTU分类中丰度最大的序列作为代表序列,与Greengenes数据库[13]进行比对,生成OTU分类表格。挑选出TOP100的OTU通过Figtree软件[14]对进化树进行呈现。同时,采用相同分类算法,将100%相似水平下的代表序列归为一个分类单元OTU,并挑选OTU分类中丰度最大的序列作为代表序列,与Greengenes数据库[13]进行比对,生成100%相似水平下OTU分类表格。
在97%相似度进行的OTU分类条件下,抽样计算了不同深度的Alpha多样性指数,其中,Chao指数在生态学中常用来估计物种总数;Gini-simpson和Shannon Wiener指数,即估算样品中微生物的多样性指数,反应样品中微生物群落的丰富度和均匀度。Good's coverage指数反应样品中微生物群落内的物种丰度性程度。
1.5 耐药絮凝活性菌的鉴定
在分离筛选得的50株耐药絮凝菌中,取两株代表性耐药絮凝活性菌,用奥林巴斯CX41生物显微镜观察两株菌的形态特征。两株耐药絮凝活性菌的DNA按照DNA提取试剂盒(TianGen,中国)方法进行提取,细菌的16S rDNA PCR扩增采用GeneAmp®9700 PCR 仪(ABI,美国);PCR 反应体系包括 1 μL 的 10 μM 引物 27F(5'-AGAGTTTGATCCTGGCTCAG-3'),1 μL 的 10 μM 引物 1492R(5'-TACGGCTACCTTGTTACGACTT-3'),1 μL DNA 模板,1 μL 的 2xTaq Master Mix 溶液(Vazyme,南京),加 ddH2O 将反应体系至30 μL。PCR 反应条件为:95 °C 预变性 5 min,95 °C 变性 30 s,55 °C 退火 30 s,72 °C 延伸 1 min,变性到延伸反应需35个循环,最后反应在72°C修复延伸7 min,并在4°C条件下终止反应。PCR产物通过1%的琼脂糖凝胶进行纯化,进行上机测序。将16S rDNA测序结果分别在EzBioCloud数据库中进行比对分析,采用MAGA6.0软件将两株耐药絮凝活性菌和EzBioCloud数据库中与菌株相对应的前20个同源性较高的模式菌株进行系统发育关系进化树的构建。
2 结果与讨论
2.1 耐药絮凝活性菌群的微生物多样性分析
样品GNHY.B通过高通量测序共得到30 168条优化序列,平均长度分布在440~449 bp。在97%相似性水平上,样品共得到967个OTUs,挑选出丰度最高的前TOP100 OTUs构建了系统进化树,如图1所示,并计算出TOP100的OTUs所占丰度值。样品的Alpha多样性指数分别以1 000为一个步长,在每个步长进行10次重复抽取序列来计算。由表1可知,Good’s Coverage指数为0.975 2,指数越接近1说明测序深度已经基本覆盖到样品的所有物种;表1中的Shannon Wiener与Gini-Simpson指数值分别为2.65和0.59,根据Shannon Wiener与Gini-Simpson指数的计算方法[15-16]可知,均低于相对理论计算值,表明样品GNHY.B中的微生物多样性较低。
经测定的菲律宾蛤仔黏附污泥耐药细菌群落的原始数据提交在 NCBI(National Center of Biotechnology Information)数据库中,登录号为SRP138342。
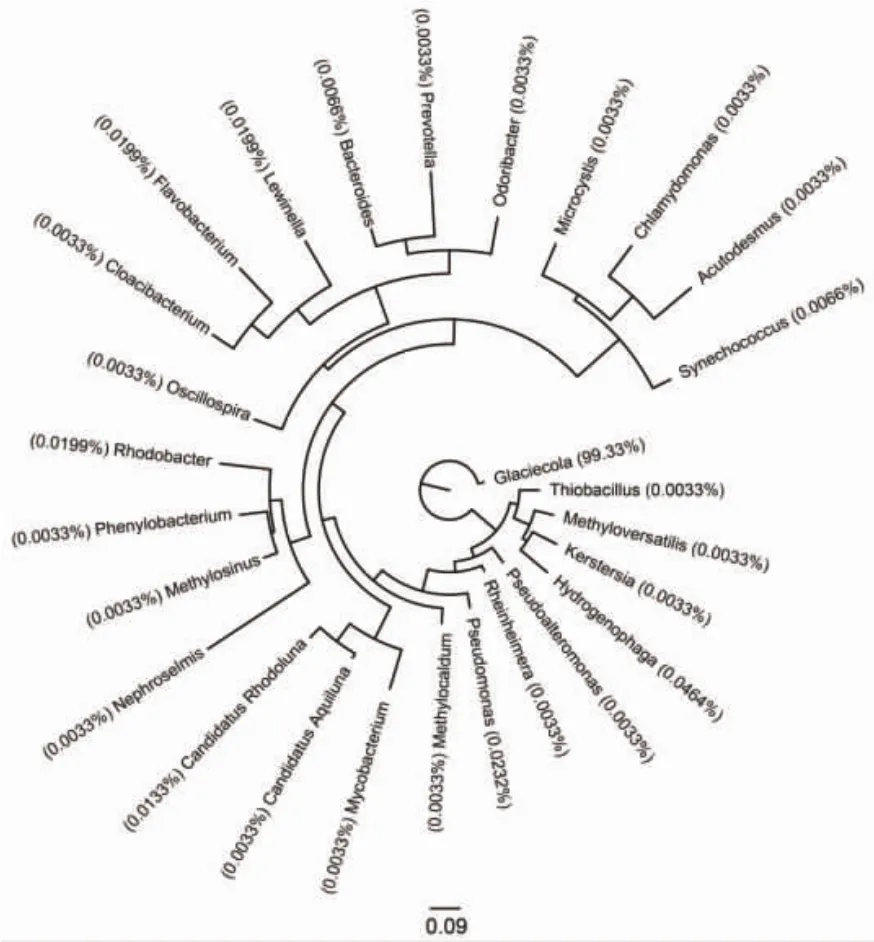
图1 耐药絮凝活性菌群TOP100 OTUs的物种发育进化树Fig.1 The phylogenic tree of TOP100 OTUs in drugresistant bioflocculant-producing bacterial community

表1 耐药絮凝活性菌群的微生物多样性指数Tab.1 Richness and diversity estimation of drug-resistant bioflocculant-producing bacterial community based on MiSeq sequencing
2.2 耐药絮凝活性菌群的微生物组成分析
根据97%相似度进行的OTU分类学结果,分析样品GNHY.B中细菌群落的相对丰度。结果表明,样品GNHY.B共覆盖7个门,以变形菌门Proteobacteria为主,所占比例为99.71%;共包含21个纲,37个目,57个科和56个属,以变形菌门Proteobacteria中的γ-变形菌纲Gammaproteobacteria,交替单胞菌目Alteromonadales,交替单胞菌科Alteromonadaceae,Glaciecola属为主,各鉴定水平下的代表群落均占样品相对丰度的99%以上,如图2所示为样品GNHY.B在属的水平下的群落结构分布图。鉴定结果中虽然显示出样品中含有其他物种,但其丰度值总和占样品相对丰度不到1%的比例,因此,耐药絮凝活性菌样品GNHY.B主要以最高丰度的细菌属类Glaciecola为主。样品GNHY.B在100%相似度下进行OTU分类的结果表明,最高丰度群落为变形菌门Proteobacteria中的属类Glaciecola,所占比例为99.33%,与样品GNHY.B在97%相似度进行OTU分类的结果相同。鉴定结果再次表明了样品GNHY.B微生物群落结构的单一性,并初步推断在抑制细菌的蛤仔暂养系统中,所收集的菲律宾黏附污泥样品筛选出的50株耐药絮凝活性菌大部分可能属于同一物种。
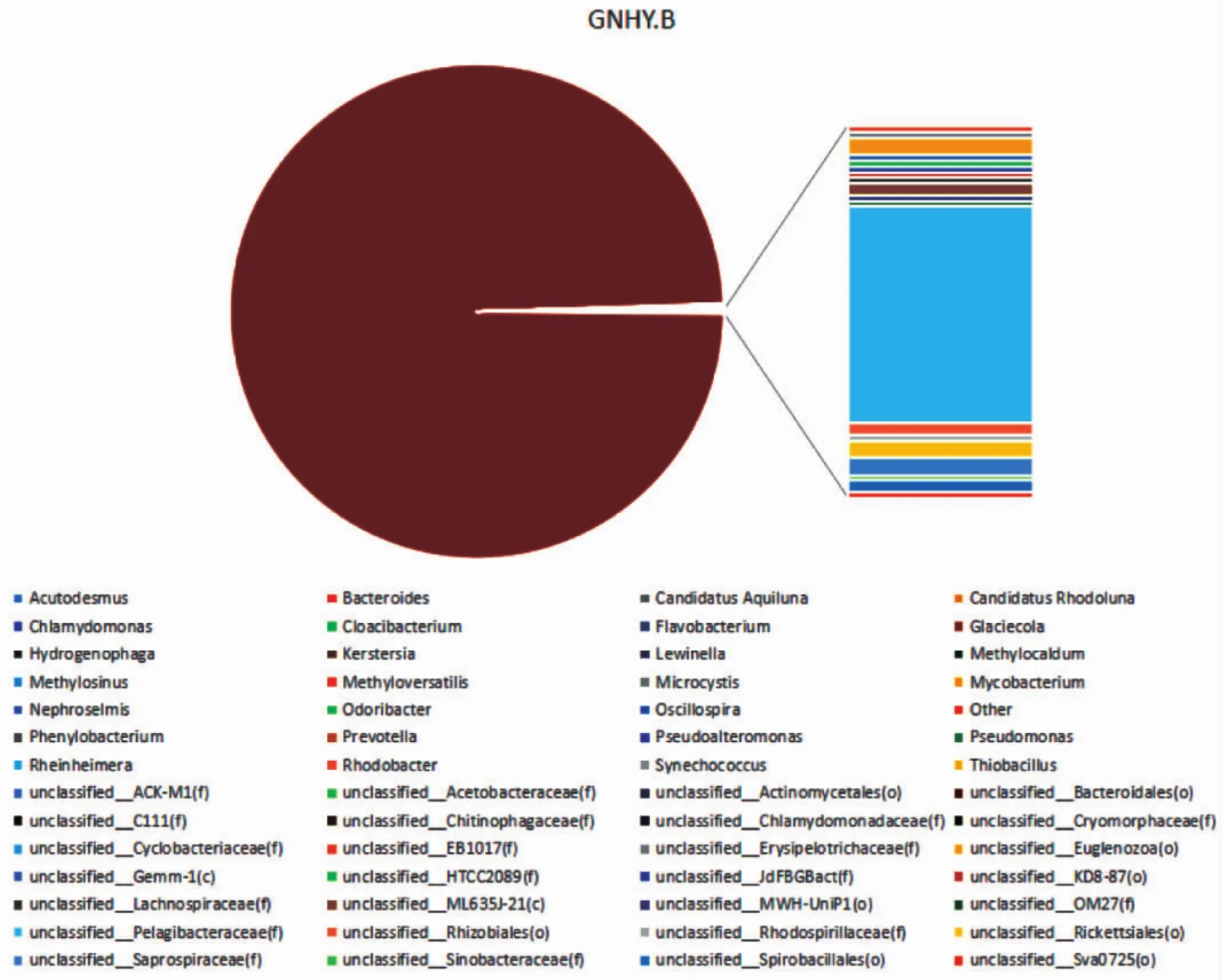
图2 耐药絮凝活性菌群属水平下的微生物群落结构分布图Fig.2 Microbial community structure distribution of drug-resistant bioflocculant-producing bacterial community at genera level by 16S rDNA sequencing
2.3 耐药絮凝活性菌的鉴定结果
耐药絮凝活性菌GHNY-1与GHNY-2的菌落形态特征均为小圆形、凸起、乳白色微黄、边缘整齐、平滑;通过细胞的简单染色与显微镜观察,耐药絮凝活性菌GHNY-1与GHNY-2的细胞形态如图3所示,均为卵形细胞。16S rDNA测序结果在EzBioCloud数据库进行同源性比对可知,耐药絮凝活性菌GHNY-1、GHNY-2与菌株Paraglaciecola chathamensis的同源性分别为99.24%和99.09%。如图4所示,为耐药絮凝活性菌GHNY-1、GHNY-2和EzBioCloud数据库中相应的前20个同源性较高的模式菌株的系统发育关系,其中,系统发育树中最接近的进化枝为相应相似性最高的模式菌株Paraglaciecola chathamensis。由于SHIVAJI,et al[17]在2014年已将属Glaciecola重新划分为两个属,即Glaciecola与Paraglaciecola,其中,Paraglaciecola chathamensis为 Glaciecola chathamensis重新鉴定后的物种;原物种Glaciecola chathamensis首次发现于太平洋的沉积物中,是一种胞外多糖产生菌,在海水配制的固体培养基上形成平滑,凸起,无色素的菌落,为卵形或弯曲的杆状细胞[18]。结果表明,耐药絮凝活性菌GHNY-1、GHNY-2的菌落形态和细胞形态与物种Glaciecola chathamensis具有较高的相似性,与其重新鉴定后的物种Paraglaciecola chathamensis的同源性最高,均在99%以上,因此,均被鉴定为Paraglaciecola sp.。
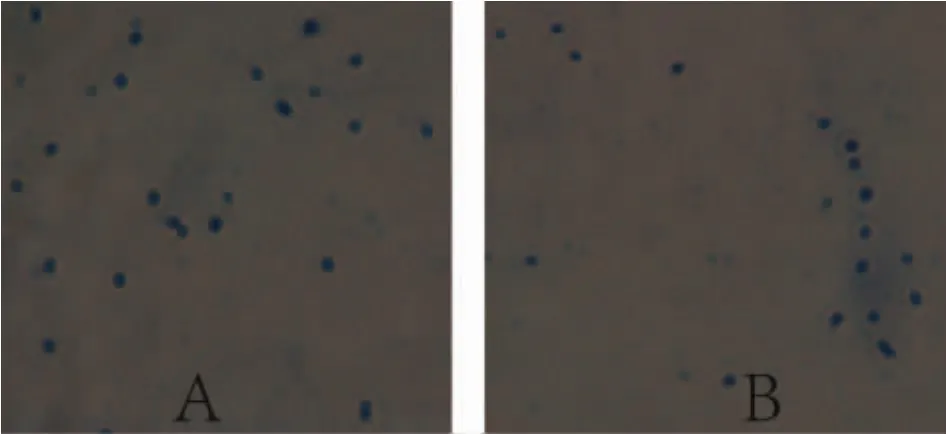
图3 耐药絮凝活性菌GHNY-1(A)与GHNY-2(B)的细胞形态图Fig.3 Cell Morphology images of drug-resistant bioflocculant-producing bacteria GHNY-1(A)and GHNY-2(B)
将耐药絮凝活性菌GHNY-1、GHNY-2的16S rDNA序列提交到GenBank数据库中,登录号分别为:MH261367,MH261368。
3 结论
以菲律宾蛤仔抗生素胁迫暂养下的黏附污泥中分离出的50株耐药絮凝活性菌为研究对象,利用Illumina高通量测序方法研究了其微生物组成,同时,对代表性的耐药型絮凝活性菌进行形态学与16S rDNA鉴定,结果表明,以97%相似度进行OTU分类,Alpha多样性指数表明耐药絮凝活性菌的微生物多样性较低,微生物群落组成分析表明耐药絮凝活性菌以属Glaciecola为主,所占丰度比例为99.33%;以100%相似度再次进行OTU分类,鉴定结果与97%相似度下的OTU分类结果和物种组成相同,初步表明耐药絮凝活性菌的物种组成主要为同一物种;选取耐药絮凝活性菌GHNY-1和GHNY-2,将其16S rDNA测序结果分别在EzBioCloud数据库进行同源性比对可知,同源性最高的模式菌株均为Paraglaciecola chathamensis,该模式菌株为Glaciecola chathamensis重新鉴定后的新物种,同时物种Glaciecola chathamensis与耐药絮凝活性菌GHNY-1和GHNY-2的菌落形态与细胞形态方面展现出了高度相似性,因此均被鉴定为Paraglaciecola sp.。本研究证明了菲律宾蛤仔抗生素胁迫暂养下的黏附污泥具有絮凝活性,在生物因素方面主要是由于耐药絮凝活性菌Paraglaciecola sp.的存在,同时也从耐药菌出现的角度证实了细菌是菲律宾蛤仔黏附污泥活性成分的产生源。
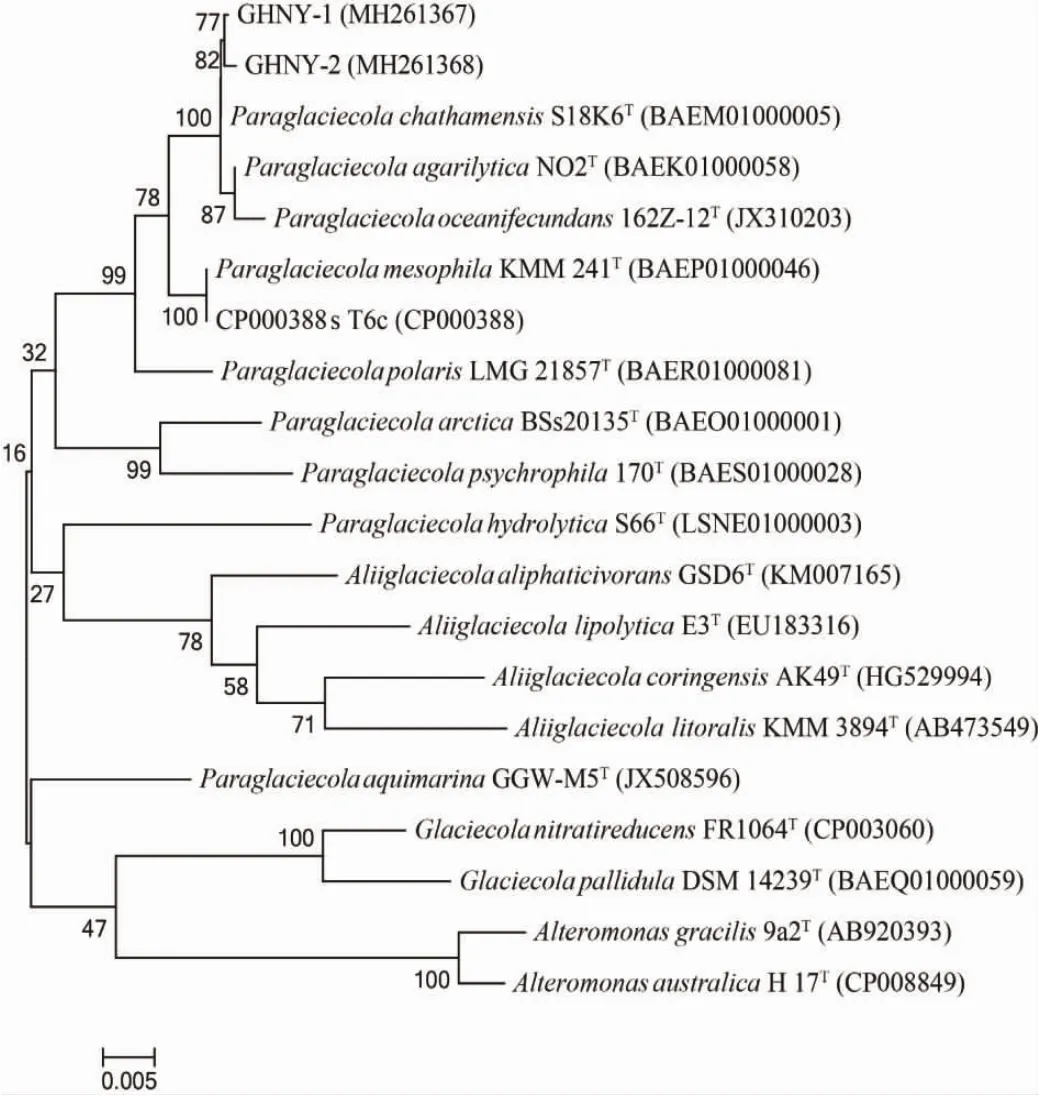
图4 菌株GHNY-1,GHNY-2与其在EzBioCloud数据库中前20个同源性较高的模式菌株的系统发育关系Fig.4 The phylogenetic relationship of the strains GHNY-1,GHNY-2 and their top 20 closely related type strains identified in the EzBioCloud's database
