山黧豆CASase基因DNA全长序列的扩增及CRISPR/Cas9敲除载体的构建
2018-08-10陶英杰刘凤娟毕春晓任雪冰曲瑞红李科友徐全乐
陶英杰,刘凤娟,毕春晓,任雪冰,曲瑞红,李科友,徐全乐
(西北农林科技大学 生命科学学院,陕西 杨凌712100)
山黧豆(LathyrussativusL.)具有与大豆相媲美的蛋白含量,是山黧豆属(Lathyrus)187种作物中最具经济价值和广泛种植的栽培品种[1],其发达的根系、与根瘤菌共生等生物学特征,使得山黧豆有极强的耐贫瘠性和抗旱性[2]。然而,和大多数豆科作物一样,其体内Cys、Met等含硫氨基酸水平较低[3-4]。这与其存在内源毒素β-N-草酰-L-α,β-二氨基丙酸(β-N-oxalyl-L-α,β-diaminopropionic acid,β-ODAP)一起被认为是山黧豆神经性中毒的2个并行因素[5]。实际上,β-ODAP的生物合成与含硫氨基酸的生物合成密切相关[6-7]。β-ODAP生物合成的直接前体是β-异噁唑啉-5-酮-2-L-丙氨酸[β-(isoxazoline-5-on-2-yl)-L-alanine,BIA],BIA由异噁唑啉-5-酮(isoxazolin-5-one)与O-乙酰丝氨酸(O-acetylserine,OAS)在β-腈基丙氨酸合成酶(β-cyanoalanine synthase,CASase)催化下生成[8]。同时,OAS还可以在半胱氨酸合成酶(cysteine synthase,CSase)催化下生成Cys,这导致β-ODAP的积累与Cys的合成呈负相关。因而,同属β取代基丙氨酸合成酶家族(β-substituted alanine synthase,BSAS family)的CSase和CASase在此过程中可能扮演着重要作用[5,9]。尤其是CASase,在山黧豆β-ODAP与硫、氮代谢平衡中可能起到分子开关的作用[5]。
研究表明,山黧豆的CASase活性与其苗期BIA的迅速积累呈显著正相关[10-11]。CASase基因表达水平也与β-ODAP积累模式相符[5,12]。根据山黧豆CASase基因的cDNA序列推测CASase蛋白氨基酸序列与大豆、拟南芥的CASase蛋白序列分别具有88%和79%的一致性,差异主要体现在N端的信号肽序列,且三维结构高度相似[5,13-14]。这种结构的保守性对于依赖于辅因子磷酸吡哆醛(pyridoxal phosphate,PLP)的BSAS家族发挥生物学功能至关重要[14]。在大豆CASase中,PLP的结合位点是Lys95[14]。构建大豆CASase蛋白的K95A点突变体发现,突变体蛋白结构中的吡啶环相对于野生型发生了15°的位移,并导致了CASase活性的丧失[14]。据此构建山黧豆CASase蛋白K95A、K95E和K95R点突变体,发现CASase活性完全丧失[5]。说明山黧豆与大豆CASase功能相似,均可能在硫代谢中扮演重要作用,是山黧豆β-ODAP与硫代谢平衡中的关键酶。对CASase的遗传操作是在增加含硫氨基酸含量的同时,降低β-ODAP含量的一条有效途径[5,15-16]。因而,利用CRISPR/Cas9敲除山黧豆CASase基因将是有益的尝试。本研究克隆了山黧豆CASase基因DNA序列全长,并据此构建了CRISPR/Cas9敲除载体,采用叶盘转化法敲除山黧豆CASase基因,以期为筛选“低毒、高硫”山黧豆品系奠定基础。
1 材料与方法
1.1 材 料
将山黧豆种子置于含蛭石、腐殖土、珍珠岩(三者体积比为1∶1∶1)的混合基质中,23 ℃覆膜萌发4 d。采集黄化苗根部组织,备用。
1.2 方 法
1.2.1 基因组DNA的提取 采用CTAB法[17]提取黄化苗根部组织基因组DNA。用NanoDrop 1 000 (Thermo Scientific,USA)微量核酸密度仪检测DNA质量,用1%琼脂糖凝胶电泳检测DNA完整性。
1.2.2CASase基因的PCR扩增及序列测定 根据CASase基因cDNA序列(GenBank登录号:KJ563188)设计引物:P1.5′-GGCGAATTCCTCGGAACTGTGGAAGAAAT-3′;P2.5′-CGGGATCCATCACCTTCCTCAAGCGAACCTC-3′,引物由Takara公司合成。以1.2.1提取的DNA为模板,进行PCR反应。反应体系50 μL,包括Takara LATaq酶(5 U/μL)0.5 μL,10×LA PCR Buffer Ⅱ(含Mg2+)5 μL,dNTP Miture(各2.5 mmol/L)8 μL,模板DNA 2.5 ng,上、下游引物各2 μL,ddH2O补足至50 μL。将该反应混合物短暂离心至管底,于Jena EasyCycler PCR仪(耶拿,德国)上进行反应。降落PCR反应条件为:94 ℃ 5 min;94 ℃ 30 s,70 ℃ 30 s(-0.5 ℃/循环),72 ℃ 2 min 15 s,30个循环;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 2 min 30 s,20个循环;72 ℃ 10 min。PCR产物经1%琼脂糖凝胶纯化、切胶回收后送宝生物(大连)公司测序。
1.2.3CASase基因序列分析、sgRNA设计及体外活性检测 利用BLAST确定上述测序结果同源性,用Mega 4.0构建系统发育树,利用在线软件Augustus进行外显子与内含子分析;利用sgRNACas9设计sgRNA序列并进行脱靶分析,优选5个潜在sgRNA靶位点。应用luciferase SSA重组法(single-strand annealing recombination assay)对优选的5个sgRNA(表1)进行体外活性初筛。用HEK293A铺 24孔板,当细胞汇合度达50%左右时用lipofectamine 2000转染。转染后48 h收取细胞,按双荧光素酶报告基因检测试剂盒(碧云天)说明书检测双荧光的活性。用萤火虫荧光素酶和海肾荧光素酶活性的比值(Dual-Luciferase Reporter,DLR)表示sgRNA活性,该值越大,表示活性越强。以不添加sgRNA和pcDNA3.1-Cas9载体的转染细胞为阴性对照,以添加100 ng pSSA-CLTA1、0.5 μg sgRNA-CLTA1的转染细胞为阳性对照。
1.2.4 CRISPR/Cas9基因敲除载体的构建 选取活性较高的4个sgRNA靶位点分别各自设计1对20 bp左右的Oligo DNA,包含BamHⅠ和XbaⅠ酶切位点(表1)。使用BamHⅠ和XbaⅠ双酶切pPLatU6-sgRNA载体,将上述Oligo DNA退火后连接到pPLatU6-sgRNA(Biomics Custom)质粒中。

表1 本研究设计的sgRNA Oligo DNA序列Table 1 Designed Oligo DNA sequences of sgRNA in the study
注:sgRNA模板序列中下划线为PAM序列;Oligo DNA中5′和3′端下划线为接头序列,其中包含BamHⅠ和XbaⅠ酶切位点。
Note:The underline in sequence of sgRNA template is PAM,the nuderline of Oligo DNA are adapters,includingBamHⅠ andXbaⅠ sites.
采用同尾酶法,将重组的pPLatU6-sgRNA载体进行NheⅠ和KpnⅠ双酶切,回收小片段,将其与经XbaⅠ和KpnⅠ双酶切的pPLHACas9载体进行T4 DNA酶连接,转化大肠杆菌DH5α,22 ℃培养1 h后,涂Kan+平板筛选,得到的重组载体pPLHACas9,即为构建好的CRISPR/Cas9植物基因敲除载体。
1.2.5 农杆菌转化 将验证阳性的重组载体pPLHACas9,采用CaCl2冻融法转化到农杆菌GV3101中。随机挑选阳性克隆,采用引物AtU6-Fwd和M13fwd进行菌落PCR鉴定,其中引物序列分别为:AtU6-Fwd.5′-AGTCGAAGTAGTGATTGGATC-3′;M13fwd.5′-GTAAAACGACGGCCAGT-3′。20 μL PCR体系如下:2×EsTaq mix 10 μL,ddH2O 7.5 μL,模板1 μL,引物各0.75 μL。PCR程序如下:94 ℃ 7 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 10 s,30个循环;72 ℃ 2 min。PCR产物预期大小为188 bp。
2 结果与分析
2.1 山黧豆CASase基因DNA全长克隆
利用CTAB法提取萌发4 d山黧豆黄化苗根部DNA,根据山黧豆CASase基因cDNA全长序列(GenBank登录号:KJ563188)设计引物进行PCR扩增,1%琼脂糖凝胶电泳检测显示,在2~4 kb有1条明显亮带(图1)。
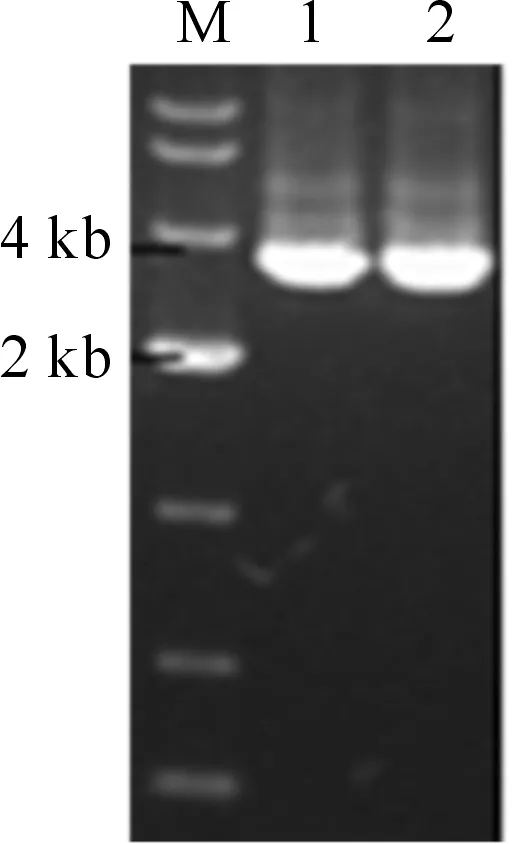
M.DL10000 Marker;1,2.PCR扩增产物M.DL10000 Marker;1,2.PCR products图1 山黧豆CASase基因的PCR扩增Fig.1 Amplification of CASase gene in L.sativus via PCR
PCR产物经切胶回收后测序显示,所获片段长度为3 143 bp。在NCBI上进行序列比对发现,该序列编码山黧豆CASase,与目标基因一致。利用Mega 4.0构建系统进化树发现,山黧豆CASase基因与蒺藜苜蓿、鹰嘴豆、大豆、菜豆等物种的CASase基因聚在一支,而拟南芥、水稻和玉米的CSase基因聚在另一支,其中山黧豆CASase与蒺藜苜蓿和鹰嘴豆亲缘关系最近(图2)。

图2 山黧豆与其他物种CASase基因的系统进化树分析Fig.2 Homology analysis of DNA sequences of CASase gene from L.sativus and other plant species
2.2 山黧豆CASase基因序列分析及sgRNA的体外活性检测
利用在线软件Augustus对山黧豆CASase基因序列分析发现,该序列包含10个外显子(Exon)、8个内含子(Intron)和1个外显子间的间隔序列(图3)。以山黧豆CASase基因前4个外显子为靶序列区间,预测共有45个潜在sgRNA靶位点,对其进行脱靶分析,从中优选出5个sgRNA靶位点,与可能的8个山黧豆半胱氨酸合成酶家族基因序列进行比对发现,潜在sgRNA靶位点在上述基因中的保守性较低,可以特异性地作用于山黧豆CASase基因,其具体位置见图4。
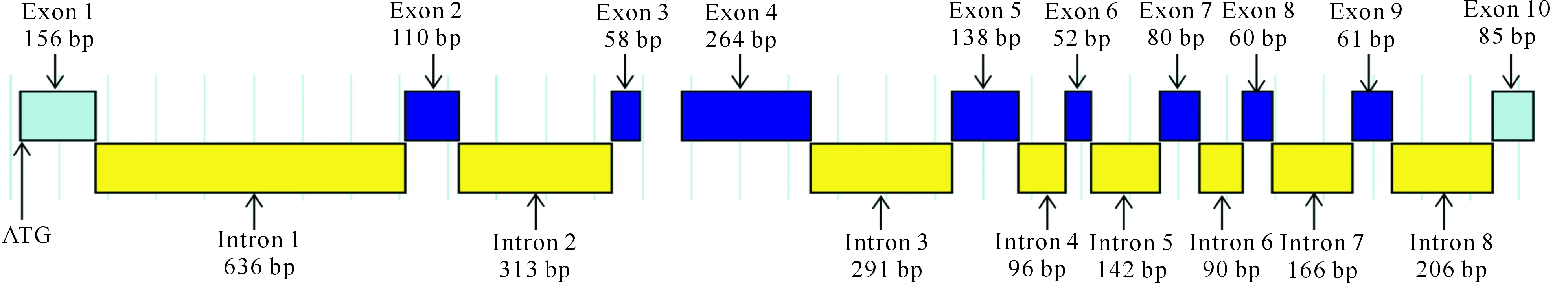
图3 山黧豆CASase基因的gDNA结构Fig.3 gDNA structure of CASase gene in L.sativus

阴影间隔表示不同的外显子,方框表示sgRNA靶位点所在位置Shade interval represents different exons.The box means the candidate targeting sequence sgRNA图4 sgRNA靶位点在山黧豆CASase基因中所处的位置Fig.4 Location of candidate targeting sequence of sgRNA in CASase gene of L.sativus
进一步应用luciferase SSA重组法对设计的5个sgRNA进行体外活性初筛,双荧光检测结果(图5)表明,5个sgRNA均有效剪切了相应的靶序列,其体外活性大小依次为sgRNA20r>sgRNA68r>sgRNA225f≈sgRNA256f>sgRNA396f。
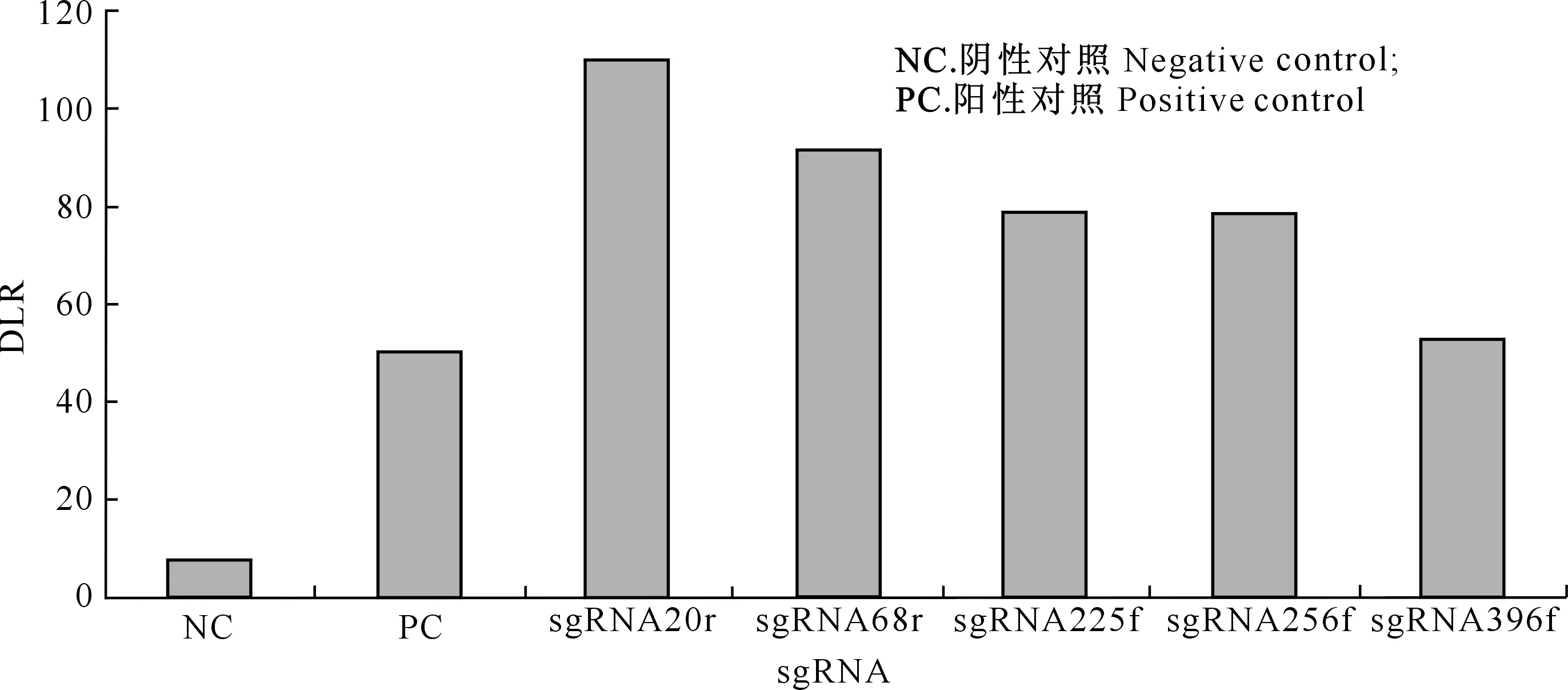
图5 山黧豆5条sgRNA体外活性的双荧光酶报告基因检测Fig.5 In vitro activity analysis of five sgRNAs from L.sativus via luciferase SSA
2.3 山黧豆CRISPR/Cas9基因敲除载体的构建
取活性较高的sgRNA20r、sgRNA68r、sgRNA225f和sgRNA256f,退火连接到经BamHⅠ和XbaⅠ双酶切的pPLatU6-sgRNA质粒中。将重组pPLatU6-sgRNA载体进行NheⅠ和KpnⅠ双酶切,结果(图6-A)显示获得长度为413 bp的片段。再利用XbaⅠ(与NheⅠ为同尾酶)和KpnⅠ双酶切pPLHACas9载体,结果显示酶切成功(图6-B)。
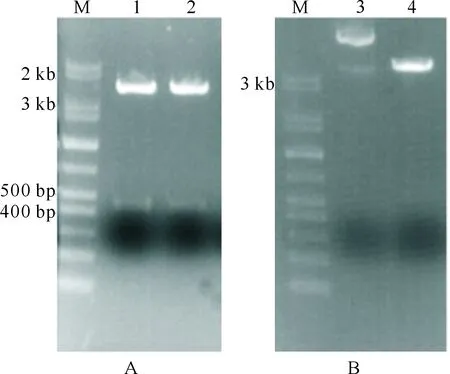
M.1 kb plus DNA Marker;1,2.pPLatU6-sgRNA载体的NheⅠ和KpnⅠ双酶切产物;3.pPLHACas9载体对照;4.pPLHACas9载体的XbaⅠ和KpnⅠ双酶切产物M.1 kb plus DNA Marker;1,2.pPLatU6-sgRNA restricted by NheⅠ and KpnⅠ;3.Control of pPLHACas9;4.pPLHACas9 restricted by XbaⅠ and KpnⅠ图6 山黧豆pPLatU6-sgRNA载体(A)和pPLHACas9载体(B)的双酶切检测Fig.6 Restriction analysis of pPLatU6-sgRNA vector via NheⅠ and KpnⅠ (A) and pPLHACas9 vector via XbaⅠ and KpnⅠ (B)
凝胶回收上述目标片段,插入到双酶切后的pPLHACas9载体中并转化大肠杆菌DH5α,22 ℃培养1 h后,涂Kan+平板筛选。挑选阳性克隆将其转化到农杆菌GV3101并进行菌落PCR鉴定,结果(图7)表明,pPLHACas9-sgRNA20r载体、pPLHACas9-sgRNA68r载体、pPLHACas9-sgRNA225f载体、pPLHACas9-sgRNA256f载体在200 bp左右都有特征条带,说明上述载体均构建成功。
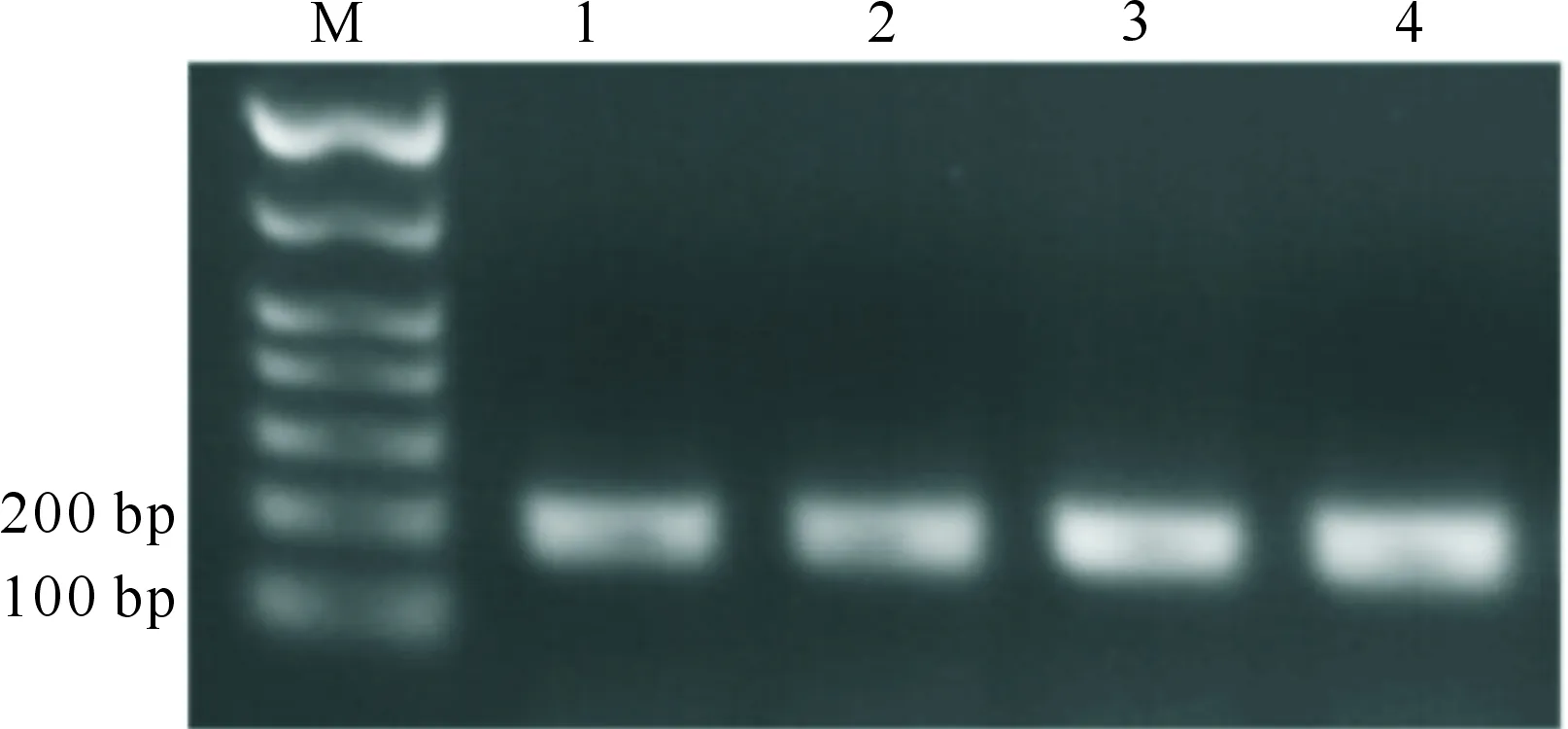
M.1 kb plus DNA Marker;1.pPLHACas9-sgRNA20r载体;2.pPLHACas9-sgRNA68r载体;3.pPLHACas9-sgRNA225f载体;4.pPLHACas9-sgRNA256f载体M.1 kb plus DNA Marker;1.pPLHACas9-sgRNA20r;2.pPLHACas9-sgRNA68r;3.pPLHACas9-sgRNA225f;4.pPLHACas9-sgRNA256f图7 山黧豆4个sgRNA靶位点载体转化农杆菌后的阳性克隆PCR结果Fig.7 PCR identification of the Agrobacterium strain conta-ining CRZSPR/Casq vectors of CASase gene in L.sativus
3 讨 论
有关山黧豆毒素β-ODAP的分离纯化、定量检测、毒理机制、生物合成及其与抗逆性的关系,已经取得了一系列的研究成果[4,18]。近期的研究表明,β-ODAP与谷氨酸竞争性结合离子型受体α-氨基-3-羟基-5-甲基-4-异恶唑丙酸(α-amino-3-hydroxy-5-methyl-4-isoxazole propionic acidreceptor,AMPAR),这会过度激活胞内钙离子流的信号级联反应,导致细胞过表达细胞膜表面的β1整合蛋白;并可进一步导致黏着斑激酶(FAK)的磷酸化和桩蛋白过表达,使得肌动蛋白微丝上大量聚合黏着斑单体,干扰细胞微丝的装配,使其严重变形并最终破坏细胞骨架[19]。这些研究加深了对β-ODAP生物学功能的理解,然而,对于β-ODAP含量的分子调控机制仍然知之甚少。
Tripathy等[20]对具有不同β-ODAP含量的山黧豆进行大量杂交后发现,至少有2个位点在β-ODAP的生物合成中具有重要作用。研究表明,山黧豆与硫营养比较丰富的谷类作物混合食用可以明显减少中毒的发生[21],而去除培养基中的Cys和Met等含硫氨基酸则会加剧β-ODAP对神经元细胞培养株系的损伤[22]。结合β-ODAP的生物合成途径,Xu等[5]认为,CASase是调控山黧豆毒素β-ODAP含量与硫代谢平衡的关键酶。由于CASase活性与β-ODAP的直接前体BIA的积累量呈显著正相关[10-11],因而,在基因水平上敲除或下调CASase基因可能会使β-ODAP含量下调,同时升高含硫氨基酸的水平。张明科等[16]构建了山黧豆CASase基因RNAi载体,Li等[23]建立了山黧豆的高效离体再生体系,这均有利于调控CASase基因表达的进一步研究。
本研究获得了山黧豆CASase基因3 143 bp的DNA全长序列,设计并检测了5个sgRNA靶位点;利用酶切连接构建了4个sgRNA靶位点的CRISPR/Cas9基因敲除载体,为进一步敲除山黧豆CASase基因,筛选“低毒、高硫”山黧豆品系奠定了基础。
