生物信息学方法筛选原发性肝癌早期诊断相关miRNA
2018-06-27
空军军医大学(第四军医大学)唐都医院传染病科/全军感染病诊疗中心,陕西 西安 710038
【Abstract】ObjectiveTo screen miRNAs by bioinformatics for the early diagnostic tests of primary liver cancer.MethodsThe data of miRNA expression in primary liver cancer tissues were downloaded from the Cancer Genome Atlas (TCGA) database. The expression levels of miRNAs were compared by groupedt-test. miRNAs with a significantly difference of more than 2-time expression levels were enrolled. Target genes of enrolled miRNAs were analyzed based on the miRNA-target analysis tool miRWalk 2.0, and common mutation genes related to primary liver cancer were acquired from c-Bioportal database. MiRNAs whose target genes contained liver cancer related mutation gene were selected, some of which were identified as target miRNAs combined with literature reports.ResultsThe TCGA data included 372 cases of primary liver cancer tissues and 50 cases of normal liver tissues with 1 881 miRNA detected. Forty-one miRNAs increased to more than 2 times in liver cancer tissues, and 115 decreased to more than 1/2. Sixty-eight common mutant genes of primary liver cancer (incidence >5%) were obtained from the c-Bioportal.ConclusionThe bioinformatics method based on TCGA database can easily and reliably screen target miRNAs for follow-up study, which has high popularization value.
【Keywords】 Bioinformatics; Primary liver cancer; Early diagnosis; MicroRNA
miRNA是高度保守、19~25核苷酸长度的单链非编码RNA,参与调控机体发育、细胞增殖/分化、细胞死亡、代谢等生物学过程。miRNA种类众多,数据库miRBase 2014年数据表明,仅在人(homo sapiens,HSA)中经测序确定的成熟miRNA序列就有2 588条。miRNA通过与信使RNA调控区的3′或5′非编码区序列特异性互补结合,阻断mRNA翻译或促进mRNA降解,使相应靶基因沉默或低表达,发挥基因抑制作用。一种miRNA可作用于多个靶基因,同一个基因也可受多种miRNA调节,这种网络式相互作用增大了miRNA生物学功能研究的难度,目前主要利用miRNA缺失、低表达和过表达的动物模型和细胞系研究miRNA的确切功能。随着miRNA生理功能研究的持续深入,更多新的发现帮助人们理解生命活动,辅助人类疾病诊疗的基础研究。
鉴于miRNA广泛、多样地参与生理、病理过程,近年来越来越多的相关研究聚焦于miRNA在疾病发生、发展、诊断、治疗及预后中的作用。体外实验、动物实验及患者体内miRNA表达水平变化等研究发现,miRNA在许多疾病的发病过程中发挥重要作用,如恶性肿瘤、心血管疾病、糖尿病、肥胖、神经系统疾病等。如何将miRNA与疾病的临床管理联系起来是研究热点,主要方向是恶性肿瘤的早期诊断、慢性疾病的治疗监测、预后预测等[6]。近年来,miRNA作为原发性肝癌无创诊断标志物的研究很多,显示出良好的临床前景,但不同miRNA的诊断效能高低不一,选择合适的miRNA十分重要[7]。本文拟利用生物信息学分析,建立一套简便而可靠的方法,筛选合适的miRNA用于原发性肝癌的早期诊断。
1 材料与方法
1.1材料网络在线数据库:miRNA基本信息数据库miRBase(http://www.mirbase.org/),miRNA靶点预测数据库miRWalk 2.0(http://zmf.umm.uni-heidelberg.de/apps/zmf/mirwalk2/),肿瘤基因组数据库TCGA(The Cancer Genome atlas,https://cancergenome.nih.gov/)。工具网站:癌症基因数据分析的可视化工具c-Bioportal(cBio Cancer Genomics Portal,http://www.cbioportal.org/),TCGA数据库RNA数据可视化及生存分析工具OncoLnc(http://www.oncolnc.org/)。数据分析及辅助工具:统计学分析软件SPSS 22.0(美国IBM公司),文献搜索引擎PubMed(https://www.ncbi.nlm.nih.gov/pubmed/)。
1.2 方法
1.2.1 原发性肝癌相关miRNA发掘:(1)miRNA数据下载:从TCGA数据库获取原发性肝癌组织miRNA表达情况数据;(2)筛选差异表达的miRNA:TCGA数据库的原发性肝癌患者miRNA表达情况由新一代测序技术(next-generation sequencing,NGS)得到,样本分为两组,一组为原发性肝癌癌组织中miRNA新一代测序结果,另一组为癌旁正常肝组织miRNA新一代测序结果。成组t检验进行miRNA表达水平的比较,P<
0.05为差异有统计学意义,获得表达水平差异2倍以上的miRNA。
1.2.2 原发性肝癌相关突变基因分析:c-Bioportal上获取原发性肝癌常见突变基因,结合文献报道,筛选出发生频率高于5%的突变基因。
1.2.3 确定目标miRNA:将上述得到的miRNA在靶点预测工具miRWalk 2.0分别进行分析,得到一系列可能与miRNA发生相互作用的基因,与上一步骤筛选出的原发性肝癌相关高频突变基因比较。对于原发性肝癌中miRNA表达变化2倍以上者,若其靶基因中含原发性肝癌高频突变基因,则定为目标miRNA。
2 结果
2.1原发性肝癌组织中表达水平显著变化的miRNA分析TCGA数据库中得到的原发性肝癌miRNA表达谱数据,共有1 881条miRNA序列,原发性肝癌癌组织372例,正常对照肝组织50例。NGS检测得到的miRNA表达水平数据数值变异很大,波动于0至107之间,数值分布不对称,不利于组间比较。将所有数据进行以10为底的对数转换(lgX),分布对称性明显改善。与正常肝组织相比,原发性肝癌组织中表达升高至2倍以上的miRNA有41个,降低至1/2以下的miRNA有115个(见表1)。
2.2原发性肝癌相关突变基因原发性肝癌相关突变基因以c-Bioportal数据库为主,结合相关参考文献进行查找,表2列举了发生率5%以上的原发性肝癌相关突变基因。

表1 差异表达的miRNATab 1 Deregulated miRNAs
注:(1)miRNA表达差异为原发性肝癌组织与癌旁组织相比较;(2)表中有“/”连接者表示多个前部相同的miRNA。
2.3miRNA靶点预测与目标miRNA确定在miRWalk 2.0中分析上述表达差异2倍以上的miRNA,得到其靶基因。筛选所得的靶点需要经至少一种检测方法的检验并有正式发表文献支持,其中高等级证据包括报告基因分析、蛋白质免疫印迹试验、qPCR,证据较弱的检测方法有基因芯片检测、NGS等。miRNA靶基因中含有原发性肝癌相关突变基因者初步作为候选miRNA。将候选miRNA在PubMed逐一检索,结合检索结果确认候选miRNA作为待研究miRNA的可靠性。
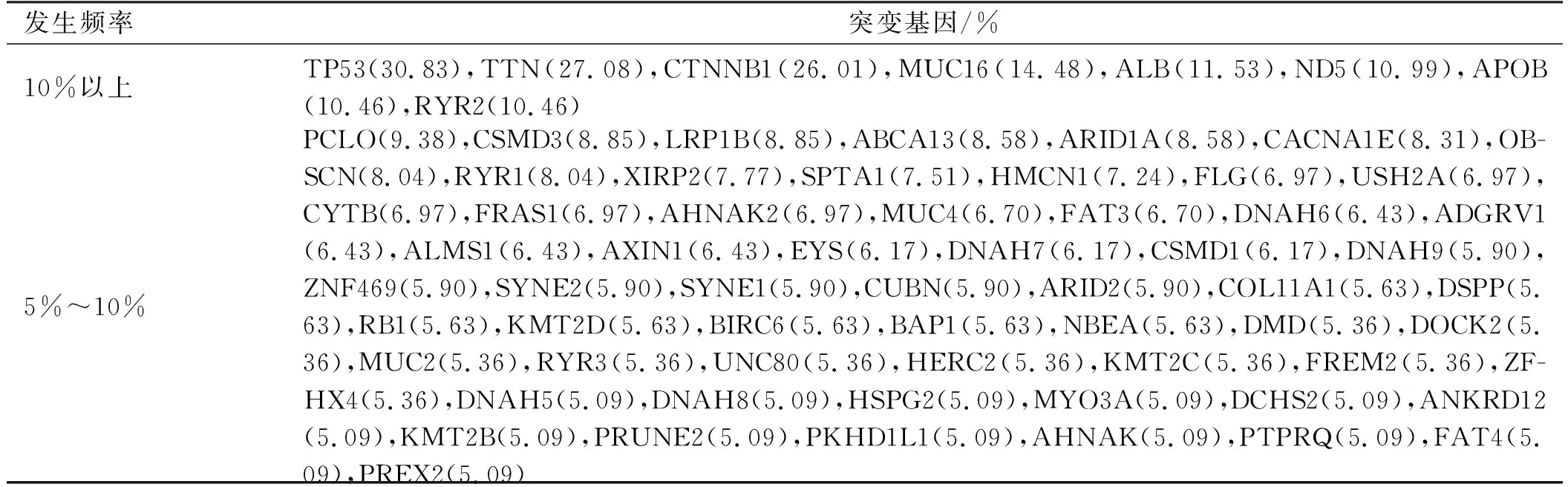
表2 原发性肝癌相关高频突变基因Tab 2 High-frequency gene mutations associated with primary liver cancer
3 讨论
miRNA与原发性肝癌的关系一向是热门课题,每年均有大量文献发表。miRNA种类繁多,广泛参与机体生理功能、病理变化,但仅有部分miRNA表现出明显组织或细胞特异性[8]。研究原发性肝癌早期诊断相关的miRNA,首先需要寻找目标miRNA。合适的目标miRNA一般需具备以下特点:(1)在原发性肝癌组织中表达水平显著异于正常肝组织;(2)miRNA直接参与或间接涉及原发性肝癌发病机制、侵袭或转移过程;(3)对肝癌细胞系中miRNA表达水平人为干预可抑制或促进癌细胞生长、增殖或恶性表现[9]。其中,第一条最为重要,也是筛选目标miRNA的第一步。
寻找新的miRNA一般使用高通量的检测方法如NGS,可极大提高工作效率,但费用高昂,数据分析复杂,一般需要大型项目支持[10]。选择原发性肝癌诊断相关miRNA时,很多研究者参考已有miRNA信息,使用高通量检测方法微芯片技术从肝癌细胞系或患者癌组织、体液中筛选目的miRNA,获取一个或多个miRNA用于后续分子机制或诊断试验等研究[11]。但该研究方式缺陷显而易见。首先,微芯片技术成本不低,资金不充裕的情况下很难进行大样本筛查,得到的miRNA差异表达结果可靠性不高。其次,微芯片技术灵敏性高,但假阳性亦很常见,筛选试验中往往得到大量差异表达的miRNA,需要对结果进行合理取舍及进一步筛选[12]。第三,原发性肝癌发病机制复杂,涉及的突变基因众多,上下游受影响的miRNA数量难以预估,每一个原发性肝癌患者或每一株肝癌细胞系的miRNA表达谱均可能互不相同。因此,寻找简便、可靠的筛选目标miRNA的方法在miRNA临床研究中意义重大。
TCGA是美国国立癌症研究所(National Cancer Institute,NCI)和美国国立人类基因组研究所(National Human Genome Research Institute,NHGRI)联合推进的癌症基因组图谱计划,通过大规模的基因组测序和分析技术,获取多种癌症的基因表达、DNA甲基化、基因突变、miRNA表达等信息,以期全面理解恶性肿瘤发病、进展的分子机制。TCGA有一套完善而严谨的工作流程,包括样本及入组患者或健康志愿者临床信息的收集、分类、处理、质检和储存,高通量技术方法进行基因组鉴定、测序,专门的数据整理和分析中心,全流程保证获得数据的准确、可靠[13]。包括原发性肝癌在内,TCGA目前已经收集了61个部位的恶性肿瘤的基因组数据资料,且相当多的数据公开无偿使用,在国际上知名度相当高。除数据质量过硬以外,大样本量、含详细临床资料和持续随访也是TCGA数据的典型特点。然而,NGS技术产生的数据量大而复杂,通常需生物信息学专业人士处理,无编程基础的非专业人员一般难以分析利用NGS原始数据[14]。近年来,基于TCGA数据进行二次开发的可视化在线工具大量涌现,如c-Bioportal、OncoLnc和MEXPRESS等,使得无生物信息学知识背景的研究者也可轻松使用TCGA数据进行课题研究[15]。目前,利用TCGA公开数据筛选原发性肝癌相关miRNA也是较常用手段[16],我们认为,该方法可能比用小样本肝癌患者或多种肝癌细胞系进行miRNA筛选中更有效率、更准确。
在探索miRNA对原发性肝癌诊断价值的研究中,筛选相关性强、特异性好的miRNA作为血清学诊断标志物至关重要[17]。为使筛选的miRNA与原发性肝癌相关性更强,除要求miRNA在肝癌组织中表达水平较正常肝组织变化2倍以上,尚需其与至少一个原发性肝癌突变基因发生相互作用,而这种相互作用需要较强的证据支持。为此,我们在利用TCGA数据得到差异表达的miRNA后,进一步在miRNA靶点预测在线工具miRWalk 2.0上大规模寻找备选miRNA的可能作用靶点,并将靶点与原发性肝癌高频突变基因进行比对,有重合者列为重点考虑miRNA,以期找到与原发性肝癌有强相关性的miRNA[18]。miRWalk 2.0是一个miRNA靶点预测工具,除自身的算法工具以外,其整合了其他12个miRNA靶点预测项目的资源[DIANA-microTv4.0、DIANA-microT-CDS、miRanda-rel2010、mirBridge、miRDB4.0、miRmap、miRNAMap、doRiNA(PicTar2)、PITA、RNA22v2、RNAhybrid2.1和Targetscan6.2],提供miRNA在启动子、编码序列、5′非翻译区和3′非翻译区上结合位点的计算和预测,并从miRTarBase、PhenomiR、miR2Disease和HMDD获取miRNA-靶点相互作用的实验验证证据[19]。在miRWalk 2.0上将前述差异表达2倍以上的miRNA进行检索,若得到的靶点中包含原发性肝癌相关高频突变基因且得到高强度实验证据,则确认为候选miRNA。将候选基因miRNA在PubMed逐一检索,浏览文献摘要,确认候选miRNA作为原发性肝癌无创诊断标志物的可行性。
目前已发表的关于miRNA诊断原发性肝癌的研究较多,且在持续增加,由于单个miRNA对原发性肝癌的诊断敏感性较差,准确性也不尽如人意,目前研究者越来越倾向于使用多种miRNA联合诊断,且常常联合血清AFP,以提高诊断灵敏性和特异性[20]。综合考虑临床应用的经济性和减少实验操作误差,早期诊断模型中miRNA数量不宜过多,参考既往研究[20-21]结果,联合3~4种miRNA建立原发性肝癌诊断模型比较理想。本文利用网络数据库TCGA中原发性肝癌相关miRNA数据进行生物信息学分析,结合肝癌突变基因及miRNA-靶基因相互作用数据,筛选miRNA进行后续原发性肝癌的早期诊断研究,方法简便,涉及统计学技术不复杂,值得推广。缺点在于TCGA数据来自于原发性肝癌组织,据此筛选的miRNA是否在循环中的表达水平也有显著差异,需要在临床样本中进一步确认。
[1] AMBROS V. The functions of animal microRNAs [J]. Nature, 2004, 431(7006): 350-355. DOI: 10.1038/nature02871.
[2] KOZOMARA A, GRIFFITHS-JONES S. miRBase: annotating high confidence microRNAs using deep sequencing data [J]. Nucleic Acids Res, 2014, 42(Database issue): D68-D73. DOI: 10.1093/nar/gkt1181.
[3] IWAKAWA H O, TOMARI Y. The functions of microRNAs: mRNA decay and translational repression [J]. Trends Cell Biol, 2015, 25(11): 651-665. DOI: 10.1016/j.tcb.2015.07.011.
[4] BUSHATI N, COHEN S M. MicroRNA functions [J]. Annu Rev Cell Dev Biol, 2007, 23: 175-205. DOI: 10.1146/annurev.cellbio.23.090506.123406.
[5] KUMAR S, VIJAYAN M, BHATTI J S, et al. MicroRNAs as peripheral biomarkers in aging and age-related diseases [J]. Prog Mol Biol Transl Sci, 2017, 146: 47-94. DOI: 10.1016/bs.pmbts.2016.12.013.
[6] BERINDAN-NEAGOE I, MONROIG P C, PASCULLI B, et al. MicroRNAome genome: a treasure for cancer diagnosis and therapy [J]. CA Cancer J Clin, 2014, 64(5): 311-336. DOI: 10.3322/caac.21244.
[7] YANG N, EKANEM N R, SAKYI C A, et al. Hepatocellular carcinoma and microRNA: new perspectives on therapeutics and diagnostics [J]. Adv Drug Deliv Rev, 2015, 81: 62-74. DOI: 10.1016/j.addr.2014.10.029.
[8] LONDIN E, LOHER P, TELONIS A G, et al. Analysis of 13 cell types reveals evidence for the expression of numerous novel primate- and tissue-specific microRNAs [J]. Proc Natl Acad Sci U S A, 2015, 112(10): E1106-E1115. DOI: 10.1073/pnas.1420955112.
[9] WU H, TAO J, LI X, et al. MicroRNA-206 prevents the pathogenesis of hepatocellular carcinoma by modulating expression of met proto-oncogene and cyclin-dependent kinase 6 in mice [J]. Hepatology, 2017, 66(6): 1952-1967. DOI: 10.1002/hep.29374.
[10] DE RIE D, ABUGESSAISA I, ALAM T, et al. An integrated expression atlas of miRNAs and their promoters in human and mouse [J]. Nat Biotechnol, 2017, 35(9): 872-878. DOI: 10.1038/nbt.3947.
[11] WEN Y, HAN J, CHEN J, et al. Plasma miRNAs as early biomarkers for detecting hepatocellular carcinoma [J]. Int J Cancer, 2015, 137(7): 1679-1690. DOI: 10.1002/ijc.29544.
[12] FIEGLER H, REDON R, ANDREWS D, et al. Accurate and reliable high-throughput detection of copy number variation in the human genome [J]. Genome Res, 2006, 16(12): 1566-1574. DOI: 10.1101/gr.5630906.
[13] WANG Z, JENSEN M A, ZENKLUSEN J C. A practical guide to the Cancer Genome Atlas (TCGA) [J]. Methods Mol Biol, 2016, 1418: 111-141. DOI: 10.1007/978-1-4939-3578-9_6.
[14] SONG K, REN J, REINERT G, et al. New developments of alignment-free sequence comparison: measures, statistics and next-generation sequencing [J]. Brief Bioinform, 2014, 15(3): 343-353. DOI: 10.1093/bib/bbt067.
[15] GAO L, ZHANG L J, LI S H, et al. Role of miR-452-5p in the tumorigenesis of prostate cancer: a study based on the Cancer Genome Atl (TCGA), Gene Expression Omnibus (GEO), and bioinformatics analysis [J]. Pathol Res Pract, 2018.[Epub ahead of print]. DOI: 10.1016/j.prp.2018.03.002.
[16] HUNG C H, HU T H, LU S N, et al. Circulating microRNAs as biomarkers for diagnosis of early hepatocellular carcinoma associated with hepatitis B virus [J]. Int J Cancer, 2016, 138(3): 714-720. DOI: 10.1002/ijc.29802.
[17] SHEN S, LIN Y, YUAN X, et al. Biomarker microRNAs for diagnosis, prognosis and treatment of hepatocellular carcinoma: a functional survey and comparison [J]. Sci Rep, 2016, 6: 38311. DOI: 10.1038/srep38311.
[18] DWEEP H, STICHT C, PANDEY P, et al. miRWalk--database: prediction of possible miRNA binding sites by "walking" the genes of three genomes [J]. J Biomed Inform, 2011, 44(5): 839-847. DOI: 10.1016/j.jbi.2011.05.002.
[19] DWEEP H, GRETZ N. miRWalk2.0: a comprehensive atlas of microRNA-target interactions [J]. Nat Methods, 2015, 12(8): 697. DOI: 10.1038/nmeth.3485.
[20] ZUO D, CHEN L, LIU X, et al. Combination of miR-125b and miR-27a enhances sensitivity and specificity of AFP-based diagnosis of hepatocellular carcinoma [J]. Tumour Biol, 2016, 37(5): 6539-6549. DOI: 10.1007/s13277-015-4545-1.
[21] ZHUANG C, JIANG W, HUANG D, et al. Serum miR-21, miR-26a and miR-101 as potential biomarkers of hepatocellular carcinoma [J]. Clin Res Hepatol Gas, 2016, 40(4): 386-396. DOI: 10.1016/j.clinre.2015.11.002.
doi:10.3969/j.issn.1006-5709.2018.06.004
