靶向加工番茄elF4E1的CRISPR/Cas9载体有效性验证
2018-04-17付紫梅邵冬南郝小军崔百明向本春
付紫梅,袁 伦,邵冬南,郝小军,崔百明,向本春
(1.石河子大学生命科学学院/石河子大学农业生物技术重点实验室,新疆石河子 832003;2.威斯腾生物医药科技有限责任公司,重庆 400039;3.新疆绿洲农业病虫害治理与植保资源利用自治区普通高校重点实验室/石河子大学农学院,新疆石河子 832003)
0 引 言
【研究意义】新疆是我国最大的加工番茄种植基地,加工番茄作为新疆经济的支柱性产业之一,对新疆经济发展有着举足轻重的影响[1]。然而病毒病害一直以来都是影响加工番茄产量和品质的限制性因素,马铃薯Y病毒(PotatoVirusY, PVY)是马铃薯 Y病毒属(Potyviruses)的典型代表,同时也是感染新疆加工番茄的主要病毒种类之一[2]。利用基因工程的手段,从分子水平上培育新的抗病品种,是需要迫切解决的问题。【前人研究进展】CRISPR/Cas9(clustered regularly interspaced short palindromic repeat-associated protein9)作为继锌指核酸酶(Zinc-finger nucleases,ZFN)、转录激活因子样效应子核酸酶(transcription activator-like effector nucleases,TALENs)之后的第三代基因组编辑技术,自2013年被报道以来,由于简便、高效的特点,在动植物基因组编辑中被广泛的应用[3-4],到目前为止,已在水稻、土豆、玉米、番茄等经济作物功能基因的编辑中取得显著成效[5-8]。Mccallum C M等[9]通过TILLING(Targeting Induced Local Lesions IN Genomes)技术靶向番茄eIF4E基因,获得了抗PVY的eIF4E功能失活突变体。Mazier M等[10]通过RNA干扰的手段下调eIF4E1和eIF4E2基因,使得番茄植株对马铃薯Y病毒属病毒获得了更广谱的抗性。然而利用新兴的基因组编辑技术CRISPR/Cas9靶向编辑PVY抗病相关基因eIF4E1(eukaryotic translation initiation factors 4E),从分子水平上改良加工番茄品种的研究却鲜有报道。【本研究切入点】翻译起始因子是一个小的多基因家族,在功能上具有冗余[11],在番茄当中已鉴定出的3个成员,分别是eIF4E1、eIF4E2和eIF(iso)4E,其中eIF4E1和eIF4E2编码eIF4E蛋白,eIF(iso)4E编码eIF(iso)4E蛋白[12]。宿主细胞mRNA既能够利用eIF4E的同时也能够利用eIF(iso)4E来起始自身蛋白质的翻译,在番茄当中,马铃薯Y病毒VPg(viral genome-linked protein,VPg)已经演变为特异性结合于eIF4E来帮助病毒蛋白的翻译[10]。研究利用CRISPR/Cas9系统构建的特异性靶向编辑番茄真核翻译起始因子elF4E1基因的表达载体,在阻碍了马铃薯Y病毒VPg与加工番茄eIF4E相互作用的同时为新疆加工番茄抗病品种的培育提供了新方法。【拟解决的关键问题】通过瞬时转化的方法来快速验证靶向eIF4E1基因的CRISPR-Cas9载体的有效性,为利用CRISPR-Cas9基因组编辑技术成功敲除eIF4E1基因,获得抗PVY病毒非转基因纯合体植株提供技术支持。
1 材料与方法
1.1 材 料
1.1.1加工番茄
研究所用的加工番茄(Solanumlycopersicum)品种里格尔(87-5)购自石河子市亚心种业有限公司。
1.1.2菌株和质粒
大肠杆菌(Escherichiacoli)DH5α和根瘤农杆菌(Agrobacteriumtumefaciens)GV3101均为该实验室-80℃保存菌种。
研究所用的CRISPR/Cas9系统相关载体Px330cas9由石河子大学生命科学学院胡圣伟老师馈赠。sgRNA表达框由上海生工生物工程股份有限公司合成并克隆到pUC19载体上。
1.2 方 法
1.2.1引物设计
根据eIF4E1靶序列设计合成引物sg_4E1_1S/sg_4E1_1A,根据hcas9序列设计合成引物hCas9-S/ hCas9-A,根据eIF4E1基因序列设计检测引物DT_Sl4E1_F/ DT_Sl4E1_R。根据载体骨架设计引物Colony-F/ Colony-F,用于转化后单克隆的菌斑PCR鉴定实验。
表1实验中所用引物
Table1Sequences of primers used in this study
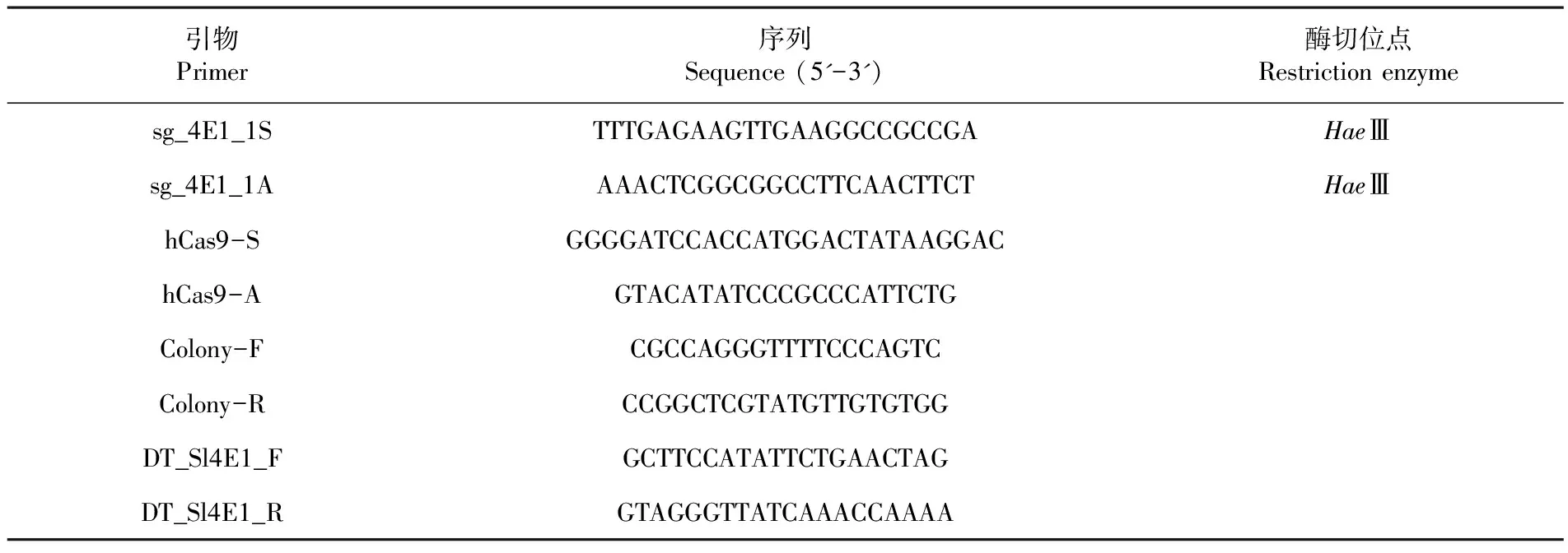
引物Primer序列Sequence(5'-3')酶切位点Restrictionenzymesg_4E1_1STTTGAGAAGTTGAAGGCCGCCGAHaeⅢsg_4E1_1AAAACTCGGCGGCCTTCAACTTCTHaeⅢhCas9-SGGGGATCCACCATGGACTATAAGGAChCas9-AGTACATATCCCGCCCATTCTGColony-FCGCCAGGGTTTTCCCAGTCColony-RCCGGCTCGTATGTTGTGTGGDT_Sl4E1_FGCTTCCATATTCTGAACTAGDT_Sl4E1_RGTAGGGTTATCAAACCAAAA
1.2.2靶序列选取
从NCBI数据库中下载eIF4E1基因序列 (NC015440),利用Vector软件在基因编码区的5'端找到NGG序列,即PAM(protospacer adjacent motifs),研究表明,番茄eIF4E1基因全长 3 340 bp,共含有5个外显子和4个内含子,根据CRISPR/Cas9系统的特异性切割原则,在综合考虑靶位点选取过程中的GC含量、鉴定过程中酶切位点的选取以及靶序列最好位于基因序列的5'等因素的情况下。研究的靶序列位于加工番茄eIF4E1基因距转录起始位点46 bp的第一个外显子上(含HaeⅢ酶切位点),并将其命名为sgR-eIF4E1:GAGAAGTTGAAGGCCGCCGA。图1

图1加工番茄eIF4E1基因靶序列示意
Fig.1The structural diagram of the Solanum lycopersicum eIF4E1 locus targeted for editing
1.2.3载体构建
将上海生工生物工程股份有限公司合成的sgRNA表达框(含番茄U6启动子)克隆到pUC19载体上,将其命名为pUC-sgRNA。以Px330为模板,hcas9-S/hcas9-A为引物进行PCR扩增,扩增产物通过NcoⅠ/BstEⅡ克隆到pCAMBIA1302载体上,通过EcoRV酶切验证后,命名为p1302-Cas9。通过引物退火的方式将sg_4E1_1S/sg_4E1_1A结合成带粘性末端的双链DNA片段后通过AarI酶克隆到pUC-sgRNA载体上,将其命名为pUC-SgR-eIF4E1。pUC-SgR-eIF4E1和p1302-Cas9载体分别用EcoRⅠ/HindⅢ双酶切,胶回收预期大小一致的片段,4℃过夜连接并转化大肠杆菌(DH5α)后,选取阳性克隆送北京华大基因公司测序验证。并将其命名为p1302-sgR-Cas9-eIF4E1。测序正确的p1302-sgR-Cas9-eIF4E1质粒电击转化农杆菌GV3101,选取阳性克隆于 -80℃甘油管中保存备用。表1,图2

图2植物表达载体p1302-sgRNA-Cas9-eIF4E1结构示意
Fig.2The structural diagram of the Plant expression vector p1302-sgRNA-Cas9-eIF4E1
1.2.4农杆菌介导番茄瞬时转化
1.2.4.1无菌苗种植
超净工作台中,将番茄种子用 2% 的次氯酸钠消毒 15 min,期间不停地摇晃,无菌水冲洗7~8次,用无菌镊子将种子接种于1/2MS培养基,28℃、黑暗条件下培养2 d,待种子发芽后,28℃、长日照下(光照16 h/黑暗8 h)培养至7~8片真叶备用。
1.2.4.2农杆菌侵染液制备
取上述 -80℃甘油管中保存的农杆菌(含重组质粒p1302-sgR-Cas9-eIF4E1),在固体LB培养基(含50 mg/L卡那霉素;50 mg/L庆大霉素;25 mg/L利福平)上划线,做好标记。28℃、黑暗条件下倒置培养2 d,挑取单斑,接种于液体LB培养基(含50 mg/L卡那霉素;50 mg/L庆大霉素;25 mg/L利福平)中,28℃,220 r/min培养2 d。将活化的菌液以1∶10的比例转移至液体LB培养基(含50 mg/L卡那霉素;50 mg/L庆大霉素;25 mg/L利福平),28℃扩大培养16~24 h至菌体生长至稳定期,OD600值为1.5。室温,4 000 g,离心 10 min,收集菌体。用1体积现配的5%蔗糖溶液悬浮菌体至无菌萌发盒中,用前加入10 μL Silwet L-77(终浓度为0.002%)。
1.2.4.3瞬时转化过程
在工作台内将无菌苗平放于含有农杆菌浸染液的萌发盒中,盖上盖。将含有菌液及番茄植株的萌发盒用隔膜真空泵在0.05 Mpa的压力下抽真空4 min。用镊子从萌发盒中取出植株放入另一个无菌萌发盒中,盖上盖,抖动,使植株表面过多的菌液滴下并用卫生纸吸干表面残余的菌液。20~25℃、黑暗条件下过夜培养后转移至1/2MS(含 300 mg/L羧苄霉素)固体培养基中,28℃、长日照下(光照16 h/黑暗8 h)培养生长。
1.2.5靶序列突变检测
以CTAB法提取的叶片基因组DNA为模版, DT_Sl4E_F和DT_Sl4E_R为引物, 94℃预变性5 min,94℃变性30 s,53℃退火30 s,72℃延伸45 s,30个循环后,72℃再延伸5 min。PCR扩增加工番茄含靶序列的eIF4E1基因部分片段。将PCR产物用回收试剂盒回收纯化后,HaeⅢ进行酶切,胶回收试剂盒回收未切开的片段与pGEM-T进行连接,连接产物转化DH5α后,用DT_Sl4E_F和DT_Sl4E_R引物(表1)进行菌斑PCR鉴定,随机挑取阳性克隆穿刺管送至北京华大生物公司测序。根据单克隆测序结果来比对分析靶序列的编辑情况。
2 结果与分析
2.1 载体的构建
利用PstⅠ酶切鉴定已构建的p1302-Cas9载体(图3A),得到9 512、1 682、1 422、1 212和24 bp大小的6条带,其特征性条带1 682、1 422和1 212 bp与预期大小一致。以sg_4E_1S和Colony-R为引物进行菌斑PCR来鉴定中间载体pUC-SgR-eIF4E1重组子,得到预期大小为150 bp的目标条带。利用特异性引物sg_4E_1A和Colony-R(表1)进行菌斑PCR鉴定植物表达载体p1302-sgR-Cas9-eIF4E1重组子,得到预期大小为374 bp的目标条带,并对阳性克隆测序验证,序列比对结果表明,p1302-sgR-Cas9-eIF4E1载体构建成功,其中sgR-eIF4E1靶序列的表达框含有番茄U6启动子,用以特异性识别和结合靶序列。Cas9表达框含有花椰菜花叶病毒(CaMV)的35S启动子和终止子,用以对靶序列进行特异性切割。提取p1302-sgR-Cas9-eIF4E1载体质粒,电击转化农杆菌GV3101,对转化的菌斑进行菌斑PCR验证,得到预期大小为374 bp的电泳条带(图3B),说明载体质粒成功转化农杆菌GV3101,可用于后续的瞬时转化实验。表1,图3

注:A:p1302-Cas9载体的PstⅠ酶切鉴定; M:DL15000;P:p1302-Cas9/PstⅠ; B:菌落PCR鉴定RISPR/Cas9重组载体转化的农杆菌菌落;M:DL2000;N:空白对照;G:GV3101,阴性对照;1~4:重组载体菌落;P:p1302-sgRNA-Cas9-eIF4E1质粒,阳性对照
Note: A:Restriction enzyme digestion of thep1302-Cas9; B:PCR verification ofAgrobacteriumtumefacienscolonies transformed with CRISPR/Cas9 recombinant vecter; M:DL2000 marker; N: Blank control; G:GV3101, negative control; 1-4: Recombinant vecterAgrobacteriumtumefacienscolony; P:p1302-sgRNA-Cas9-eIF4E1 plasmid, positive control
图3靶向eIF4E1基因载体构建
Fig.3Construction of CRISPR-Cas9 vectors targeting eIF4E1 gene
2.2 靶序列突变检测
以瞬时转化加工番茄植株和野生型加工番茄植株的基因组DNA为模板, DT_Sl4E_F和DT_Sl4E_R为引物,扩增包含靶序列的加工番茄eIF4E1基因部分片段,扩增结果(图4A),所有扩增样品均出现了与预期大小一致的408 bp的条带,PCR产物回收纯化后用HaeⅢ进行酶切(图3B),研究表明,2个瞬时转化植株相对于野生型对照均出现了HaeⅢ未切开的408 bp的条带,表明这两个植株中都有部分细胞被编辑。胶回收未切开的408 bp大小的条带与pGEM-T进行连接,连接产物转化DH5α后,随机挑取11个克隆,用DT_Sl4E_F和DT_Sl4E_R引物(表1)进行菌斑PCR鉴定(图4C),这11个克隆中有9个为阳性克隆。随后将这9个阳性克隆穿刺管送至北京华大生物公司测序。表1,图4

注:A:靶序列处eIF4E1基因片段的PCR扩增;B:HaeⅢ对PCR产物的酶切筛选;C:单克隆菌斑PCR鉴定;M:DL1000 marker;N:空白对照;W:阴性对照;P:阳性对照
Note: A: PCR amplification ofeIF4E1 gene fragment around the target sequence; B: The screening of PCR products byHaeⅢ; C: The PCR identification of Monoclonal plaque; M: DL1000 marker; N: Blank control; W: Negative control; P: Positive control
图4靶序列突变检测
Fig.4The mutation detection of target sequence
2.3 测序结果比对
将这9个克隆的靶位点处的核苷酸序列和氨基酸序列分别与野生型进行了比对,核苷酸序列比对结果(图5A),9个阳性克隆在PAM上游的第6~8个核苷酸处均发生突变,并且所有的突变均为单碱基的替换。相对应的靶位点处的氨基酸序列比对结果(图5B),研究表明,除6号克隆的氨基酸序列没有改变外,2、7、8、10、11号克隆靶位点处的丙氨酸被缬氨酸所替换,1和4号克隆靶位点处的丙氨酸被天冬氨酸替换,9号克隆的丙氨酸被苏氨酸替换。图5

注:A:核苷酸序列比对结果 (浅灰色为突变碱基位置);B:氨基酸序列比对结果(浅灰色为氨基酸突变区)
Note: A: The comparison results of the nucleotide sequence (Light gray marks the location of base mutation); B: The comparison results of the amino acid sequence (Light gray marks the region of amino acid)
图5靶位点区域核苷酸序列与氨基酸序列比对结果
Fig.5The comparison results of the nucleotide sequence and amino acid sequence at target region
3 讨 论
CRISPR/Cas9基因组编辑系统来源于细菌和古细菌,是自然界普遍存在于原核生物的一种免疫防卫系统,通过激活适应性免疫应答来抵抗质粒DNA或病毒的入侵[13]。经过人工改造后的Ⅱ型RISPR/Cas9系统在编辑过程中,sgRNA通过和靶序列的碱基配对结合到DNA上后引导Cas9核酸内切酶结合并识别靶序列下游的PAM(protospacer adjacent motifs)基序,在PAM上游约3 bp处切割DNA双链,形成平端的DNA双链断裂(double strand break,DSB),DSB在通过同源重组(homology-directed repair,HDR)和非同源性末端连接(non-homologous end joining,NHEJ)进行修复的过程中。形成靶序列处碱基的替换、插入或删除,从而形成对DNA靶位点基因编辑[14-16]。 测序结果显示所获得的9个阳性单克隆突变情况均为单碱基的替换,这与潘洪杏等[17]利用CRISPR/Cas9 系统定向敲除烟草eIF4E-6 基因后的编辑情况基本一致。推测可能是因为eIF4E1作为翻译起始因子对植物自身生长比较重要,在Cas9蛋白酶切割DNA双链后,植物出于一种防御反应优先利用同源重组来进行修复,从而出现单碱基替换的突变方式。
为了验证利用CRISPR/Cas9基因组编辑技术构建的靶向eIF4E1基因载体的有效性,研究采用了真空渗透的瞬时转化方法来对编辑情况进行评估。HaeⅢ酶切鉴定结果和序列比对结果都表明研究所采取的瞬时转化方案是可行的,这相对于采用PEG介导的原生质体的瞬时转化方法更加的方便快捷[18],所以实验所采用的瞬时转化方法可应用于CRISPR/Cas9 系统遗传转化之前的靶序列评估实验,为CRISPR/Cas9 系统在后续番茄品种改良过程中更加精准的编辑提供了方便。
4 结 论
研究利用真空渗透的瞬时转化方法对基于CRISPR/Cas9 系统构建的p1302-sgRNA-Cas9-eIF4E1植物表达载体进行了验证。从HaeⅢ酶切检测结果可以看出,未切开的408 bp条带较亮,说明这两个植株中被编辑的细胞较多,这表明利用CRISPR/Cas9系统构建的靶向编辑eIF4E1基因的载体的编辑效率较高。所获得的9个单克隆的序列比对结果表明,所有测序克隆在PAM上游6~8 bp的碱基处发生了单碱基的替换,氨基酸比对结果发现,有8个克隆在相同的位点出现了单个氨基酸的突变,表明研究构建的p1302-sgRNA-Cas9-eIF4E1能够特异性的靶向加工番茄eIF4E1。这为后续CRISPR/Cas9系统在加工番茄品种改良过程中的成功应用提供了技术支持。
参考文献(References)
[1] 姚彤,陈丰.新疆成为亚洲最大的番茄生产和加工基地[J].大陆桥视野,2012,(15):74-75.
YAO Tong, CHEN Fen. (2012). Xinjiang becomes Asia's largest Tomato production and processing Base [J].NewSilkRoadHorizon, (15):74-75. (in Chinese)
[2] 都业娟,石宝萍,李成亮,等.加工番茄病毒病田间发生情况及毒原的分子检测[J].植物保护, 2013, 39(4):110-115.
DU Ye-juan, SHI Bao-ping, LI Cheng-liang, et al. (2013). Incidence and molecular detection of virus on processing tomato in the field [J].PlantProtection, 39(4):110-115. (in Chinese)
[3] 瞿礼嘉,郭冬姝,张金喆,等.CRISPR/Cas系统在植物基因组编辑中的应用[J].生命科学,2015,27(1):64-70.
QU Li-jia, GUO Dong-shu, ZHANG Jin-zhe, et al. (2015). The application of CRISPR/Cas system in plant genome editing [J].ChineseBulletinofLifeSciences, 27(1):64-70. (in Chinese)
[4] 解莉楠,宋凤艳,张旸.CRISPR/Cas9系统在植物基因组定点编辑中的研究进展[J].中国农业科学,2015,48(9):1 669-1 677.
XIE Li-nan, ZONG Fen-yan, ZHANG Yang. (2015). Progress in Research of CRISPR/Cas9 System in Genome Targeted Editing in Plants [J].ScientiaAgriculturaSinica, 48(9):1,669-1,677. (in Chinese)
[5] Endo, M., Mikami, M., & Toki, S. (2015). Multigene knockout utilizing off-target mutations of the crispr/cas9 system in rice.Plant&CellPhysiology, 56(1): 41.
[6] Andersson, M., Turesson, H., Nicolia, A., F?lt, A. S., Samuelsson, M., & Hofvander, P. (2017). Efficient targeted multiallelic mutagenesis in tetraploid potato (solanum tuberosum) by transient crispr-cas9 expression in protoplasts:.PlantCellReports, 36(1): 1-12.
[7] Qi, W., Zhu, T., Tian, Z., Li, C., Zhang, W., & Song, R. (2016). High-efficiency crispr/cas9 multiplex gene editing using the glycine trna-processing system-based strategy in maize:BMCBiotechnology, 16(1): 58.
[8] Ron, M., Kajala, K., Pauluzzi, G., Wang, D., Reynoso, M. A., & Zumstein, K., et al. (2014). Hairy root transformation using agrobacterium rhizogenes as a tool for exploring cell type-specific gene expression and function using tomato as a model.PlantPhysiology, 166(2): 455.
[9] Mccallum, C. M., Comai, L., Greene, E. A., & Henikoff, S. (2000). Targeting induced local lesions in genomes (tilling) for plant functional genomics.PlantPhysiology, 123(2): 439.
[10] Mazier, M., Flamain, F., Nicoaï, M., Sarnette, V., & Caranta, C. (2011). Knock-down of botheif4e1 andeif4e2 genes confers broad-spectrum resistance against potyviruses in tomato.PlosOne, 6(12): e29595.
[11] Duprat, A., Caranta, C., Revers, F., Menand, B., Browning, K. S., & Robaglia, C. (2002). The arabidopsis eukaryotic initiation factor (iso)4e is dispensable for plant growth but required for susceptibility to potyviruses.PlantJournal, 32(6): 927-934.
[12] Piron, F., Nicolaï, M., Minoïa, S., Piednoir, E., Moretti, A., & Salgues, A., et al. (2010). An induced mutation in tomato eif4e leads to immunity to two potyviruses.PlosOne, 5(6): e11313.
[13] Barrangou, R., Fremaux, C., Deveau, H., Richards, M., Boyaval, P., & Moineau, S., et al. (2007). Crispr provides acquired resistance against viruses in prokaryotes.Science, 315(5819): 1,709.
[14] Cong, L., Ran, F. A., Cox, D., Lin, S., Barretto, R., & Habib, N., et al. (2013). Multiplex genome engineering using crispr/cas systems.TrendsinGeneticsTig, 32(12): 815.
[15] Ma, X., Zhu, Q., Chen, Y., & Liu, Y. G. (2016). Crispr/cas9 platforms for genome editing in plants: developments and applications.MolecularPlant, 9(7): 961-974.
[16] Jing, R. C., & Hong, L. U. (2016). The development of crispr/cas9 system and its application in crop genome editing.ScientiaAgriculturaSinica.
[17] 潘洪杏,刘侠,万秀清,等.利用CRISPR-Cas9基因组编辑技术定向敲除烟草eIF4E-6基因[J].分子植物育种, 2017,(2):538-544.
PAN Hong-xing, LIU Xia, WAN Xiu-qing, et al. (2017). Directional Knockout ofeIF4E-6 Gene Using CRISPR-Cas9 Genome Editing Technique [J].MolecularPlantBreeding, 15(2):538-544. (in Chinese)
[18] Xing, H. L., Dong, L., Wang, Z. P., Zhang, H. Y., Han, C. Y., & Liu, B., et al. (2014). A crispr/cas9 toolkit for multiplex genome editing in plants.BmcPlantBiology, 14(1): 327.
