油菜主花序角果密度及其相关性状的全基因组关联分析
2018-04-03任义英崔翠王倩唐章林徐新福林呐殷家明李加纳周清元
任义英,崔翠,王倩,唐章林,徐新福,林呐,殷家明,李加纳,周清元
(西南大学农学与生物科技学院,重庆 400715)
0 引言
【研究意义】油菜是世界上主要的油料作物,在中国是继水稻、小麦、玉米、大豆之后的第五大作物,也是中国食用植物油和动物饲料的重要来源[1-2]。角果是主要的光合器官,具有“库”和“源”的双重作用[3-4]。适宜的角果密度可以提高光能利用率,增加籽粒数量,从而提高油菜产量[5-6]。主花序角果数和主花序长是主花序角果密度的组成型性状,分别反应了主花序角果的分布情况和植株的松散程度,对油菜产量有直接或间接的影响。因此,鉴定和定位控制主花序角果密度及其相关性状的 QTL,对挖掘高产基因和培育高产品种具有重要的指导意义。【前人研究进展】角果密度、主花序角果数和主花序长的广义遗传率都较高,遗传比较稳定,受环境因素的影响较小[7-9]。余凤群等[10]发现角果密度和主花序长都受多基因控制,且控制角果密度的基因存在互补作用,控制主花序长性状的基因存在重叠作用。也有研究表明,主花序有效长、主花序角果数和角果密度受显性效应、显性与环境互作效应以及上位性效应的影响,其中主花序有效长和主花序角果数也受加性效应控制[12-14]。随着QTL定位方法和分子标记技术的飞速发展,推动了甘蓝型油菜角果相关性状QTL的研究进展。CHEN等[15]检测到15个与主花序角果密度有关的QTL,单个位点的表型贡献率在 2.79%—7.59%,其中 A06、C05、C06和C08上的4个QTL在2个群体之间共有;发现13个与主花序长度有关的QTL,单个位点的QTL贡献率在2.58%—9.22%,在2个群体中检测到7个重叠的QTL。高必军[16]在第12连锁群上检测到1个与主花序角果密度相关的QTL,解释17.3%的表型变异;5个与主花序有效角果数相关的QTL,依次位于第6、12、13连锁群,单个位点可解释性状表型变异的11.25%—25%;15个控制主花序长的QTL,分别位于第1、2、6和17连锁群,每个QTL分别解释7.4%—26.6%的表型变异。阴涛[17]检测到位于A02染色体上的2个与主花序角果密度相关的QTL,单个位点分别解释5.25%和8.07%的表型变异;在A02染色体上发现1个与主花序长相关的QTL,可解释6.25%的表型变异。目前,油菜角果密度及其相关性状的QTL研究还停留在初步定位阶段,后续研究报道较少。近年来,随着一些植物全基因组测序的完成,分子标记的开发和生物信息学的迅速发展,关联分析己成为植物数量性状基因研究的热点之一。关联分析又叫连锁不平衡作图(linkage disequilibrium mapping,LD mapping),是一种以连锁不平衡为基础,根据特定的统计方法来鉴定某一自然群体内目标性状与分子标记(或候选基因)关系的定位方法,可以直接鉴定出控制目标性状的功能基因或基因内的功能多态性[18]。关联分析以自然群体为研究对象,无需构建专门的分离群体,用时少,可以节约成本;可以检测群体内与目标性状相关一个基因座位上的多个等位基因,检测量大;自然群体积累的重组信息大,分辨率高,能够精细定位目标性状,精确到单基因水平[19-20]。随着甘蓝型油菜全基因组序列的公布[21]及芸薹属 60 K SNP(single nucleotide polymorphism)芯片的开发,关联分析在甘蓝型油菜中的运用也取得了较大的成果,并鉴定出一些与重要农艺、品质性状相关联的位点[21-25]。【本研究切入点】尽管已有油菜角果密度及其相关性状的QTL的研究报道,但多数为初步定位阶段,未能进一步定位和预测候选基因。【拟解决的关键问题】本研究采用不同遗传背景和地理来源的213份甘蓝型油菜构建自然群体,结合SNP芯片数据及重庆北碚2年的表型数据,拟对主花序角果密度、主花序有效长和主花序有效角果数进行全基因组关联分析,并进一步预测与性状相关的重要候选基因,为通过分子标记辅助选择等方式提高油菜产量提供理论依据。
1 材料与方法
1.1 植物材料
213份关联分析材料为具有不同遗传背景和广泛地理来源的常规品种、品系(电子附表 1),由重庆市油菜工程技术研究中心收集并提供。
1.2 田间试验和性状调查
将213份材料分别在2014年和2015年10月种植于重庆市油菜工程技术研究中心歇马实验基地,11月份田间移栽,按完全随机区组设计,每种材料种植 3行,每行10株,行距40 cm,株距20 cm,重复2次。待油菜成熟时,选取10株长势相对一致的单株,测定各单株的主花序有效长(valid length on racemes,VLR)和主花序有效角果数(valid silique on racemes,VSR),并计算主花序角果密度(silique density on racemes,SDR)。主花序角果密度为主花序有效角果数/主花序有效长,以“个/cm”为单位;主花序有效长为从主花序底端第一个有效角果(有效角果,即角果中有饱满的种子)着生部位到顶端最后一个有效角果着生部位长度,以“厘米(cm)”为单位;主花序有效角果数为主花序上着生的有效角果数,以“个”为单位。表型数据采用Excel 2016软件进行初步整理,取2个重复的平均值,计算每个性状的平均数、标准差和变异系数;采用 DPS7.05软件进行统计分析和性状间的Spearman相关性分析。
1.3 基因型测定与分析
利用油菜60 K Illumina Infinium SNP芯片对213份材料进行基因型分析,该芯片包括覆盖甘蓝型油菜全基因组的52 157个SNP标记。采用 Genome Studio(Illumina公司)软件对213份油菜种质进行SNP基因型的检测,挑选出杂合率(heterozygosity,H)、缺失率(miss rate,M)小于 20%,同时最小等位基因频率(minor allele frequency,MAF)大于10%[26],且均匀分布于染色体上的23 767个高质量SNP标记用于后续全基因组关联分析。
1.4 群体结构与亲缘关系分析
利用 Structure 2.3.4软件对该群体进行群体结构分析[27],设置亚群数目K为1—10,5次模拟运算,模拟参数迭代(length of burn-in period)和蒙特卡罗迭代(markov chain monte carlo,MCMC)都设置为1×105次循环,在混合模型和频率相关模型下运算。输出的后验概率值结果输入STRUCTURE HARVESTER(http://taylor0.biology.ucla.edu/structureHarvester/),计算2个连续的后验概率值的变化速率(Δk)和每个材料分别来自1—10亚群的Q值,以Q值0.8为分界线,最终确定群体的数目和结构[28]。
利用 Tassel 5.1.0软件对自然群体进行亲缘关系(relative kinship)评估,计算亲缘关系K矩阵[29]。亲缘关系代表两特定材料之间的遗传相似度与任意材料之间遗传相似度的相对值,因此当材料之间的亲缘关系值为负值时,直接将其定义为0[20]。
1.5 连锁不平衡分析
利用Tassel 5.1.0分析连锁不平衡(LD)在甘蓝型油菜各染色体上的分布,绘制各染色体的LD衰减图[31],LD类型参数设置为 Full Matrix,以决定系数(coefficient of determination)r2=0.2为衰减阈值,计算显著关联SNP所在染色体的LD衰减距离,用于候选基因预测和功能注释分析。
1.6 最优模型的选择和全基因组关联分析
将基因型数据导入Tassel5.1.0后进行主成分分析(PCA矩阵),再将Q和K矩阵导入Tassel5.1.0,以Q、K和PCA矩阵作协变量,采用基于一般线性模块(general linear model,GLM)的GLM、Q和PCA模型和混合线性模块(mixed linear model,MLM)的K、Q+K和PCA+K共6种模型进行关联分析。以ggplot2[30]绘制Quantile-Quantile散点图(QQ plot)表示不同模型的检测效力,以实际P值与期望P值最接近的模型为每个性状GWAS分析的最佳模型。最后基于最优统计模型,对群体内主花序角果密度及其相关性状进行关联检测。用P值来衡量关联标记的显著性,显著关联SNP阈值设为1/23767=4.21×10-5。利用qqman[31]绘制Manhattan图显示关联分析检测到的与目标性状显著相关的标记位点。
1.7 候选基因分析
在关联分析结果的基础上,用与性状显著关联的SNP所在染色体上r2=0.2时的衰减距离为候选基因的LD区间。根据这个区间在油菜基因组中的位置,以已公布的甘蓝型油菜“Darmor-Bzh”的基因组注释信息(http://www.Genoscope.cns.fr/brassicanapus)分析区间内的基因,并与拟南芥的基因序列进行 BLAST比对,以同源性最高的拟南芥基因注释候选基因功能,筛选可能控制目标性状的候选基因。
2 结果
2.1 表型统计分析
连续2年(2015年和2016年)分别对213份油菜种质的主花序角果密度、主花序有效长和主花序有效角果数的表型进行考察和统计分析(表1、图1),结果表明,3个性状在2年内品种间差异均达到极显著水平,变异系数介于15.38%—26.42%,变幅较大。正态检验(表1)和频数分布图(图1)也表明,目标性状2年Shapiro-Wilk检验的W值均在0.991—0.996,且 P>0.05,符合正态分布;两年 3个性状都呈连续性分布,符合典型的数量性状特点,适合进行GWAS分析。3个性状的广义遗传率分别为68.06%、67.47%和69.05%,也表明其主要受遗传因素影响。

表1 甘蓝型油菜主花序角果密度、主花序有效长和主花序有效角果数的统计分析Table 1 Statistical analysis of SDR, VLR and VSR in B. napus
2.2 油菜主花序角果密度及其相关性状的相关性分析
2015年,主花序角果密度与主花序有效长、主花序有效角果分别存在统计学意义上的极显著负相关和显著正相关,相关系数分别为-0.265和 0.144;2016年,主花序角果密度与主花序有效长具有统计学意义上的有极显著负相关。两年主花序角果密度与主花序有效长呈极显著负相关,意味着主花序增长,但没有相应地增加有效角果数,因此,研究主花序角果密度对增加主花序有效角果数,提高油菜产量有重要意义。
2.3 群体结构与亲缘关系分析
选择8 923个在染色体上均匀分布且最小等位基因频率大于0.3的SNP标记,利用软件Structure2.3.4对每个可能的K值模拟运算,输出结果分析后验概率LnP(D)值得到方差以及变化速率,当K=2时,模型中显示△K有最大变化(图2-A)。213份甘蓝型油菜分为P1和P2 2个亚群,P1亚群包含50份材料(23.5%),绝大多数源自于国内的湖北省、春性油菜和少量的半冬性材料及国外引进的材料;P2亚群包含163份材料(76.5%),主要是中国的半冬性油菜品系(电子附表1)。基于数学模型对群体的类群划分,基本与油菜的地理栽培属性一致。亲缘关系分析显示群体中约89.74%的材料之间的亲缘关系值小于0.2(图2-B),其中约有59.91%材料的亲缘关系值为0,约12.01%材料的亲缘关系值介于0—0.05。说明整个自然群体材料之间的亲缘关系较远,对关联分析结果影响较小。

图1 自然群体主花序角果密度、主花序有效长和主花序有效角果数的频次分布Fig. 1 Frequency distribution of SDR, VLR and VSR from the natural population

表2 主花序角果密度及其相关性状表型间的相关性Table 2 Correlation of phenotype among SDR and its component traits in two years

图2 213份甘蓝型油菜的群体结构和亲缘关系分析Fig. 2 Analysis of population structure and relative kinship in 213 B. napus
2.4 连锁不平衡在A、C基因组中的衰减
对A基因组的10条染色体(A01—A10)和C基因组的9条染色体(C01—C09)分别绘制带平滑线的LD衰减散点图(图3)。由图3可以看出A和C基因组的 r2随着遗传距离的增加而下降,衰减距离也各不同。在r2的阈值为0.2标准下(表3),A基因组的平均衰减距离为450 kb,其中A08染色体的衰减速度最慢,衰减距离最大,约为1 350 kb;A02和A04染色体衰减速度最快,衰减距离约为200 kb。C基因组的平均衰减距离为950 kb,其中C02的衰减距离最大,约为1 400 kb;C07染色体的衰减距离最小,约为500 kb。A基因组的衰减距离整体比C基因组的衰减距离小得多。这可能与中国半冬性甘蓝型油菜A基因组在育种中发生大规模重组,打破连锁不平衡有关。

表3 连锁不平衡在A、C基因组中的衰减距离Table 3 The Linkage disequilibrium attenuation decline in A and C genome
此外,A和C基因组中有些染色体在进入衰减距离后仍存在不同程度和数量的波峰(图 3)。A基因组中的A06染色体在1 800 kb左右有明显的峰,A01、A04、A05、A08和A10染色体在1 400—1 900 kb范围内均出现了明显且数量不同的峰。C基因组C07染色体在1 800和3 300 kb左右出现了明显的峰,除C04、C05和C09染色体外的其他染色体也在不同的遗传距离上出现了不同程度和数量的波峰。表明A和C基因组中同一条染色体上即使是距离较远的2个标记之间,也可能存在一定的连锁不平衡。
2.5 油菜主花序角果密度相关性状的6种模型比较
从图4可以看出,可能由于群体较小以及目标基因座与群体结构高度相关,除了2016年的主花序角果密度外,其他性状的K、K+Q和K+PCA模型都表现出一定的假阴性。在 2015年的主花序角果密度(图4-A)和2016年的主花序有效长(图4-D)检测到Q模型有效地控制了假阳性和假阴性结果的出现;2015年的主花序有效长(图4-C)以PCA为最佳模型;2015和2016年的主花序有效角果数(图4-E和图4-F),MLM模型下的K、K+Q和K+PCA模型虽然表现为假阴性,但它们的P值较Q和GLM模型更接近期望值,因此,选用K+Q作为主花序有效角果数的最优模型;2016年的主花序角果密度(图4-B)MLM模型下的K+Q和K+PCA模型均能较好地控制假阳性,但K+Q模型检测到P值更接近期望值。

图3 连锁不平衡在A、C基因组不同染色体的衰减Fig. 3 The linkage disequilibrium decline in different chromosomes for A and C genome
2.6 全基因组关联分析
GWAS分析2年数据共检测到17个SNP位点与主花序角果密度及其相关性状关联(表4和图5)。与主花序角果密度关联的SNP标记位点有7个,其中2015年环境中,仅在 A03染色体上检测出Bn-A03-p15240794与主花序角果密度关联(图5-A),P值为1.03×10-5,可解释11.76%的表型变异;在2016年环境中,检测到6个与主花序角果密度性状相关的SNP位点(图5-B),分布于染色体A01、A03、A04、A10和C01上,可解释11.34%—15.96%的表型变异。与主花序有效长关联的SNP标记位点共有9个,其中2015年环境中,在C03和C09染色体上各检测出1个显著关联的SNP位点(图5-C),可分别解释10.67%和9.67%的表型变异;在2016年环境中,检测到7个与主花序有效长相关的SNP位点(图5-D),除C05染色体上检测到1个位点,其他6个关联位点都在C09染色体的6.5—6.8 M区域被检测到,平均相距约50 kb,表型贡献率在10%—13.10%;仅在2016年环境中,检测到1个在A01染色体上与主花序有效角果数相关的SNP位点(图5-F )。
2.7 候选基因预测
基于甘蓝型油菜参考基因组序列,对 LD区间的候选基因进行分析,筛选出了22个与主花序角果密度及其相关性状有关的候选基因(表 5)。通过扫描与主花序角果密度显著关联SNP标记的LD区域,共筛选到12个重要候选基因;在与主花序有效长关联SNP标记的LD区域内扫描到8个可能候选基因;在主花序有效角果数相关 LD区间筛选出 2个候选基因。其中,BnaA01g16940D、BnaC01g38800D和 BnaA04g09170D等主要通过调控赤霉素和生长素等内源激素的合成和信号转导来控制主花序角果密度及其相关性状,BnaA01g16970D、BnaA03g29180D、BnaA03g29810D、BnaC01g39680D和BnaC03g32770D通过对分生组织或花分生组织的调控来改变性状表型,BnaC09g18690D和BnaC09g09210D等主要通过控制细胞分裂生长等过程改变性状形态;而BnaA01g22310D在拟南芥中的同源基因 AT1G60420编码 NRX1,具有典型的硫氧还蛋白(TRX)活性位点(WCG/PPC),在植物体内以二聚体的形式被发现,nrx1突变体表现花粉生育力减弱,导致最终角果数目显著减少[32-33]。总之,这些基因主要与赤霉素和生长素等内源激素的合成和信号转导、分生组织或花分生组织以及细胞分裂生长等过程有关,它们可能通过上述功能影响油菜花序或角果的发育,导致主花序角果密度及其相关性状的表型变异。

图4 6种统计模型对主花序角果密度及其相关性状进行关联分析的QQ图Fig. 4 Quantile-quantile plots of estimated -Iog10(p) from association analysis using six models in SDR and its component traits
3 讨论
角果密度是油菜理想株型的重要因素之一,其构成性状(主花序长度和主花序有效角果数)是影响单株产量的重要因素,主要受多基因控制,遗传率高[5-10]。本研究利用60 K SNP芯片对213份油菜的主花序角果密度及其相关性状进行GWAS分析,两年的主花序角果密度及其相关性状的标记并没有发现重复,且标记数目也相差较大,这可能是两年环境差异较大引起的。本研究对主花序角果密度关联分析发现,两年都在A03染色体上检测出显著关联的SNP,且标记之间相距4.4 Mb,说明在A03染色体上有控制主花序角果密度的主效基因;CHEN等[15]也在 A03染色体上检测到与主花序角果密度关联的 QTL。本研究两年在 C09染色体上检测到与主花序有效长显著关联位点成簇分布,与段秀建[60]在 C09上发现的位点相距约4.4 Mb,这表明在C09染色体上有控制主花序有效长的主效QTL。孙美玉[61]在两年三环境中重复检测到A01染色体上与主花序有效角果数相关的QTL,与该性状连锁的3个标记(GSSR134、BrSF162-9和BrSF49-32)距离较近,仅相距1 Mb左右,与本研究
发现的 Bn-Scaffold000582-p3247位点也是相距约 1 Mb,认为在 A01染色体的该区段内存在控制主花序有效角果数的主效QTL。

表4 两年主花序角果密度及其相关性状的显著关联标记Table 4 Significance association markers of SDR and its component traits in two years

表5 主花序角果密度及其相关性状的候选基因Table 5 A summary of candidate genes associated with SDR and its component traits
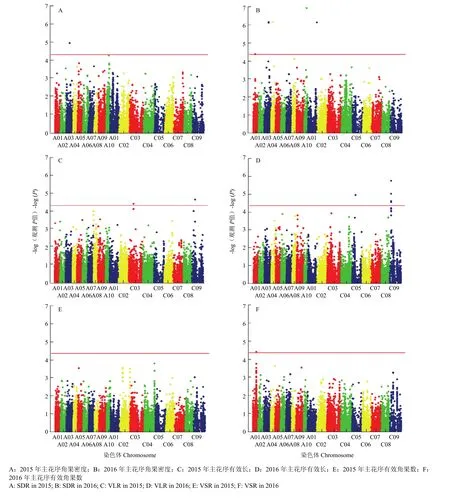
图5 2年主花序角果密度及其相关性状的Manhattan图Fig. 5 Manhattan plot of SDR and its component trait in two year
通过关联分析,扫描与性状显著关联SNP位点的 LD区域,借助拟南芥基因组数据库,筛选到了22个与性状相关的候选基因,其中包含多个位于关联 SNP位点处或在位点附近的基因。这些基因部分与赤霉素和生长素等内源激素的合成和信号转导、分生组织或花分生组织以及细胞分裂生长等过程有关。如BnaA01g16940D与拟南芥AT4G28160同源,在花序中表达,编码的富含脯氨酸糖蛋白家族蛋白是细胞壁的组成成分,参与对赤霉素刺激的反应,诱导茎伸长和花发育,通过调控最终角果数目和花序长影响角果密度[33-34];C01染色体的BnaC01g39480D与拟南芥的AT3G08510(ATPLC2)同源,编码磷酸肌醇特异性磷脂酶C(PI-PLC)催化磷脂酰肌醇4,5-二磷酸水解成肌醇 1,4,5-三磷酸和二酰基甘油,参与生长素的生物合成和信号转导,是雌配子发生和胚胎发育所必需的,杂合子plc2-2突变体有角果数较少和胚胎在球状期前停滞与细胞分裂异常的表型[49-50];BnaA01g16970D与拟南芥ULT1同源,编码 B-box 结构域富含 Cys(半胱氨酸)蛋白,是分生组织细胞积累在花序和花分生组织的负调节器;ult1突变体使花序分生组织扩大,产生额外的花和花器官,并在花分生组织中表达降低[35-36];A03染色体的BnaA03g29810D,在拟南芥中的同源基因AT3G07050编码类核干细胞因子1(nucleostemin- like 1,NSN1),于花序分生组织和花原基中高水平表达,是维持花序分生组织和花器官发育所必需的;NSN1杂合和纯合植株通过形成花冠花而终止花序发育,过表达导致顶端优势丧失和缺陷花的形成[42-43]。虽然,基因与环境互作导致环境特异关联SNP很难通过分子标记辅助选择促进其他地区油菜增产,但通过基因工程手段对部分高产基因进行操作很可能实现其他地区油菜产量的提升,对中国油菜生产和发展具有重要意义。
4 结论
共检测到17个SNP与主花序角果密度及其相关性状关联,其中与主花序角果密度关联的标记有 7个,与主花序有效长关联的标记有9个,与主花序有效角果数相关联的标记仅有1个,这些SNP均为环境特异位点,表明其受基因与环境互作影响。LD区间内调节内源性激素的合成和转导、植物细胞组织发生、花分生组织发育、角果数目和多器官发育基因BnaA01g16940D、BnaA03g29810D、BnaC01g39480D、BnaA01g16970D和 BnaC09g09210D等 22个候选基因,可能通过上述功能影响油菜花序或角果的发育,导致主花序角果密度及其相关性状的差异。
[1]王红, 阮晓奇. 2004年菜籽油菜粕市场行情分析. 河南畜牧兽医,2004, 25(5): 5-6.WANG H, RUAN X Q. Market analysis of rapeseed oil and rape cake in 2004. Henan Journal of Husbandry and Veterinary Medicine, 2004,25(5): 5-6. (in Chinese)
[2]曾晓波, 吴谋成, 王海英. 丙酮浸提法制取菜籽浓缩蛋白. 中国粮油学报, 2001, 16(4): 10-13.ZENG X B, WU M C, WANG H Y. Preparation of concentrated rapeseed protein by extraction with acetone. Chinese Cereals and Oils Association, 2001, 16(4): 10-13. (in Chinese)
[3]SCHILTZ S, MUNIER-JOLAIN N, JEUDY C, BURSTIN J, SALON C. Dynamics of exogenous nitrogen partitioning and nitrogen remobilization from vegetative organs in pea revealed by15N in vivo labeling throughout seed filling. Plant Physiology, 2005, 137(4):1463.
[4]YANG P, SHU C, CHEN L, XU J, WU J, LIU K. Identification of a major QTL for silique length and seed weight in oilseed rape(Brassica napus L.). Theoretical and Applied Genetics, 2012, 125(2):285-296.
[5]浦惠明, 龙卫华, 高建芹. 油菜全程机械化生产配套农艺技术研究:Ⅰ.不同播期和密度对直播油菜产量和经济性状的影响. 江苏农业科学, 2009(3): 48-51.PU H M, LONG W H, GAO J Q. The research of complete mechanized production matching agronomic techniques in rapeseed:Ⅰ.Different planting date and density on yield and economic characters of rape. Jiangsu Agricultural Sciences, 2009(3): 48-51. (in Chinese)
[6]曾川, 刘成家, 徐洪志, 黄涌, 伊淑丽. 油菜株型育种研究进展].中国农学通报, 2014(12): 14-18.ZNEG C, LIU C J, XU H Z, HUANG Y, YI S L. The research progress about plant type breeding of rape. Chinese Agricultural Science Bulletin, 2014(12): 14-18. (in Chinese)
[7]安彩泰, 孙万泉, 李学才. 甘蓝型油菜数量性状的遗传研究: Ⅲ.六个农艺性状的遗传分析. 甘肃农大学报, 1988(4): 58-65.AN C T, SUN W Q, LI X C. Genetic studies on quantitative characters in Brassica napus L.: Ⅲ.Genetic analysis of six agronomic characters.Journal of Gansu Agricultural University, 1988(4): 58-65. (in Chinese)
[8]李栒, 官春云. 油菜主要性状遗传力和遗传相关的初步研究. 遗传,1981, 3(3): 24-27.LI X, GUAN C Y. The preliminary study on heritability and genetic correlation of major characters in Brassica napus. Hereditas(Beijing),1981, 3(3): 24-27. (in Chinese)
[9]江满霞, 高冰可, 卢列健, 张小元, 阳太羊. 13个油菜品种的农艺性状遗传力和相关性分析. 浙江农业科学, 2015, 56(4): 472-474.JIANG M X, GAO B K, LU L J, ZHANG X Y, YANG T Y.Heritability and correlation analysis of agronomic characters in 13 Rape. Journal of Zhejiang Agricultural Sciences, 2015, 56(4):472-474. (in Chinese)
[10]余凤群, 金明源, 肖才升, 王蕾. 甘蓝型油菜 DH 群体几个数量性状的遗传分析. 中国农业科学, 1998, 31(3): 44-48.YU F Q, JIN M Y, XIAO C S, WANG L. Genetics analysis of several quantitative traits of doubled haploid population in Brassica napus L..Scientia Agricultura Sinica, 1998, 31(3): 44-48. (in Chinese)
[11]张书芬, 宋文光, 任乐见, 田保明, 文雁成, 刘建民, 王建平. 甘蓝型双低油菜数量性状的遗传力及基因效应. 中国油料作物学报,1996(3): 1-3.ZHANG S F, SONG W G, REN L J, TIAN B M, WEN Y C, LIU J M,WANG J P. Heritability and gene effects of quantitative characters in"Double-Low" Rapeseed(Brassica napus L.).Oil Crops of China,1996(3): 1-3. (in Chinese)
[12]王瑞, 李加纳, 唐章林, 谌利, 张学昆. 甘蓝型黄籽油菜产量性状的遗传分析. 中国农学通报, 2004, 20(5): 37-38.WANG R, LI J N, TANG Z L, SHEN L, ZHANG X K. Genetic analysis for yield characters in yellow-seeded rapessed lines(Brassica napus L.). Chinese Agricultural Science Bulletin, 2004, 20(5): 37-38.(in Chinese)
[13]王俊生, 田建华, 张继澍, 李殿荣, 袁志发. 紧凑型油菜株型性状的遗传及其与主要产量性状的相关性研究. 西北农林科技大学学报(自然科学版), 2005, 33(6): 7-12.WANG J S, TIAN J H, ZHANG J P, LI D R, YUAN Z F. Study on the heredity of plant-type traits in compact rapeseed lines and the genetic correlation with yield traits. Journal of Northwest A & F University(Natural Science Edition), 2005, 33(6): 7-12. (in Chinese)
[14]王会. 油菜重要性状的ADAA遗传模型分析及SNP标记预测杂种优势研究[D]. 北京: 中国农业科学院, 2014.WANG H. Genetic analysis of important traits using ADAA model and prediction of heterosis using SNP markers in Brassica napus L..[D]. Beijing: Chinese Academy of Agricultural Sciences, 2014. (in Chinese)
[15]CHEN W, ZHANG Y, LIU X P, CHEN B Y, TU J X, FU T D .Detection of QTL for six yield-related traits in oilseed rape ( Brassica napus ) using DH and immortalized F2, populations. Theoretical and Applied Genetics, 2007, 115(6): 849-858.
[16]高必军. 甘蓝型油菜napin基因启动子的克隆与几个重要农艺性状的初步QTL定位[D]. 雅安: 四川农业大学, 2007.GAO B J. Cloning of the napin gene promoter and preliminary QTL of some agronomical ImPort traits in Brassica napus [D].Ya’an:Sichuan Agricultural University, 2007. (in Chinese)
[17]阴涛. 甘蓝型油菜株高和角果长及其相关性状 QTL定位[D]. 重庆: 西南大学, 2015.YIN T. QTL mapping of major plant-type traits related to plant height and silique length in Brassica napus L.[D]. Chongqing: Southwest University, 2015. (in Chinese)
[18]FLINT-GARCIA S A, THORNSBERRY J M. Structure of linkage disequilibrium in plants. Annual Review of Plant Biology, 2003, 54(4):357-374.
[19]FLINT-GARCIA S A, THUILET A C, YU J, GAEL P, SUSAN M,ROMERO, SHARON E, MITCHELL, JOHN D, STEPHEN K,MAJOR M. GOODMAN, BUCKLER E S. Maize association population: A high-resolution platform for quantitative trait locus dissection. The Plant Journal, 2005, 44(6): 1054-1064.
[20]YU J, BUCKLER E S. Genetic association mapping and genome organization of maize. Current Opinion in Biotechnology, 2006, 17(2):155-160.
[21]LI F, CHEN B, XU K, GAO G, YAN G, QIAO J, LI J, LI H, LI L,XIAO X, ZHANG T Y, NISHIO T S, WU X M. A genome-wide association study of plant height and primary branch number in rapeseed (Brassica napus). Plant Science, 2015, 242: 169-177.
[22]段继凤. 甘蓝型油菜含油量性状全基因组关联分析[D]. 武汉: 华中农业大学, 2013.DUAN J F. Genome-wide association mapping of oil content trait in rapeseed (Brassica napus L.) [D]. Wuhan: Huazhong Agricultural University, 2013. (in Chinese)
[23]HUMBERTO A, GAJARDO, BENJAMIN W, BRAULIO S C, ERIN E, HIGGINS, ISOBEL A P, PARKIN, ROD J. SNOWDON, MARIA L, FEDERICO L, INIGUEZ L. Association mapping of seed quality traits in Brassica napus L. using GWAS and candidate QTL approaches. Molecular Breeding, 2015, 35: 143.
[24]LIU S, FAN C, Li J, CAI G J, YANG Q Y, WU J, YI X Q, ZHANG C Y, ZHOU Y M. A genome-wide association study rKINeveals novel elite allelic variations in seed oil content of Brassica napus.Theoretical and Applied Genetics, 2016, 129(6): 1203-1215.
[25]卢坤, 王腾岳, 徐新福, 李加纳, 唐章林, 曲存民, 贺斌, 梁颖. 甘蓝型油菜结角高度与荚层厚度的全基因组关联分析. 作物学报,2016(3): 344-352.LU K, WANG T Y, XU X F, LI J N, TANG Z L, QU C M, HE B,LIANG Y. Genome-wide association analysis of height of podding and thickness of pod canopy in Brassica napus. Acta Agronomica Sinica, 2016(3): 344-352. (in Chinese)
[26]LIU S, FAN C, LI J, CAI G, YANG Q, WU J, YI X, ZHANG C,ZHOU Y. A genome-wide association study reveals novel elite allelic variations in seed oil content of Brassica napus. Theoretical and Applied Genetics, 2016, 129(6): 1203-1215.
[27]PRITCHARD J K, STEPHENS M, ROSENBERG N A, DONNELLY P. Association mapping in structured populations. American Journal of Human Genetics, 2000, 67(1): 170-181.
[28]EVANNO G, REGNAUT S, GOUDET J. Detecting the number of clusters of individuals using the software structure: A simulation study.Molecular Ecology, 2005, 14: 2611-2620.
[29]BRADBURY P J, ZHANG Z, KROON D E, CASSTEVENT T M,RAMDOSS Y, BUCKLER E S. TASSEL: Software for association mapping of complex traits in diverse samples. Bioinformatics, 2007,23(19): 2633-2635.
[30]GINESTET C. ggplot2: Elegant graphics for data analysis. Journal of the Royal Statistical Society: Series A (Statistics in Society), 2011,174(1): 245.
[31]TURNER S D. qqman: An R package for visualizing GWAS results using Q-Q and manhattan plots. The preprint server for Biology, 2014:005165.
[32]MARCHAL C, VALERIE D H , BARIAT L, SIALA W, BELIN C,SAEZ-VASQUEZ JULIO S V, RIONDET C, REICHHELD J P.NTR/NRX define a new thioredoxin system in the nucleus of Arabidopsis thaliana cells. Molecular Plant, 2014, 7(1): 30-44.
[33]PHILLIPS A L, HUTTLY A K. Cloning of two gibberellin-regulated cDNAs from Arabidopsis thaliana by subtractive hybridization:Expression of the tonoplast water channel, gamma-TIP, is increased by GA3. Plant Molecular Biology, 1994, 24(4): 603-615.
[34]SHI X J, SERGEY M, SHERON D O, CAROLINE B, ROCHUS B F,LUKAS S, ASAPH A. SHINE transcription factors act redundantly to pattern the archetypal surface of Arabidopsis Flower Organs. PLoS Genetics, 2011, 7(5): e1001388.
[35]CARLES C C, CHOFFNES-INADA D, REVILLE K, KVIN L,JENNIFER C, FLETCHERl. ULTRAPETALA1 encodes a SAND domain putative transcriptional regulator that controls shoot and floral meristem activity in Arabidopsis. Development, 1991, 132(5): 897-911.
[36]FLETCHER J C. The ULTRAPETALA gene controls shoot and floral meristem size in Arabidopsis. Development (Cambridge, England),2001, 128(8): 1323-1333.
[37]JAYAWEERA T, SIRWARDANA C, DHARMASIRI S, QUINT M,QUINT M, GRAY W M, DHARMASIRI N. Alternative splicing of Arabidopsis IBR5 pre-mRNA generates two IBR5 isoforms with distinct and overlapping functions. PLoS ONE, 2014, 9(8): e102301.
[38]NADELLA V, SHIPP M J, MUDAY G K, WYATT S E. Evidence for altered polar and lateral auxin transport in the gravity persistent signal(gps) mutants of Arabidopsis. Plant Cell & Environment, 2006, 29(4):682-690.
[39]WITHERS J C, SHIPP M J, RUPASINGHE S G, POORNIMA SUKUMAR, SCHULER M A, MUDAY G K, WYATT S E. Gravity persistent signal 1 (GPS1) reveals novel cytochrome P450s involved in gravitropism. American Journal of Botany, 2013, 100(1): 183-193.
[40]DOMELAS M C, VAN LAMMEREN A A, KREIS M. Arabidopsis thaliana SHAGGY-related protein kinases (AtSK11 and 12) function in perianth and gynoecium development. The Plant Journal, 2000,21(5): 419-429.
[41]YOUN J H, KIM T W, KIM E J, BU S, KIM S K, WANG Z Y, KIM T W. Structural and functional characterization of Arabidopsis GSK3-like kinase AtSK12. Molecules and Cells, 2013, 36(6): 564-570.
[42]WANG X M, GINGRICH D K, DENG Y F, HONG Z L. A nucleostemin-like GTPase required for normal apical and floral meristem development in Arabidopsis. Molecular Biology of the Cell,2012, 23(8): 1446-1456.
[43]WANG X, XIE B, ZHU M, ZHANG Z M, HONG Z L. Nucleosteminlike 1 is required for embryogenesis and leaf development in Arabidopsis. Plant Molecular Biology, 2012, 78(1): 31-44.
[44]HUTCHISON C E, LI J, ARGUESO C, Monica GONZALEZ M,LEWIS M W, MAXWELL B B, PERDUE T D, SCHALLER G E,Jose M. ALONSO J M, ECKER J R, KIEBERA J J. The Arabidopsis histidine phosphotransfer proteins are redundant positive regulators of cytokinin signaling. The Plant Cell, 2006, 18(11): 3073-3087.
[45]STOWEEVANS E L, HARPER R M, MOTCHOULSKI A V,LISCUM E. NPH4, a conditional modulator of auxin-dependent differential growth responses in Arabidopsis. Plant Physiology, 1998,118(4): 1265-1275.
[46]HARPER R M, STOWEEVANS E L, LUESSE D R, MUTO H,TATEMATSU K, WATAHIKI M K, YAMAMOTO K, LISCUM E.The NPH4 locus encodes the auxin response factor ARF7, a conditional regulator of differential growth in aerial Arabidopsis tissue.The Plant Cell, 2000, 12(5): 757-770.
[47]LECHNER E, XIE D S, GRAVA S, PIGAGLIO E, PLANCHAIS S,MURRAY J A H, PARMENTIER Y, MUTTRER J, DUBREUCQ B,SHEN W H, GENSCHIK P. The AtRbx1 protein is part of plant SCF complexes, and its down-regulation causes severe growth and developmental defects. Journal of Biological Chemistry, 2002, 277(51):50069-50080.
[48]QU J, KANG S G, HAH C, JANG J C. Molecular and cellular characterization of GA-Stimulated Transcripts GASA4 and GASA6 in Arabidopsis thaliana. Plant Science, 2016, 246: 1-10.
[49]LI L, HE Y, WANG Y, ZHAO H J, CHEN X, YE T T, WU Y X, WU Y.Arabidopsis PLC2 is involved in auxin-modulated reproductive development. The Plant Journal, 2015, 84(3): 504-515.
[50]FINO L M D, D’AMBROSIO J M, TEJOS R, WIIK R V,LAMATTINA L, MUNNIK T, PAGNUSSAT G C, LAXALT A M .Arabidopsis phosphatidylinositol-phospholipase C2 (PLC2) is required for female gametogenesis and embryo development. Planta,2016: 1-12.
[51]SAIGA S, FURUMIZU C, YOKOYAMA R, KURATA T, SATO S,KATO T, TABATA S, SUZUKI M, KOMEDA Y. The Arabidopsis OBEROLN1 and OBERON2 genes encode plant homeodomain finger proteins and are required for apical meristem maintenance. Development,2008, 135(10): 1751-1759.
[52]MIZUNO S, OSAKABE Y, MARUYAMA K, ITO T, OSAKABE K,SATO T, SHINOZAKI K, YAMAGUCHI-SHINOZAKI K. Receptorlike protein kinase 2 (RPK 2) is a novel factor controlling anther development in Arabidopsis thaliana. The Plant Journal, 2007, 50(5):751-766.
[53]RIBONE P A, CAPELLA M, CHAN R L. Functional characterization of the homeodomain leucine zipper I transcription factor AtHB13 reveals a crucial role in Arabidopsis development. Journal of Experimental Botany, 2015, 66(19): 5929-5943.
[54]SHI Y F, WANG D L, WANG C, CULLER A H, KREISER M A,SURESH J, COHEN J D, PAN J, BAKER B, LIU J Z. Loss of GSNOR1 function leads to compromised auxin signaling and polar auxin transport. Molecular Plant, 2015, 8(9): 1350-1365.
[55]GALLA G, ZENONI S, MARCONI G, MARCONI G, BOTTON A,PINOSA F, CITTRRIO S, RUPETRI B, PALME K, ALBERTINI E,PEZZZOTTI M, MAU M, SHARBEL T F, STORME N D, GEELEN D, BARCACCIA G. Sporophytic and gametophytic functions of the cell cycle-associated Mob1 gene in Arabidopsis thaliana L.. Gene,2011, 484(1/2): 1-12.
[56]CUI X, GUO Z, SONG L, WANG Y, CHENG Y. NCP1/AtMOB1A plays key roles in auxin-mediated Arabidopsis development. PLoS Genetics, 2016, 12(3): e1005923.
[57]KATO H, MOTOMURA T, KOMEDA Y, SAITO T, KATO A.Overexpression of the NAC transcription factor family gene ANAC036 results in a dwarf phenotype in Arabidopsis thaliana.Journal of Plant Physiology, 2010, 167(7): 571-577.
[58]MVSSAR K J, KANDASSAMY M K, MCKINNEY E C,MEAGHRR R B. Arabidopsis plants deficient in constitutive class profilins reveal independent and quantitative genetic effects. BMC Plant Biology, 2015, 15(1): 177-193.
[59]MIKKELSEN M D, NAUR P, HALKIER B A. Arabidopsis mutants in the C-S lyase of glucosinolate biosynthesis establish a critical role for indole-3-acetaldoxime in auxin homeostasis. The Plant Journal,2004, 37(5): 770-777.
[60]段秀建. 油菜株型相关性状的全基因组关联分析[D]. 重庆: 西南大学, 2015.DUAN X J. Genome-wide association analysis of plant-type related traits in rapeseed (Brassica napus L.) [D]. Chongqing: southwest university, 2015. (in Chinese)
[61]孙美玉, 华玮, 刘静, 王新发, 刘贵华, 王汉中. 甘蓝型油菜主花序有效角果数QTL定位. 中国油料作物学报, 2013, 35(1): 1-7.SUN M Y, HUA W, LIU J, WANG X F, LIU G H, WANG H Z. QTLs for effective silique number of main inflorescence on rapeseed( Brassica napus L.). Chinese Journal of Oil Crop Sciences, 2013,35(1): 1-7. (in Chinese)
