湘鄂地区三叶木通野生资源的RAPD亲缘关系分析
2018-03-01李红英王晓辉覃大吉向极钎1,杨永康1,陈菲菲1,万海英1,周敏2,朱麟1
李红英 王晓辉 覃大吉 向极钎1,杨永康1,陈菲菲1,万海英1,周 敏2,朱 麟1

摘要:采用RAPD分子标记技术对湘鄂地区6个不同区域的野生三叶木通[Akebia trifoliata(Thunb.) Koidz.]进行了遗传多样性研究。从50个随机引物中筛选出10个条带清晰、多态性明显并且重复性好的引物,共扩增出86条DNA片段,其中73条为多态性条带,占总扩增带数的84.9%。利用NTSYS软件进行聚类分析,以相似系数0.578为标准,可将所有品种分为3类,第Ⅰ大类包括恩施红庙(紫色型)和湖南古丈(麻色型);第Ⅱ类包括建始花坪、巴东茶店子和宣恩椒园;恩施太阳河的独立分为第Ⅲ类。
关键词:三叶木通[Akebia trifoliata(Thunb.) Koidz.];野生资源;RAPD;亲缘关系
中图分类号:Q37 文献标识码:A
文章编号:0439-8114(2018)23-0148-05
DOI:10.14088/j.cnki.issn0439-8114.2018.23.035 开放科学(资源服务)标识码(OSID):
三叶木通[Akebia trifoliata(Thunb.) Koidz.]隶属于木通科木通属,俗称预知子、八月瓜,落叶藤木,长可达10 m。多生长于海拔350~1 600 m的山地、山坡、山谷、溪边、密林或疏林灌丛较阴湿地。分布于长江流域各省以及甘肃、河北等省。小枝褐色或暗褐色,疏生淡黄褐色皮孔;茎枝无毛。三叶复叶,小叶卵形,边缘具波状圆齿或浅裂,侧脉一般5~6对,顶叶较小,基部偏斜。总状花序,花单性,暗紫红色。果为肉质果,椭圆形,稍弯曲,种子多数,扁平卵圆形,黑色。花期5月,果期8—9月。本种果实含有糖类,种子含脂肪油43%。根、藤茎及果实、种子均可入药。种子可榨油;果实熟后可食用。
RAPD是美国的Williams与Welsh 2个研究小组于1990年同时提出的1种DNA分子标记技术[1,2]。RAPD技术建立在PCR技术基础上,用一系列(通常数百个)不同的随机排列碱基序列的寡核苷酸单链(一般为8~10 bp)作为引物,对所研究的基因组DNA进行单引物扩增,然后用凝胶电泳分离扩增片段,经染色来显示扩增DNA片段的多态性,扩增片段多态性反映了基因组相应区域的DNA多态性[3]。相对于传统的酶学、RFLP等方法,RAPD标记具有操作快速、简便、多态性丰富、适应性广等诸多优点,现已广泛应用于遗传图谱构建、基因定位,特别是遗传多样性的检测[4]和品种分类、亲缘关系鉴定和病毒检测、优良性状及抗体筛选等方面[5]。由于RAPD标记技术应用随机引物,当对大量试验材料进行研究时,往往首先要对引物进行筛选[6]。为此,试验以湘鄂6个不同区域的三叶木通为材料,通过RAPD标记技术对50个10碱基的随机引物进行筛选,以期为应用RAPD标记技术进行三叶木通的遗传多样性分析、品种分子鉴别、构建遗传指纹图谱及分子标记辅助育种等研究奠定基础。
1 材料与方法
1.1 试验材料
1.1.1 供试样品 供试样品为2017年收集的湘鄂6个不同地区三叶木通类群的鲜叶,具体信息见表1。
1.1.2 主要仪器 高压蒸汽灭菌锅(上海东亚容器01J2003-04)、超低温冰箱(SANYO MDF-392)、冰箱(Hair 208K/ANCINA)、制冰机(SCOTSMAN DA24845-3)、PCR擴增仪(德国Eppendorf 5331);高速低温离心机(德国Eppendorf 5417R)、电泳仪(北京六一DYY-4C)、涡旋振荡器(美国Scientific Industries Vortex-Genie 2)、电子天平(赛多利斯Secura?誖)、凝胶成像系仪器(美国伯乐 Gel Doc 200)、超微量紫外可见分光光度计(北京普析TU-1810)。
1.1.3 引物及试剂 试验所设计的RAPD引物由上海生工生物工程技术服务有限公司合成,DNA提取试剂盒、PCR扩增所用试剂购于宝日医生物技术有限公司。
1.2 试验方法
1.2.1 基因组DNA的提取 三叶木通DNA提取使用TaKaRa MiniBEST Plant Genomic DNA Extration Kit。取叶片100 mg,液氮研磨后加入500 μL的Buffer HS I和10 μL的50×DTT Buffer混匀,然后加入10 μL的RNase A(10 mg/mL),充分振荡混匀,于56 ℃水浴温育10 min;加入62.5 μL的Buffer KAC(Buffer HS I或Buffer HS II的1/8体积),充分混匀,冰上放置5 min;12 000 r/min离心5 min,取上清,加入与上清液等体积的Buffer GB,充分混匀,将Spin Column安置于Collection Tube,溶液移至 Spin Column中(由于溶液较多,一般需要分两次过柱,每次过柱的体积量不要超过700 μL),12 000 r/min离心1 min,弃滤液;将500 μL的Buffer WA加入至Spin Column中,12 000 r/min离心1 min,弃滤液;将700 μL的Buffer WB加入至Spin Column中,12 000 r/min离心1 min,弃滤液(沿Spin Column管壁四周加入Buffer WB,这样有助于完全冲洗沾附于管壁上的盐分);重复上一个步骤;将Spin Column安置于Collection Tube上,12 000 r/min离心2 min;将Spin Column安置于新的1.5 mL的离心管上,在Spin Column膜的中央处加入30~50 μL的Elution Buffer或灭菌水,室温静置5 min(将 Elution Buffer或灭菌水加热至65 ℃时使用有利于提高洗脱效率);12 000 r/min离心2 min洗脱DNA。用超微量紫外可见分光光度计和1.3%琼脂糖凝胶电泳检测DNA样品的纯度与浓度。
1.2.2 RAPD PCR扩增 反应体系总体积25 μL,含有1 μL基因组DNA(30 ng),1 μL引物(5 μmol/L),2.5 μL 10×PCR buffer反应缓冲液,2 μL MgCl2(25 mmol/L),2 μL dNTP(2.5 mmol/L),0.2 μL Taq酶,用ddH2O补充体积至25 μL。反应程序:94 ℃预变性5 min;94 ℃变性1 min,35.5 ℃退火1 mins,72 ℃延伸1 min,38个循环;72 ℃延伸10 min,4 ℃保存。扩增产物使用1.3%的琼脂糖凝胶电泳分离,电压110 V,电泳35 min,在凝胶成像仪上观察照相、记录。
1.3 数据分析
根据各分子在相同电泳迁移率(相同分子量片段)的有无,统计得到所有条带的二元数据,有DNA扩增条带记为“1”,无带记为“0”,利用Ntsys计算出遗传相似系数、构建聚类图。
2 结果与分析
2.1 三叶木通基因组DNA的提取结果
由图1可知,提取的DNA经1.3%琼脂糖凝胶电泳检测,条带清晰,表明DNA质量满足后续试验的要求。
2.2 引物扩增结果
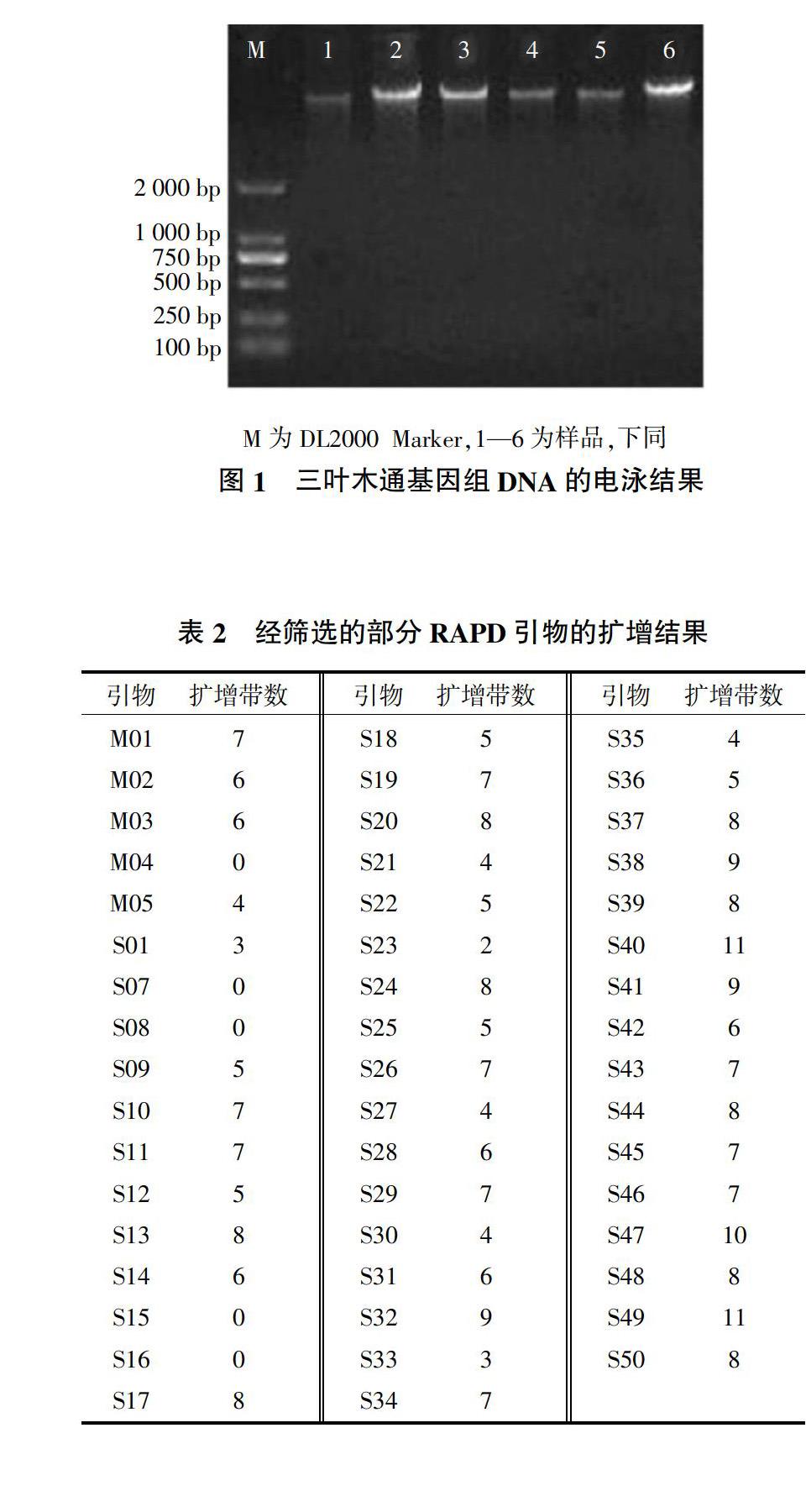
进行引物的筛选也是进行RAPD-PCR扩增的关键,本试验选取了上海生工生物工程技术服务有限公司合成的50种随机引物进行筛选,在合适的PCR扩增条件下,一些引物如M04、S07、S08、S15、S16无扩增条带,而S11、S13、S29、S32、S40、S43、S44、S47、S49、S50扩增谱带清晰且多态性好扩增范围广泛,都在7条带及以上,所以选取这10种引物用于亲缘关系分析(表2)。
2.3 亲缘关系分析
在建立合适的RAPD反应体系的基础上,从50个10 bp随机引物中选用最好的引物S11、S13、S29、S32、S40、S43、S44、S47、S49、S50用于正式扩增,对6个供试的三叶木通叶片基因组DNA扩增出10张指纹图谱,共获得86条DNA谱带,其中73条多态性带,占总扩增带数的84.9%,每个引物扩增的带数在7~11条,平均为8.6条,扩增出的DNA片段大小在100~1 000 bp,表3为所选的10种引物扩增结果。
图2至图11为引物S11、S13、S29、S32、S40、S43、S44、S47、S49、S50的RAPD扩增图谱。10个指纹图谱综合分析结果表明,湖南古丈与恩施太阳河、宣恩椒园的共同点位很少,扩增的条带少,它们的亲缘关系较远,而恩施红庙与湖南古丈、建始花坪与巴东茶店子的扩增带的相似性很大且谱带清晰,所扩增的片段都在100~1 000 bp,它们的亲缘关系较近。
2.4 遗传相似性分析
利用RAPD标记扩增DNA片段,通过Ntsys计算出遗传相似系数(GS)。RAPD标记揭示的6个地区三叶木通资源间的遗传相似系数为0.434 1~0.682 9(表4),平均值為0.558 5。其中建始花坪(JS)与巴东茶店子(BD)GS值最大,为0.682 9,湖南古丈(HN)与恩施太阳河(TYH)GS最小,为0.434 1。
2.5 聚类结果分析
由图12可知,以相似系数0.578为标准,可将6个不同区域的三叶木通分为3大类,第Ⅰ大类包括恩施红庙自选品种紫色型(ES)和湖南古丈麻色型(HN),它们之间的基因组差异不是很明显,这很可能是由于自选品种恩施红庙紫色型多年来对湖南古丈三叶木通进行的品种选育和野转家栽培造成的;第Ⅱ类包括宣恩椒园(XE)、巴东茶店子(BD)和建始花坪(JS);恩施太阳河(TYH)独立分为一类。
3 小结
RAPD标记技术是出现最早的分子标记技术之一,其操作简便、成本较低及不需要DNA的序列材料等特点是其他标记技术无法比拟的[7]。但RAPD标记也存在某些不足,其中最主要的是其灵敏度高,受反应条件影响较大,检测的重复性较差,因此,研究者在利用RAPD标记技术进行遗传多样性分析时一定要严格控制试验条件,采用稳定优化的反应体系方能取得理想的试验结果[8]。同时,在运用RAPD标记技术时,由于每个随机引物并不适合于所有物种,因此对引物的筛选是非常必要的,筛选出多态性丰富、稳定性好的随机引物是整个试验成功的关键[9]。
田宗城等[10]以七叶木桶、三叶木通、白木通、五叶木通等4个种的12个材料为研究对象,用RAPD技术对其进行亲缘关系分析,从40个随机引物(10mer)中筛选出5个(S30、S168、S11、S12、S96),对所有供试材料进行扩增,共获得5张指纹图谱,共在52个位点上扩增出条带,平均每个引物扩增10.4个,多态位点43个,占总带数的82.7%。而本试验以湘鄂地区6个不同区域的三叶木通为供试材料,更为系统地分析了三叶木通遗传多样性,从50个随机的RAPD引物中成功筛选出10条多态性丰富、条带清晰且可重复性好的有效引物,10条引物共扩增出83条DNA条带,其中84.9%为多态性条带,这为应用RAPD标记技术快速、准确地进行三叶木通遗传多样性分析和品种分子鉴别等研究奠定了良好基础。
三叶木通分布于中国河北、山西、山东、河南、陕西南部、甘肃东南部至长江流域各省区。可见它分布遍布全国各地,样品采集应该来源于全国各地,而试验不足之处在于样品采集的局限性,只收集了鄂湘6个不同区域,对三叶木通遗传多样性的研究不是很全面,但是却为三叶木通种子资源的筛选与开发利用奠定了一定的基础。
参考文献:
[1] WILLIAMS J G K,KUBELIK A R,LIVAK K J,et al. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J].Nucleic Acids Research,1990,18(22):6531-6535.
[2] WELSH J,HONEYCUTT R J,MCCLELLAND M,et al. Parentage determination in maize hybrids using the arbitrarily primed polymerase chain reaction(AP-PCR)[J].Theor Appl Genet,1991, 82:473-476.
[3] 陈名红,熊华斌,李成云.分子标记在百合属植物遗传多样性研究中的应用[J].生物技术通报,2011(12):65-69.
[4] 赵 锦,刘 孟,吕增仁,等.RAPD技术在植物遗传多样性研究中的应用[J].河北农业大学学报,2000,23(1):25-28.
[5] 邹喻萍.系统与进化植物学中的分子标记[M].北京:科学出版社,2001.
[6] CHARLES J S,FRED J M. A quick and versatile method for plant DNA extraction from small amounts of tissue[J].Nucleic Acids Res,1990,18:6531-6535.
[7] 周 娟,罗 育,吴耀生,等.8份广西产绞股蓝种质的RAPD引物筛选及其遗传多样性分析[J].基因组学与应用生物学,2010, 29(2):273-278.
[8] 张美勇,上官凌飞,徐 颖,等.核桃RAPD引物筛选及品种间亲缘关系研究[J].中国农学通报,2010,26(2):84-90.
[9] 何庆元,玉永雄,王松华,等.根瘤菌RAPD引物筛选及条件优化[J].核农学报,2007,21(2):132-135.
[10] 田宗城,李 峰,王文龙,等.木通属植物的RAPD研究[J].湖南文理学院学报(自然科学版),2005,17(2):40-42.
