建立我国规模化无菌动物高效研究应用体系
2017-12-22曾本华魏泓
曾本华,魏泓
(第三军医大学基础部实验动物学教研室,重庆 400038)
研究进展
建立我国规模化无菌动物高效研究应用体系
曾本华,魏泓*
(第三军医大学基础部实验动物学教研室,重庆 400038)
无菌动物与宏基因组技术是驱动人体健康微生物组研究的两大动力。没有无菌动物菌群(株)移植模型,就无法确立菌群与疾病的因果关系;没有无菌动物,就没有菌群与人体疾病研究飞速发展的今天与明天。无菌动物应用已形成有菌与无菌动物比较、菌群(株)移植、基因工程动物无菌化、无菌动物发育四种通用研究模式,研究模式的标准化将加大无菌动物研究应用规模及速度。本课题组经过十几年努力,已建成国内体量大、技术体系稳定、服务单位多、具有较大影响力的无菌动物平台,为国内生物医学、畜牧、食品微生物组科学问题解决提供了有力支撑。但是,目前我国无菌动物规模小、效率低、供用平台与应用条件分离,无菌动物基础理论与技术体系尚待发展,难以满足日益增长的应用需求。亟待建立规模化无菌动物高效研究应用体系,以适应我国微生物组研究高速发展。
无菌动物;微生物组;动物模型
人体被认为是由宿主细胞和共生细菌组成的超级生物体(superorganism)。菌群数量约为宿主细胞数量的10倍,其基因数量约为宿主基因的100倍[1]。宿主基因组和菌群宏基因组共同决定人体健康状态和疾病表型,人的生老病死无不受肠道菌群的影响[2]。正常菌群具有定植抗力、屏障、代谢、免疫等生理功能,而菌群紊乱则可导致肥胖、糖尿病、自闭症、抑郁症等多种疾病[3-5]。菌群与疾病关系研究领域被评为2011、2013年全球十大科学进展。
解析人体这一超级生物体的功能,无菌动物(germ-free animal,GF animal)是必不可少的工具。无菌动物体内外没有任何可检测到的活的细菌、病毒和寄生虫动物,没有任何微生物干扰研究,已成为单菌、多菌、菌群与宿主关系研究的不可替代研究工具[6]。本课题组已建立国内体量较大、技术体系稳定、服务单位广、较具影响力的无菌动物平台。但目前无菌动物相关模型制备效率低、基本理论及制备技术基础研究不足,已成为制约我国菌群与疾病关系研究的瓶颈问题。只有建立我国规模化无菌动物高效研究应用体系,才能满足我国生命科学、畜牧兽医学、食品科学等对无菌动物及其模型研究需求。
1 无菌动物在人体微生物组研究中不可或缺
要彻底解析人体这一超级生物体的功能,需无菌动物技术与遗传工程技术的集成、整合,从而从宿主(基因组)及共生菌群(宏基因组)两个层面完整、深入研究整个有机体,二者缺一不可。没有基于无菌动物技术的菌群敲除及其菌群移植,就无法研究菌群及宏基因组的功能;同样,没有基因敲除、转基因技术,则无法研究基因功能。鉴于无菌动物的特殊价值,利用无菌动物发表SCI论文近年来在国内外快速增加(以无菌小鼠为例,见图1)。
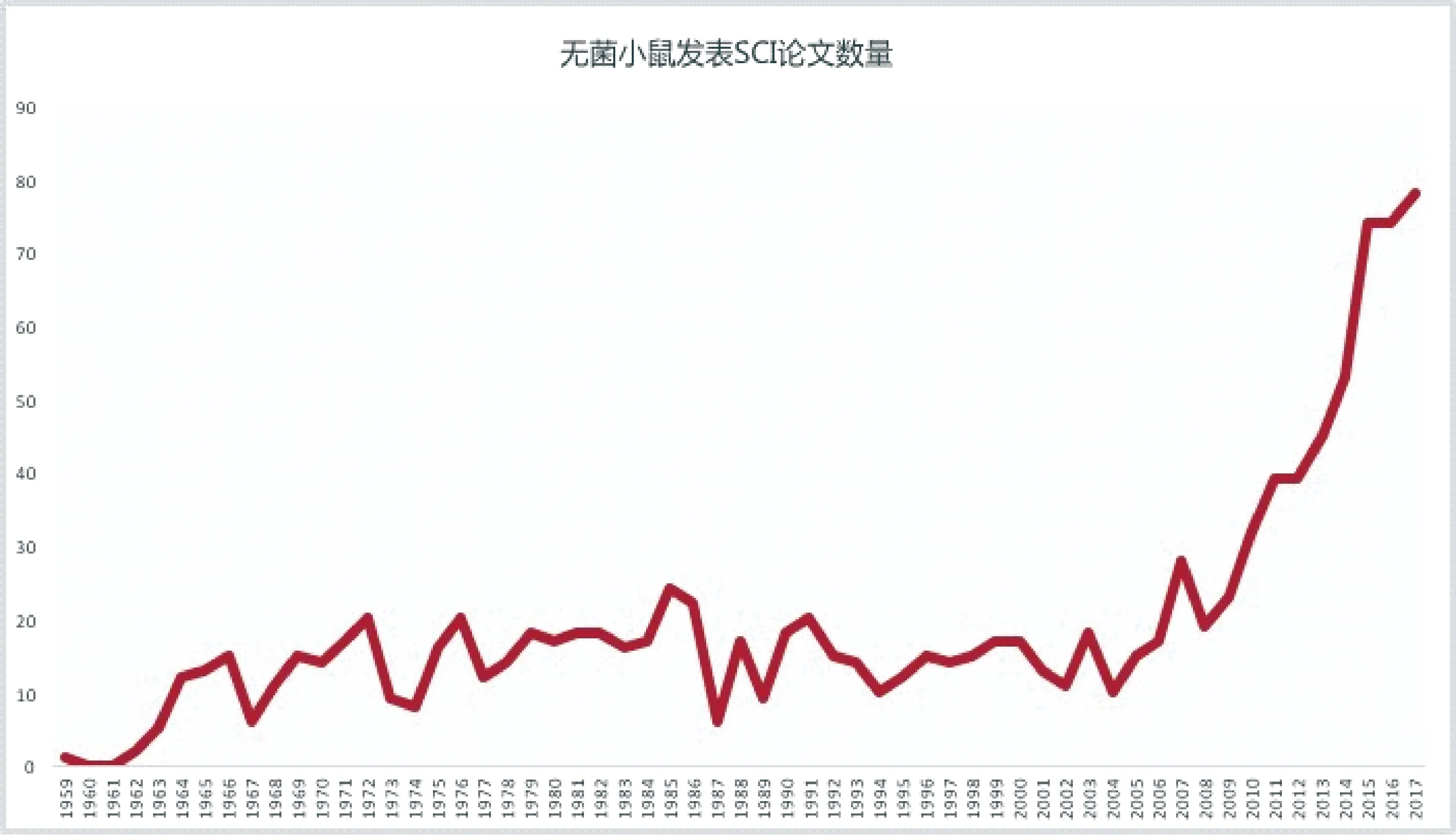
注:NCBI数据库检索题目或摘要有“germ-free mice”、“germ-free mice”或“germ-free mice”的文献;检索截止日期2017年3月28日;2017年文献数量以1月1日~3月28日为基数估算。图1 无菌小鼠应用发表SCI论文快速增加Note.Searching literature in the title or abstract with “germ-free mice”,“germ-free mice”,or "germ-free mice" in NCBI database; deadline to March 28.The data of 2017 is estimated based on the data from January 1 to March 28.Fig.1 Rapid increase of published SCI papers on germ-free mice
宏基因组技术和无菌动物技术是推动人体微生物组的两大关键技术。宏基因组技术解决了大部分肠道菌群不能培养无法清晰解析的技术瓶颈,而无菌动物技术则解决了清晰研究菌群或菌株对机体作用影响的技术难题(图2)。自出生起,人体便开始定植各种微生物,并陪伴人的一生。而根据科学研究的单变量原则,要阐明菌群这一变量对宿主的影响,必须要对菌群这一变量进行人工设定才能完成,而在人体显然无法完成这样的菌群人工设定。无菌动物是完成单一菌群或菌株设定及解析的唯一工具。
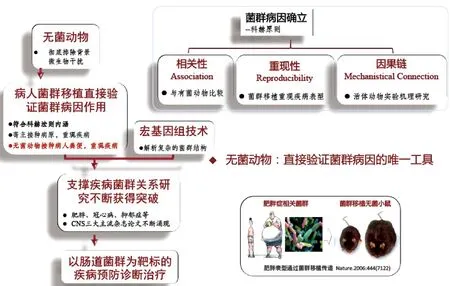
图2 无菌动物技术是菌群与疾病因果关系确立唯一工具Fig.2 Germ-free animal technology is the only tool to establish the causal relationship between microbiota and disease
根据疾病病因确定的科赫氏法则,确定肠道菌群作为某种疾病发生发展的关键因素,需要从相关性、重现性、分子证据链三方面进行分析及研究(图2)[6]。以本课题组与重庆医科大学谢鹏课题组合作的实验为例(图5)[5]。根据基于菌群源性代谢物在抑郁症患者体内异常的线索,进一步研究发现患者抑郁症患者菌群中Actinobacteria菌门丰度升高,Bacteroidetes菌门丰度降低,提示菌群紊乱与抑郁症的疾病表型存在相关性。为进一步证实菌群紊乱与抑郁症发生的因果关系,我们将抑郁症患者粪便移植无菌小鼠,发现小鼠重现与患者相似的抑郁样行为,同时抑郁症患菌群在无菌小鼠体内定植后与健康志愿者菌群在无菌小鼠体内定植后结构差异显著。提示通过菌群移植,可以在无菌小鼠体内重现抑郁症的疾病表型。接受抑郁症病人菌群移植的小鼠与接受健康人菌群移植小鼠比较,表现为碳水化合物、氨基酸代谢紊乱,为抑郁症表型在无菌小鼠体内重现提供了分子证据链。
2 无菌动物应用研究已形成通用模式
无菌动物应用模式化推动人体微生物组研究模式化,产生无菌动物规模化需求。无菌小鼠应用研究形成了四种通用模式(如图3所示),即:无菌动物与正常动物比较、无菌动物菌群移植、无菌动物与遗传工程技术结合、无菌动物不同发育阶段比较)[7]。各种研究模式并非孤立、矛盾,可交叉综合应用于研究,为综合研究及明确菌群在疾病发生发展中作用提供了不可替代的独特技术途径及研究模式。
2.1 无菌动物与正常菌群动物比较
为了区别于将无菌动物重新定植获得菌群的动物,习惯将正常饲养自然定植菌群的动物称为正常菌群动物(CON-R)。既可以通过无菌动物与正常菌群动物比较,间接证明有无菌群存在对机体的影响。也可以比较无菌动物与正常菌群动物对疾病诱导,间接证明有无菌群存在对疾病敏感性的影响。本课题组与中国科学院生物物理所刘志华研究员合作,通过无菌小鼠与SPF小鼠比较发现肠道共生菌可以选择性指导宿主细胞的物质分选,肠道肥大细胞溶菌酶释放必须依赖于肠道菌群存在(图4)[8]。
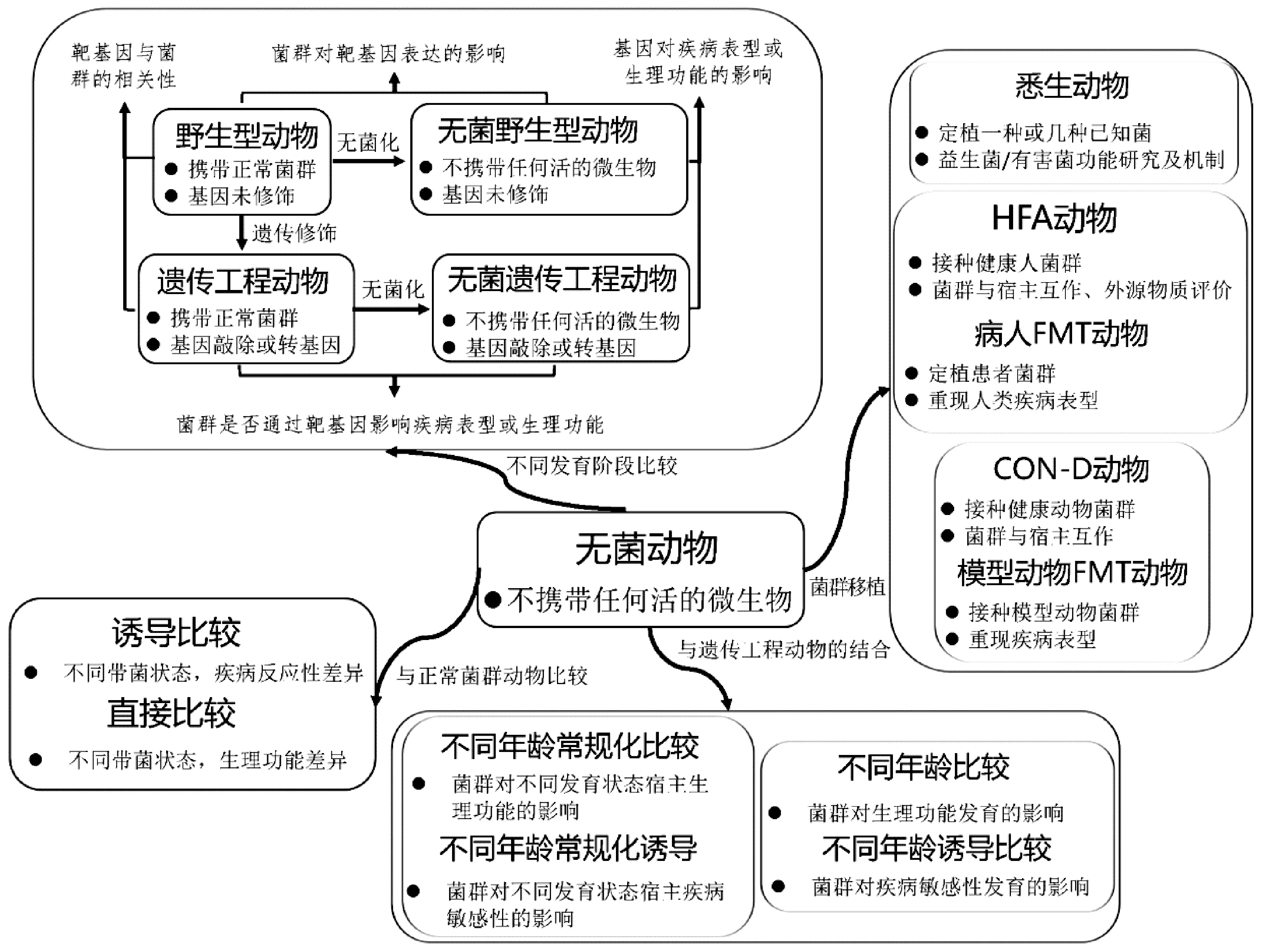
图3 无菌动物应用研究的四种通用模式Fig.3 Four commonly used models for the application studies of germ-free animals
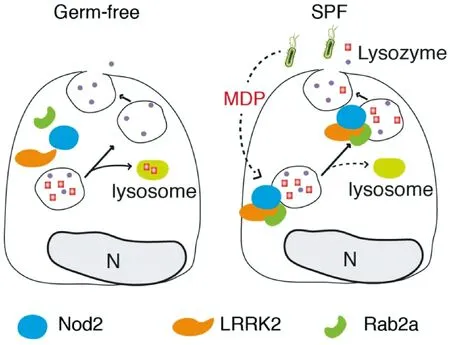
图4 无菌动物与SPF动物比较研究证实溶菌酶释放必须依赖于肠道菌群存在Nat Immunol.2015, 16(9):918-926Fig.4 A schematic working model for bacterially directed lysozyme sorting
2.2 无菌动物菌群移植
以菌株或人、动物不同健康状态、不同病程、不同处理的菌群移植于无菌动物,研究菌群重构后对机体的影响,直接证实菌群的功能。当移植物为一种或几种菌株时,获得的动物为悉生动物(gnotobiotic animal,GN),用于研究益生菌或有害菌功能及作用机制。当移植物为健康人菌群时,获得的动物为人源菌群动物(human flora associated animal,HFA),用于人肠道菌群与宿主相互作用或模拟人肠道菌群用于替代人进行外源物质如抗生素残留等评价。当移植物为健康的同种动物菌群时,获得的动物习惯称为常规化动物(conventionalized animal,CON-D),用于研究人肠道菌群与宿主相互作用。当移植物为病人或疾病动物模型菌群时,获得的动物习惯称为菌群移植动物(fecal microbiota transplantation animal,FMT),用于重现人类疾病,直接验证菌群病因作用。本课题组与香港中文大学于君教授、沈祖尧院士课题组合作,证实结直肠癌病人菌群移植无菌小鼠后,可促进其肠癌发生[17];本课题组与重庆医科大学谢鹏教授合作,利用无菌小鼠移植抑郁症患者菌群,证实肠道菌群紊乱是抑郁症发生的关键因素(图5)[5]。上述菌群移植疾病模型的研究,使我国在肠道菌群与抑郁、肠癌重大疾病发生发展的研究中,形成开拓性成果,产生学术影响。
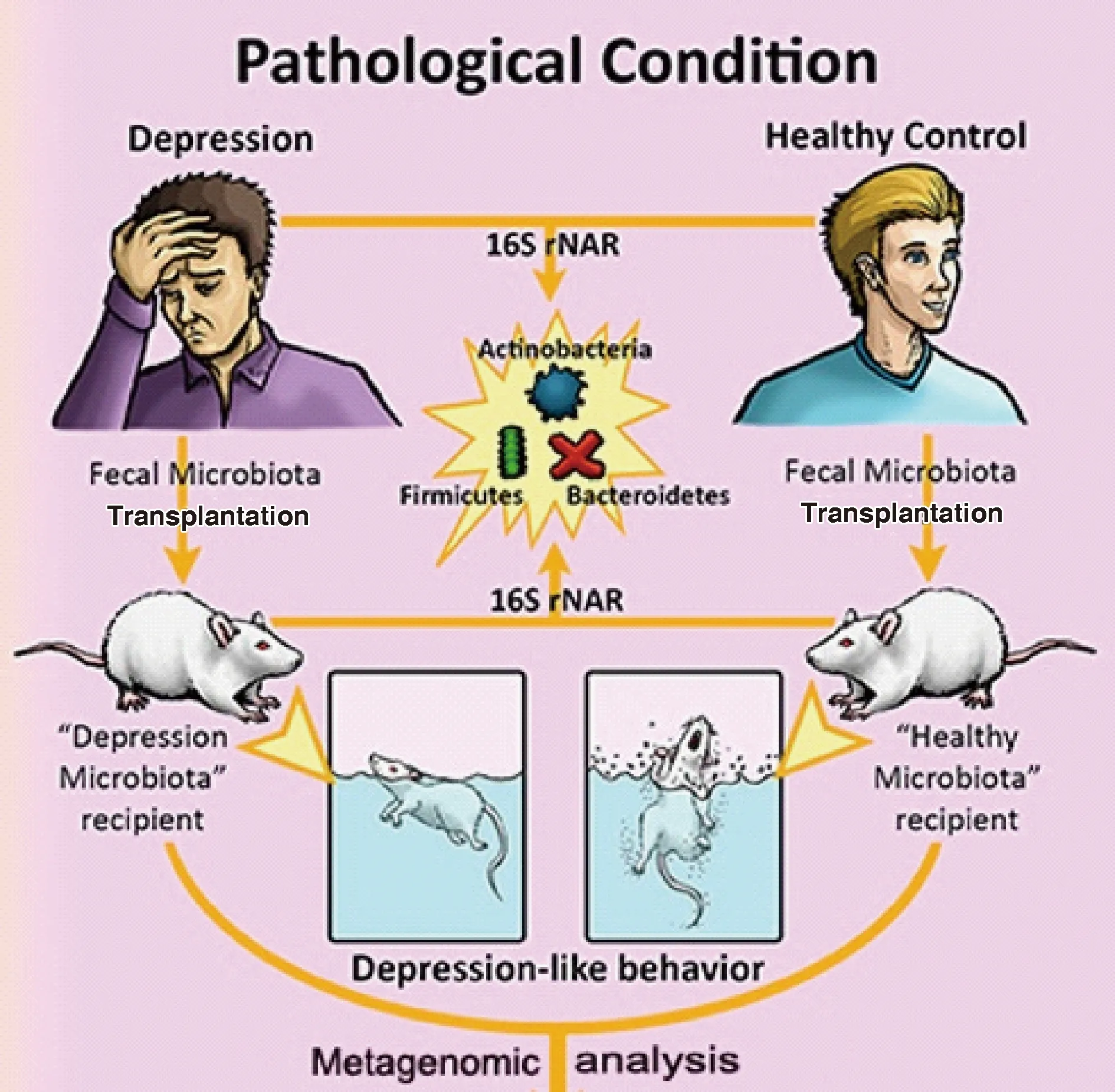
图5 菌群移植无菌小鼠模型证实肠道菌群紊乱是抑郁症发生关键因素(重庆医科大学谢鹏课题组提供图片)Fig.5 The model of microbiota transplantation in the germ-free mice confirmed that the disorder of dysbacteriosis is the key factor of depression (the picture courtesy of Professor Xie Peng’s research group in Chongqing Medical University)
HFA与病人FMT动物除移植物健康状态不同外,其移植程序也有很大差异。HFA动物一般要求菌群达到稳态;而FMT动物一般以出现疾病表型,在不给予致病因子刺激的情况下移植后观察时间过长,虽然菌群达到稳态但往往会导致疾病表型消失,可能与宿主对菌群修饰有关。
2.3 无菌动物与遗传工程动物的结合
遗传工程动物是研究特定基因功能的必备工具,将无菌动物技术与遗传工程技术结合,可阐明菌群与基因互作对相应疾病表型的影响。通过遗传工程动物菌群结构变化可以观察宿主基因改变对菌群的影响,以及基因引起的表型变化与菌群之间的相关性。通过遗传工程动物无菌化后表型的变化,可以观察菌群是否通过靶基因影响表型。通过观察无菌遗传工程动物及野生型动物比较,排除菌群干扰,从而阐明基因改变对表型的影响。而野生型动物无菌化后可以观察菌群对特定基因表达的影响。
2.4 无菌动物不同发育阶段比较
通过无菌动物不同发育阶段比较研究,阐述菌群对某种生理功能发育或疾病表型的影响。将不同发育阶段的无菌动物和正常菌群动物比较,观察菌群存在与否对特定生理功能发育的影响;将不同发育阶段的无菌动物和正常菌群动物进行诱导,观察菌群存在与否对疾病敏感性的影响;将不同发育阶段的无菌动物常规化,观察菌群对不同发育状态宿主生理功能的影响;将不同发育阶段的无菌动物常规化后进行诱导,观察菌群对不同发育状态宿主疾病敏感性的影响。
3 第三军医大学无菌动物平台为我国微生物组研究发挥了重要支撑作用
通过长期、大量的无菌动物培育研究及其模型应用研究,第三军医大学基础部实验动物学教研室建成了国内体量较大、技术体系稳定、服务单位多、具有一定影响力的无菌动物平台。
在连续两轮973课题、863课题、国家自然科学基金、国家支撑计划、国家重点研发计划等的资助下,第三军医大学自2006年开展无菌小鼠研究工作,于2009年首次获得无菌昆明小鼠种群。突破高效无菌剖腹产生物净化技术、小鼠人工乳配方,独创无菌小鼠自动哺乳技术,建立了无菌小鼠制备关键技术体系。形成了完备的无菌小鼠制备、繁育、质控、应用体系,持续繁育无菌小鼠8年,年生产供应2000余只。突破无菌近交系小鼠制备繁育技术瓶颈,获得无菌小鼠品系十余个。
第三军医大学利用无菌小鼠技术体系与中国科学院生物物理所、香港中文大学、重庆医科大学、浙江大学、华南农业大学等40余个单位开展了合作研究,有力推动了我国菌群与疾病关系研究。在免疫学[8,9,20-22]、结直肠癌[17]、神经科学及抑郁症[5,12,19]、肝病[14]、营养代谢[11]、食品科学[16,18]、类风湿关节炎[23]、畜牧兽医学[10,13,15]等多个学科获得重要进展。
第三军医大学为清华大学、中山大学等提供了基于无菌动物的高水平技术服务,为国内一批重大科学问题解决提供了关键技术支撑[24,25]。为清华大学提供技术服务,利用无菌小鼠模型与基因表达谱分析手段,证明了肠道微生物对RALDH+DC以及淋巴结发育的影响[24]。
4 建立我国规模化无菌动物高效研究应用体系
4.1 我国无菌动物研究应用存在的问题
4.1.1 无菌动物制备效率低
我国无菌动物制备效率低、规模小、种类少,不能满足我国日益增长的应用研究需求。
4.1.2 无菌动物供应与应用分离
我国无菌动物制备供应平台与应用研究平台分离,制约无菌动物推广应用。
4.1.3 无菌动物尚未形成整体研究应用规模
尽管我国在抑郁症、结肠癌、溶菌酶释放调节等领域取得了重大进展,但相对于华盛顿大学Jeffrey Gordon等利用无菌动物及宏基因组集成技术体系整体推进的国际先进水平具有明显差距,有待加强整体化推进规模。
4.2 建立我国规模化无菌动物高效研究应用体系
4.2.1 构建无菌动物高效制备技术体系
建立国际领先的规模化无菌动物制备、研究、应用体系,支撑我国菌群与疾病、微生物组研究的创新发展。
4.2.2 建立基于无菌动物技术的整合研发平台
构建无菌动物制备、实验、表型检测集成综合无菌动物试验平台,通过核心技术平台凝聚项目、人才、资源以服务全国,适应中国微生物组计划发展趋势。
4.2.3 积极推动基于无菌动物技术的科学发现与研发评价
以无菌动物为核心模型,推动重大疾病肠道菌群因果关系确证导向的科学发现,服务于功效菌株体内筛选评价、肠道菌群靶标产品的安全性有效性评价、粪菌移植临床治疗的安全性有效性评价等基础研究、应用研发、产品评价。
4.2.4 拓展无菌猪、无菌仓鼠等无菌动物种类,满足科学研究中对不同种类无菌动物的个性化需求
本课题组已获得国家自然科学基金“以肠道菌群为靶点的拟人化仓鼠冠心病模型建立”(81770434)资助,进行无菌仓鼠制备研究。与重庆市畜牧科学院葛良鹏研究员课题组联合获得重庆市科技研发基地能力提升项目“无菌动物应用示范平台”(cstc2015pt-nsjg80003)资助,进行无菌猪及无菌小鼠繁育研究。
[1] Eckburg PB,Bik EM,Bernstein CN,et al.Diversity of the human intestinal microbial flora [J].Science,2005,308(5728): 1635-1638.
[2] Nicholson JK,Holmes E,Kinross J,et al.Host-gut microbiota metabolic interactions [J].Science,2012,336(6086): 1262-1267.
[3] Ridaura VK,Faith JJ,Rey FE,et al.Gut microbiota from twins discordant for obesity modulate metabolism in mice [J].Science,2013,341(6150): 1241214.
[4] Hsiao EY,McBride SW,Hsien S,et al.Microbiota modulate behavioral and physiological abnormalities associated with neurodevelopmental disorders [J].Cell,2013,155(7): 1451-1463.
[5] Zheng P,Zeng B,Zhou C,et al.Gut microbiome remodeling induces depressive-like behaviors through a pathway mediated by the host’s metabolism [J].Mol Psychiatry,2016,21(6): 786-796.
[6] Zhao L.The gut microbiota and obesity: from correlation to causality [J].Nat Rev Microbiol,2013,11(9): 639-647.
[7] 曾本华,魏泓.无菌动物与微生态学专题教学改革实践 [J].中国微生态学杂志,2017,29(3): 354-356.
[8] Zhang Q,Pan Y,Yan R,et al.Commensal bacteria direct selective cargo sorting to promote symbiosis [J].Nat Immunol,2015,16(9):918-926.
[9] Cao S,Su X,Zeng B,et al.The gut epithelial receptor LRRC19 promotes the recruitment of immune cells and gut inflammation [J].Cell Rep,2016,14(4): 695-707.
[10] Ren W,Yin J,Xiao H,et al.Intestinal microbiota-derived GABA mediates interleukin-17 expression during enterotoxigenic Escherichia coli infection [J].Front Immunol,2017,7: 685.
[11] Zhong CY,Sun WW,Ma Y,et al.Microbiota prevents cholesterol loss from the body by regulating host gene expression in mice [J].Sci Rep,2015,5: 10512.
[12] Zeng L,Zeng B,Wang H,et al.Microbiota modulates behavior and protein kinase C mediated cAMP response element-binding protein signaling [J].Sci Rep,2016,6: 29998.
[13] Yan H,Diao H,Xiao Y,et al.Gut microbiota can transfer fiber characteristics and lipid metabolic profiles of skeletal muscle from pigs to germ-free mice [J].Sci Rep,2016,6: 31786.
[14] Wei Y,Zeng B,Chen J,et al.Enterogenous bacterial glycolipids are required for the generation of natural killer T cells mediated liver injury [J].Sci Rep,2016,6: 36365.
[15] Diao H,Yan HL,Xiao Y,et al.Intestinal microbiota could transfer host gut characteristics from pigs to mice [J].BMC Microbiol,2016,16(1): 238.
[16] Zhang X,Zeng B,Liu Z,et al.Comparative diversity analysis of gut microbiota in two different human flora-associated mouse strains[J].Curr Microbiol,2014,69(3): 365-373.
[17] Wong SH,Zhao L,Zhang X,et al.Gavage of fecal samples from patients with colorectal cancer promotes intestinal carcinogenesis in germ-free and conventional mice[J].Gastroenterology,2017,doi: 10.1053/j.gastro.2017.08.022.
[18] Wang L,Zeng B,Zhang X,et al.The effect of green tea polyphenols on gut microbial diversity and fat deposition in C57BL/6J HFA mice [J].Food Funct,2016,7(12): 4956-4966.
[19] Chen JJ,Zeng BH,Li WW,et al.Effects of gut microbiota on the microRNA and mRNA expression in the hippocampus of mice [J].Behav Brain Res,2017,322(Pt A): 34-41.
[20] Wang L,Zeng B,Zhang X,et al.rip2 is required for nod2-mediated lysozyme sorting in Paneth cells [J].J Immunol,2017,198(9): 3729-3736.
[21] Jiang W,Wang X,Zeng B,et al.Recognition of gut microbiota by NOD2 is essential for the homeostasis of intestinal intraepithelial lymphocytes [J].J Exp Med,2013,210(11): 2465-2476.
[22] Su X,Yan H,Huang Y,et al.Expression of FABP4,adipsin and adiponectin in Paneth cells is modulated by gut Lactobacillus [J].Sci Rep,2015,5: 18588.
[23] Liu X,Zeng B,Zhang J,et al.Role of the gut microbiome in modulating arthritis progression in mice [J].Sci Rep,2016,6: 30594.
[24] Zhang Z,Li J,Zheng W,et al.Peripheral lymphoid volume expansion and maintenance are controlled by gut microbiota via RALDH+dendritic cells [J].Immunity,2016,44(2): 330-342.
[25] Wang D,Xia M,Yan X,et al.Gutmicrobiota metabolism of an thiocyanin promotes reverse cholesterol transport in mice via repressing miRNA-10b [J].Circ Res,2012,111(8): 967-981.
Establishmentofahighlyeffectivelarge-scalegerm-freeanimalefficiencyresearchandapplicationsysteminChina
ZENG Ben-hua,WEI Hong*
(Department of Laboratory Animal Science,College of Basic Medical Sciences,Third Military Medical University,Chongqing 400038,China)
Germ-free animals and metagenomic technology are the two major wheels for the study of microbiome in human health.Without a transplantation model of germ-free animals inoculated with microbiota (bacterial strains),it is impossible to establish the causal relationship between microbiota and diseases.Without germ-free animals,there will be no rapid development of research on the relationship between microbiota and human diseases today and tomorrow.Application of germ-free animals has formed four kinds of general research models,including comparison of conventional and germ-free animals,bacteria (strain) transplantation with germ-free animals,rederivation of germ-free genetic engineering animals,and development of germ-free animals.Standardization of research mode will increase the scale and speed of application of germ-free animals.After ten years of efforts,Professor Wei Hong’s group has built the largest scale,the most stable technical system,most widely used platform of germ-free animal in our country,which have provided a strong support in our country for scientific problem solving in the fields including animal husbandry,biomedicine,and food microbiology.At present,the germ-free animals are in small scale,low in efficiency,and the platform is separated from the application conditions.The basic theory and technical system of germ-free animals are still to be developed,and it is difficult to meet the growing demand for application.Establishment of an efficient large-scale research and application system of germ-free animals will meet the needs of rapid development of microbiome research in China.
Germ-free animal; Microbiome; Animal model
WEI Hong.E-mail: weihong63528@163.com
国家自然科学基金面上项目(No.81770434,No.8137090);973课题(No.2013CB532406);重庆市科技研发基地能力提升项目(No.cstc2015pt-nsjg80003)。
曾本华(1980-),男,博士,从事无菌动物及其模型研究。Email:gfanimal@163.com
魏泓,男,博士,教授,博士生导师,从事无菌动物与遗传工程动物模型研究。Email:weihong63528@163.com
Q95-33
A
1005-4847(2017) 06-0648-06
10.3969/j.issn.1005-4847.2017.06.011
2017-10-11
