基于de novo高通量测序的曼氏无针乌贼(Sepiellajaponica)ESTs中微卫星位点筛选与特征分析*
2017-12-14吕振明刘立芹陈永久郭宝英董迎辉吴常文
吕振明 侯 龙 龚 理 刘立芹 陈永久 郭宝英董迎辉 吴常文
(1.浙江海洋大学 海洋生物种质资源发掘与利用国家地方联合工程实验室 舟山 316022;2.浙江万里学院 浙江省水产种质资源高效利用技术研究重点实验室 宁波 315100)
基于de novo高通量测序的曼氏无针乌贼(Sepiella
japonica)ESTs中微卫星位点筛选与特征分析*
吕振明1侯 龙1龚 理1刘立芹1陈永久1郭宝英1董迎辉2①吴常文1
(1.浙江海洋大学 海洋生物种质资源发掘与利用国家地方联合工程实验室 舟山 316022;2.浙江万里学院 浙江省水产种质资源高效利用技术研究重点实验室 宁波 315100)
对均一化转录组测序获得的 47604个曼氏无针乌贼的微卫星序列进行分析, 结果表明, 乌贼转录组中微卫星位点丰富, 每1402nt的EST中就有一段不小于12nt长度的微卫星序列。单碱基重复是EST微卫星序列的主要形式(38.69%), 其次依次为三碱基(31.14%)、二碱基(26.35%)、四碱基(3.29%)、五碱基(0.38%)、六碱基(0.14%)重复, 短序列类型占微卫星总量的 96.18%。同碱基类型的微卫星序列组成又存在差异, AC(54.51%)和 AG(31.22%)是最常见的二碱基重复序列; 而AGC(16.37%)和 AAC(14.06%)是最常见的三碱基重复序列。通过引物设计和体系优化, 共筛选到了24对多态性微卫星位点, 对来自福建漳州海域的35只野生曼氏无针乌贼(Sepiella japonica)个体进行群体遗传学检测, 结果表明, 每个位点检测到的等位基因数3—10个不等(均值5.9个); 平均杂合度观测值(Ho)和期望值(He)分别为0.518和0.681。除2个位点外所有位点的多态信息含量(PIC)都大于0.5。Hardy-Weinberg平衡检测表明, 仅4个位点有显著偏离(P<0.05), 未检测到连锁不平衡现象。这表明转录组高通量测序技术适于乌贼微卫星位点的开发, 获得的SSR标记可用于今后乌贼资源遗传评估和科学有效管理过程。
曼氏无针乌贼; 转录组; EST序列; 微卫星
曼氏无针乌贼(Sepiella japonica)属软体动物门、头足纲、十腕目、乌贼科、无针乌贼属种类, 是我国重要的海洋渔业资源之一, 20世纪70年代, 我国曼氏无针乌贼年产量曾达 6万多吨, 是我国著名的“四大海产”之一(董正之, 1988)。但自20世纪80年代以来,由于过渡捕捞或海域环境的破坏, 其资源量严重衰退, 几近枯竭的边缘(Lüet al, 2016)。为了恢复我国曼氏无针乌贼的渔业资源和产量, 近年来我国陆续攻克了其规模化苗种繁育和养殖技术, 人工养殖已在国内沿海省份悄然兴起, 而其人工增殖放流更是在国内得到迅猛开展, 并取得显著成效(Jianget al,2014; Yanet al, 2016)。然而, 为更好地开展曼氏无针乌贼的繁养及增殖养护工作, 迫切需要对其现有种群的种质状况和遗传组成进行精确的了解。
微卫星标记技术是研究物种种质状况和遗传组成的重要方法, 由于其高度多态性和共显性等优势而被广泛运用于物种的群体遗传结构(Anet al,2014)、种质状况评估(Lüet al, 2013)及遗传改良研究中(Fujiet al, 2007)。近年来, 有关曼氏无针乌贼微卫星标记的开发已有少量相关报道(Wuet al, 2010; Guoet al, 2013), 但仍需要开发出更多的微卫星标记用于其种质和遗传组成的评估, 同时以往的研究均以DNA建库和探针杂交富集法进行微卫星的筛选(Zaneet al, 2002), 但该方法不仅费时、费力, 而且需要预先对物种的基因组信息有较好的了解(Wanget al,2012)。近年来, 基于二代高通量测序的出现为快速、高效的进行微卫星的开发提供了可能(Csencsicset al,2010), 该技术可通过一次高通量的测序产生海量的微卫星序列, 因而被广泛运用于许多非模式动植物的微卫星开发中(Yuet al, 2011; Zhuet al, 2012)。特别是转录组高通量测序技术, 由于成本低, 微卫星序列检出率高, 因而被广泛运用于包括鱼类(Teacheret al,2012)、虾蟹类(Maet al, 2014)、贝类(Patnaiket al,2016)等水生生物的微卫星标记开发中, 目前采用高通量测序进行乌贼等头足类动物的微卫星标记开发还未见相关报道, 本研究拟采用转录组测序技术从海量的 EST序列中筛选微卫星序列, 分析其序列组成和特征, 开发多态性较强的微卫星位点用于其种群遗传变异分析, 相关研究成果将为今后我国曼氏无针乌贼资源科学合理保护和开发奠定基础。
1 材料与方法
1.1 材料
用于转录组de novo高通量测序分析的曼氏无针乌贼(Sepiella japonica)鲜活样品取自浙江省海洋水产研究所西闪养殖基地。活体解剖后取肌肉、肝脏、鳃、脑、性腺等组织, 迅速放入液氮中冷冻保存备用。用于多态微卫星位点分离与鉴定及群体遗传多态度检测的曼氏无针乌贼样本取自福建漳州海域的自然群体,共计35只, 取肌肉组织用70%的酒精保存备用。
1.2 方法
1.2.1 转录组 EST微卫星序列获取 曼氏无针乌贼组织RNA的提取、cDNA文库的构建参照Teacher等(2012)的方法进行。构建好的 cDNA文库在 CBot上进行簇生成反应(TruSeq PE Cluster Kit V3-cBot-HS,Illumina)后, 采用Illumina Hiseq 2000高通量测序平台(TruSeq SBS KIT-HS V3, Illumina) 进行双末端测序, 由深圳华大基因科技有限公司完成。获得的clean reads进行De novo 序列拼接和组装, 采用MISA软件对拼接得到的序列进行微卫星的搜索, SSR位点确认设置条件为单碱基类型重复至少12次, 2碱基类型重复至少 6 次, 3、4碱基类型重复至少5次, 6碱基以上类型重复至少4次。对转录组EST微卫星的类型, 各微卫星类型比例及序列特征等进行分析和统计。
1.2.2 多态微卫星位点分离与鉴定 选取二、三碱基重复单元为主的微卫星序列, 采用 primer3设计微卫星引物, 引物合成由上海英俊生物技术有限公司完成。微卫星位点分离时DNA的提取采用传统的酚、氯仿抽提法; PCR扩增采用 25μL的反应体系, 内含50ng基因组DNA, 1×buffer (Promega, USA), 2mmol/L MgCl2, 0.2mmol/L dNTPs, 0.2μmol/L各引物和1UTaq酶(Promega, USA)。反应程序条件为94℃预变性5min;94℃变性1min, 50—65℃退火1min, 72℃延伸1min,35个循环; 72℃延伸 10min, 各引物退火条件在50—65℃范围内进行优化。利用 10个个体进行多态性标记的检测, PCR产物采用毛细管电泳检测, 用Genemapper 3.5 (Applied Biosystem)分析产物长度。
1.2.3 群体遗传多态度检测与统计 采用筛选出的多态性微卫星引物对采自福建漳州天然海域的 35只曼氏无针乌贼进行SSR扩增, 产物经电泳、分型后,采用ARLEQUIN3.5.1.3 (Excoffieret al, 2010)软件统计位点等位基因数(NA)、观测杂合度(Ho)、预期杂合度(He)、多态信息含量(PIC)等遗传变异参数。采用CERVUS3.0.3 (Marshallet al, 1998)和GENEPOP 4.0.10 (Raymondet al, 1995)检测各位点等位基因分布是否符合 Hardy-Weinberg平衡(HWE)及各位点是否存在连锁不平衡。
2 结果
2.1 曼氏无针乌贼转录组EST中微卫星的序列特征
采用 Illumina测序和 De novo拼接后共获得了85674个Unigene, 总长66760294nt, 共搜索到47604个微卫星序列, 平均每1402nt的EST序列中就含有1个至少12nt的微卫星序列, 微卫星分布相当丰富。对微卫星的组成类型和数量进行分析, 结果表明: 单碱基重复是曼氏无针乌贼转录组 EST最主要的微卫星类型(38.69%); 其次为三碱基(31.14%)和二碱基(26.35%)重复类型, 上述三种短序列重复类型占微卫星总量的 96.18%。而长序列类型微卫星相对占比很低, 四碱基(3.29%)、五碱基(0.38%)、六碱基(0.14%)的重复类型总和只占微卫星总量的3.82%, 如图1所示。各碱基类型的微卫星中, 不同序列组成的微卫星数量迥异。如图2所示, 二碱基类型中, AC(54.51%)和 AG(31.22%)是最常见的二碱基重复形式, 两者在二碱基类型中的占比达 85.73%; 而 CG(0.10%)的重复形式最少, 较低频率的GC类型可能与胞嘧啶易甲基化, 使其因脱氨基作用而变成胸腺嘧啶有关(Schorderetet al, 1992)。而三碱基类型中各碱基组成形式差异相对较小, AGC(16.37%)和AAC(14.06%)是最常见的三碱基重复形式, 其次为 ATC(13.72%)、ACC(13.07%)、AAT(10.67%)、ACT(10.27%)、AGG(9.62%)、AAG(9.52%),而 ACG(1.99%)和 CCG(0.72%)类型则最少。
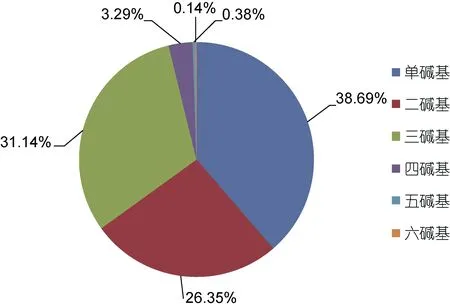
图1 曼氏无针乌贼转录组EST中1-6碱基类型微卫星的组成Fig.1 The percentages of mono-, di-, tri-, tetra-, quad-, penta-,and hexa-nucleotide repeats in SSR motif sequences in S.japonica transcriptome EST
各碱基类型的微卫星在重复次数变异上也存在巨大差别。如表1所示, 二碱基类型中重复次数变异6—11次不等, 而三碱基类型的重复次数变异范围为5—8次; 四碱基和五碱基类型的重复次数仅5—6次和4—5次; 六碱基类型中未发现有重复次数的变异,均为重复4次, 表明各碱基类型随着碱基数的增加存在的变异也逐渐减少。同时, 转录组中微卫星均以最低重复数的序列最多, 如在二碱基类型中, 最低重复数 6的微卫星数量最多, 占所有二碱基类型的35.04%。三碱基类型中, 最低重复数5的微卫星数量最多, 占所有三碱基类型的53.85%。随着重复数的增加, 相应微卫星的数量逐级减少。

图2 曼氏无针乌贼转录组EST中二、三碱基类型微卫星的序列组成情况Fig.2 Observed counts of identified microsatellite loci for different repeat sequence motifs of di- and trinucleotide repeats in S.japonica transcriptome EST
2.2 曼氏无针乌贼多态微卫星位点的分离及多态性分析
共筛选了121对二、三碱基类型为主的微卫星引物, 仅 65对引物能稳定扩增出清晰、可辨的微卫星条带, 其中具多态位点的微卫星数达24对。采用24对微卫星多态位点对采自福建漳州海域的35只曼氏无针乌贼个体进行遗传变异和多态性检测, 结果如表2所示: 24对引物共扩增得到142个等位基因, 每个位点的等位基因数(Na)3—10不等, 平均 5.92个等位基因。杂合度观测值(Ho)在 0.030—0.857之间, 均值为 0.518, 杂合度预期值(He)在 0.420—0.826之间,均值为 0.681。多态信息含量(PIC)在 0.3863—0.8728之间。除位点CL2235-2 (PIC=0.3863)、CL899(0.4009)外, 所有位点的 PIC值都大于 0.5, 显示了较高的多态性。二、三碱基类型微卫星相比, 二碱基微卫星标记的多态性无论是位点等位基因数(Na)、杂合度(He)、多态信息含量(PIC)等参数都明显比三碱基高, 并且在He和PIC上的差异达到显著(P<0.05)。不同重复次数的微卫星相比, 重复次数越高的微卫星其多态性也越高, 但仅在位点等位基因数(Na)差异上达到显著水平(P<0.05), 如表3所示。经检测, 有4个位点明显偏离Hardy-Weinberg平衡(P<0.05); 经连锁不平衡检测, 所有位点均未检测到明点的连锁不平衡现象(P>0.05)。
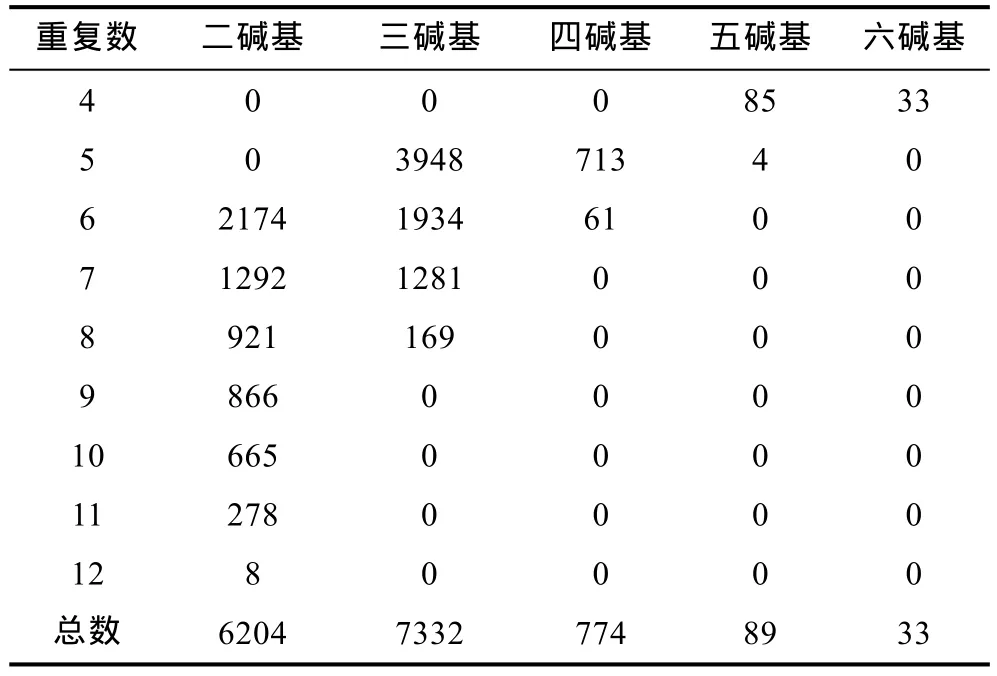
表1 曼氏无针乌贼转录组EST中各种微卫星类型的重复数变异Tab.1 The repeating number of di-, tri-, quad-, penta-, and hexa-nucleotide repeats in SSR motif sequences in S.japonica transcriptome EST
3 讨论
从本研究的结果可以看出, 基于转录组的高通量测序是非常快速、高效的海洋头足类微卫星分离方法。本研究之前, 仅有约 25对曼氏无针乌贼的微卫星标记被成功开发(Wuet al, 2010; Guoet al, 2013),而利用de novo转录组高通量测序技术, 我们共获得了85674个Ungene, 基因序列总长66760294nt, 共搜索到47604个微卫星序列, 平均每1402nt的EST就有一个不少于 12nt的微卫星序列。大部分序列都含有完整的适于微卫星引物设计的两翼序列, 并可用于今后的微卫星标记大规模开发中, 这些微卫星序列几乎是之前采用DNA建库和探针杂交富集法获得的微卫星序列的上千倍。这也许就是为什么近年来高通量测序技术越来越多地被用于微卫星标记开发之中的原因。
除快速、高效之外, 高通量测序技术还提供了传统方法无法提供的微卫星基因组序列特征, 而这些微卫星基因组序列信息可为更有效地筛选微卫星目标序列、设计引物, 更快速地筛选高多态性的微卫星标记提供了良好的基础。如由研究结果可知, 一、二、三碱基是最主要基因组微卫星碱基类型, 四、五、六碱基则相对稀少; 呈现出随着碱基数量的增加, 微卫星数量逐步减少的现象, 这与已知的许多真核生物的基因组微卫星组成情况类似(Chistiakovet al, 2006;Zhuet al, 2012), 这就提示我们在曼氏无针乌贼微卫星标记规模化开发时, 二、三碱基的微卫星类型可能是最主要的筛选对象。同时二、三碱基的微卫星类型相比, 二碱基微卫星的重复次数从6—12共7种类型,而三碱基微卫星的重复次数仅 5—8共 4种类型, 二碱基比三碱基微卫星具有更多的变异类型, 暗示二碱基也可能比三碱基微卫星标记具有更高的多态性,而该结果也得到了后续实验结果的证实, 二碱基微卫星无论在He和 PIC上都显著高于三碱基(P<0.05),这也提示为了更有效地筛选多态性微卫星标记, 二碱基类型将是首选的筛选对象。另外, 不同重复次数的微卫星相比, 重复次数越高的微卫星其多态性也越高, 如图2所示。该结果与前人在哺乳动物包括人类中的研究结果相似(Kelkaret al, 2008), 这也告诉我们, 在曼氏无针乌贼的微卫星开发中, 选择重复次数高的微卫星将是提高微卫星多态性的有效方法。
本研究共筛选出 24对多态性的微卫星位点, 并用于检测来自福建漳州的曼氏无针乌贼自然群体的遗传多态性。结果表明, 漳州曼氏无针乌贼自然群体仍存在较高的遗传多样性。24个位点的等位基因数(NA)3—10不等, 平均5.92个等位基因。杂合度观测值(Ho)在 0.030—0.857之间, 杂合度预期值(He)在0.420—0.826之间, 多态信息含量(PIC)在 0.3863—0.8728之间。该结果与Guo等(2013)以DNA测序为基础的生物素磁珠法开发的微卫星标记多态性大致相当, 而略低于 Wu等(2010)以尼龙膜杂交法开发的微卫星标记的多态性。但应该指出的是Guo等(2013)研究的群体是福建宁德的自然群体, 与本研究的福建漳州自然群体所处海区接近, 而 Wu等(2010)研究的群体为广东湛江群体。该结果说明, 采用转录组测序技术获得的微卫星多态性也可与DNA测序获得的微卫星多态性相当, 而与过去认为的编码区域的微卫星多态性一般较非编码区域的微卫星多态性低的看法相异(Mochmannet al, 2004; Pearsonet al, 2005),而与Kelkar等(2008)哺乳动物如人与大猩猩中的结果类似。因此该结果再一次说明转录组测序技术是一种高效快捷的微卫星标记开发方法。经检测24个微卫星位点无明显的连锁不平衡现象(P>0.05), 仅 4个显著偏离Hardy-Weinberg平衡(P<0.05), 这些新开发的多态性微卫星位点可为今后曼氏无针乌贼渔业资源的科学管理和保护提供有力的遗传学评估手段。
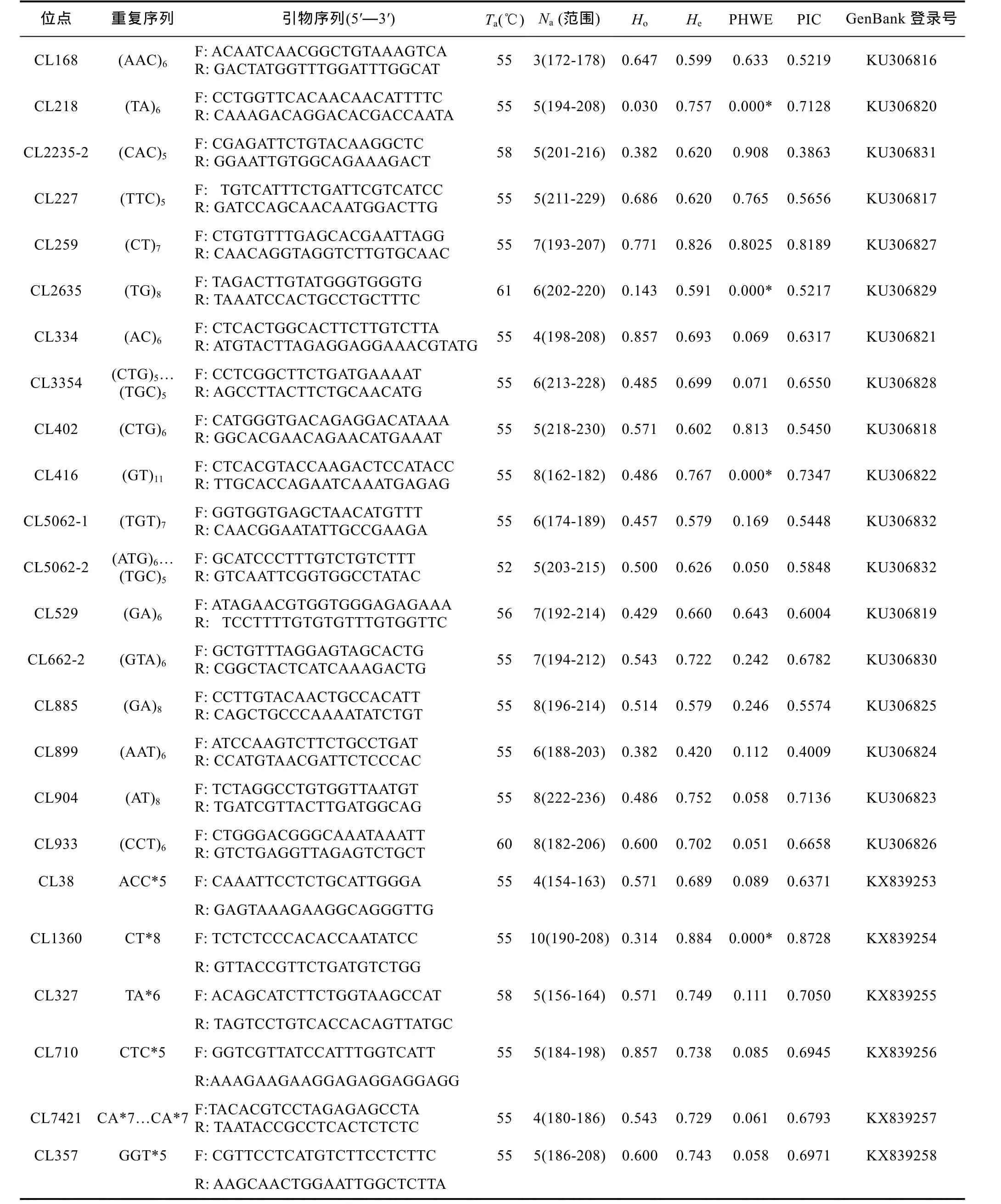
表2 曼氏无针乌贼24对微卫星多态位点特征Tab.2 Characteristics of 24 microsatellite loci developed for S.japonica

表3 不同碱基数和重复次数对微卫星多态性的影响Tab.3 The influence of repeat motif and repeat number on the polymorphic level of microsatellite markers
4 结论
本研究的结果表明, 基于转录组的高通量测序技术是海洋头足类微卫星标记高效开发的理想方法。与传统的DNA建库和探针杂交富集法开发微卫星标记相比, 其不仅快速、高效, 而且开发的微卫星多态性也与传统方法相当。本研究筛选出的24对多态性微卫星位点可用于今后乌贼资源遗传评估和科学有效管理过程。
董正之, 1988.中国动物志: 软体动物门, 头足纲.北京: 科学出版社
An H S, Lee J W, Kim H Yet al, 2014.Population genetic structure of the sea bass (Lateolabrax japonicus) in Korea based on multiplex PCR assays with 12 polymorphic microsatellite markers.Genes & Genomics, 36(2): 247—259
Chistiakov D A, Hellemans B, Volckaert F A Met al, 2006.Microsatellites and their genomic distribution, evolution,function and applications: a review with special reference to fish genetics.Aquaculture, 255(1—4): 1—29
Csencsics D, Brodbeck S, Holderegger R, 2010.Cost-effective,species-specific microsatellite development for the endangered dwarf bulrush (Typhaminima) using next-generation sequencing technology.Journal of Heredity,101(6): 789—793
Excoffier L, Lischer H E L, 2010.Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows.Molecular Ecology Resources,10(3): 564—567
Fuji K, Hasegawa O, Honda Ket al, 2007.Marker-assisted breeding of a lymphocystis disease-resistant Japanese flounder (Paralichthys olivaceus).Aquaculture, 272(1—4):291—295
Guo B Y, Qi P Z, Zhu A Yet al, 2013.Isolation and characterization of new polymorphic microsatellite markers from the cuttlefishSepiella maindroni(Cephalopoda;Sepiidae).Genetics and Molecular Research, 12(3):2376—2379
Jiang L H, Zhu A Y, Wu C Wet al, 2014.Tetracycline immersion tagging of cuttlefish,Sepiella japonica, Larvae.Journal of the World Aquaculture Society, 45(3): 342—349
Kelkar Y D, Tyekucheva S, Chiaromonte Fet al, 2008.The genome-wide determinants of human and chimpanzee microsatellite evolution.Genome Research, 18(1): 30—38
Lü Z M, Li H M, Liu L Qet al, 2013.Rapid development of microsatellite markers from the large yellow croaker(Pseudosciaena crocea) using next generation DNA sequencing technology.Biochemical Systematics and Ecology, 51: 314—319
Lü Z M, Liu W, Liu L Qet al, 2016.Cloning, characterization,and expression profile of estrogen receptor in common Chinese cuttlefish,Sepiella japonica.Journal of Experimental Zoology Part A, 325(3): 181—193
Ma H Y, Jiang W, Liu Pet al, 2014.Identification of transcriptome-derived microsatellite markers and their association with the growth performance of the mud crab(Scylla paramamosain).PLoS One, 9(2): e89134
Marshall T C, Slate J, Kruuk L E Bet al, 1998.Statistical confidence for likelihood-based paternity inference in natural populations.Molecular Ecology, 7(5): 639—655
Mochmann L H, Wells R D, 2004.Transcription influences the types of deletion and expansion products in an orientation-dependent manner from GAC·GTC repeats.Nucleic Acids Research, 32(15): 4469—4479
Patnaik B B, Wang T H, Kang S Wet al, 2016.Sequencing,de novoassembly, and annotation of the transcriptome of the endangered freshwater pearl bivalve,Cristaria plicata,provides novel insights into functional genes and marker discovery.PLoS One, 11(2): e0148622
Pearson C E, Edamura K N, Cleary J Det al, 2005.Repeat instability: mechanisms of dynamic mutations.Nature Reviews Genetics, 6(10): 729—742
Raymond M, Rousset F, 1995.An exact test for population differentiation.Evolution, 49(6): 1280—1283
Schorderet D F, Gartler S M, 1992.Analysis of CpG suppression in methylated and nonmethylated species.Proceedings of the National Academy of Sciences of the United States of America, 89(3): 957—961
Teacher A G F, Kähkönen K, Merilä J, 2012.Development of 61 new transcriptome-derived microsatellites for the Atlantic herring (Clupea harengus).Conservation Genetics Resources, 4(1): 71—74
Wang J J, Yu X M, Zhao Ket al, 2012.Microsatellite development for an endangered breammegalobrama pellegrini(Teleostei, Cyprinidae) using 454 sequencing.International Journal of Molecular Sciences, 13(12):3009—3021
Wu C W, Chi C F, He G Yet al, 2010.Isolation via enrichment and characterization of ten polymorphic microsatellite loci in the cuttlefish,Sepiella maindronide Rochebruns.Acta Oceanologica Sinica, 29(6): 121—124
Yan Y J, Wang T M, Liu Wet al, 2016.Identification and expression profile of the gonadotropin-releasing hormone receptor in common Chinese cuttlefish,Sepiella japonica.Journal of Experimental Zoology Part A, 325(7): 453—466
Yu J N, Won C, Jun Jet al, 2011.Fast and cost-effective mining of microsatellite markers using NGS technology: an example of a Korean water deerHydropotes inermis argyropus.PLoS One, 6(11): e26933
Zane L, Bargelloni L, Patarnello T, 2002.Strategies for microsatellite isolation: a review.Molecular Ecology, 11(1):1—16
Zhu H, Senalik D, McCown B Het al, 2012.Mining and validation of pyrosequenced simple sequence repeats (SSRs)from American cranberry (Vaccinium macrocarponAit.).Theoretical and Applied Genetics, 124(1): 87—96
ISOLATION AND ANALYSIS ON EST MICROSATELLITES OFSEPIELLA JAPONICABY DE NOVO HIGH-THROUGHPUT TRANSCRIPTOME SEQUENCING
LÜ Zhen-Ming1, HOU Long1, GONG Li1, LIU Li-Qin1, CHEN Yong-Jiu1, GUO Bao-Ying1,DONG Ying-Hui2, WU Chang-Wen1
(1.National and Provincial Joint Laboratory of Exploration and Utilization of Marine Aquatic Genetic Resources,Zhejiang Ocean University,Zhoushan316022,China; 2.Zhejiang Key Laboratory of Aquatic Germplasm Resources,Zhejiang Wanli University,Ningbo315100,China)
A total of 47604 microsatellite sequences fromSepiella japonicatranscriptome was obtained and analyzed by next-generation sequencing.The results show that there were abundant SSR sequences in transcriptome ofS.japonicaand, in average, 1402nt EST sequence would contain a SSR in length of no less than 12 nucleotides.The mono-nucleotides SSR is the main type of microsatellites (38.69%), followed by tri-nucleotide (31.14%), di-nucleotide (26.35%),quad-nucleotide (3.29%), penta-nucleotide (0.38%), and hexa-nucleotide (0.14%) microsatellites.The shorter repeats accounted for 96.18% of the total quantity of the microsatellite sequences.Among di-nucleotide repeats, AC (54.51%) and AG (31.22%) were most common, while among tri- nucleotide repeats, AGC (16.37%) and AAC (14.06%) were most frequent.Twenty-four polymorphic microsatellites were isolated and tested in 35 cuttlefish individuals collected from a natural population in Zhangzhou sea area, Fujian province.The results show that the number of alleles per locus ranged from 3 to 10 and the mean allelic richness was 5.9; the mean observed heterozygosities (Ho) and expected heterozygosities(He) were 0.518 and 0.681, respectively.The polymorphic information content (PIC) values of all loci, except for two, were above 0.5.A Hardy-Weinberg equilibrium test revealed significant deviation in 4 of the 24 microsatellite loci after sequential Bonferroni corrections (P<0.05).Linkage disequilibrium was not observed between any pair of loci, indicating that the markers were independent.Therefore, this microsatellite isolation method is cost and time effective in comparison to traditional approaches.The SSR markers isolated will be useful in future’s genetic evaluation process of this species,which is essential for a sound management of cuttlefish fishery resources.
Sepiella japonica; transcriptome; EST sequence; microsatellites
Q789; S931
10.11693/hyhz20170300079
* 国家自然科学基金项目, 41406138号; 浙江省自然科学基金项目, LY130190001号; 浙江省水产种质资源高效利用技术研究重点实验室开放课题, KF201-6008号。吕振明, 博士研究生, 教授, E-mail: nblzmnb@163.com
① 通讯作者: 董迎辉, 博士, 副教授, E-mail: 15067427669@126.com
2017-03-29, 收修改稿日期: 2017-04-23
