中国鹿类动物分类及系统进化研究进展
2017-11-17刘汇涛董依萌周永娜邢秀梅杨福合
刘汇涛 董依萌,2 王 磊 周永娜 邢秀梅* 杨福合*
(1.中国农业科学院特产研究所,特种经济动物分子生物学国家重点实验室,长春,130112;2.吉林农业大学,长春,130118)
中国鹿类动物分类及系统进化研究进展
刘汇涛1董依萌1,2王 磊1周永娜1邢秀梅1*杨福合1*
(1.中国农业科学院特产研究所,特种经济动物分子生物学国家重点实验室,长春,130112;2.吉林农业大学,长春,130118)
中国的鹿类资源十分丰富。过去国内外学者对鹿类动物的分类主要通过生物学特征和染色体核型进行研究。近年来,随着分子生物学技术及分子进化理论的快速发展,应用基因组(核、线粒体)对鹿类动物的研究逐渐开展起来,这在一定程度上对传统分类进行了修正和补充。由于传统分类易受主观和各种客观因素的限制(如难以获得足够数量和种类的标本),不同学者利用分子生物学分类使用的基因各不相同,因此,目前鹿类动物的分类和系统进化研究还存在着一些争议和不同的意见,如麝、狍、毛冠鹿等的归属仍没有统一定论,有关麝科、鹿科中亚科之间的系统进化关系还有待于进一步阐明。本文对中国鹿类动物分类和系统进化的研究进展进行综述,并提出展望,旨在为中国鹿类动物的分类及系统进化研究提供参考。
鹿;分类;进化;系统发育
鹿类动物是指反刍亚目的鹿上科(Super Cervidae或Cervoidea),包括麝科(Moschidae)和鹿科(Cervidae)动物。中国拥有丰富的鹿类资源,中国鹿种占全球的41.7%,从古化石鹿类到现生鹿类均为世界上种类最多、最有特色的地区。盛和林等认为中国现存的鹿类动物共21种,其中鹿上科动物20种,鼷鹿上科的鼷鹿(Tragulusjavanicus)1种[1]。吴家炎和王伟认为包括鼷鹿科(Tragulidae)在内的现代鹿类共3科,4亚科,23种,38亚种。麝科在世界上仅产5种,主要分布在中国,据调查,从我国东北部到西南部分布有3~5种麝科动物。鹿科的3个亚科中,獐亚科(Hydropotinae)在世界上仅产1种2亚种,原产地主要分布在中国和朝鲜半岛地区,其中分布区较多的指名亚种(Hydropotesinermisinermis)分布于中国长江流域。麂亚科(Muntiacinae)在世界有8种,中国有6种,所以,麂亚科麂族(Muntiacini)的分布中心也在中国。鹿亚科(Gervinae)共16种,其中7种分布于中国[2]。鹿族(Cervini)4属中,除Dama属以外,其他3属均分布于中国,其中白唇鹿(Cervusalbilostris)是中国特产。鹿属的进化舞台在中国,标志着各种、各亚种的发展进化舞台遍布于整个中国[1]。鹿科中还有1个美洲鹿亚科(Odocoileinae),共有17种,主要产在欧亚北部及美洲新大陆,属于美洲鹿亚科的3个族在中国都有分布,狍(Capreioluscapreolus)、驼鹿(Alcesalces)、驯鹿(Rangifertarandus)3种分布于中国大陆中部以北的地域[2]。此外,作为鹿类祖先型的鼷鹿仍然残存于云南省,表明在中国大陆存在着几乎所有鹿的类群,显示出世界其他地域所不具有的特色。
国内外学者通过传统形态学、细胞生物学、分子生物学对中国鹿类动物的系统发育和起源进化展开了研究,但是,目前还存在着一些争议和不同的意见,如麝科与牛科(Bovidae)、鹿科亲缘关系的远近问题,狍、毛冠鹿(Elaphoduscephalophus)等的归属问题仍没有统一定论,麝科、鹿科中有些种及种下分类单元的系统发育地位仍有待于进一步阐明。本文对中国鹿类动物系统发育的国内外研究进展进行了综述,旨在为中国鹿类动物的分类及起源进化研究提供参考。
1 中国鹿类动物的历史分类
1.1 鹿类动物分类方法的发展
过去,人们对中国鹿类动物起源和系统进化研究主要依据形态学[1,3]和染色体学[4-5]。很多学者根据鹿类动物及其化石的主要生物学特征(角的有无和形态、体型和体重、头骨和炮骨、泪窝和腺体的有无及特征、齿式和毛色特征、行为特征等)进行了传统分类。在形态学方面,研究人员使用的分类性状主要是表型和生理性状,其中颅骨衍生物(牙齿、角等)是系统分类的重要特征之一。盛和林等[1]认为鹿类动物的化石记录虽能较真实地反映其进化历史,但由于化石的分类基准大都使用角或容易变异的骨的形态,因此很容易造成异议。很多学者也认为仅仅通过角来对鹿类动物进行分类很容易受主观因素的影响,而且反刍亚目中有些有蹄类动物可能经受了很强的选择压力,具有性二态的颅骨衍生物是生态和行为的原因[6-7]。Geist等指出偶蹄类的比较形态学在分类和进化研究中并不总是可靠的,因为这些动物的形态学特征随着环境条件变化而产生很大差异,所以结果不能被普遍接受[8]。Emerson 和Tate指出鹿类动物形态学研究所依据的角形和皮毛颜色等特征容易受到趋同(convergence)或平行进化(parallel evolution)的影响[9]。
近30多年来,很多学者在传统分类基础上引入了细胞学和分子生物学以及计算机技术对鹿类动物进行分类研究。王宗仁等利用染色体核型研究了鹿科动物的进化[10]。兰宏等利用RFLP法对麝和麂的分歧时间进行了分析[11]。李明等利用细胞色素b基因片段序列分析了林麝(Mouchusberezovskii)、獐(Hydropotesinermis)、小麂(Muntiacusreevesi)和水鹿(Cervusunicolor)的遗传差异[12]。Su等从分子水平提出麝类为单系群[13]。Gilbert等利用两个线粒体蛋白编码基因和两个核内含子对25个鹿种的系统发生、分类与进化进行了研究[14]。Hassanin等通过分析线粒体全基因组研究了鹿类动物的系统发育[15],Zhang等基于线粒体全基因组对GenBank中已有线粒体全长的19种鹿类物种进行了系统发育和进化分析[16]。刘学东、施燕峰和刘忠权等分别应用线粒体12S rRNA、D-loop区和ATP8 基因研究了狍的归属问题[17-19]。Randi等利用线粒体控制区序列研究了麋鹿的分类地位[20]。Miyamoto等利用12S rRNA的序列研究了獐、麂、鹿及美洲鹿间的关系[21-22]。Kuznetsova等利用线粒体12S rRNA和16S rRNA基因以及核基因β-血影蛋白的区域对鹿科动物的进化关系进行了分析[23]。Guha等通过分析16S rRNA和细胞色素b基因研究了牛科、鹿科、麝科的系统发育地位[24]。分子生物学分类方法在一定程度上对传统分类进行了修正和补充,使鹿类动物的分类更加趋向于精确。
1.2 鹿类动物的几种分类观点
1.2.1 Ohtaishi和盛和林提出的鹿类动物分类
Ohtaishi 和盛和林[1]认为反刍亚目(Ruminantia)的分类系统应该分为鼷鹿上科(Traguloidea)及鹿上科(Cervoidea);在鹿上科下分麝科、鹿科;鹿科下分獐亚科、鹿亚科(Cervinae)、美洲鹿亚科;将鹿亚科、麂亚科合并,降为“族(Tribe)”,分为麂族(Muntiacini)和鹿族(Cervini);在亚科的归属上去掉麂亚科而保留鹿亚科,将两个重要亚科的种属合并在一起(图1)。该系统是依据Groves的系统分类思想,这种分类系统是否适当尚待进一步求证。

图1 中国偶蹄目和鹿上科的分类系统 (引自盛和林,1992)[1]Fig.1 The classification of Artiodactyla and Cervoidea in China (cited from Sheng Helin,1992)[1]
1.2.2 Simpson提出的反刍亚目下的分类系统
在很长一段时间内麝都作为鹿科下的1个亚科(麝亚科,Moschinae)。我国很多动物学专著都沿用了Simpson提出的哺乳动物分类系统,长期以来一直为分类学工作者所用,1971年Simpson又提出了自己的修改方案。随着新资料的积累和发现,特别是分类学本身的新进展,又提出了很多重要的修改意见[25]。与Othaishi分类系统不同的是,该系统将鹿科分为4个亚科,分别为麝亚科、麂亚科、鹿亚科和空齿鹿亚科(Odocoileinae),麝亚科位于鹿科之下而并没有与鹿科并列为麝科,麂升为亚科与鹿亚科并列,獐亚科降为獐族(Hydropotini),位于空齿鹿亚科之下(图2)。

图2 反刍亚目下的分类系统(引自Simpson,1945)[25]Fig.2 The classification of Ruminantia(cited from Simpson,1945)[25]
1.2.3 近10年利用分子生物学方法提出的鹿类动物分类
Gilbert[14]利用2个线粒体蛋白编码基因和2个核基因内含子对25个鹿种的系统发生、分类与进化进行了研究,为了使分类和系统发育相一致,其提出了一种新的分类方法,将鹿科动物分为2个亚科和5个族,一个为鹿亚科,包括鹿族和麂族2个族,鹿族分为鹿属(Cervus)、轴鹿属(Axis)、黇鹿属(Dama)和泽鹿属(Rucervus),其中将麋鹿(Elaphurusdavidianus)并入了鹿属,将沼泽鹿(Cervusduvauceli)并入了泽鹿属,麂族分为麂属(Muntiacus)和毛冠鹿属(Elaphodus);另一个为狍亚科(Capreolinae),包括狍族(Capreolini)、驼鹿族(Alcini)和空齿鹿族(Odocoileini),其中将狍属(Capreolus)和獐属(Hydropotes)并入了狍族,将驯鹿属(Rangifer)和美洲鹿属(Americangenera)并入了空齿鹿族(图3)。Hassanin等通过分析线粒体全基因组研究了偶蹄目(Artiodactyla)动物的系统发育树[15](图4),其构建的系统发育树强烈支持Gilbert提出的分类方法,Zhang等基于线粒体全基因组对GenBank中已有线粒体全长的19种鹿类物种进行了系统发育和进化分析,结果系统发育树支持鹿科动物为单系群,鹿科被分为Plesiometacarpalia和Telemetacarpalia两个亚科,以及鹿、麂、獐和空齿鹿4个族[16]。

图3 基于线粒体基因和核基因构建并合成的鹿科动物系统发育树(引自Gilbert,2006)[14]Fig.3 Synthetic tree of the family Cervidae based on mitochondrial and nuclear gene sequences(cited from Gilbert,2006)[14]
2 鹿类动物分类仍然存在的争议
2.1 麝的分类地位
麝(Moschusspp.)是偶蹄目反刍亚目(Ruminantia)新反刍下目(Pecora)中的小型有蹄类动物,是亚洲地区特有类群。过去许多国外学者根据局部地区的少量标本的研究发表了多种观点。中国学者是在20世纪50年代后期才开始研究讨论麝的分类问题。
麝曾被作为鹿科的1个亚科存在,Zittel(1893年)首次提出麝亚科[26],随后Simpson[25]再次确认麝为鹿科的1个亚科,高耀亭和Romer也支持此观点[27-28]。Strirton(1944年)最早提出麝科[26],Flerov通过麝的形态学研究指出麝应为反刍目动物中1个独立的科[29],Corbet等[30]认为应将麝另立一个独立的科-麝科,并得到众多学者的认可[1,3,31-32]。在形态学上麝作为1个独立的科比较明确,在分子生物学上也有许多证据可以印证。兰宏等利用RFLP法对麝和麂的mtDNA进行了分析得出麝和麂是在600万年前后开始分歧的[11]。李明对线粒体DNA上得到367 bp的细胞色素b基因序列分析得出麝与獐、麂和鹿的遗传差异在12.53%~14.44%之间处在科间变化范围之内,在分子水平上进一步表明麝应作为一个独立科[12]。Su等进一步从分子水平提出麝类为单系群[13],支持麝类独立为科。
目前麝科作为独立科的观点占据优势,但是持有两种观点的分类学者都承认麝与鹿科动物相比,具有一个非常特殊的位置。麝没有角,却生有一个麝香腺,麝有胆囊,其他鹿种却没有,它们的腿与其他鹿种也不相同。
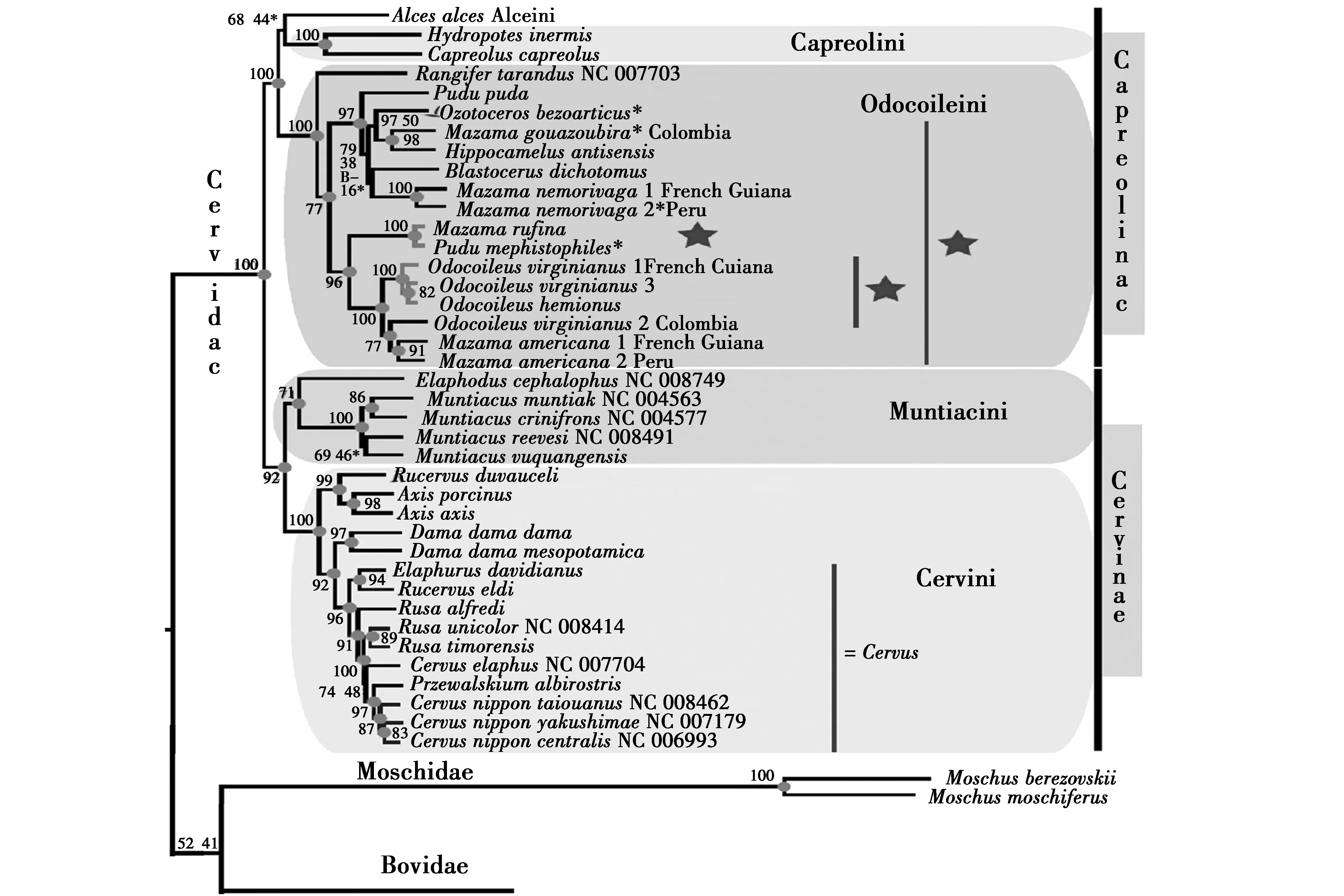
图4 基于线粒体基因组构建的偶蹄目动物系统发育树(引自Hassanin A,2012)[15]Fig.4 Phylogeny of Cetartiodactyla based on complete mitochondrial genomes(cited from Hassanin A,2012)[15]
2.2 麝属内种的分类
现生麝科动物仅有1属尚无争议,但因为麝科动物的外形极其相似,所以其种及种下划分一直存在争议。目前主要存在以下3种观点。
第一种观点:麝属仅有1种。许多学者支持麝属仅1种的观点,最初Lyedkker[26](1915年)认为原麝(Moschusmoschiferus)为1种,3个亚种。陈服官等[33]根据众多的标本,从颅全长、体色以及按照种与亚种的划分理论将我国的麝定为1种,即原麝。Sokolov和Prikhod’ko[34]根据麝科在细胞核型上无明显的种间差异,头骨属渐变群变异,控制色型的基因十分相似等依据,又支持了这一观点。该观点将麝分为2个类群,7个亚种。
西伯利亚类群:
指名亚种M.m.moschiferus
锡霍特亚种M.m.turowi
北极亚种M.m.arcticus
萨哈林亚种M.m.sachalinesis
喜马拉雅类群:
远东亚种M.m.parvipes
高山麝亚种M.m.chrysogaster
喜马拉雅亚种M.m.leucogaster
第二种观点:麝属有4种或6种。高耀亭和Romer[27-28]、Flerov[29]根据外形、头骨和距骨的形态特点毛被结构和色型,结合地理分布和生态学资料等确定中国的麝是3个独立的种,分别为原麝、林麝(Moschusberezovskii)和马麝(Moschuschrysogaster)。蔡桂全和冯祚建[35]从体色和头骨特征上又提出将喜马拉雅麝(Moschusleucogaster)定为一独立种,李致祥[36]发表了黑麝(Moschusfuscus)。李明等和Su等根据分子系统学方法将安徽麝提升为独立的种[13,37]。Groves等[31]根据形态学认为麝属可分为5种或6种。至此,麝属被认为有6种。但是Ohatishi和Gao认为黑麝和喜马拉雅麝可能是马麝的亚种[38],由于二者的标本不足,其分类地位有待于进一步研究。
原麝MoschusmoschiferusLinnaeus,1758
指名亚种M.m.moschiferusLinnaeus,1758
东北亚种M.m.parvipesHollister,1911
林麝M.beerzovskiiFlerov 1929
指名亚种M.b.beerzovskiiFlerov,1929
云贵亚种M.b.yunguiensisWang et Ma,1993
滇西北亚种M.b.bijiangensisWang et Li,1993
马麝(高山麝)M.chrysogasterHodgson,1839
黑麝M.fuscusLi,1981
喜马拉雅麝M.leucogasterHodgson,1839
安徽麝M.anhuiensisWang,Hu & Yan,1982
第三种观点:麝属分为3种。盛和林等通过分析吻长、鼻骨长及泪骨缝长认为中国麝至多可分4个种,即马麝、黑麝、喜马拉雅麝及原麝。因为林麝和原麝的头骨结构非常一致,无明显差异,二者的分布区自北至南没有重叠,个体自北向南逐步小型化,而且2种麝的染色体数目相等,染色体G带高度一致,所以认为林麝只能作为原麝的1个亚种[1]。盛和林和刘志宵通过对麝类颅骨外形、骨片形态、度量和比例等各种性状的比较研究,运用主成分分析和判别分析,并结合前人研究资料和分子生物学数据,对麝科的分类作了进一步研讨后认为中国麝类均可区分为高山麝型和原麝型2个类群,原麝群包括林麝和原麝2种,高山麝(Moschuschrysogaster)群为单型种,共分为3种。盛和林修改了原来认为林麝为原麝的1个亚种的主张,并认为原麝和林麝之间存在一定的分化,可被识别为2个近缘种,安徽麝和黑麝并不是独立的种,而应属于林麝,喜马拉雅麝趋向于高山麝型[26]。Groves和冯祚建[39]、Corbet等[30]以及Wang 等[32]从毛皮质地、体色、体型大小、头骨大小、眼框形状以及栖息环境等特征也将安徽大别山麝定为林麝的1个亚种。
原麝类群:
原麝MoschusmoschiferusLinnaeus,1758
指名亚种M.m.moschiferusLinnaeus,1758
东北亚种M.m.parvipesHollister,1911
林麝MoschusberezovskiiFlerov,1929
川西亚种(指名亚种)M.b.beerzovskiiFlerov,1929
安徽亚种M.b.anhuiensisWang,Hu & Yan,1982
云贵亚种M.b.yunguiensisWang et Ma,1993
滇西北亚种M.b.bijiangensisWang et Li,1993
越北亚种M.b.caobangisDao,1969
高山麝类群Moschuschrysogaster
横断山亚种M.c.sifanicusBuechner,1891
甘肃亚种(暂定)M.c.cansus
喜马拉雅麝M.c.leucogasterHodgson,1839
2.3 狍的分类地位
王宗仁等通过分析染色体核型认为狍的性染色体与空齿鹿亚科的其他属相似,但与鹿亚科的明显不同[10]。因此,虽然狍的分布很广,几乎占据东半球的北温带,但细胞遗传学与形态分类方面的结果一致,清楚地表明它应该归入分布在北美和南美的空齿鹿亚科。Douzery等利用线粒体控制区研究指出狍与獐的关系很近,且推测出二者的分化时间在8.7~10.4百万年前[40]。刘学东利用线粒体12S rRNA基因对中国鹿类动物的系统发育进行研究表明:獐与鹿亚科的种间距离为7.69%~8.22%;与鹿亚科的鹿、毛冠鹿的距离为7.82%和9.53%;与美洲鹿亚科的驯鹿、驼鹿的距离为6.99%和8.63%。有趣的是,狍与獐的距离很近,为6.39%和6.51%,小于狍与同亚科的驼鹿(8.73%、8.68%)及驯鹿的距离(8.03%、8.15%),而且在12S rRNA分子系统树中獐与狍形成1个单系群,与细胞色素b的分子系统树具有同样的拓扑结构。因此,刘学东建议将狍从美洲鹿亚科转入獐亚科,立为其中的1个独立的属(狍属)[17]。施燕峰等通过对鹿科动物线粒体控制区序列分析与系统进化研究表明獐与狍聚为一支[18]。刘忠权等通过对线粒体ATP8基因分析也建议狍应从美洲鹿亚科转入獐亚科[19]。
2.4 毛冠鹿的分类地位
毛冠鹿别名青麂,体型似麂,很像黑麂(Muntiacuscrinifrons),但额顶部有马蹄形黑色冠毛(黑麂为棕色)[1]。盛和林等把毛冠鹿和麂类归到鹿亚科中,但很多古生物学者把麂亚科作为独立的1个亚科,并把毛冠鹿包含到该亚科中[41]。事实上,毛冠鹿正如形态学上的特点一样,在线粒体16S rRNA 基因水平上也是介于鹿类和麂类与獐和狍之间,比鹿类和麂类稍为原始一些(自信值为49%)[42],而线粒体ATP和12S rRNA基因分析认为毛冠鹿介于鹿类与麂类之间,同样自信值低于50%[17,19]。因此,无论从形态学或分子生物学上,提示毛冠鹿的地位都有其特殊性,至于到底将其是归于鹿亚科还是麂亚科,还需要更多的研究样本和更多的遗传信息。
2.5 鹿亚科内属的分类
王宗仁等认为黇鹿属和斑鹿属在染色体核型方面的差异并不比现存鹿属内各个种间的差异更大[10],这似乎支持应将这2个属归为鹿属。
Emerson等认为鹿亚科动物不是单源的,鹿属分为2枝,一枝包含北美马鹿(英文名Wapiti,拉丁名Cervuselaphus)、欧洲赤鹿(英文名Red deer,拉丁名Cervuscanadiensis)和梅花鹿(Cervusnippon),另一枝包含水鹿(英文名Sambar deer,拉丁名Cervusunicolor)和鬣鹿(又名爪哇鹿,英文名Rusa deer,拉丁名Cervustimorensis)。麋鹿与赤鹿、马鹿和梅花鹿的遗传关系较近,水鹿和鬣鹿与豚鹿(Axisporcinus)属和斑鹿(Cervusaxis)属成员间的关系比二者与所在同一属其他成员间的关系更近[9]。
Grubb和Geist等认为麋鹿是独立的属[43-44],但有学者认为麋鹿应该并入鹿属[9,45-46]。Randi等利用线粒体控制区序列对25种鹿科动物的系统发育关系进行分析后也认为麋鹿应该并入鹿属[20]。刘向华等根据线粒体Cytb基因分析认为斑鹿属可能并非单系发生,应将豚鹿并入鹿属,麋鹿属与鹿属有较近的进化关系,也应并入鹿属,归并后的鹿属为单系发生[47]。
Polziehn等利用线粒体DNA对欧洲赤鹿和北美/亚洲马鹿进行了系统发育分析[48],通过简约法和距离法2种分析方法表明赤鹿和马鹿起源于单一祖先,但用最大似然法分析表明,赤鹿与梅花鹿和马鹿形成姐妹群,线粒体细胞色素b和控制区的分析结果是一致的,因此,建议重新考虑马鹿的分类地位。
3 鹿类动物进化关系研究
3.1 麝、麂、鹿、獐间的进化关系
兰宏等利用RFLP 法分析认为麝和麂在600万年左右开始分歧[11]。Miyamoto等利用12S rRNA基因对獐、麂、鹿及美洲鹿间的关系研究发现,麂和鹿的关系最近并组成姐妹群[21-22]。李明等的研究结果也认为麂与鹿的关系最近,而与麝的关系最远,且麝是最先分化出来的,麂和鹿是最后分化的[12]。根据哺乳动物细胞色素b进化速度每百万年为2.5%[49]可知,麝科与鹿科的分歧时间在500~600万年左右,而鹿科的3个亚科间的分歧时间在350~500万年间,并且麂与鹿是最后分化的,这同时说明了鹿与麂的亲缘关系最近,与利用12S rRNA基因的研究结果一致[22]。古生物学家通过鹿类化石研究也持相似观点,认为现生鹿类动物中麝继承了古鹿类的原始特征,是最原始的,獐是麂的祖先型,而鹿是最后发展起来的一个类群[1,27]。从角的进化来看,在现生鹿类动物中角是从无到有且逐渐扩大的[50],现生獐虽然无角,但在其大型化过程中有角,只是在小型化时期失去了角[28],其祖先可能是中新世前期欧洲的Dremotherium[1]。因此从角的有无来看,獐比麝进步,而麂和鹿则是进化程度较高的。李明等认为麝最先从鹿类动物的祖先种分化出来,其次是獐、麂与鹿组成姐妹群,它们共同组成了一个单系群,在600万年前它们有一共同祖先[12]。
3.2 麝科与鹿科、牛科的进化关系
在麝科与鹿科、牛科的进化关系问题上,大多数形态学家认为麝科与鹿科的亲缘关系更近[3,51-55]。Su等[13]利用细胞色素b基因全长研究了麝科动物的系统发育,结果表明,麝科动物为单源群,其与鹿科动物的关系比与牛科动物的关系更近。
目前有很多学者与传统形态分类持相反的观点,认为麝科与牛科的亲缘关系比与鹿科的更近,而且多数分子证据也支持这种观点。Hassanin等[54]研究反刍动物系统发育时指出,麝科、牛科、鹿科共聚为一支,但麝与牛的距离比麝与鹿的距离更近。Kuznetsova等对鹿科动物及其他偶蹄动物进行系统发育分析认为,鹿科与牛科为姐妹群,麝科应该归到牛上科[23]。Fernández和Vrba对现生以及新近绝灭的共197 种反刍动物进行研究认为新反刍下目分为3 个总科:长颈鹿总科Giraffoidea、鹿总科Cervoidea和牛总科Bovoidea,其中,鹿总科则包括鹿科和麝科,支持麝与鹿有更近的亲缘关系[56]。Guha等也认为麝科与牛科的距离比麝科与鹿科的距离更近[24]。Agnarsson和May-Collado以奇蹄目动物为外群,用Cytb基因对包括麝及2种新近绝灭的物种(Mouse Goat,Irish Elkor Giant Deer)共266种偶蹄目动物的系统进化关系研究发现,牛科与麝科为姐妹群,支持麝与牛科的进化关系比与鹿科的更近[57]。Peng等[58]测定了林麝全线粒体序列,并分别运用MP、Bayesian 2种方法对牛科、鹿科、麝科3个类群共23个物种的系统发生关系进行分析,结果支持麝科为鹿科的姐妹群。Hassanin和Vislobokova等通过分析线粒体全基因组研究认为麝科与牛科的亲缘关系比麝科与鹿科的亲缘关系更近[15,59]。Cernohorska等[60]通过细胞学染色体荧光原位杂交技术也进一步证明麝科与牛科的亲缘关系比麝科与鹿科的亲缘关系更近。
还有一些学者认为麝科是三者中最原始的进化枝,是牛科和鹿科二者共同的姐妹群。Hassanin和Douzery[54]以鼷鹿科为外群,应用48个形态学特征重新分析了包括麝科动物在内的新反刍下目5个类群23个物种的系统进化关系,结果表明,麝科与鹿科和牛科的系统进化呈现一种三分的进化关系:麝科或者与鹿科的亲缘关系近,或者与牛科的亲缘关系近。O’Leary和Gatesy应用635个表型性状数据分析了包括现生种及化石种在内的偶蹄目动物的系统进化,研究结果与上述麝科、鹿科、牛科间的系统进化关系不同,在5个类群中,麝科为最原始的类群,最先分歧,然后依次是鹿科和叉角羚科,牛科与长颈鹿科的关系最近,叉角羚科是它们二者的姐妹群[55]。
3.3 鹿属动物进化关系
王宗仁等[5]认为现生鹿属动物中水鹿是最原始的,而白唇鹿与梅花鹿几乎同时从水鹿的祖先种分化而来的,马鹿则是最后分化出来的。大泰司纪之根据形态特征研究指出水鹿和坡鹿(Rucervuseldii)是近缘的,白唇鹿的祖先是水鹿,梅花鹿和马鹿是近缘种,且马鹿是从梅花鹿中分化出来的[1]。李明等[37]利用DNA序列分析中国4种鹿属动物的系统进化和分歧时间得出,在鹿属动物的系统进化上梅花鹿与马鹿的关系最近,且它们组成一个单系群,而水鹿是最原始的。水鹿与坡鹿、马鹿和梅花鹿的分歧时间在240~280万年前左右;梅花鹿和马鹿是在160万年前左右分歧的,与Cook[61]得出的梅花鹿和马鹿分歧时间为70~190万年前的结论基本一致。从它们的分歧时间来看大约是在更新世期间,这也基本上符合根据古生物学和古地理学以及细胞遗传学研究得出在上新世末期至更新世初期的结论[5,50]。
Cook等利用线粒体细胞色素b和控制区基因研究了中国东北梅花鹿、日本本州岛北部梅花鹿、日本本州岛南部梅花鹿、越南梅花鹿、台湾梅花鹿系统发育,支持梅花鹿是单源的[62]。
Ludt等利用细胞色素b基因研究了马鹿的系统地理学,将世界马鹿分为3个种群,分别为西部种群(包括西欧、巴尔干、中东和非洲)、东部种群(包括亚洲美洲北部、南亚和东亚)和一个分布在中亚地区的原始种群,并认为马鹿起源于今天兴都库什山脉附近的中亚地区[63]。
4 小结
偶蹄目中的鹿科动物种数不太多,但现存的鹿种却能表现它几乎全部系统发生的阶段。鹿类动物的系统学研究对我们认识进化的规律是一个无可取代的珍贵实验材料。中国的鹿类资源十分丰富,从鹿类进化观点来看,中国大陆被称作“鹿类进化的展览场”,是一个极其珍贵的鹿类进化的研究地,在中国研究鹿类动物的系统发育问题拥有良好的条件。但是,目前中国鹿类动物的分类和系统进化还存在着一些争议,尽管近些年很多学者通过分子生物学方法对传统分类进行了一定的修正和补充,但毕竟有些研究只是利用线粒体或者核基因的部分序列来研究分析,而且样本不全面和方法不统一问题导致不同学者使用同一片段所获得试验结果也不尽一致。因此,利用更加全面的鹿类动物资源样本,通过线粒体DNA全序列甚至全基因组,结合古生物学和动物地理学等来综合分析是研究中国鹿类动物系统发育的一个发展方向。
[1] 盛和林.中国鹿类动物[M].上海:华东师范大学出版社,1992:1-251.
[2] 吴家炎,王伟.中国白唇鹿[M].北京:中国林业出版社,1999:31-68.
[3] Groves C P,Grubb P.Relationships of living deer in biology and management of the Cervidae[M].Washington DC:Smithsonian Institution Press,1987.
[4] Neitzel H.Chromosome evolution of Cervidae karyotypic and molecular aspects[M]//Obe G,Basler A.Cytogenetics.Berlin:Springer-Verlag,1987:90-112.
[5] 王宗仁,杜若甫.鹿属动物的染色体组型及其进化[J].遗传学报,1982,9(11):24-31.
[6] Scott K M,Janis C M.Relationships of the ruminantia(Artiodactyla)and an analysis of the characters used in ruminant taxonomy[J].Mammal Phylogeny,1993,2:282-302.
[7] Janis C M.The interrelationship of higher ruminant families:with special emphasis on the member of the Cervoidea[J].American Museum Novitates,1987,2893:1-85.
[8] Geist V.Deer of the world:their evolution,behaviour,and ecology[M].Stackpole Books,1998.
[9] Emerson B C,Tate M L.Genetic analysis of evolutionary relationships among deer(Subfamily Cervinae)[J].Journal of Heredity,1993,84(4):266-273.
[10] 王宗仁,杜若甫.鹿科动物的染色体组型及其进化[J].动物学报,1983,29(3):214-222.
[11] 兰宏,施立明.麂属(Muntiacus)动物线粒体DNA多态性及遗传分化[J].中国科学(B辑),1993,23(5):489-497.
[12] 李明,盛和林,玉手英利,等.麝、獐、麂和鹿间线粒体DNA的差异及其系统进化研究[J].兽类学报,1998,18(3):184-191.
[13] Su B,Wang Y X,Lan H,et al.Phylogenetic study of complete cytochromebgenes in musk deer(GenusMoschus)using museum samples[J].Molecular Phylogenetics and Evolution,1999,12(3):241-249.
[14] Gilbert C,Ropiquet A,Hassanin A.Mitochondrial and nuclear phylogenies of Cervidae(Mammalia,Ruminantia):systematics,morphology,and biogeography[J].Molecular Phylogenetics and Evolution,2006,40(1):101-117.
[15] Hassanin A,Delsuc F,Ropiquet A,et al.Pattern and timing of diversification of Cetartiodactyla(Mammalia,Laurasiatheria),as revealed by a comprehensive analysis of mitochondrial genomes[J].Comptes Rendus Biologies,2012,335(1):32-50.
[16] Zhang W Q,Zhang M H.Phylogeny and evolution of Cervidae based on complete mitochondrial genomes[J].Genetics and Molecular Research:GMR,2012,11(1):628-635.
[17] 刘学东.中国鹿类动物线粒体12S rRNA基因序列的比较分析及分子系统发育研究[D].哈尔滨:东北林业大学,2003.
[18] 施燕峰,单祥年,李健,等.鹿科动物线粒体控制区序列分析与系统进化[J].遗传学报,2004,31(4):395-402.
[19] 刘忠权,韩梅,黄建伟.麋鹿的线粒体ATP8基因及分类地位探讨[J].盐城师范学院学报:自然科学版,2008,26(1):126-131.
[20] Randi E,Mucci N,Claro‐Hergueta F,et al.A mitochondrial DNA control region phylogeny of the Cervinae:speciation inCervusand implications for conservation[J].Animal Conservation Forum,2001,4(1):1-11.
[21] Miyamoto M M,Kraus F,Ryder O A.Phylogeny and evolution of antlered deer determined from mitochondrial DNA sequences[J].Proceedings of the National Academy of Sciences,1990,87(16):6127-6131.
[22] Miyamoto M M,Kraus F,Laipis P J,et al.Mammal phylogeny[M].Heidelberg :Springer-Verlag,1993:268-281.
[23] Kuznetsova M V,Kholodova M V,Danilkin A A.Molecular phylogeny of deer(Cervidae:Artiodactyla)[J].Genetika,2005,41(7):910-918.
[24] Guha S,Goyal S P,Kashyap V K.Molecular phylogeny of musk deer:a genomic view with mitochondrial 16S rRNA and cytochromebgene[J].Mol Phylogenet Evol,2007,42(3):585-597.
[25] Simpson G G.The principles of classification and a classification of mammals[J].American Museum of Natural History,1945,85(3):1-307.
[26] 盛和林,刘志霄.中国麝科动物[M].上海:上海科学技术出版社,2007.
[27] 高耀亭.中国麝的分类[J].动物学报,1963,15(3):479-487.
[28] Romer A S.Vertebrate paleontology[M].Chicago:University of Chicago Press,1966.
[29] Flerov K K.Musk deer and deer[M]// Fauna of the USSR.Mammals.Moscow:The Israel Program for Scientific Translations,1952.
[30] Corbet G B,Hill J E.The mammals of the indomalayan region:a systematic review[M].Natural History Museum Publications,Oxford University Press,1992:252-253.
[31] Groves P C,Wang Y,Grubb P.Taxonomy of musk-deer,genus Moschus(Moschidae,Mammalia)[J].Acta Theriologica Sinica,1995,15(3):181-197.
[32] Wang Y X,Ma S L,Li C Y.The taxonomy,distribution and status of forest musk.deer in China[M].Elsevier Science Publishers,1993:126-134.
[33] 陈服官,闵芝兰,黄洪富,等.陕西省秦岭大巴山地区兽类分类和区系研究[J].西北大学学报:自然科学版,1980(1):137-147.
[34] Sokolov V E,Prikhod’ko V I.Taxonomy of the musk deer(Artiodactyla,Mammalia)[J].Izv Akad Nauk Ser Biol,1998(1):37-46.
[35] 蔡桂全,冯祚建.喜马拉雅麝在我国的发现及麝属的分类探讨[J].动物分类学报,1981,6(1):106-110.
[36] 李志祥.中国麝一新种的记述[J].动物学研究,1981,2(2):157-161.
[37] 李明,王小明,盛和林,等.四种鹿属动物的线粒体DNA差异和系统进化关系研究[J].动物学报,1999,45(2):99-105.
[38] Ohtaishi N,Gao Y.A review of the distribution of all species of deer(Tragulidae,Moschidae and Cervidae)in China[J].Mammal Review,1990,20(2/3):125-144.
[39] Groves,冯祚建.安徽省麝的分类地位[J].兽类学报,1986(2):105-106.
[40] Douzery E,Randi E.The mitochondrial control region of Cervidae:evolutionary patterns and phylogenetic content[J].Mol Biol Evol,1997,14(11):1154-1166.
[41] Webb S D,Taylor B E.The phylogeny of hornless ruminants and a description of the cranium of Archaeomeryx[M].American Museum of Natural History,1980,167:117-158.
[42] 刘忠权.基于线粒体16S rRNA基因探讨鹿科动物系统发生关系[J].四川动物,2010,29(5):509-512.
[43] Grubb P.Artiodactyla:Cervidae.In mammal species of the World[M].Washington,DC:Smithsonian Institution Press,1993.
[44] Geist V.Deer of the World.Their evolution,behavior and ecology[M].Mechanicsburg,PA:Stackpole Books,1998.
[45] Cronin M A.Mitochondrial-DNA phylogeny of deer(Cervidae)[J].Journal of Mammalogy,1991,72(3):553-566.
[46] Cronin M A,Stuart R,Pierson B J,et al.K-casein gene phylogeny of higher ruminants(Pecora,Artiodactyla)[J].Mol Phylogenet Evol,1996,6(2):295-311.
[47] 刘向华,王义权,刘忠权,等.从cytb基因序列探讨鹿亚科动物的系统发生关系[J].动物学研究,2003,24(1):27-33.
[48] Polziehn R O,Strobeck C.A phylogenetic comparison of red deer and wapiti using mitochondrial DNA[J].Mol Phylogenet Evol,2002,22(3):342-356.
[49] Meyer A,Wilson A C.Origin of tetrapods inferred from their mitochondrial DNA affiliation to lungfish[J].Journal of Molecular Evolution,1990,31(5):359-364.
[50] Flerov K K.Morphology and ecology of Cervidae during its evolution[J].J.Paleontology Translation,1957,1(2):2-16.
[51] Matthew W D,Colbert E H.A phylogenetic chart of the Artiodactyla[J].Journal of Mammalogy,1934,15(3):207-209.
[52] Leinders J J M,Heintz E.The configuration of the lacrimal orifices in pecorans and tragulids(Artiodactyla,Mammalia)and its significance for the distinction between Bovidae and Cervidae[J].Beaufortia,1980,30(7):155-160.
[53] Janis C M,Scott K M.The phylogeny of the Ruminantia(Artiodactyla,Mammalia)[M]//The Phylogeny and Classification of the Tetrapods,Mammals.Oxford UK:Clarendon Press,1988.
[54] Hassanin A,Douzery E J P.Molecular and morphological phylogenies of Ruminantia and the alternative position of the Moschidae[J].Systematic Biology,2003,52(2):206-228.
[55] O’Leary M A,Gatesy J.Impact of increased character sampling on the phylogeny of Cetartiodactyla(Mammalia):combined analysis including fossils[J].Cladistics,2008,24(4):397-442.
[56] Fernández M H,Vrba E S.A complete estimate of the phylogenetic relationships in Ruminantia:a dated species-level supertree of the extant ruminants[J].Biological Reviews,2005,80(2):269-302.
[57] Agnarsson I,May-Collado L J.The phylogeny of Cetartiodactyla:the importance of dense taxon sampling,missing data,and the remarkable promise of cytochromebto provide reliable species-level phylogenies[J].Mol Phylogenet Evol,2008,48(3):964-985.
[58] Peng H,Liu S,Zeng B,et al.The complete mitochondrial genome and phylogenetic analysis of forest musk deer(Moschusberezovskii)[J].Journal of Natural History,2009,43(19/20):1219-1227.
[59] Vislobokova I A,Lavrov A V.The earliest musk deer of the genusMoschusand their significance in clarifying of evolution and relationships of the family Moschidae[J].Paleontological Journal,2009,43(3):326-338.
[60] Cernohorska H,Kubickova S,Kopecna O,et al.Molecular cytogenetic insights to the phylogenetic affinities of the giraffe(Giraffacamelopardalis)and pronghorn(Antilocapraamericana)[J].Chromosome Res,2013,21(5):447-460.
[61] Cook C E.The historical biogeography and phylogeny of sika deer(Cervusnippon)in East Asia[D].Berkeley:University of California,1993:141.
[62] Cook C E,Wang Y,Sensabaugh G.A mitochondrial control region and cytochrome b phylogeny of sika deer(Cervusnippon)and report of tandem repeats in the control region[J].Mol Phylogenet Evol,1999,12(1):47-56.
[63] Ludt C J,Schroeder W,Rottmann O,et al.Mitochondrial DNA phylogeography of red deer(Cervuselaphus)[J].Mol Phylogenet Evol,2004,31(3):1064-1083.
Research Progress on Taxonomy and Phylogeny of Deer in China
Liu Huitao1Dong Yimeng1,2Wang Lei1Zhou Yongna1Xing Xiumei1*Yang Fuhe1*
(1.State Key Laboratory for Molecular Biology of Special Animals,Institute of Special Animal and Plant Sciences,Chinese Academy of Agricultural Sciences, Changchun,130112,China;2.Jilin Agricultural University,Changchun,130118,China)
The resources of deer species are very rich in China.In the past,the classification of deer was mainly based on biological characteristics and chromosome karyotype.In recent years,with the development of molecular biology techniques and the theory of molecular evolution,the application of the genome(nuclear,mitochondrion)research of deer has been carried out,which partly modified and supplemented the traditional classification.The traditional classification is subject to the limitation of subjective and objective factors such as insufficient numbers of specimens and species.Different researchers classify deer based on different genes in study.Therefore,many problems remain concerning the classification and evolution of China’s deer,such as the taxonomy of musk deer,roe deer,and tufted deer,and the phylogeny of sub families of Moschidae and Cervidae.We reviewed the taxonomy and phylogeny of China’s deer in this paper.This review will serve as a reference for future research of the taxonomy and phylogeny of China’s deer.
Deer;Taxonomy;Evolution;Phylogeny
稿件运行过程
2016-11-07
修回日期:2017-02-27
发表日期:2017-08-10
S858.25 Q343
A
2310-1490(2017)03-514-10
中国农业科学院科技创新工程(CAAS-ASTIP-2017-ISAPS);特种动物种质资源共享平台(201601)
刘汇涛,男,32岁,助理研究员;主要从事鹿类动物遗传资源研究。E-mail:liuhuitao0508@126.com
*通讯作者:邢秀梅, E-mail:xingxiumei2004@126.com;杨福合,E-mail:yangfh@126.com
