变异链球菌低pH感应系统的构建
2017-06-19康迪李雨庆周学东
康迪 李雨庆 周学东
口腔疾病研究国家重点实验室,国家口腔疾病临床研究中心,四川大学华西口腔医院,成都 610041
·基础研究·
变异链球菌低pH感应系统的构建
康迪 李雨庆 周学东
口腔疾病研究国家重点实验室,国家口腔疾病临床研究中心,四川大学华西口腔医院,成都 610041
目的 构建变异链球菌低pH感应系统,原位可视地检测其所处环境的pH。方法 通过聚合酶链反应(PCR)和基因重组技术,克隆得到唾液链球菌57.I的ureⅠ基因启动子片段(pureⅠ)及绿色荧光蛋白(GFP)的编码基因gfp,经过酶切后将两个基因片段连接融合,然后再经双酶切将融合片段与大肠杆菌-变异链球菌穿梭载体pDL278连接,构建出重组质粒pDL278-pureⅠ-gfp。将重组质粒转化到变异链球菌UA159中,使用荧光显微镜观察其在不同pH条件和不同处理时间的单位面积荧光强度。结果 目的基因pureⅠ和gfp扩增片段大小分别为450 bp和717 bp,与预期大小相符。构建的重组质粒pDL278-pureⅠ-gfp测序结果与数据库比对完全一致。重组变异链球菌低pH报告菌株PCR扩增片段与预期结果相符。在一定范围内,变异链球菌低pH感应系统单位面积荧光强度随pH值的降低和处理时间的延长而增强。结论 本研究成功构建了变异链球菌低pH感应系统,同时验证了唾液链球菌酸诱导启动子pureⅠ能在变异链球菌中正常发挥功能,为今后研究菌斑生物膜中原位pH的动态变化提供了新方法。
变异链球菌; 唾液链球菌; 绿色荧光蛋白; 尿素酶; 启动子
龋病是口腔常见的感染性疾病之一。牙菌斑生物膜的形成是龋病形成的首要因素,而变异链球菌则是牙菌斑中主要的致龋菌之一[1-2]。变异链球菌等产酸细菌通过分解糖类产酸,降低菌斑局部pH使牙面脱矿,最终形成龋损[3]。这些酸性代谢产物是造成牙齿脱矿最直接的原因[4]。目前,检测菌斑基质中pH值主要采用pH染料、微电极等方法,但这些测量方法常为非原位测量的方法,易带入其他混杂因素[4-5]。
唾液链球菌是口腔中的主要产碱菌,在酸性环境中可被诱导产碱,以调节环境pH[6-7]。产生此特性的分子机制是:在高pH环境下,CodY蛋白与尿素酶基因ureⅠ启动子(promoter of ureaseⅠ,pureⅠ)结合,阻遏下游基因表达;在低pH环境下,CodY蛋白从pureⅠ上解离,启动下游基因表达[6]。绿色荧光蛋白(green fluores-cence protein,GFP)常作为报告基团,用于监控细胞组织动态变化、基因表达、蛋白定位等[8]。本研究拟将pureⅠ与GFP编码基因gfp连接并转入变异链球菌中,构建变异链球菌低pH感应系统,从而原位指示变异链球菌所处环境的pH状态,以准确探讨菌斑生物膜中原位pH的动态变化。
1 材料和方法
1.1 材料
大肠杆菌-变异链球菌穿梭载体pDL278(四川大学口腔疾病研究国家重点实验室提供);限制性核酸内切酶BamHⅠ、SacⅠ和SalⅠ,T4 DNA连接酶(Takara公司,日本),KOD-Plus DNA聚合酶(Toyobo公司,日本);细菌基因组DNA抽提试剂盒、DNA凝胶回收试剂盒、柱式质粒小量抽提试剂盒(北京天根生化科技有限公司);其余试剂均为国产或进口分析纯试剂。实验引物合成及DNA序列测定由上海生工生物工程股份有限公司完成。
1.2 菌种和培养基
唾液链球菌57.I、变异链球菌UA159、含有GFP编码基因gfp的变异链球菌菌株(四川大学口腔疾病研究国家重点实验室保存),大肠杆菌DH5α感受态细胞(北京天根生化科技有限公司);脑心浸液(brain heart infusion,BHI)培养基及LB(Luria-Bertani)培养基(Sigma公司,美国)。50%BHI培养基配制方法:将BHI培养基与无菌水等体积混合,调节pH=7.5,使用0.22 μm孔径滤器过滤。
1.3 引物
根据唾液链球菌基因组序列设计并合成pureⅠ特异性引物,其中上游引物包含SacⅠ的限制性酶切位点,下游引物包含BamHⅠ的限制性酶切位点;根据Clonetech公司的pGFP载体序列设计并合成gfp基因特异性引物,其中上游引物包含BamHⅠ的限制性酶切位点,下游引物包含SalⅠ的限制性酶切位点。引物序列如表1所示。

表 1 pureⅠ和gfp基因特异性引物Tab 1 Gene-specific primers for pureⅠand gfp
1.4 目的基因的扩增
将唾液链球菌及含有GFP编码基因gfp的变异链球菌菌株分别接种于BHI培养基琼脂平板,37 ℃厌氧培养箱内(体积分数10%H2,5%CO2及85%N2)培养24 h。挑取单菌落于BHI液体培养基中增菌过夜。4 000 r·m in-1离心10 m in收集细菌至1.5 m L EP管中。使用细菌基因组DNA抽提试剂盒,分别提取唾液链球菌以及含GFP编码基因gfp的变异链球菌基因组DNA。分别以上述的DNA为模板进行聚合酶链反应(polymerase chain reaction,PCR)扩增,反应条件:94 ℃预变性5 m in,进入热循环(98 ℃变性30 s,55 ℃退火30 s,68 ℃延伸30 s),共35个循环,最后68 ℃再延伸8 m in。通过2%琼脂糖凝胶电泳检测PCR扩增产物,DNA凝胶回收试剂盒回收PCR产物中的目的基因片段。
1.5 含目的基因重组质粒的构建与验证
将扩增得到的目的基因片段用限制性核酸内切酶BamHⅠ酶切并使用2%琼脂糖凝胶电泳及DNA凝胶回收试剂盒回收酶切后的目的基因片段。将回收后的目的基因片段用T4 DNA连接酶16 ℃连接16 h。使用1.5%琼脂糖凝胶电泳对连接产物进行分析,并切取1 200 bp大小的条带进行DNA回收。以回收产物作为模板,pureⅠ-SacⅠ及gfp-SalⅠ作为引物,PCR扩增融合连接体。反应条件:94 ℃预变性5 min,进入热循环(98 ℃变性30 s,55 ℃退火30 s,68 ℃延伸1 m in),共35个循环,最后68 ℃再延伸8 min。将上述融合片段与质粒pDL278分别使用SacⅠ及SalⅠ双酶切。将酶切产物进行琼脂糖凝胶电泳后,使用DNA凝胶回收试剂盒回收酶切后的核酸片段。使用T4 DNA连接酶将上述两片段在16 ℃下连接16 h。将连接产物转化到大肠杆菌DH5α感受态细胞中,涂布含有100 μg·m L-1壮观霉素的LB培养基琼脂平板,37 ℃有氧条件下培养24 h,挑选阳性克隆,增菌培养,提取质粒进行PCR和测序验证。验证正确的重组质粒命名为pDL278-pureⅠ-gfp,并冻存留用。
1.6 变异链球菌低pH感应系统的构建
将变异链球菌UA 159接种于BHI培养基琼脂平板,37 ℃厌氧培养箱内培养。1 d后传代,经涂片镜检及生化鉴定后,再次传代过夜培养。使用BHI培养基将菌液稀释20倍,厌氧条件下培养至吸光度OD600值为0.2。取500 μL上述菌液置于无菌EP管中,加入变异链球菌感受态刺激肽(competence stimulating peptide,CSP)使其终质量浓度为1 μg·m L-1,37 ℃孵育10 m in。向上述菌液中加入pDL278-pureⅠ-gfp并使其终质量浓度为2.5 μg·m L-1,厌氧条件下培养2.5 h。取100 μL菌液涂布含有1 g·L-1壮观霉素的BHI培养基琼脂平板并于厌氧条件下培养。48 h后挑选壮观霉素阳性克隆进行增菌培养。使用菌液进行PCR验证,验证正确的重组菌株加入甘油后于-80 ℃冻存。
1.7 变异链球菌低pH感应系统的验证
将重组变异链球菌菌株接种于含有终质量浓度为15 g·L-1酸碱缓冲剂Pipes(Sigma公司,美国)和1 g·L-1壮观霉素的50%BHI培养基中,37 ℃厌氧培养箱内培养过夜,菌液备用。将9个灭菌后的玻璃小圆片分别置于24孔板的9个孔内,并向其中加入2 m L上述混合培养液及20 μL上述菌液,37 ℃厌氧培养箱内培养14 h后,分别置于pH值为7.5、5.5、3.5的BHI培养基中处理1、30、60 m in。然后将各小圆片取出置于载玻片上,向小圆片中央滴加一滴无荧光镜油,小心盖上盖玻片。于奥林巴斯正置荧光显微镜spgr-b通道下观察上述样本,每个样本随机选取5个视野并使用ImagePro-Plus计算其单位面积荧光强度,并使用SNK检验分析组间差异。
2 结果
2.1 目的基因扩增产物的鉴定及gfp和pureⅠ基因片段的连接
PCR产物琼脂糖凝胶电泳结果显示,在约700 bp和450 bp处分别有特异性单一PCR产物条带,与预期大小(717 bp和450 bp)一致(图1)。将上述基因片段通过BamHⅠ酶切位点酶切后连接,连接产物琼脂糖凝胶电泳显示,在约450、700、900、1 200、1 400 bp处均可见清晰条带(图2)。其中,1 200 bp处条带大小符合预期。回收该条带内DNA并将其作为模板,以pureⅠ-SacⅠ和gfp-SalⅠ为引物,行PCR扩增。扩增产物电泳结果显示,在约1 200 bp处见特异性单一PCR产物条带,与预期大小一致(图3)。2.2 重组质粒pDL278-pureⅠ-gfp的构建和鉴定
将融合片段pureⅠ-gfp与载体pDL278通过SacⅠ及SalⅠ酶切位点双酶切后连接,并将连接体系转化入大肠杆菌感受态细胞DH5α中,涂布壮观霉素抗性平板,挑选阳性菌落,增菌后提取质粒。重组质粒及其PCR产物经琼脂糖凝胶电泳结果显示,约7 900 bp处可见特异性条带,与预期重组质粒大小一致(图4),在约1 200 bp处可见特异性单一PCR产物条带,与pureⅠ-gfp融合片段预期的大小一致(图4)。经过对该PCR产物进行DNA序列测定和比对,确定该融合片段的DNA序列与数据库中的序列100%一致。
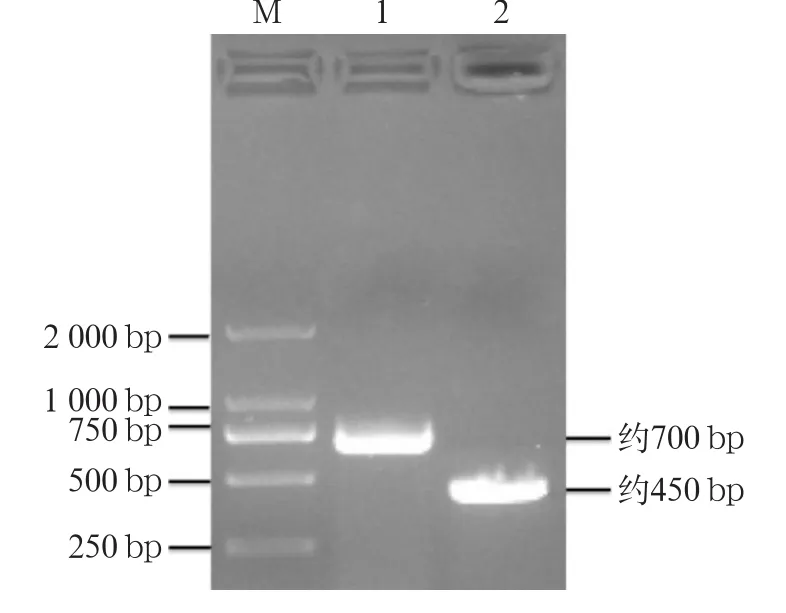
图 1 gfp和pureⅠ基因PCR扩增产物Fig 1 PCR products of gfp and pureⅠ genes

图 2 gfp和pureⅠ基因的连接产物Fig 2 Connection products of gfp and pureⅠgenes

图 3 gfp和pureⅠ基因融合片段的PCR扩增产物Fig 3 PCR products of the connected segment of gfp and pureⅠ genes

图 5 重组变异链球菌PCR验证产物琼脂糖凝胶电泳结果Fig 5 Agarose gel electrophoresis results of PCR products of recom- binant Streptococcus mutans
2.3 变异链球菌低pH报告菌株的构建及鉴定
将经验证的重组质粒转化入经诱导的变异链球菌细胞中,涂布壮观霉素抗性平板,共挑选出6株变异链球菌壮观霉素阳性克隆,PCR产物琼脂糖凝胶电泳结果显示,6个样本均在约1 200 bp处可见特异性单一PCR产物条带,与pureⅠ-gfp融合片段预期大小一致(图5)。

图 4 重组质粒及其PCR验证产物琼脂糖凝胶电泳结果Fig 4 Agarose gel electrophoresis results of recombinant plasm ids and PCR products
2.4 变异链球菌低pH报告菌株感应不同pH能力比较
为了验证变异链球菌低pH报告菌株感应不同pH的能力,本研究分别对比了在pH值为7.5、5.5、3.5以及处理时间为1、30、60 m in时变异链球菌低pH报告菌株的单位面积荧光强度。不同pH和处理时间变异链球菌低pH感应系统荧光图片见图6,单位面积荧光强度统计图见图7。

图 6 不同pH和处理时间变异链球菌低pH感应系统荧光图 荧光显微镜 × 100Fig 6 The scene of the low-pH-sensing system in different pH and different processing time fluorescence microscope × 100
当pH为7.5和5.5时,随处理时间的延长,单位面积荧光强度明显升高(P<0.05);当pH值为3.5时,处理1 min与30 min单位面积荧光强度无明显差异(P> 0.05),但均较处理60 min时低(P<0.05)。不同pH处理相同时间时,单位面积荧光强度随pH值的降低而升高(图7)。

图 7 不同pH和处理时间变异链球菌低pH感应系统单位面积荧光强度统计图Fig 7 Fluorescence intensity statistical figure of the low-pH sensing system in different pH and different processing time
3 讨论
变异链球菌是牙菌斑生物膜中的重要成员,该菌能够代谢糖类产酸,降低局部pH,抑制其余细菌(如血链球菌)的生长,在竞争中逐渐成为优势菌。同时变异链球菌具有耐酸性,可以在低pH的环境中正常生长,并且产生更多的酸,使环境始终处于低pH状态[9-10]。牙菌斑生物膜形成迅速且较难清除,其中的微生物代谢产生的乳酸等酸性物质不易流出亦不易被唾液中和,故可造成牙菌斑底部附着牙面的局部长期低pH环境[1]。龋病的发生就始于局部低pH环境造成的牙体脱矿。由此可见,相对于细菌本身而言,其产酸造成的菌斑附着表面的低pH环境才是龋病发生的直接因素。如何直观准确地反映菌斑pH显得尤为重要。本实验构建的低pH感应系统以致龋微生物——变异链球菌作为报告菌,可用于与龋相关的微生物及微生态研究,不带入其他影响因素,使用范围广。
目前,生物膜pH的测量方法大致有以下几种。1)接触式电极测量法[5]:将微电极插入待测部位进行探测。2)取样检测法[5]:将待测部位菌斑收集起来,再通过pH计进行检测。这两种方法简单、便宜、灵活,但具有测量时会扰乱待测部位生物膜,测试时间不连续,唾液缓冲作用影响大,非原位检测等缺点。3)内在电极测量法[5]:将微电极事先置于未形成生物膜的物体表面(如义齿基托、培养皿底部等),然后置于患者口内或接入细菌,即可通过无线电技术连续长期地检测pH。这种方法不扰乱生物膜的形成且为原位检测,但其价格昂贵、操作复杂。4)pH染料法[11-12]:使用商品化的pH荧光染料对生物膜进行染色和检测。此方法中pH染料染色的操作易受生物膜厚度和表面粗糙程度等因素的影响,造成实验误差。总体来说,生物膜pH的测量方法不尽如人意,研究者们渴望寻得一种方便、廉价、连续原位测量且误差较小的测量方法。本实验构建的变异链球菌低pH感应系统,于生物膜原位检测pH,检测方法操作简单,可最大程度还原生物膜局部的真实pH高低状态,减少人为因素带入的影响。
唾液链球菌尿素酶编码基因ureⅠ的启动子pureⅠ酸诱导性强,机制较清楚。CodY蛋白与RNA聚合酶竞争pureⅠ上的结合位点,而pH的高低则决定CodY蛋白与结合位点的结合与解离[6]。本课题组发现,变异链球菌中的CodY蛋白与唾液链球菌中的CodY蛋白高度同源,且有研究者将该启动子转入格氏链球菌中并验证了该启动子在链球菌属其他菌种中也可以正常发挥酸诱导特性[13]。Li等[14]的研究中展望了使用该启动子连接一段报告基因以制作生物膜pH探针的应用前景。GFP具有荧光稳定,非种属特异,不影响细胞正常生理功能,可用于活细胞直接观测等特点,其编码基因常被作为报告基因用于启动子功能验证,报告菌株构建,蛋白质定位以及分泌的检测等[8,15-17],也曾被用于变异链球菌中研究gtfB基因的表达调控[18]。故本实验将启动子pureⅠ与gfp基因连接转入变异链球菌中,从而构建受环境pH调节荧光强弱的变异链球菌低pH报告系统。该系统感受环境pH的受体位于核酸层面,调控精度高,从而提高了此系统的准确性。
本研究在变异链球菌低pH感应系统的验证中发现,培养14 h后的生物膜样本已检测出较弱荧光,但荧光强度明显低于处理后。笔者认为,造成此现象的原因可能是:1)由于变异链球菌代谢产酸,虽已使用50%BHI培养基降低营养,并于培养基中加入酸碱缓冲剂Pipes,但经检测培养14 h后培养基仍处于弱酸性;2)质粒在变异链球菌中为多拷贝状态,而变异链球菌基因组codY基因为单拷贝。变异链球菌产生的CodY蛋白不足以在接近中性pH的环境下将pureⅠ启动子完全阻遏,故仍可有GFP的合成。本课题组后期实验计划将融合片段pureⅠ-gfp整合到变异链球菌基因组中,以期具有更精确灵敏的pH感应作用。
综上所述,本研究成功构建了变异链球菌低pH感应系统。该系统可在一定的范围内原位反映生物膜局部pH状态,具有一定的应用前景。同时,本研究验证了唾液链球菌尿素酶基因的启动子pureⅠ在变异链球菌中可以正常发挥酸诱导功能,为日后进一步将该启动子应用于变异链球菌基因表达调控的相关研究提供了理论基础。
[1] Bowen WH. Dental caries—not just holes in teeth! A perspective[J]. Mol Oral M icrobiol, 2016, 31(3):228-233.
[2] Bowen WH, Koo H. Biology of Streptococcus mutans-derived glucosyltransferases: role in extracellular matrix formation of cariogenic biofilms[J]. Caries Res, 2011, 45(1):69-86.
[3] Svensäter G, Borgström M, Bowden GH, et al. The acidtolerant m icrobiota associated with plaque from initial caries and healthy tooth surfaces[J]. Caries Res, 2003, 37(6):395-403.
[4] Bowen WH. The stephan curve revisited[J]. Odontology, 2013, 101(1):2-8.
[5] Preston AJ, Edgar WM. Developments in dental plaque pH modelling[J]. J Dent, 2005, 33(3):209-222.
[6] Huang SC, Burne RA, Chen YY. The pH-dependent expression of the urease operon in Streptococcus salivarius is mediated by CodY[J]. Appl Environ M icrobiol, 2014, 80(17): 5386-5393.
[7] 周双双, 郑欣, 周学东, 等. 菌斑生物膜产碱代谢与龋病[J]. 国际口腔医学杂志, 2016, 43(5):573-577. Zhou SS, Zheng X, Zhou XD, et al. Relationship of alkali production by plaque biofilm and dental caries[J]. Int J Stomatol, 2016, 43(5):573-577.
[8] Bizzarri R, Serresi M, Luin S, et al. Green fluorescent protein based pH indicators for in vivo use: a review[J]. Anal Bioanal Chem, 2009, 393(4):1107-1122.
[9] Klein M I, Hwang G, Santos PH, et al. Streptococcus mutansderived extracellular matrix in cariogenic oral biofilms[J]. Front Cell Infect M icrobiol, 2015, 5:10.
[10] 高敬, 黄文明. 变异链球菌耐酸毒力因子质子移位膜ATP酶在龋病进展中的动态变化[J]. 华西口腔医学杂志, 2016, 34(2):200-204. Gao J, Huang WM. Dynam ic changes of aciduric virulence factor membrane-bound proton-translocating ATPase of Streptococcus mutans in the development of dental caries [J]. West Chin J Stomatol, 2016, 34(2):200-204.
[11] Xiao J, Klein M I, Falsetta ML, et al. The exopolysaccharide matrix modulates the interaction between 3D architecture and virulence of a mixed-species oral biofilm[J]. PLoS Pathog, 2012, 8(4):e1002623.
[12] DePedro HM, Urayama P. Using lysosensor yellow/blue DND-160 to sense acidic pH under high hydrostatic pressures[J]. Anal Biochem, 2009, 384(2):359-361.
[13] Chen YY, Betzenhauser MJ, Burne RA. cis-Acting elements that regulate the low-pH-inducible urease operon of Streptococcus salivarius[J]. M icrobiology, 2002, 148(Pt11):3599-3608.
[14] Li YH, Chen YY, Burne RA. Regulation of urease gene expression by Streptococcus salivarius grow ing in biofilms [J]. Environ M icrobiol, 2000, 2(2):169-177.
[15] Choi WG, Swanson SJ, Gilroy S. High-resolution imaging of Ca2+, redox status, ROS and pH using GFP biosensors[J]. Plant J, 2012, 70(1):118-128.
[16] Walker CL, Lukyanov KA, Yampolsky IV, et al. Fluorescence imaging using synthetic GFP chromophores[J]. Curr Opin Chem Biol, 2015, 27:64-74.
[17] Josenhans C, Friedrich S, Suerbaum S. Green fluorescent protein as a novel marker and reporter system in Helicobacter sp[J]. FEMS M icrobiol Lett, 1998, 161(2):263-273.
[18] Yoshida A, Kuram itsu HK. Streptococcus mutans biofilm formation: utilization of a gtfB promoter-green fluorescent protein (PgtfB::gfp) construct to monitor development[J]. M icrobiology, 2002, 148(Pt 11):3385-3394.
(本文编辑 吴爱华)
Construction of a low-pH-sensing system in Streptococcus mutans
Kang Di, Li Yuqing, Zhou Xuedong. (State Key Laboratory of Oral Diseases, National Clinical Research Center for Oral Diseases, West China Hospital of Stomatology, Sichuan University, Chengdu 610041, China)
ObjectiveTo construct a low-pH-sensing system in Streptococcus mutans (S. mutans) and to visually detect the pH in situ.MethodsPromoter of ureaseⅠ(PureⅠ) and green fluorescence protein (gfp) DNA fragments were amplified by polymerase chain reaction (PCR) from the genome of Streptococcus salivarius 57.I and S. mutans containing the gfp fragment. The two amplified DNA fragments were ligated together and further integrated into pDL278 to construct the recombinant plasm id pDL278-pureⅠ-gfp. This recombinant plasm id was then transformed into S. mutans UA159 cells. Subsequently, the intensity of the optical density per unit area of the low-pH-sensing system was measured and compared under different pH conditions and different processing times.ResultsPureⅠ and gfp DNA fragments were amplified successfully with the correct molecule sizes (450 and 717 bp, respectively). The recombinant plasmid pDL278-pureⅠ-gfp was constructed and further verified by PCR and sequencing. The intensity of the optical density per unit area of the low-pH-sensing system increased with decreasing pH and increasing processing time.ConclusionA low-pH-sensing system was constructed successfully in S. mutans. Our research verified that pureⅠ of Streptococcus salivarius can function well in S. mutans as an acid induced promoter, and provided a new method of detecting the pH of plaque biofilms in situ.
Streptococcus mutans; Streptococcus salivarius; green fluorescence protein; urease; promoter
R 780.2
A
10.7518/hxkq.2017.03.003
Supported by: National Natural Science Foundation of China (81430011, 31200985). Correspondence: Zhou Xuedong, E-mail: zhouxd@scu.edu.cn.
2016-10-25;
2016-12-12
国家自然科学基金(81430011,31200985)
康迪,博士,E-mail:juejiangkang@gmail.com
周学东,教授,博士,E-mail:zhouxd@scu.edu.cn
