基于COI及RAG2基因序列15种金线鱼科鱼类分子系统进化关系*
2017-06-19梁日深陈冬青苏国茂吴灶和邹记兴
梁日深, 陈冬青, 苏国茂 , 周 萌, 吴灶和, 邹记兴
(1.仲恺农业工程学院动物科技学院,广东 广州 510225; 2.华南农业大学海洋学院,广东 广州 510642)
基于COI及RAG2基因序列15种金线鱼科鱼类分子系统进化关系*
梁日深1, 陈冬青1, 苏国茂1, 周 萌1, 吴灶和1, 邹记兴2
(1.仲恺农业工程学院动物科技学院,广东 广州 510225; 2.华南农业大学海洋学院,广东 广州 510642)
本研究基于线粒体DNACOI及核DNARAG2基因部分序列分析了印度-太平洋金线鱼科4属15种鱼类分子系统进化关系,综合2基因序列采用最大似然法构建了系统进化树。研究获得15种金线鱼科鱼类COI基因序列651 bp及RAG2基因序列726 bp,2基因片段均不存在插入与缺失。构建的进化树上,(1)15种金线鱼科鱼类种内不同个体间均聚为一支,形成物种内的单系,节点支持率为100%。(2)金线鱼科4个属主要分成2个分支,金线鱼属与锥齿鲷属聚为一支,眶棘鲈属与副眶棘鲈属聚为另一支,与形态分类一致。(3)对于金线鱼属物种间的聚类情况,六齿金线鱼与深水金线鱼,印度洋金线鱼与金线鱼,苏门答腊金线鱼与日本金线鱼均紧密聚成姐妹分支,红金线鱼与姬金线鱼分类地位较为原始,位于上述金线鱼大分支的基部。而该物种间聚类结果与Mohd在形态上根据尾鳍上下叶的长度性状把金线鱼分成2大组群的结果不同。对于该分歧是由于分子进化速率与表型进化速率不一致导致,还是体色斑纹、鳍条数等性状不能作为区分金线鱼属鱼类分类关系的有效指标,还需今后进一步的研究。
金线鱼科;系统发育;COI基因;RAG2基因
1 研究背景
金线鱼科(Nemipteridae)隶属鲈形目(Perciformes)鲈亚目(Percoidei),主要分布于印度-西太平洋亚热带、热带海区。目前世界金线鱼科主要分为金线鱼属(Nemipterus)、锥齿鲷属(Pentapodus)、眶棘鲈属(Scolopsis)、副眶棘鲈属(Parascolopsis)和裸颞鲷属(Scaevius)5个属,共64种,我国记载有4属约30余种,主要分布在南海[1-5]。金线鱼科属内种间形态差异很小,基于传统形态学的分类手段,许多物种的分类关系存在很大的争议[2-7]。如目前形态学对金线鱼科所划分的4个属中,金线鱼属与锥齿鲷属形态较为接近,眶棘鲈属与副眶棘鲈属形态也异常相似,过去的分类资料曾认为副眶棘鲈属应归为眶棘鲈属中[2,4-5],而另有资料却认为副眶棘鲈属可单独形成一个属[3]。在金线鱼属内种间的分类中,有研究依据金线鱼尾鳍上下叶长度是否一致将其分为2大类,上叶长度与下叶一致的归为一类,上叶延长大于下叶的归为另一类[8],而另有资料却依据体表条纹颜色、大小、数量或鳍条数目来划分[2-3,6],导致金线鱼属鱼类种间的分类关系无法统一,而进一步的分子水平的研究报道相对较少[9-10]。形态分类混乱,物种分类不清已严重制约了金线鱼科种质资源的正确开发与有效利用。
近年来,随着分子生物技术的发展,物种的分类也逐渐由形态学向分子水平的方向前进。物种进化本质上是基因组的进化,物种分类在DNA水平上更精确地反应物种之间的聚类关系。线粒体DNA、核DNA等不同类型的基因可作为有效的遗传标记应用在物种系统分类研究中,有效解决许多鱼类形态分类上悬而未决的难题[11-13]。线粒体DNA中细胞色素C氧化酶亚基I(COI)基因,进化速率适中,在鱼类分子系统进化研究中已被验证为是可靠的遗传标记。同时,其5端一段长度为648 bp的片段,能在基因水平上成功区分物种,可作为物种分子鉴定有效的DNA条形码标准序列,为生物的分类提供一种快速简便、可信可靠的分类方法[14]。重组激活基因(Recombination Activating Genes,RAGs)是脊椎动物特异性免疫反应的关键基因,由RAG1和RAG2 2个基因组成[15-16]。其中,RAG2序列相对保守,适用于分子系统进化分析,已广泛应用于各种鱼类系统分类研究[17-19]。线粒体DNACOI及核DNARAG2 2基因序列各有特点,可应用于科、属、种等分类阶元的研究。两基因相结合,优势互补,将提供更多更完善的基因信息与数据,更有效的解决物种分类问题。
本研究通过测定和比较金线鱼科4个属15种29个个体线粒体DNACOI及核DNARAG2基因部分序列,共同分析这15种金线鱼科鱼类分子系统分类关系。同时,验证COI基因在金线鱼属鱼类DNA条形码识别技术的有效性,为该类群物种的分类争议提供分子水平的依据。
2 材料与方法
2.1 材料来源
金线鱼科实验样品主要采集于印度-西太平洋沿海区域,我国分布的种类主要采集于南海,国外样品来源于南非水生物多样性研究院及新喀里多尼亚泻湖水族馆,样品材料依照《中国鱼类检索系统》进行形态学鉴定与分类。样品的分类信息、采集地及编号见表1。
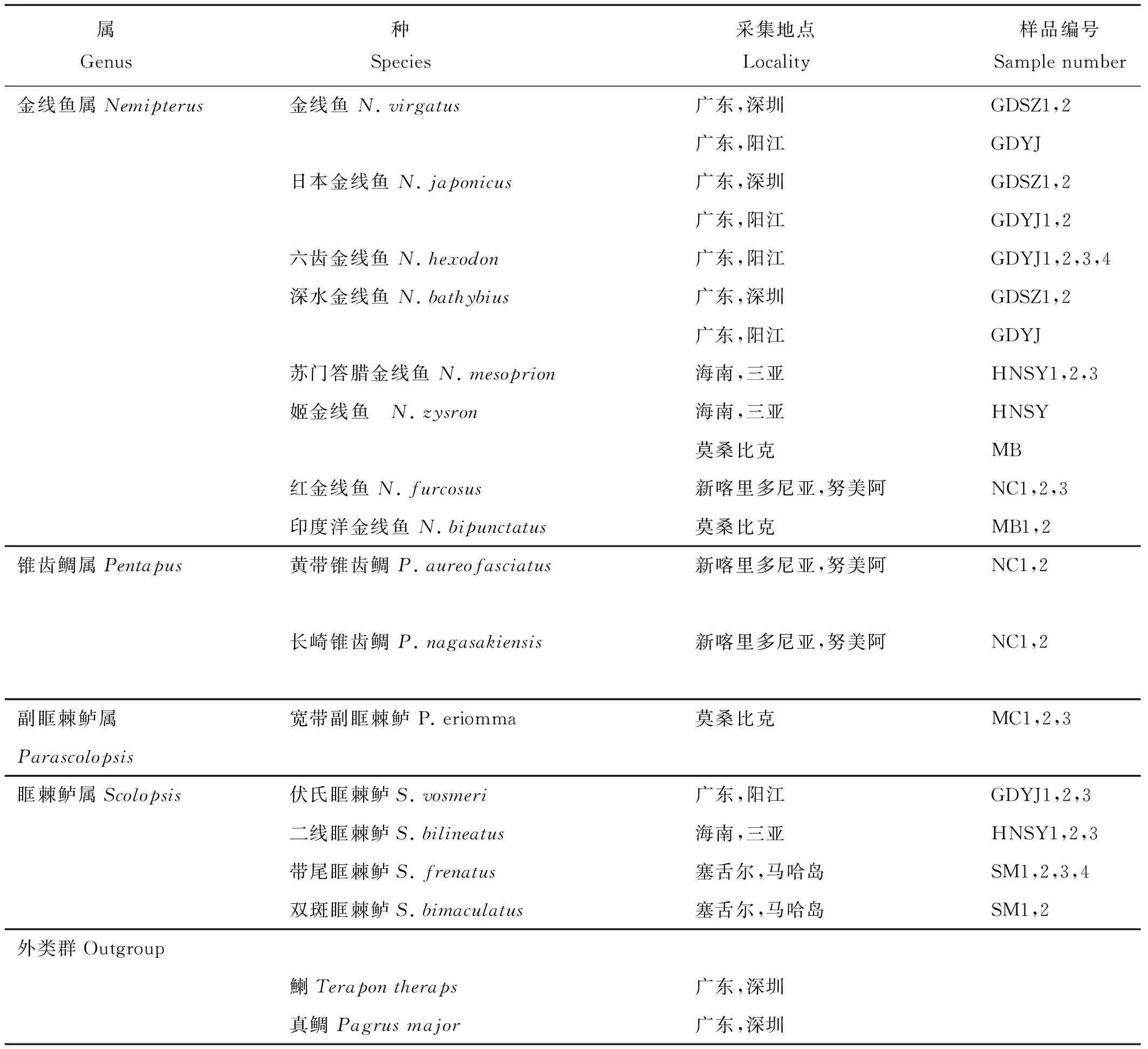
表1 金线鱼科15种鱼类样品基本信息
2.2 总基因组DNA的提取
基因组DNA提取采用动物组织DNA提取试剂盒(天根生化科技有限公司)进行提取。提取的DNA溶解于100 μL洗脱缓冲液中, 经1.5%琼脂糖凝胶电泳检测,-20℃保存备用。
2.3 PCR扩增和测序
实验用的COI及RAG2基因片段的PCR扩增引物分别参考国外相关文献进行设计,由上海英骏生物公司合成。PCR反应体系为50 μL,其中10×缓冲液5 μL, dNTPs (各2.5 mmol/L)2 μL,上下游引物各1 μL,Ex Taq酶(1 U/μL) 2 μL。引物序列如下:
COI上游引物: 5- TCA ACY AAT CAY AAA GAT ATY GGC AC-3。
COI下游引物: 5- ACT TCY GGG TGR CCR AAR AAT CA-3[20]。
RAG2上游引物: 5’-GAG GGC CAT CTC CTT CTC CAA-3’。
RAG2下游引物: 5’-GAT GGC CTT CCC TCT GTG GGT AC-3’[21-22]。
PCR反应条件如下:对于COI基因,先94 ℃预变性5 min,随后运行35个循环(94 ℃变性30 s,55℃退火30 s,72℃延伸1 min),最后72 ℃延伸10 min;对于RAG2基因,除了退火温度58 ℃ 30 s,其他条件与COI基因一致。
COI与RAG22基因扩增结束后,电泳检测,纯化回收,产物送上海英骏生物技术有限公司进行双向测序。
2.4 数据分析
所得序列采用 NCBI 的Blastn 工具(http://www.ncbi.nlm.nih.gov/blast)进行在线比对,验证序列是否为所得目的片段。利用Clustal W[23]对所有序列进行排序并辅以手工校正。利用 MEGA5.0[24]计算核苷酸组成、碱基变异及转换颠换信息,基于K-2-P双参数模型计算样品种内、种间及属间遗传距离。利用COI,RAG2及COI+RAG2整合序列,以鯻科及裸颊鲷科鱼类作为外类群,基于最大似然法(Maximum likelihood,ML)构建分子系统进化树,进化树采用重复抽样分析(Bootstrap analysis)1 000次的方法检验各分支的置信度。
3 结果分析
3.1 序列分析
扩增得到的15种金线科鱼类COI基因为651 bp的片段,碱基组成A、T、G、C 平均含量分别为 23.3%、30.6%、18.6%、27.4%,其中 A+T 含量(53.9%)略高于G+C 含量(46.0%)。无碱基的插入、缺失,编码227个氨基酸。在密码子第一位,G-1含量最高(30.7%);密码子第二位,T-2含量(41.0%)明显高于其他3种,A-2,G-2较低,约15%;密码子第三位T-3含量最高(32.0%),G-3含量仅有9.6%(见图1)。可见碱基A、T、G、C的含量在密码子不同位置差异很大,使用频率存在偏倚。此外,在长度为651 bp序列中,15种金线科鱼类共有保守位点413个,约63.4%;变异位点238个,约占36.6%;简约性信息位点214个,约占32.9%。
RAG2基因部分序列比对分析得到一致序列为729 bp,编码243个氨基酸。碱基A、T、G、C平均含量分别为20.5%,21.5%,27.9%和30.2%,显示A碱基相对缺乏,其中A + T的含量(42.0%)明显低于G + C含量(58.1%)。密码子第1,第2位四种碱基含量均相差不大,而密码子第3位四种碱基含量有较大差异,其中C-3含量最高,为42.9%,而A-3含量最低,为9.9%,表现出反A偏倚(见图2)。序列的保守位点579个,约占79.4%;变异位点150个,约占20.6%;简约性信息位点93个,约12.8%。可知,与COI基因相比,RAG2基因相对较为保守,物种间序列差异性较小。

图1 COI基因3个位置密码子的碱基组成

图2 RAG2基因3个位置密码子碱基组成
3.2 种间及种内的遗传距离
利用MEGA5.0基于Kimura 2-parameter模型计算出15种金线鱼科鱼类COI及RAG2基因遗传距离如表2、3所示。在COI基因中,物种种内遗传距离为0.000~0.004,平均遗传距离0.002,低于Hebert等[25]所推荐的物种鉴定最小种间遗传距离值0.020。种间遗传距离为0.129~0.257,遗传距离最大在苏门答腊金线鱼与双斑眶棘鲈之间,最小在日本金线鱼与苏门答腊金线鱼之间,平均遗传距离0.203,均高于Hebert等[25]设定的0.020的遗传差异值。种间平均遗传距离是种内遗传距离的101.5倍。显示COI基因序列在金线鱼科物种具有足够大的种间差异区分各物种。对于物种数量最多的金线鱼属中,遗传距离范围为0.129~0.226,其中最大是六齿金线鱼与苏门答腊金线鱼,最小是日本金线鱼与苏门答腊金线鱼。
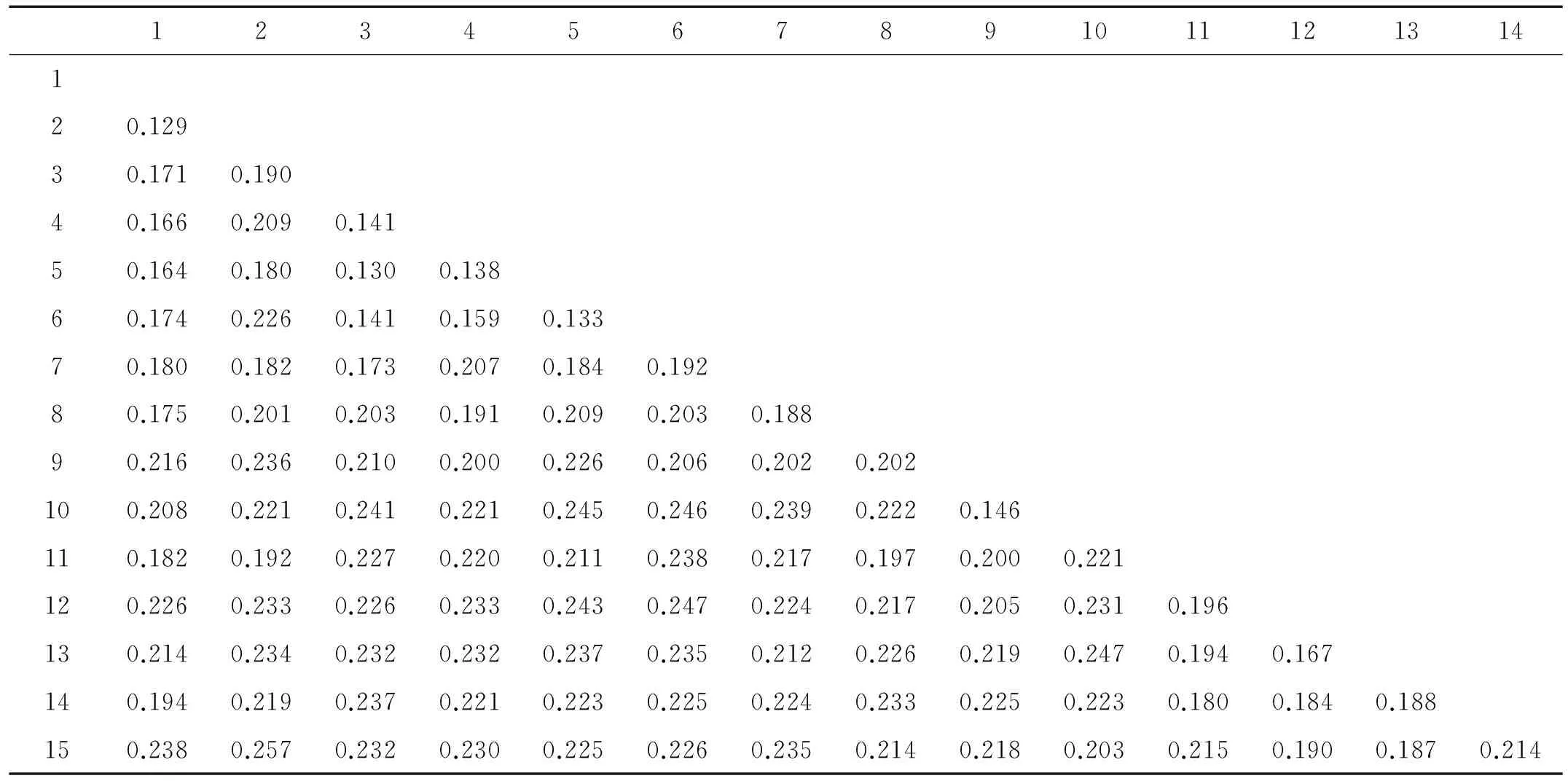
表2 15种金线鱼科鱼类COI基因遗传距离(Kimura-2模型)
注: 1.日本金线鱼N.japonicus; 2.苏门答腊金线鱼N.mesoprion; 3.印度洋金线鱼N.bipunctatus; 4.金线鱼N.virgatus; 5.深水金线鱼N.bathybius; 6.六齿金线鱼N.hexodon; 7.红金线鱼N.furcosus; 8.姬金线鱼N.zysron; 9.黄带锥齿鲷P.aureofasciatus; 10.长崎锥齿鲷P.nagasakiensis; 11.宽带副眶棘鲈P.eriomma; 12.伏氏眶棘鲈S.vosmeri; 13.二线眶棘鲈S.bilineatus; 14.带尾眶棘鲈S.frenatus; 15.双斑眶棘鲈S.bimaculatus

表3 15种金线鱼科鱼类RAG基因遗传距离(Kimura-2模型)
注: 1.日本金线鱼N.japonicus; 2.苏门答腊金线鱼N.mesoprion; 3.印度洋金线鱼N.bipunctatus; 4.金线鱼N.virgatus; 5.深水金线鱼N.bathybius; 6.六齿金线鱼N.hexodon; 7.红金线鱼N.furcosus; 8.姬金线鱼N.zysron; 9.黄带锥齿鲷P.aureofasciatus; 10.长崎锥齿鲷P.nagasakiensis; 11.宽带副眶棘鲈P.eriomma; 12.伏氏眶棘鲈S.vosmeri; 13.二线眶棘鲈S.bilineatus; 14.带尾眶棘鲈S.frenatus; 15.双斑眶棘鲈S.bimaculatus
在RAG2基因中,15种金线鱼科鱼类种内遗传距离范围在0.000~0.004之间,平均遗传距离为0.001。种间遗传距离范围在0.015~0.097之间,平均遗传距离为0.065。可知,RAG2基因序列差异性比COI基因要小。其中,遗传距离最小的是黄带锥齿鲷与长崎锥齿鲷,遗传距离最大是六齿金线鱼与带尾眶棘鲈。在金线鱼属内部,遗传距离范围为0.018~0.059,其中最大是六齿金线鱼与红金线鱼,最小是金线鱼与印度洋金线鱼。
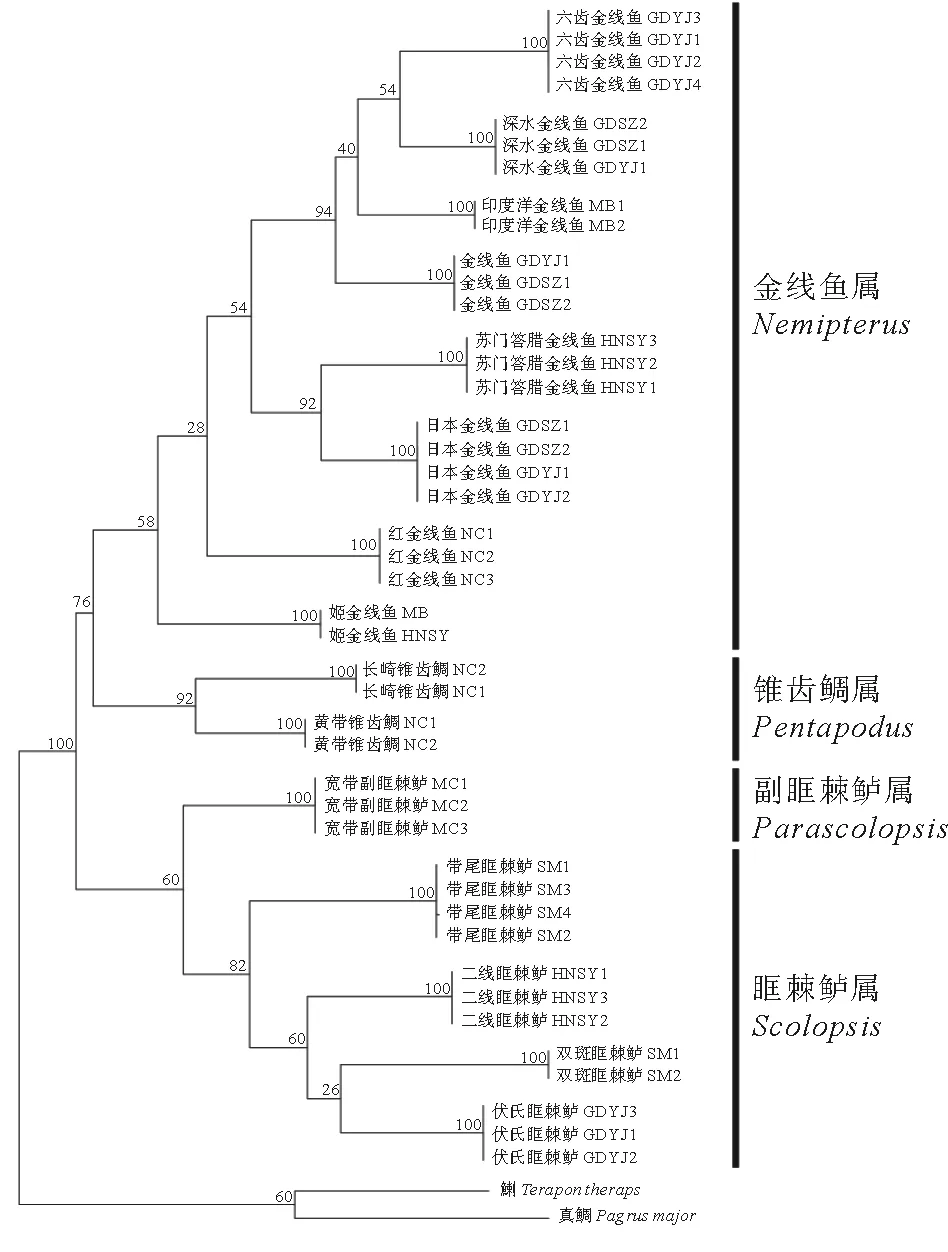
图3 基于COI序列利用最大似然法构建的分子系统树
3.3 分子系统进化树的构建
基于所得COI、RAG2及COI+RAG2整合序列,以鲈形目中鱼类中真鲷和鯻作为外类群进行系统进化分析,采用最大似然法ML法分别构建了3棵系统进化树,结果见图3~5。3棵系统进化树的拓扑结构基本一致,具有较高的节点支持率。这里我们主要基于COI+RAG2整合序列构建的进化树进行分析。进化树上,金线鱼科鱼类种内不同个体均聚为一支,形成物种内单系,节点支持率为100%。金线鱼科4个属主要分成2个分支,金线鱼属与锥齿鲷属聚为一支,眶棘鲈属与副眶棘鲈属聚为另一支。对于各物种间的聚类情况,在金线鱼属中,六齿金线鱼与深水金线鱼,印度洋金线鱼与金线鱼,苏门答腊金线鱼与日本金线鱼均两两紧密聚成姐妹分支,红金线鱼与姬金线鱼分类地位较为原始,位于上述金线鱼大分支的某端,其中姬金线鱼最先分化出来,位于整个金线鱼属类群底部。
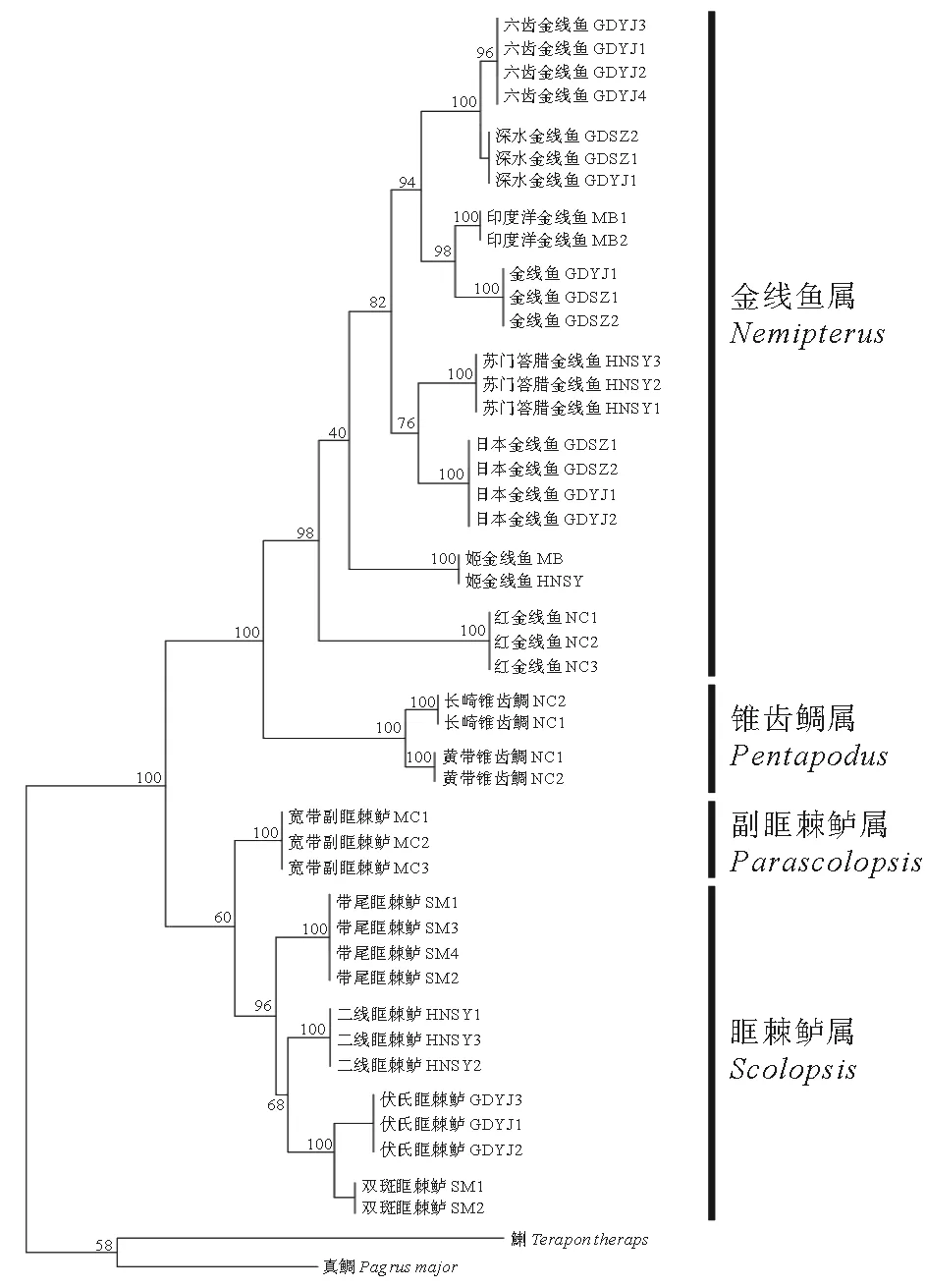
图4 基于RAG2序列利用最大似然法构建的分子系统树
4 讨论
COI及RAG2是鱼类分子系统发育研究常用的标记基因,国内外已广泛应用并有效解决许多鱼类系统分类问题。COI基因进化速率适中,主要应用于分类阶元较低,如属内种间以及种内样品分类关系;RAG2进化比较保守,应用于种级以上分类阶元的研究。本研究基于COI、RAG2及COI+RAG2整合序列,分别构建了金线鱼科鱼类分子系统进化树,3个基因得出系统树的拓扑结构基本一致,仅少数节点分枝及支持率有区别,如金线鱼、印度洋金线鱼的分类位置,说明COI及RAG2基因均能有效揭示金线鱼科物种间的分类关系。COI+RAG2整合序列结合两者基因的序列信息,优势互补,并丰富了可分析的位点数目,得到支持率更高,金线鱼科物种聚类关系更清晰更真实的系统进化树。
关于金线鱼科属级阶元的分类,不同的资料存在争议。目前国内外主流的分类趋向认为金线鱼科分为金线鱼属、锥齿鲷属、眶棘鲈属、副眶棘鲈属、裸颊鲷属5个属[3,6,26-27]。其中金线鱼属与锥齿鲷属形态较为接近,其身体大都呈长梭形,前鳃盖骨均被鳞片,部分种类尾鳍上叶有丝状延长等[3,26]。眶棘鲈属与副眶棘鲈属形态也异常相似,过去的分类资料曾认为副眶棘鲈属应归为眶棘鲈属中[2,4-5],近期部分学者基于前者的第一眶下骨棘短小,将其独立出来[3],也有学者发现二者在颌骨构造、上侧肋骨数目、眶下骨以及第二臀鳍鳍棘发达程度等方面均存在明显的差异,认为副眶棘鲈属可单独形成一个属[26]。本研究的进化树上,金线鱼科4个属以高支持率分为2个主要的分支,金线鱼属和锥齿鲷属聚为一支,眶棘鲈属和副眶棘鲈属聚为另外一支,与形态学大分类结果一致。同时,进化树上,副眶棘鲈属单独形成一支,没有聚类于眶棘鲈属种类的类群中,初步支持上述副眶棘鲈属可单独形成一个属的分类观点。

图5 基于COI+RAG2整合序列15种金线鱼科鱼类最大似然法(ML)分子系统树
金线鱼属是金线鱼科中物种数量最多的一个属。形态上对金线鱼属鱼类分类的依据主要体型体色、斑纹的差异以及尾鳍上叶末端是否延长等。Mohd等[8]曾依据金线鱼尾鳍上下叶长度是否一致将其分为两大类群,上叶长度与下叶一致的归为一类,上叶存在延长大于下叶的归为另一类。本研究的进化树上,六齿金线鱼与深水金线鱼,印度洋金线鱼与金线鱼,苏门答腊金线鱼与日本金线鱼两两紧密聚成姐妹分支,形态上,六齿金线鱼、印度洋金线鱼尾鳍上叶均不存在丝状延长,苏门答腊金线鱼具丝状延长或延长不明显。可知,在进化树上没有形成以尾鳍上叶末端是否延长的为区分标准的2大分支,与前期形态学研究结论有一定的出入,但与近年的部分系统分类学研究结果相似。如宁平[28]基于16S rRNA分子标记分析的金线鱼科鱼类结果也没有形成以尾鳍上下叶形态不一致的两大类群,尾鳍上下叶不一致的种类相互交错聚类在一起,本研究前期基于核DNA S7基因分析的7种金线鱼属鱼类系统进化研究也得出相似的结果[10]。
在上述形成的多个姐妹分支组合中,部分组合与形态学的分类特征一致[3,8],如日本金线鱼与苏门答腊金线鱼,两者形态尾鳍上叶长于下叶,肩部具有红色斑点,背鳍中间有纵行斑纹,体侧有数条纵带等。Mohd等[8]分析了11种金线鱼的形态数据,曾依此依据将两者归为一类群。Murty[29]比较形态学研究也得出相似的结果。但也有部分聚类结果与形态学分类有出入,如金线鱼与印度洋金线鱼,金线鱼的尾鳍上叶丝状延长,体侧具数条明显的黄色纵带,而印度洋金线鱼不具有该典型特征。另外,金线鱼具有一可与其他金线鱼属鱼类明显区分的典型特征——其臀鳍鳍条数目为8,其它金线鱼臀鳍鳍条均为7。部分资料根据这一特点把金线鱼最先划分出来[3]。在本研究的系统进化树上,金线鱼并没有独立形成一支,而是与印度洋金线鱼聚在一起,位于整个金线鱼属类群内部,处于中间的进化位置。由此可知,部分金线鱼属鱼类的分子分类与形态学分类结果存在出入,这在宁平的研究中也出现该情况,至于该分歧是分子进化速率与表型进化速率不一致导致,还是体色、花纹、鳍条数等主要性状不能作为分清金线鱼属鱼类分类关系的有效形态指标,还需今后更深一步的研究。
[1] Russell B C. Nemipterid fishes of the world[J]. FAO Species Catalogue, 1990, 125(12): 1-49.
[2] 成庆泰, 郑葆珊. 中国鱼类系统检索[M]. 北京:科学出版社, 1987: 339-341. Cheng Q T, Zheng B S. Systematic Synopsis of Chinese Fishes [M]. Beijing: Science Press, 1987: 339-341.
[3] 沈世杰. 台湾鱼类志[M]. 台北:台湾大学动物学系出版, 1993: 360-363. Shen S J. Fishes of Taiwan [M]. Taibei: Taiwan University Press. 1993: 360-363.
[4] 陈清潮. 南沙群岛至华南沿岸的鱼类(上)[M]. 北京:科学出版社, 1997:114. Chen Q C. Fishes from Nansha Islands to South China Coastal Waters 1 [M]. Beijing: Science Press,1997: 114.
[5] 刘瑞玉. 中国海洋生物名录[M]. 北京:科学出版社, 2008: 886-1066. Liu R Y. Checklist of Marine Biota of China Seas [M]. Beijing: Science Press, 2008: 886-1066.
[6] Nelson J S. Fishes of the World[M]. New York: John Wiley & Sons Inc, 2006: 624.
[7] Russell B C. A review of the threadfin breams of the genusNemipterus(Nemipteridae)from Japan and Taiwan, with redescription of a new species[J]. Japanese Jounral of Ichhtyology, 1993, 39(4): 295-310.
[8] Mohd Z M S, Mohd A A, Abu K M M. Colour pattern as an additional aid to the identification ofNemipterusspeciesand their relationship[J]. Pertanika, 1990, 3(1): 17-26.
[9] Menezes M, Arolkar U, Rathod V. 2002. Genetic relationships among five Nemipterid fish species from the Indian coast using allozyme analysis[J]. Journal of Fish Biology, 2010, 60(5): 1326-1330. [10] 梁日深, 王超, 邹青, 等. 基于S7核糖体蛋白基因部分序列的7种金线鱼属鱼类分子系统进化关系[J]. 中国水产科学, 2013, 20(3):506-513. Liang R S, Wang C, Zou Q, et al. Molecular phylogenetic relationship among seven Nemipterus species based on partial S7 ribosomal protein gene sequences[J]. Journal of Fishery Sciences of China, 2013, 20(3): 506-513.
[11] Bloom D D, Weir J T, Piller K R, et al. Do freshwater fishes diversify faster than marine fishes? A testusing state-dependent diversification analyses and molecular phylogenetics of new world silversides (atherinopsidae) [J]. Evolution, 2013, 67 (7): 2040-2057.
[12] DiBattista J D, Rocha L A, Hobbs J-PA, et al. When biogeographical provinces collide: hybridization of reef fishes at the crossroads of marine biogeographical provinces in the Arabian Sea[J]. Journal of Biogeography, 2015, 42: 1601-1614.
[13] Liang R S, Wang C, Zou Q, et al. Molecular phylogenetic relationships of some common sweetlips (Haemulidae: Plectorhynchinae) and the synonyms controversy of two Plectorhinchus species[J]. Mitochondrial DNA, 2016, 27(3): 2209-2214.
[14] Icolas H, Robert H, Louis B. Identification Canadian freshwater fishes through DNA barcodes[J]. Plos One, 2008, 3(6): 1-8.
[15] Van G D, Mcblane J F, Ramsden D A, et al. Initiation of V( D) J recombination in a cell-free system[J]. Cell, 1995, 81: 925-934.
[16] Market E, Papavasiliou F N. V (D) J recombination and the evolution of the adaptive immune system[J]. Plos Biology, 2003, 1(1): e16.
[17] Rocha L A, Lindeman K C, Rocha C R, et al. Historical biogeography and speciation in the reef fish genus Haemulon (Teleostei: Haemulidae)[J]. Molecular Phylogenetics and Evolution, 2008, 48: 918-928.
[18] Cramer C A, Bonatto S L, Reis R E. Molecular phylogeny of the Neoplecostominae and Hypoptopomatinae (Siluriformes: Loricariidae) using multiple genes[J]. Molecular Phylogenetics and Evolution, 2011, 59(1), 43-52.
[19] Martinez-Takeshita N, Purcell C M, Chabot C L, et al. A tale of three tails: Cryptic speciation in a globally distributed marine fish of the genus Seriola[J]. Copeia, 2015, 103(2): 357-368.
[20] Ward R D, Zemlak T S, Innes B H, et al. DNA barcoding Australia’s fish species[J]. Philosophical Transactions of the Royal Society B, 2005, 360: 1847-1857.
[21] Rocha L A, Lindeman K C, Rocha C R. Historical biogeography and speciation in the reef fish genus Haemulon (Teleostei: Haemulidae) [J]. Molecular Phylogenetics and Evolution, 2008, 48: 918-928.
[22] Lovejoy N R. Reinterpreting recapitulation: Systematics of needlefishes and their allies ( Teleostei: Belonidae)[J]. Internation Journal of Organic Evolution, 2000, 54(4): 1349-1362.
[23] Thompson J D, Higgins D G, Gibson T J. CLUSIAL W: Improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position specific gap penalties and weight matrix choice[J]. Nucleic Acids Research, 1994, 22(22): 4673-4680.
[24] Tamura K, Peterson D, Peterson N, et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739.
[25] Hebert P D N, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society of London, Series B, 2003a, 270(1512): 313-321.
[26] Edwards A J. Family Nemipteridae. An annotated and illustrated catalogue of Nemipterid species known to date FAO Fisheries Synopsis 125, Vol. 12[M]//. Russell B C. FAO Species Catalogue. Rome: FAO, 1990: 149.
[27] Nakabo T. Fishes of Japan: With Pictorial Keys to the Species[M]. Tokyo: Tokai University Press, 2002: 1800.
[28] 宁平. 中国金线鱼科鱼类分类,系统发育及动物地理学研究[D]. 青岛: 中国科学院研究生院(海洋研究所), 2012: 134. Ning P. Taxonomy, Molecular Phylogeny and Zoogeography of the Family Nemipteridae (Pisces: Perciformes) in China Seas[D]. Qingdao: Graduate University of Chinese Academy of Sciences (Institute of Oceanology), 2012: 134.
[29] Murty V S. Nemipterus mesoprion (Bleeker 1853) (Nemipteridae Pisces), a new record from the seas around India[J]. Indian Journal of Fisheries, 1981, 25: 207-213.
责任编辑 高 蓓
Molecular Phylogenetic Relationship of 15 Nemipteridae Fishes Based on MitochondrialCOIand NuclearRAG2 Gene Sequnces
LIANG Ri-Shen1, CHEN Dong-Qing1, SU Guo-Mao1, ZHOU Meng1, WU Zao-He1, ZOU Ji-Xing2
(1.College of Animal Technology, Zhongkai University of Agriculture and Engineering, Guangzhou 510225, China; 2.College of Marine Science, South China Agricultural University, Guangzhou 510642, China)
Fishes of the family Nemipteridae, belong to the suborder Percoidei of order Perciformes, which are mainly found in the oceanic waters of tropical, subtropical areas throughout the Indo-Western Pacific. Morphologically, classification and identification within Nemipteridae remains problematic because of their spectacular morphological diversification. Currently, few comprehensive molecular studies was concentrated on the Nemipteridae group to resolve the confused classificational issues. In the present study, molecular phylogenetic relationships among 15 Nemipteridae fish of 4 genera were analyzed based onCOIandRAG2 genes and the phylogenetic tree were constructed using maximum likelihood method. The sequences ofCOIandRAG2 genes were determined to be 651 bp and 726 bp, respectively, with no insertion and deletion existed. In the phyogenetic tree, (1) Individuals in each species were clustered together as a monophyletic group, with high bootstrap support value of 100%; (2) In the family Nemipteridae, four genera were divided into two major group, one group composed ofNemipterusandPentapodus, another group composed ofScolopsisandParascolopsis. (3) Within the genusNemipterus,N.japonicusandN.mesoprion,N.virgatusandN.bipunctatus,N.bathybiusandN.hexodonwere cluster tightly as sister species, indicating their close relationships with each other,N.zysronandN.furcosuswere first separated and rooted at the base of the above bigNemipterusgroup. The phylogenetic result was inconsistent with Mohds’ of hypothesis of dividingNemipterusspecies into two morphologically distinct groups according to the length of upper and lower of their caudal fin lobes, because some species in the same clusters share different characters of . Besides, the relationships of some clusters were somewhat different from the morphological classifications. To this divergence, it was due to the inconsistence between the molecular evolution rate and phenotypic evolution rate, or the morphological characters of color, stripes, shapes, fin ray numbers etc could not be used as effective morphological index to distinguish the taxonomic relationships of the Nemipterus fish, further investigation will be needed.
Phylogeny; nemipteridae;COIgene;RAG2 gene
广东省自然科学基金项目(2016A030310236);广东省教育厅青年创新人才类项目(2014KQNCX16);广东省大学生创新创业训练计划项目(201511347028)资助 Supported by the Natural Science Foundation of Guangdong Province(2016A030310236);Foundation for Young Talents in Higher Education of Guangdong(2014KQNCX164);Undergraduate Training Program for Innovation and Entrepreneurship(201511347028)
2016-12-12;
2017-04-12
梁日深(1984-),男,讲师,研究方向:水生生物资源及保护利用。E-mail: cheetahliang@126.com
Q953
A
1672-5174(2017)08-074-08
10.16441/j.cnki.hdxb.20160407
梁日深,苏国茂,陈冬青, 等. 基于COI及RAG2基因序列15种金线鱼科鱼类分子系统进化关系[J]. 中国海洋大学学报(自然科学版), 2018, 47(8): 74-81.
LIANG Ri-Shen, CHEN Dong-Qing, SU Guo-Mao, et al. Molecular phylogenetic relationship of 15 nemipteridae fishes based on mitochondrialCOIand nuclearRAG2 gene sequnces [J]. Periodical of Ocean University of China, 2018, 47(8): 74-81.
