基于生物加工的PET功能修饰的进展
2017-05-05李育强王宏阳巩继贤季玮玮李辉芹
李育强+王宏阳+巩继贤+季玮玮+李辉芹+李政+李秋瑾+张健飞

摘要:PET材料的功能化和高性能化日益受到重视,生物加工是进行聚酯功能化改性及环保处理的一种有效方法。本文综述了用于PET生物加工的两种方式的特性及应用情况,着重介绍了3种不同形态的PET进行生物水解表面改性修饰的过程,以及PET的物理特征、结构和水解产物等对其表面改性修饰能力的影响,最后对聚酯生物加工处理的前景做了展望。
关键词:PET;生物加工;表面改性;水解酶
中图分类号:TS101.4;Q819 文献标志码:A
Functional Modification of PET Based on Biological Processing
Abstract: Functionalization and high-performance have become increasingly important to PET, while biological processing is an effective way for realizing functional modification and environmental-friendly treatment of PET. In this paper, the characteristics and applications of two PET biological processing methods were reviewed by elaborating on the process of surface modification of four different forms of PET via biological hydrolysis as well as the impact of the physical characteristics and structure of PET and the hydrolysis products on the surface modification ability. The prospects of PET biological processing were also discussed.
Key words: PET; biological processing; surface modification; hydrolytic enzyme
生物加工作為生物技术的一个重要发展方向,在材料加工和处理方面开始发挥日益重要的作用。基于生物加工的PET表面功能化修饰,本质上是通过生物催化反应使底物表面发生有限分解,从而产生极性基团。聚合物底物的大分子链应尽可能地从中间断裂,而不是发生端基的分解。因此,对实现PET材料的表面功能化处理而言,关键在于大分子链发生分解的部位。当前,生物催化降解已经扩展到能够以高分子聚合物作为底物的生物降解,生物表面改性也从单一性能发展为多种性能得到优化。
涤纶具有诸多优良性能,已被大量用于衣料、床上用品、各种装饰布料以及其他工业纤维品中,但也有不足之处,相对于棉织物其在生物加工方面的生物抛光以及功能改性方面的研究进程缓慢。目前,有报道Yoshida S等从几百种自然菌中筛选出一种细菌能够直接以PET为主要能源和碳源,将PET有效转化为环保单体,为PET在生物加工处理方面提供了良好契机。
本文将有针对性地总结PET生物加工的两种主要方法,以及PET的纤维、膜和颗粒等3种不同形态结构的生物加工表面修饰能力的进展,希望通过此研究能进一步改善生物催化降解以及改性聚酯过程。
1 PET生物加工方法
被用于PET表面功能化修饰的生物加工技术主要有酶和菌的生物加工两种方法。
1.1 酶
目前PET进行生物加工处理所用的水解酶包括脂肪酶、角质酶、酯酶和蛋白酶,均能水解酯键,其中最常用的是脂肪酶和角质酶。
脂肪酶(lipase,EC 3.1.1.3)全称三酰基甘油酰基水解酶。除了酶的一般特性外,其还具有独特的性质:只能在异相系统即水-油界面或有机相中作用,对水溶性底物没有催化作用。结构上,绝大多数微生物脂肪酶有一个“盖子”结构(lid),覆盖在脂肪酶活性中心上方,当脂肪酶与疏水界面接触时,“盖子”打开,活性中心才会外露。与化学碱处理相比,脂肪酶在相对低的浓度(0.01 g/L)、温和的反应温度(25 ℃)和较短的反应时间(10 min)下就能改善涤纶织物的润湿性和吸水性能。同时脂肪酶对涤纶表面进行降解后,纤维表面变得比较粗糙,水的接触角变小,表面形成极性基团羟基(—OH)和羧基(—COOH),亲水性得到显著改变。
角质酶(cutinase,EC 3.1.1.74)是一种丝氨酸酯酶,已证实至少有22种植物病原真菌能产生角质酶。角质酶的活性中心位于整个结构的顶端,并暴露于溶剂中,没有盖子结构遮挡,因此没有界面活化现象。角质酶的分子量普遍较小;最适温度为30 ~ 60 ℃;最适pH值偏碱性,通常为 8 ~ 10,当pH值低于 7 时酶的活性急剧下降。据文献报道,源于真菌F.solani的角质酶在PET生物处理中运用较多。Nimchua等采用源于不同菌属的角质酶在相同条件下处理PET纤维,检测对苯二甲酸(TA)的生成量,发现相同酶活性下两种角质酶释放的TA量相差两倍,因此同类酶来源不同则催化效率也有差异。
1.2 菌
在最初对菌的研究中,人们的焦点主要集中于从对酯键有水解作用的商品化的脂肪酶或酯酶中筛选出可用于催化PET水解的酶,但效果均不理想。于是人们转向环境微生物,用PET单体及其模拟物作为底物,进行相关菌株的筛选与分离。尽管PET被看作“不可降解的聚酯”,但有学者采用源于Trichosporonh和Arthrobacter的菌株作用于PET纤维的无定形区,8 周后能使纤维失重约15%;之后,Müller等使用放射菌类嗜热裂孢菌(Thermobifida fusca)解聚PET膜和颗粒,在55 ℃下其腐蚀速度能达到每周 8 ~ 17 μm。目前,日本学者筛选的Ideonella sakaiensis 201-F6细菌在30 ℃下作用 6 周后基本能使PET膜(结晶度为1.9%)完全降解。目前用于处理PET的菌株,从种类上看涵盖了细菌、放线菌、真菌和霉菌。同时微生物菌处理PET进行生物分解,是伴随菌的生长和底物分解的生物过程,远比直接以酶为生物催化剂的过程复杂。
2 不同形态的PET生物加工
2.1 PET膜的生物处理
对于PET膜的处理,酶的选择也是一个重要因素。Eberl等使用角质酶催化水解PET膜后有TA、单(2-羟乙基)对苯二甲酸(MHET)产物生成,而经脂肪酶处理没有这些产物生成,表明其不能催化水解PET膜。
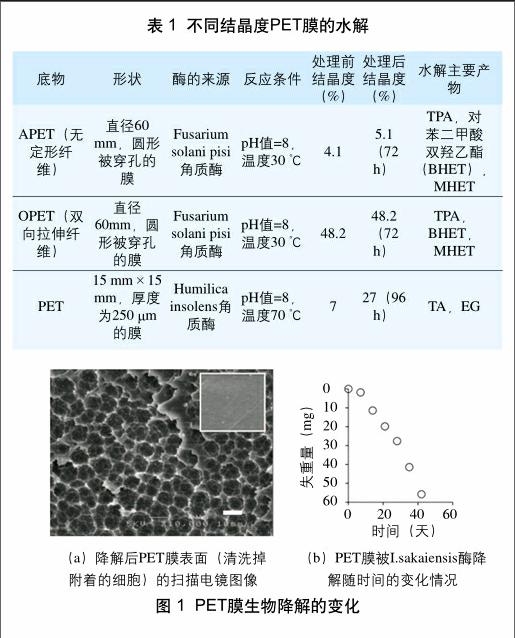
PET的结晶度很大程度上限制了其生物加工能力。为消除结晶度的影响,张健飞等采用自制的无结晶区域存在但高分子结构没有改变的PET膜,经过 3 个月的生物加工后,其薄膜表面有明显的孔洞刻蚀的痕迹出现。Ronkuist等和Vertommen等探究了几种角质酶水解不同结晶度的PET膜,其处理效果如表 1 所示。结晶度低的PET在一定的生物加工处理后,其结晶度有较明显增加,而结晶度高的PET却没有增加,说明结晶度是一个重要的影响因素。此外降解发生的温度与聚酯结晶部分的熔融温度有关,酶在接近聚酯玻璃化温度条件下催化处理低结晶度(7%)的PET膜,96 h后其失重率可达97%,并且降解的水溶性产物仅有TA和乙二醇(EG);同时角质酶作用于结晶度为7%的PET的活性比作用于结晶度为35%的PET要高10倍。因此,当酶有较好的热稳定性时,其水解效果更好。最近,日本京都理工学院的Yoshida S等搜集了带有沉积物、土壤、污水以及来自PET瓶回收站点活性污泥的250份PET残片样品,从中筛选出了能够以低结晶度PET膜(结晶度为1.9%)为碳源和能源的微生物Ideonella sakaiensis 201-F6。图 1(a)对比了PET膜在经酶处理前后的SEM扫描图像,发现经过一定时间处理后PET膜大部分能被I.sakaiensis酶降解;图 1(b)则进一步表明在30 ℃下培养 6 周后PET膜基本完全降解。综上可以看出,PET膜生物可降解性受到其化学结构、结晶度和分子量的影响。
2.2 PET颗粒的生物处理
随着微纳米技术的发展,科研工作者们尝试将PET制备成微纳米颗粒,增加与酶和菌株的接触面积。对于粒径为 4 ~ 6 mm的PET颗粒,酶在最适条件下处理 8 h后能释放出芳香族化合物。酶法水解不溶性聚酯是一个表面侵蚀的过程,因为酶分子的大小无法直接渗透进入聚合物内部,因此可以通过扩大聚酯的可利用表面来大大提高降解的速度,PET颗粒的粒径尺寸越小,则PET粒子与酶接触比表面积越大。
Herog等人使用脂肪酶法水解了几种粒径在50 ~ 250 nm的脂肪族和芳香族聚酯纳米粒子,可能是PET小尺寸粒子有较高的比表面积,相比膜的降解速度酶水解聚酯纳米粒子的速度明显提高。Wei等对比了由PET颗粒、膜和纤维所制成的纳米粒子的水解速率和水解吸附平衡常数,得出PET的生物降解能力主要受聚酯链段的流动性影响较大,因为链段的流动性决定了酶与酯键的亲和力和可接近性,从而决定了酶水解聚酯的速度,而链的流动性又与材料的结晶度相关。而Barth等发现水解中间产物MHET和BHET对水解酶水解PET纳米粒子有抑制作用。MHET进一步水解生成TA的速度比生成BHET慢得多,这是在PET纳米粒子降解过程中限制水解酶降解效率的主要因素。此外酶与纳米粒子之间的大小比例也会影响降解速率,纳米粒子表面的强曲率可能会影响酶的吸附,从而影响其催化降解。
2.3 PET纤维的生物处理
目前PET纤维的表面功能化主要采用碱减量法,生物加工的方法只是作为辅助手段。随着对PET纤维生物加工研究的逐步深入,发现酶水解使纤维表面的羟基和羧基基团的数量增多,从而增加吸水性;而碱处理没有羟基和酸性基团出现。从酶的角度来看,其种类影响PET的降解修饰效果。李旭明等人用脂肪酶(L3126)处理涤纶织物,发现一定条件下脂肪酶能有效催化涤纶织物水解,但效果不理想,仅对涤纶表面产生一定侵蚀,但能有效改善其亲水性。同时,Akbar等研究了脂肪酶水解对涤纶长丝纱的改性作用,酶水解后,涤纶纱的失重率小于0.5%,强度损失达10%,断裂延伸率下降7% ~ 25%,水吸收率升高达50%;酶水解仅限于纤维表面,与纤维的拉伸率相比,纤维表面积对酶水解作用的影响更大。相比角质酶,Nimchua等采用源于Fusarium oxysporum(尖孢镰刀菌属)strain LCH1和Fusarium solani(腐皮镰刀菌)两种真菌的角质酶,在相同浓度、pH值为 7、30 ℃下分别处理PET织物(20 cm×2 cm),经过长达168 h的处理后,PET织物的吸水性能发生一些变化,如表 2 所示(结果以10 min内水的升高高度表示)。
从纤维角度来看,起初针对纤维状的PET采用天然微生物降解,从电镜上观察虽有较明显的刻蚀痕迹,但其降解率很低。由于PET结构紧密同时纤维的表面光滑,因而酶难以攻击和降解其结构,因而形成的侵蚀迹象较浅,降解作用有限。后来经过筛选和诱导,酶能够对纤维表面产生明显均匀的刻蚀效果。Wang等利用DTP、BHET和聚酯短纤维作为诱导物,使米曲霉菌(Aspergillus oryzae)产生脂肪酶来水解PET织物底物,发现BHET的诱导效果最好,PET经过其诱导酶处理后表面产生很多的羧基基团,反应后底物中有块状PET、TA产生并有单(2-羟基乙基)对苯二甲酸酯(mono(2-hydroxyethyl)terephthalate,MHT)生成。发现酶优先水解PET的无定形区域,释放出大量的MHET、TA和BHET;而半结晶纤维只有MHET和TA释放出来,由于在无定形区聚酯链具有高彈性,因而酶的活性点容易接近聚酯链。Alisch等研究发现酶与纤维的作用在温度接近纤维的玻璃化温度(大约80 ℃)时,PET链容易移动到聚合物表面,因而认为较高的温度有利于催化水解PET。生物加工处理后PET酯键基团减少,末端亲水基团(—OH、—COOH)增加,使纤维表面具有亲水性,涤纶织物的抗污性、润湿性和染色性能均得到不同程度的改善。纤维的动电电荷、纤维粘着性以及纤维表面键的稳定性,都是不容忽视的影响因素。
3 结语
生物加工作为一种清洁环保工艺,在许多领域已得到广泛应用。当前,业界虽然在通过PET生物加工赋予PET材料功能化方面取得了一定进展,如聚合物底物表面形成新的基团,能够增加材料表面的亲水性,但仍有一些问题亟待解决。目前处理PET所用的商业酶制剂并不是单独以PET作为底物而设计的酶,因而有待发现水解PET效果更佳、具有高活性并能够进入PET内部进行降解的新酶。酶催化处理温度通常低于PET的玻璃化转变温度,可添加有效的助剂以降低聚酯的玻璃化转变温度,并在低温条件下使非结晶部位的纤维易于接近。从PET材料的特性来看,其分子链上存在一定量的酯键,内部具有结晶区,熔融温度较高,因此可通过优化PET分子结构、降低聚酯的聚合度和结晶度等方式从而使其得到良好的生物降解性能,其他还包括增加底物与微生物的接触面积以及与微生物的亲和性等方法。
参考文献
[1] 巩继贤,王宏阳,李辉芹,等. 聚酯生物加工技术研究进展[J].纺织学报,2016,37(3):166-172.
[2] 李旭明,师利芬,钱志华,等. 脂肪酶处理对涤纶织物亲水性能的改善[J]. 纺织学报,2012,33(4):91-94.
[3] Yoshida S, Hiraga K, Takehana T, et al. A bacterium that degrades and assimilates poly(ethylene terephthalate)[J]. Science,2016,351(6278):1196-1199.
[4] 王小花,洪枫,陆大年,等. 脂肪酶在纺织工业中的应用[J]. 毛纺科技,2005(6):22-24.
[5] Eberl A, Heumann S, Brückner T, et al. Enzymatic surface hydrolysis of poly(ethylene terephthalate)and bis(benzoyloxyethyl)terephthalate by lipase and cutinase in the presence of surface active molecules[J]. Journal of Biotechnology, 2009, 143(3):207-212.
[6] Hsieh Y L, Cram L A. Enzymatic hydrolysis to improve wetting and absorbency of polyester fabrics[J]. Textile Research Journal, 1998, 68(5):311-319.
[7] 武荣瑞. 聚酯的生物改性[J]. 高分子通报,2010(11):36-47.
[8] Nimchua T, Punnapayak H, Zimmermann W. Comparison of the hydrolysis of polyethylene terephthalate fibers by a hydrolase from Fusarium oxysporum LCH I and Fusarium solani f. sp.pisi[J]. Biotechnology Journal, 2007, 2(3):361-364.
[9] Ronkvist A M, Xie W, Lu W, et al. Cutinase-catalyzed hydrolysis of poly(ethylene terephthalate)[J]. Macromolecules, 2009, 42(14):5128-5138.
[10] Zhang J, Gong J, Shao G, et al. Biodegradability of diethylene glycol terephthalate and poly(ethylene terephthalate) fiber by crude enzymes extracted from activated sludge[J]. Journal of Applied Polymer Science,2006, 100(5):3855-3859.
[11] Müller R, Schrader H, Profe J, et al. Enzymatic degradation of poly(ethylene terephthalate):rapid hydrolyse using a hydrolase from T. fusca[J]. Macromolecular Rapid Communications,2005, 26(17):1400-1405.
[12] 張健飞,刘中君. 对苯二甲酸及聚酯膜生物降解[J]. 天津大学学报,2006(S1):324-328.
[13] Vertommen M A M E, Nierstrasz V A, Veer M V D, et al. Enzymatic surface modification of poly(ethylene terephthalate)[J]. Journal of Biotechnology,2005, 120(4):376-386.
[14] Marten E, Müller R, Deckwer W. Studies on the enzymatic hydrolysis of polyesters. II. Aliphatic–aromatic copolyesters[J]. Polymer Degradation and Stability,2005, 88(3):371-381.
[15] Alisch M, Feuerhack A, Müller H, et al. Biocatalytic modification of polyethylene terephthalate fibres by esterases from actinomycete isolates[J]. Biocatalysis and Biotransformation,2004, 22(5-6SI):347-351.
[16] Herzog K, Müller R J, Deckwer W D. Mechanism and kinetics of the enzymatic hydrolysis of polyester nanoparticles by lipases[J]. Polymer Degradation and Stability,2006, 91(10):2486-2498.
[17] Wei R, Oeser T, Barth M, et al. Turbidimetric analysis of the enzymatic hydrolysis of polyethylene terephthalate nanoparticles[J]. Journal of Molecular Catalysis B:Enzymatic,2014, 103(5):72-78.
[18] Barth M, Oeser T, Wei R, et al. Effect of hydrolysis products on the enzymatic degradation of polyethylene terephthalate nanoparticles by a polyester hydrolase from Thermobifida fusca[J]. Biochemical Engineering Journal,2015, 93(93):222-228.
[19] Khoddami A, Morshed M, Tavanai H. Effects of enzymatic hydrolysis on drawn polyester filament yarns[J]. Iranian Polymer Journal,2001, 10(6):363-370.
[20] 王小花,洪楓,陆大年,等. 涤纶的酶法改性研究进展[J]. 东华大学学报(自然科学版),2005(5):123-126.
[21] Zhang J, Wang X, Gong J, et al. A study on the biodegradability of polyethylene terephthalate fiber and diethylene glycol terephthalate[J]. Journal of Applied Polymer Science,2004, 93(3):1089-1096.
[22] Wang X, Lu D, Jonsson L J, et al. Preparation of a PET-hydrolyzing lipase from Aspergillus oryzae by the addition of bis(2-hydroxyethyl)terephthalate to the culture medium and enzymatic modification of PET fabrics[J]. Engineering in Life Sciences,2008, 8(3):268-276.
