棉花育种中黄萎病抗性连锁SSR标记辅助选择的效果
2017-03-17冯常辉张友昌李国荣宋志红孟庆忠
冯常辉+张友昌+李国荣+宋志红+孟庆忠+金利容+秦鸿德

摘要:利用长江流域棉花(Gossypium spp.)育种重要亲本苏棉12和荆55173(感黄萎病)与抗黄萎病资源Sicala V1和中21373配制两套感病×抗病杂交组合(苏棉12×Sicala V1和荆55173×中21373),在其杂交组合的自交子代群体中挑选与黄萎病抗性相关的9个SSR(Simple sequence repeat)标记实施分子标记辅助选择(Marker-assisted selection,MAS)育种。结果表明,通过单标记选择,8个SSR标记的aa与AA两种基因型个体间的黄萎病校正病情指数(Correction disease index,CDI)差异达到极显著或者显著水平。利用多元逐步回归分析得到的单标记、双标记以及多标记3种筛选方法的效果,结果表明合理采取多个有效标记同时筛选能够提高选择的准确度。
关键词:棉花(Gossypium spp.);黄萎病;标记辅助选择育种;简单重复序列
中图分类号:S562 文献标识码:A 文章编号:0439-8114(2016)23-6045-05
DOI:10.14088/j.cnki.issn0439-8114.2016.23.008
Abstract: The self-pollinated progenies derived from two cotton (Gossypium spp.) crosses(Jing55173×Zhong21373 and Sumian12×Sicala V1) of the susceptible cultivars(Sumian12 and Jing55173) and the highly tolerant cultivars(Sicala V1 and Zhong21373) to Verticillium wilt were used for resistance breeding with marker-assisted selection(MAS). MAS of these progeny populations was put forward by nine simple sequence repeats(SSR) which related to quantitative trait locis(QTL) for Verticillium wilt resistance. For eight of nine SSRs,the difference of correction disease index(CDI) between aa and AA genotype individuals reached either significant or highly significant levels. The effects among single marker, two markers and multi-markers on MAS of cotton were compared using three makers that were selected by multiple stepwise regression analysis, the results showed that selective effects were enhanced through multi-markers way.
Key words: cotton(Gossypium spp.); Verticillium wilt; marker-assisted selection breeding; simple sequence repeats
黄萎病是棉花(Gossypium spp.)高产和稳产的主要障碍,棉花黄萎病防治问题仍旧是世界性难题[1]。抗性品种的选育是最有效途径。然而,抗病性既受环境条件的影响,又受制于寄主与病原菌之间的互作。育种进程势必会因为误判病情而受到耽误[2]。
分子标记辅助选择(Molecular marker assisted selection,MAS)可以将传统表型选择转变为直接选择基因型[3]。MAS抗病育种在水稻上已有不少成功先例[4],在小麦[5]、玉米[6]等作物上也有少量成功报道。MAS育种也在棉花上陆续开展,集中在纤维品质、抗虫、抗病方面。郭旺珍等[7]使用MAS聚合了高强纤维QTL(来源于品系7235)和抗虫基因,培育出新品系南农85188。石玉真等[8]利用与高强纤维QTL紧密连锁的2个SSR标记在不同群体中进行MAS,快速改良了sGK321和sGK9708的纖维强度。祁伟彦等[9]对以中植372为亲本的各世代材料,利用黄萎病抗性连锁SSR标记跟踪检测,筛选得到抗性强的后代,进而培养出抗病优质新品种。
湖北省的黄萎病育种受到气候限制,相对黄河流域棉区和新疆棉区品种而言,湖北省棉花品种对黄萎病的抗性水平偏低,影响了湖北省高产品种在北方棉区的推广使用。本研究试图在抗病育种程序中,利用连锁SSR标记对部分已经定位的黄萎病抗性QTL进行MAS,验证SSR的MAS效果,筛选获得可以应用于棉花黄萎病抗性MAS中的SSR标记及其组合,并初步探讨影响黄萎病抗性MAS育种效率的因素,为黄萎病抗性的MAS育种提供理论依据。
1 材料与方法
1.1 材料
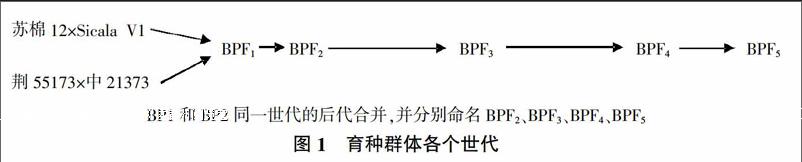
苏棉12和荆55173是长江流域棉花育种的重要亲本,丰产性和广适性较好,属于感黄萎病类型;Sicala V1和中21373是从国外和黄河流域引进的抗黄萎病资源。本研究选用上述材料作为亲本,配制两套感病×抗病杂交组合,分别为组合BP1(苏棉12×Sicala V1)和组合BP2(荆55173×中21373)(图1)。BP1F1和BP2F1连续自交,各自获得不同世代,包括F2、F3、F4、F5。
1.2 黄萎病抗性鉴定
黄萎病抗性鉴定试验采用2种不同方式,并设置3次重复。温室里利用伤根接菌的方法鉴定黄萎病抗性。当棉苗长至二叶一心时,使棉苗外围根系受到损伤(伤根程度尽量一致)后定量注射浓度2.48×107个/mL、具有中致病力的黄萎病菌液(病原菌从湖北省天门市发病棉区采集后分离出单菌落)18 mL。然后置于温度为27 ℃,光/暗条件为16 h/8 h的温室里继续培养,定期浇水保持一定湿度。接菌后第30天,棉苗黄萎病病情逐渐稳定,第33天进行病情调查。大田里采用人工接种黄萎病菌的方式。移栽时先将营养钵置于浓度为1.03×107个/mL黄萎病液中浸泡,再移栽至大田。
1.3 抗病性调查
棉花黄萎病发病级别按0~4级划分成5级[10]。感病对照为冀棉11。校正病情指数(Correction disease index,CDI)=个体实际病情指数×50.00/感病对照实际病情指数。其中,实际病情指数=Σ(各级病株数×该病级值)/(调查总株数×4)×100。病级(Disease grade,DG)=Σ(各级病株数×该病级值)/调查总株数。
1.4 SSR标记和基因型检测
DNA抽提采用改良的CTAB法[11]。SSR标记的扩增、电泳和染色程序参照孔祥瑞等[12]和张军等[13]的方法。为了便于统计分析,对于共显性标记,电泳条带与中21373相同时,个体基因型记作AA,否则记作Aa或aa;对于显性标记,电泳条带与中21373相同时,个体基因型记作Aa,否则记作aa。从已报道的36个黄萎病抗性相关SSR标记[14-19]中,选择在2个群体的4个亲本中具有多态性的9个SSR标记进行MAS。其中标记BNL1053检测到2个多态性位点,分别记作BNL1053-1和BNL1053-2(表1)。
1.5 黄萎病抗病性状的SSR标记辅助选择
子代群体BPF2(共680个)依照大田产量(主要考察结铃性)初步选择之后得到BPF3(含75个株行)。然后在海南繁殖加代得到BPF4。本轮标记辅助选择过程通过比较个体不同位点基因型与抗病亲本基因型差异,淘汰差异十分明显的个体。BPF5利用SSR标记着重选育抗或者较抗黄萎病的材料(图1)。
1.6 统计分析
使用DPS软件进行相关分析和多元逐步回归分析,利用Excel 2003进行t检验。温室能为棉苗发病创造良好的发病条件[20],其鉴定结果比大田更稳定,因此回归分析对象定为个体校正病情指数(YCDI),因子为SSR标记(见1.4)基因型值(Xmarker)。其中,AA、Aa和aa 3种基因型分别赋值为“2”、 “1” 和“0”。根据拟合模型中所保留的因子数命名为拟合模型1~8。
2 结果与分析
2.1 黄萎病抗性鉴定结果
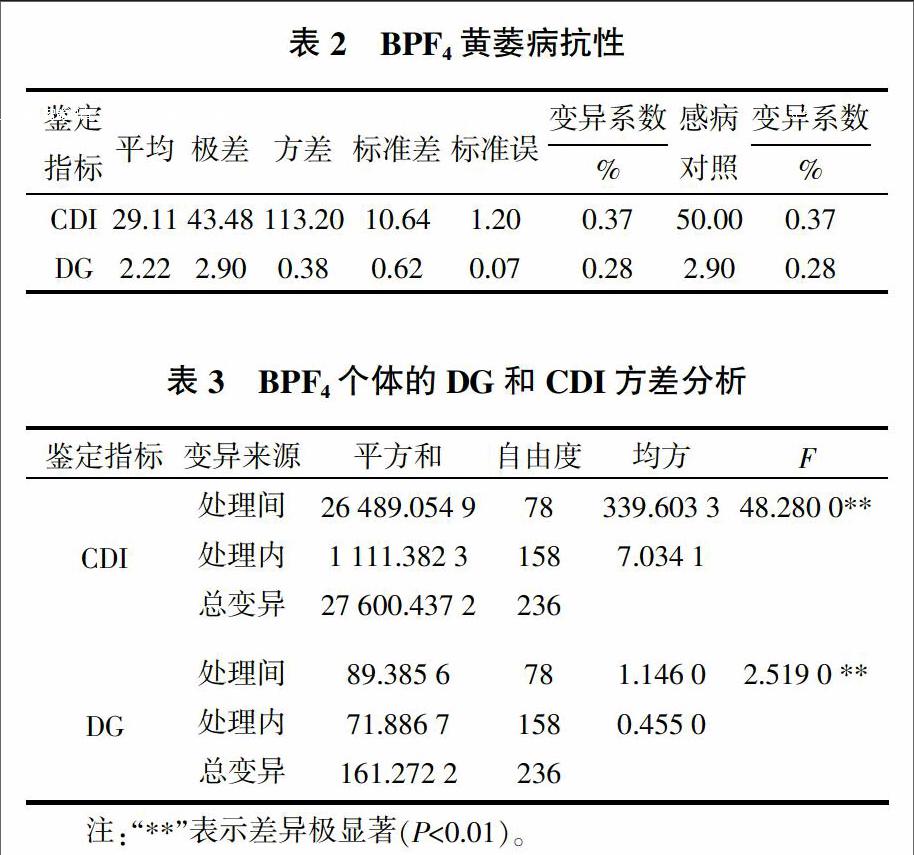
该育种群体抗病性状变异程度较大,病情离散分布。田间抗病性状变异程度略低于室内(表2)。另外,方差分析结果也表明BPF4个体之间的抗病性差异极显著(表3)。
2.2 连锁SSR标记与黄萎病抗性的多元逐步回归分析
用于MAS的9个SSR标记位点的基因型值(Xmarker)对校正病情指数(YCDI)产生极显著的负向影响(表4)。经逐步回归分析法扣除其中1~8个标记位点的影响,建立Xmarker与YCDI的拟合模型。当扣除6个标记位点时,YCDI与拟合模型中的各个因子的偏相关系数(r)全部达到显著水平,而且其调整相关系数(Ra)为0.71,也十分接近最高值0.72,直观地反映了XNAU1167、XNAU5467、XNAU3053对黄萎病YCDI产生更重要的影响。因此确定YCDI=47.036 481 7-8.245 074 816 XNAU1167-4.120 554 754 XNAU5467-4.587 778 119XNAU3053为较优拟合模型。
2.3 单标记对黄萎病抗性选择的效果
利用NAU1167、NAU3053、NAU1225、NAU5428、NAU2859、NAU5120共6个标记中任一标记筛选后,基因型aa与AA类型间CDI差异均达到极显著水平(表5)。利用NAU5467和BNL1053-1两个标记中任一标记筛选后,基因型aa与AA类型间差异仅达到显著水平。标记NAU1167、NAU5467、NAU3053在DNA电泳检测时表现为共显性,根据不同个体的标记基因型能将BPF4及其亲本分为3类,分别属于aa(a1a1、a2a2、a3a3)、Aa(A1a1、A2a2、A3a3)和AA(A1A1、A2A2、A3A3)基因型。标记NAU5467在基因型aa与Aa类型间进行t测验时不显著,但是在aa与AA类型间达到显著水平,同时在AA与Aa类型间达到极显著水平。标记NAU1167和NAU3053在不同类型之间进行t测验时都达到极显著水平。
2.4 双标记、多标记对黄萎病抗性選择的效果
BPF4及其亲本经NAU1167/NAU5467、NAU1167
/NAU3053、NAU5467/NAU3053双标记筛选后,aaaa与AAAA类型间个体黄萎病CDI的差异都达到极显著水平(表5)。经三标记同时筛选后,aaaaaa与AAAAAA类型间个体黄萎病CDI的差异达到极显著水平,而且其均值差为25.54,大于单标记和双标记选择结果。经单标记筛选后,aa与AA类型间CDI的均值差明显低于双标记选择;经双标记筛选后,aa与AA类型间CDI的均值差低于多标记选择。由此可见,当不同标记用于黄萎病抗性辅助选择时,同时运用多个有效标记进行选择能够提高选择的准确度。
2.5 标记组合对大田黄萎病抗性选择的效果
综合以上分析结果筛选到最优标记组合为NAU1167/NAU5467/NAU3053。利用该标记在大田进行黄萎病抗性选择后,得基因型a1a1a2a2a3a3的DG值为3.60,A1A1A2A2A3A3的DG值为2.13,两者之间的均值差为1.47,抗感两基因型间差异达极显著水平。抗性较好的个体总体表现与感病对照相比,病级相差0.77。说明该标记组合的辅助选择效果在大田依然能够保持。尽管如此,由于其个体数都较少,因此还需作后续验证。
3 讨论
3.1 棉花黄萎病抗性辅助选择中有效分子标记的筛选
由于不同研究间存在材料遗传背景、发病环境条件、图谱标记密度等差异,定位结果间一致性较差。除此之外,仅仅通过作图群体估算的QTL效应仍有待验证。本次研究涉及黄萎病相关SSR标记仅有部分在MAS育种上得到验证。标记NAU5428、NAU1167、NAU5467、NAU2859均未见其应用于棉花黄萎病抗性MAS的报道。NAU5120和BNL1053已经被试图应用于MAS中,并未达到预期效果[21]。标记NAU3053和NAU1225已经被应用于分子标记辅助聚合陆地棉抗黄萎病QTL中,获得的材料聚合了多个QTLs,其抗病性与对照5026相比显著增强[16]。
QTL定位结果能否应用于实际育种中,首先需要解决其连锁标记是否有效的问题。构建群体BP的最终目标是获得产量高、黄萎病抗性与抗源亲本相当的资源材料。因此,在此类群体中实施黄萎病连锁SSR标记辅助选择,比较标记选择和接菌鉴定结果,评价各个标记辅助选择的效果。效果佳的SSR标记被他人直接利用后,MAS仍然见效。
3.2 影响黄萎病抗性辅助选择育种效率的因素
QTL效应大小及其与侧翼标记的遗传距离是影响MAS顺利、准确进行的主要因素。黄萎病抗性相关QTL分为主效和微效两种类型。李志坤等[22]利用来源于Pima90-53中的SSR标记进行辅助选择,160个BC1F1代单株材料中,平均检出率达到84.0%,还发现少许单株虽然有抗病标记但是表现感病。推断是由于标记BNL3255与目标QTL连锁不够紧密所致(其遗传距离为13.7 cM)。孔祥瑞等[12]采用分子标记辅助选择技术将与陆地棉黄萎病抗性相关的18个SSR标记用于大田辅助选择育种,其中的3个连锁SSR标记BNL2865、BNL1721、BNL3452的贡献率分别达到3.06%、13.89%、8.28%,并依据标记基因型分组的CDI结果推测BNL2865在MAS上无效。本研究中,BPF4及其亲本经标记BNL1053-1筛选后,抗感两种类型的CDI仅达到显著水平;而经BNL1053-2筛选后达不到显著水平。推测前者确实与黄萎病QTL连锁,只是连锁距离偏大,超过5 cM;后者与黄萎病性状可能无关联。此结果与前人研究结果基本一致,再次表明该标记不适用于棉花黄萎病抗性MAS[21]。标记NAU3053和NAU1167可解释黄萎病抗性变异的百分率较高,并且比NAU5467高。经标记NAU3053和NAU1167篩选后,抗感两种类型的CDI均能达到极显著水平,经标记NAU5467筛选后,抗感两种类型的CDI只能达到显著水平。从一定程度上表明了挑选与效应较大QTL相关标记是提高黄萎病抗性MAS育种效率的重要途径。
棉花抗黄萎病相关QTL或其紧密连锁标记间的相斥和相引连锁关系也是影响MAS效率的因素之一。NAU1225是通过江苏棉区培育的品系60182检测出的黄萎病抗性连锁标记,其连锁距离小[14],而在育种群体BPF4中进行辅助选择的相对效果不太明显,可能是连锁相改变引起的。只有在彻底了解其遗传机制的基础上,才能使MAS目标更明确。
未知QTL也是影响MAS效率的因素之一。多元逐步回归分析得到Ra范围为0.65~0.72,由此反映抗病性受其他未知QTL(解释的变异至少占28%)的影响。
育种实践中为选择到理想、优良的资源材料,大多需扩大群体数量。欲开展大规模MAS成本很高,但合理减少标记可以使其成本大幅下降。依据育种目标对连锁标记进行细致筛选,以运用最少的标记达到较为理想的选择效果[23]。为简化标记检测过程,在此应忽略回归分析中相关系数低的标记的选择作用,例如,标记NAU1225、NAU5428、NAU2859虽然能解释相对较大的黄萎病抗性变异或者与抗病性存在较强的关联,但因其R低于其余6个标记,也会被剔除。标记NAU5120和NAU3053同在D7染色体上,距离17.0 cM,二者可能与相同的QTL连锁。为避免MAS中标记的选择作用重叠,优先选用其中筛选效果更佳的标记NAU3053。综上所述,最终挑选出3个SSR标记(NAU3053、NAU1167和NAU5467)建立Xmarker与YCDI的拟合模型。
QTL/基因定位方法的不断革新以及棉花基因组图谱的日趋完善将使MAS现存的诸多问题不断得到解决。近年来全基因组关联分析(Genome-wide association study,GWAS)成为遗传育种领域的热点。GWAS直接利用基因本身或基因附近微小区域的分子标记与性状表型的关联来实现基因的精细定位[24]。基于SNP标记的GWAS所定位的QTL解释表型变异率高,可增强MAS的目的性和准确性。陆地棉基因组测序的完成,极大地推动了棉花的遗传改良和基础生物学研究[25]。快速、高通量分型技术——SNP芯片的应用,有助于基因精细定位以及克隆,发掘SNP位点。可以预见,寻找与黄萎病相关QTL/基因紧密连锁的SNP标记,设计配套的MAS最佳方案,势必成为真正实现棉花黄萎病MAS育种快速高效的突破口。
参考文献:
[1] 喻树迅,范术丽.我国棉花遗传育种进展与展望[J].棉花学报,2003,15(2):120-124.
[2] 闵留芳,张天真,潘家驹.有关棉花黄萎病(Verticillium dahlia Kleb.)抗性遗传研究的几个问题[J].棉花学报,1995,7(4):197-201.
[3] MOHAN M,NAIR S,BHAGWAT A,et al. Genome mapping,molecular markers and marker assisted selection in crop plants[J].Mol Breeding,1997(3):87-103.
[4] 李爱宏,潘存红,戴正元,等.以标记辅助选择改良江苏主栽粳稻品种“淮稻5 号”黑条矮缩病抗性[J].作物学报,2012,38(10):1775-1781.
[5] 高安礼,何华纲,陈全战,等.分子标记辅助选择小麦抗白粉病基因Pm2、Pm4a和Pm21的聚合体[J].作物学报,2005,31(11):1400-1405.
[6] 李常保,宋建成,姜丽君,等.玉米抗粗缩病病毒(MRDV)基因的RAPD标记及其辅助选择效果研究[J].作物学报,2002,28(4):564-568.
[7] 郭旺珍,张天真,朱协飞,等.分子标记辅助选择的修饰回交聚合育种方法及其在棉花上的应用[J].作物学报,2005,31(8):963-970.
[8] 石玉真,刘爱英,李俊文,等.与棉花纤维强度连锁的主效QTL应用于棉花分子标记辅助育种[J].分子植物育种,2007,5(4):521-527 .
[9] 祁伟彦,张永军,张天真,等.基于人工病圃筛选和分子标记辅助的棉花抗黄萎病育种方法研究与应用[J].分子植物育种,2012,10(5):607-612.
[10] 李社增,马 平,HUANG H C,等.相对病情指数划分棉花品种抗病性的统计学基础[J].棉花学报,2003,15(6):344-347.
[11] PATERSON A H,BRUBAKER C L,WENDEL J F.A rapid method for extraction of cotton(Gossypium spp.) genomic DNA suitable for RFLP or PCR analysis[J].Plant Molecular Biology Reporter,1993,11(2):122-127.
[12] 孔祥瑞,王红梅,陈 伟,等陆地棉黄萎病抗性的分子标记辅助选择效果[J].棉花学报,2010,22(6):527-532.
[13] 张 军,武耀庭,郭旺珍,等.棉花微卫星标记的PAGE/银染快速检测[J].棉花学报,2000,12(5):267-269.
[14] 蒋 锋,赵 君,周 雷,等.陆地棉抗黄萎病基因的分子标记定位[J].中国科学C辑:生命科学,2009,39(9):849-861.
[15] ZHAO Y L,WANG H M,CHEN W,et al.Genetic structure, linkage disequilibrium and association mapping of Verticillium wilt resistance in elite cotton(Gossypium hirsutum L.) germplasm population[J].Plos One,2014,9(1):e86308.
[16] 張仁善.SSR分子标记辅助聚合陆地棉抗黄萎病QTL[D].南京:南京农业大学,2010.
[17] 杨 昶.棉花抗黄萎病基因分子标记定位研究[D].南京:南京农业大学,2007.
[18] FANG H,ZHOU H P,SANOGO S,et al.Quantitative trait locus analysis of Verticillium wilt resistance in an introgressed recombinant inbred population of upland cotton[J].Mol Breeding,2014,33:709-720.
[19] WANG P,NING Z Y,LIN L,et al. Genetic dissection of tetraploid cotton resistant to Verticillium wilt using interspecific chromosome segment introgression lines[J].The Crop Journal,2014,2:278-288.
[20] 王省芬,马峙英.一种新的棉花黄萎病抗性鉴定方法[J].棉花学报,2002,14(4):231-233.
[21] LI C Q,LIU G S,ZHAO H H,et al.Marker-assisted selection of Verticillium wilt resistance in progeny populations of upland cotton derived from mass selection-mass crossing[J].Euphytica,2013,191:469-480.
[22] 李志坤,张 艳,王省芬,等.棉花抗黄萎病基因的分子标记辅助选择研究[J].河北农业大学学报,2011,34(6):2-4.
[23] 周洪昌,宋 伟,王凤格,等.玉米丝黑穗病分子标记辅助选择育种中前景引物与背景引物的筛选[J].分子植物育种,2011, 9(4):450-456.
[24] INGVARSSON P K,STREET N R.Association genetics of complex traits in plants[J].New Phytol,2011,189:909-922.
[25] ZHANG T Z,HU Y,JIANG W K, et al. Sequencing of allotetraploid cotton(Gossypium hirsutum L. acc. TM-1) provides a resource for fiber improvement[J].Nature Biotechnology,2015,33:531-537.
