基于COS Marker分析柑橘属及其近缘、远缘属植物的遗传与进化
2017-02-24王炯龚桂芝彭祝春李一兵王艳杰洪棋斌
王炯,龚桂芝,彭祝春,李一兵,王艳杰,洪棋斌
(西南大学柑桔研究所/中国农业科学院柑桔研究所/国家柑桔工程技术研究中心,重庆 400712)
基于COS Marker分析柑橘属及其近缘、远缘属植物的遗传与进化
王炯,龚桂芝,彭祝春,李一兵,王艳杰,洪棋斌
(西南大学柑桔研究所/中国农业科学院柑桔研究所/国家柑桔工程技术研究中心,重庆 400712)
【目的】研究柑橘属及其近缘、远缘属种质资源的遗传多样性及亲缘关系,为柑橘类果树的遗传和进化关系提供新的视点,也为遗传育种以及种质资源深入的收集、保护及评价利用提供新工具和参考。【方法】下载Phytozome网站公布的全部克里迈丁Conserved ortholog sequences,并在NCBI中与测序甜橙序列比对,获得具有Gap的序列,从中设计60对COS引物,筛选并利用多态性标记对柑橘属及其近缘、远缘属植物进行扩增检测,通过Structure、PowerMarker和GenAlEx等软件进行数据分析。【结果】从设计引物中筛选出谱带清晰、稳定性好、具有多态性且分布在柑橘不同连锁群的引物25对,对45份柑橘属及其近缘、远缘属种质资源进行扩增检测,共得到584条谱带,平均每对引物约为23.36条;基因多样性为0.26—0.88,平均0.49;PIC为0.26—0.87,平均0.48。群体遗传结构分析显示,在K=9时,群体结构图清晰地揭示了柑橘属及其近、远缘属植物间的遗传组成关系,可区分为宽皮柑橘类群、柚类群、枸橼类群、大翼橙类群、金柑类群、枳及澳沙檬澳圆檬类群、花椒类群、黄皮酒饼簕九里香类群以及印度野橘和莽山野柑杂合类群,类群关系与聚类分析结果总体比较一致。UPGMA聚类分析显示,COS Marker能够明确地区分柑橘属及其近缘、远缘属种质资源,柑橘属在较高分类层次与金柑属、枳属、澳沙檬属、澳圆檬属、花椒属以及黄皮、酒饼簕、九里香等近、远缘植物分离;在柑橘属内,枸橼和大翼橙类首先聚类,然后与宽皮柑橘、柚类等其他柑橘属植物聚类;宽皮柑橘类群里,道县野橘、岑溪酸橘等半野生宽皮柑橘与椪柑等栽培宽皮柑橘可以明确区分,而莽山野柑和印度野橘不在本类群。【结论】根据甜橙和克里迈丁基因测序信息设计的COS引物,在柑橘近缘、远缘属植物中均能获得有效扩增,能够有效区分柑橘属及其近缘、远缘属种质资源;莽山野柑和印度野橘的遗传组成表现杂合,聚类分析中与宽皮柑橘距离比较远,可能并非宽皮橘类群最原始类型。
柑橘;COS Marker;亲缘关系;种质资源;遗传与进化
0 引言
【研究意义】芸香科有约150属900余种植物,进化层次差异大,地域分布范围广,既有栽培价值高、种类丰富、进化程度高的柑橘属,也有种类相对单一的枳属、澳沙檬属等,以及进化层次较低的黄皮、九里香等。由于无融合生殖、属种间易于杂交、无性变异、栽培历史悠久等影响,学术界对柑橘的分类及系统演化关系并未形成被广泛接受的结论。通过开发新标记,开展柑橘属及其近、远缘属种质资源的遗传多样性研究,阐明其亲缘及进化关系,将为柑橘类果树的遗传和进化关系提供新的视点,也为遗传育种以及种质资源深入的收集、保护及评价利用提供新工具和有价值的参考。【前人研究进展】SWINGLE和REECE[1]根据雄蕊的数目和汁胞构造将多蕊橘属、澳沙檬属、澳指檬属、枳属、金柑属以及柑橘属等6属植物归为真正柑橘类果树,并将柑橘属划分为16个种,形成了一个比较有影响力的柑橘分类系统。BARRETT和RHODES[2]采用更多的形态性状进行数值分析,提出SWINGLE分类的柑橘亚属中只有柚、枸橼和宽皮柑橘3个真正生物学种,其余都是杂种起源或后生变异类型。分子标记对研究柑橘及近缘属亲缘关系提供了新的技术和视角。随着分子生物学的快速发展,不同的分子标记技术不断开发出来,并被广泛应用到柑橘及近缘属的分类和种质资源鉴定等研究中。RFLP、RAPD、AFLP[3-4]、SSR[5-6]、ISSR[7]、SRAP[8]等标记在柑橘的分类、种质资源鉴定和遗传多样性分析中得到应用。分子标记在研究柑橘及近缘属亲缘关系中,得到了与BARRETT和RHODES[2]性状数值分类法相一致的结论,如柑橘属由柚类、枸橼和宽皮橘3个种构成,但也提出了许多不一致的论断。例如,当使用不同的分子标记和聚类策略时,金柑属与柑橘属的聚类结果有很大的变化,有支持金柑属独立地位的[4,9],也有认为金柑属从属于柑橘属的[3,10]。导致不一致的原因,既与研究采用材料的代表性和全面性有关,也受到所采用的分子标记本身的影响。RFLP标记为共显性标记,但操作复杂、费用高,难以对众多代表性资源类型获得大量广泛覆盖的标记;AFLP标记为显性标记,有成簇聚集在基因组部分区域的倾向;RAPD理论上能够覆盖全部基因组,但其同样为显性标记,稳定性和产物的检测和解读存在不足;SSR标记比较高效稳定,但在遗传背景差异大时,难以确定其差异的系谱来源的一致性,进而影响对亲缘关系较远的资源类型的相互关系的准确判断。COS Marker 是基于保守直序同源序列Conserved ortholog sequence(COS)开发的一种比较新型的分子标记。COS概念首次由FULTON等[11]提出,通过对拟南芥基因组序列和番茄EST数据库进行计算比对,共鉴别出大约130 000个长度大于 1 000 bp的 EST序列,将其命名为Conserved ortholog sequence(COS),据此设计COS引物并成功应用于茄科植物的图谱比较。【本研究切入点】COS是高度保守且拷贝数低的基因序列,以PCR为基础的COS Marker为亲缘关系快速鉴定、遗传图谱构建和基因组比较等提供了有效的工具,已在不同植物中得到迅速应用,如松属[12-13]、菊属[14]、谷属[15]、烟草属[16]、蔷薇属[17]和芸苔属[18]等。而COS Marker在柑橘中的应用尚未见报道。【拟解决的关键问题】基于公布的柑橘基因组测序结果,开发一套分布于柑橘不同染色体的COS Marker,并将其运用到真正柑橘果树类的 5个近缘属植物和花椒、黄皮等5个远缘属植物的分类和进化关系研究。
1 材料与方法
试验于 2015年在中国农业科学院柑桔研究所进行。
1.1 试验材料
45份柑橘属及其近、远缘属材料(表1)均来源于国家果树种质重庆柑桔圃。

表1 用于COS Marker分析的45份材料Table 1 Forty-five Plant materials for COS Markers analysis
1.2 试验方法
1.2.1 总 DNA的提取与检测 采取试验材料的新鲜幼嫩叶片带回实验室,清水洗净表面,去除叶片主叶脉后称取1.0 g,采用CTAB法提取DNA,参照文献[5]并对其方法略做改动:研钵中加入少量PVPP与试验材料一起研磨;CTAB溶液中加入体积分数为0.2%的β-巯基乙醇;体积比为24∶1的氯仿/异戊醇抽提2次,第1次抽提时加入5 mol·L-1的醋酸钾,冰上放置5 min后10 000 r/min离心10 min;预冷的异丙醇沉淀DNA;1/10体积4 mol·L-1醋酸钠和2倍体积预冷的无水乙醇沉淀DNA。200 µL超纯水溶解DNA,经NanoDrop 2000微量紫外分光光度计检测DNA浓度和 1%琼脂糖凝胶电泳检测谱带完整度后,配制扩增检测的工作模板。
1.2.2 COS引物的开发 从Phytozome v10.0网站主页的Citrus clementina v1.0中收集下载全部已知测序克里迈丁保守直序同源序列。为了提高所设计开发引物的多态性,在 NCBI(National Center for Biotechnology Information)上利用nucleotide blast将全部下载的克里迈丁保守直序同源序列与已知测序甜橙序列进行比对,获得在启动子区或编码区内克里迈丁和甜橙具有一个或多个 Gap的保守直序同源序列275条。按目的序列应满足要求:Gap位置应处于目的序列中;均匀分布在不同染色体上,且平均每条染色体上有6—7条目的序列,运用NCBI网站的Pick Primers来设计所有满足目的序列要求的引物,且所设计开发的引物应尽量保证其具有唯一性,从275条具Gap的序列中挑选并成功设计60对引物。
按照引物类型COS,染色体(Chromosome,C)编号以及在该染色体上的设计的引物序号进行引物命名。1.2.3 COS引物的筛选及产物的检测 开发的60对COS引物均委托英潍捷基(上海)贸易有限公司进行合成。应用琯溪蜜柚、8号椪柑、梨橙2号、柠檬、金豆、飞龙枳、九里香、酒饼簕等8种代表性材料进行COS引物筛选,从中筛选出谱带清晰、稳定性好、具有多态性且分布在不同连锁群的25对COS引物。PCR反应体系为优化的 15 µL反应体系:1×PCR Buffer,1.5 mmol·L-1的Mg2+,0.2 mmol·L-1的dNTPs, 0.33 nmol·L-1的上、下游引物,1 U的Taq DNA聚合酶,DNA模板约75 ng。
PCR扩增反应在Biometra PCR仪上进行。扩增程序: 94℃预变性4 min;94℃变性45 s,64℃退火40 s,72℃延伸45 s,每次循环降低0.5℃,10个循环;94℃变性45 s,58℃退火40 s,72℃延伸 40 s,22个循环;72℃延伸5 min;最后PCR产物4℃保存。
取PCR扩增产物2 µL上样,8%非变性聚丙烯酰胺凝胶、恒定电压140 V,电泳约2 h(根据目的片段大小,电泳时间长短略有调整),银染检测并照相。1.2.4 数据处理 根据PCR扩增后电泳谱带的有无及清晰程度,按照二倍体基因型格式读取谱带,之后根据相关分析软件的要求作相应格式的转换。
应用Structure软件基于数学模型进行群体结构分析。设置MCMC(Markov Chain Monte Carlo)开始时内循环burn-in迭代为10 000次,再将burn-in迭代后的MCMC设为20 000次,亚群数量(K)设置为2—10,每个K值运行10次,运行完毕后在每个K值中取Ln P(D)最大和最小两个数值,分别绘制10个最大值和10个最小值的折线图,找出拐点,并参考系统聚类分析结果来判定合适的亚群数,选出合适的K值。当无明显拐点时,参照并修改 EVANNO等[19]、强海平等[20]、刘志斋等[21]和GARCIA等[22]的△K算法选取合适的K值。
采用PowerMarker软件进行聚类分析,计算等位基因(Allele)、基因多样性(Gene Diversity)、多态信息含量(polymorphism information content,PIC)。采用Nei’ 1983方法计算遗传距离、UPGMA法聚类,并对结果500次bootstrap重复抽样,利用MEGA绘制遗传聚类图。
运用GenAlEx软件进行主坐标轴分析。
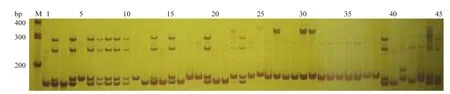
图1 引物COSC1-12的COS Marker扩增结果Fig. 1 The COS Marker amplified profile using the primer COSC1-12
2 结果
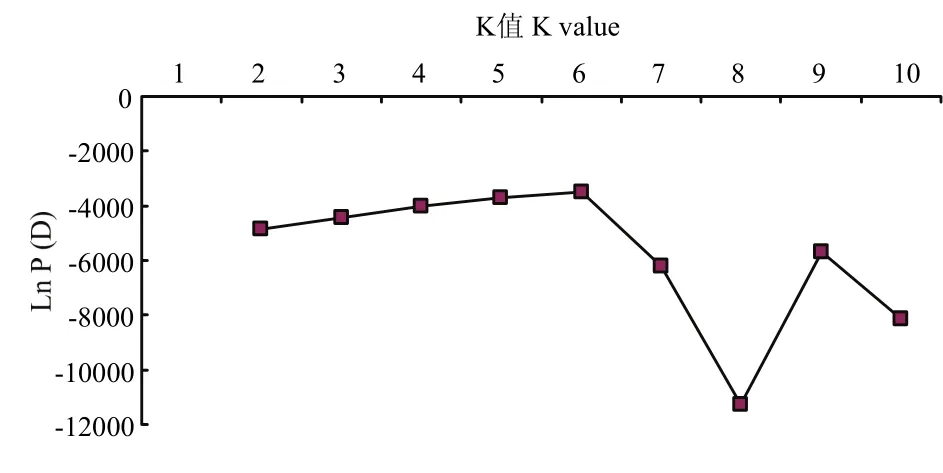
图2 不同K值对应的Ln P(D)Fig. 2 Ln P(D) of different K values
2.1 COS遗传多样性及COS 引物的分析鉴定效果
用筛选的引物对45份试验材料进行扩增,均能扩增出稳定的谱带,范围100—700 bp。25对引物获得72个标记位点,扩增出584条谱带,平均每对引物约为23.36条。扩增谱带最多的引物为COSC4-3,46条,COSC3-8扩增谱带最少,10条。25对COS引物的平均 PIC为 0.48,变化范围 0.26(COSC3-9)—0.87(COSC3-11),其中有11个COS引物的PIC≥0.50(表2)。针对所研究的45份试验材料,其平均基因多样性为0.49,变化范围处于0.26(COSC3-9)—0.88(COSC3-11),其中COSC3-11的PIC和基因多样性值最高,分别为0.87和0.88,COSC3-9的PIC和基因多样性值则最低,均为0.26。表2为25对COS引物的遗传多样性统计结果。
根据电泳谱带结果分析,COSC1-12引物能够将澳沙檬、澳圆檬区分开(图1);COSC1-12和COSC3-7引物结合能够将所有大翼橙类有效分开;COSC2-8引物能够有效区分黄皮、酒饼簕、九里香等远缘属;COSC2-8和COSC3-7引物结合能够将5种金柑属材料分开;COSC4-8引物能够将枳属两种材料区分开;COSC4-9引物能够将3种柚类区分开;COSC9-8引物能够将花椒属的两个种类分离开。由于宽皮橘材料较多,一两对引物不能有效区分,需结合多对引物才能达到满意的分类效果。从聚类分析结果看,所筛选的引物能够有效区分柑橘属及近远缘属。
2.2 群体遗传结构分析
将数据按照 Structure软件数据处理要求进行处理,找出拐点对应的K值,发现在K=6、8、9时明显出现拐点,参考UPMGA聚类分析结果,当K=9时,所有材料分为9个类群是较为合适的。
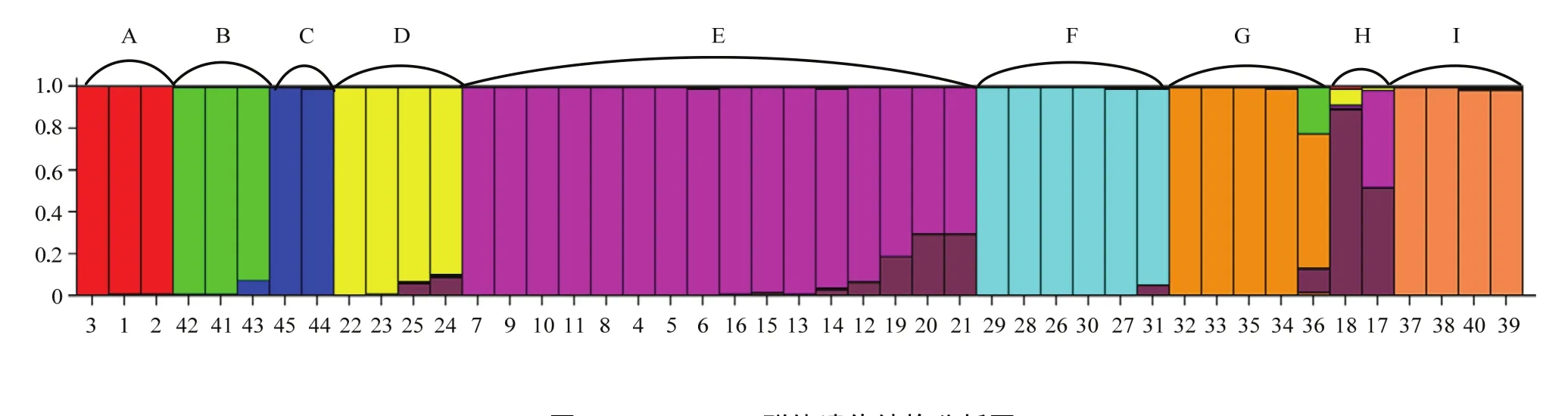
图3 Structure群体遗传结构分析图Fig. 3 The population structure map of 45 materials
应用Structure软件得到的群体遗传结构图(图3)中,将45份材料分为9个类群,分别标记为A、B、C、D、E、F、G、H、I,整体符合目前柑橘属及其近、远缘属的分类。晚白柚(1)、琯溪蜜柚(2)、岭南沙田柚(3)构成柚类一族(A),其遗传组成单一;黄皮(41)、酒饼簕(42)、九里香(43)构成远缘类群(B),黄皮和酒饼簕遗传组成单一,而九里香与花椒属(44、45)共有少量基因,显示出它们之间可能有一定的亲缘关系;花椒(44)和野花椒(45)构成外类群(C),遗传组成单一;野生香橼(22)、合江佛手(23)、柠檬(24)、塔希提莱檬(25)作为枸橼类组成一类(D),其中野生香橼和合江佛手的遗传组成无杂合现象,而柠檬和来檬则显示有外缘基因渗入,与其明显的杂种来源一致[23-24];安江红橘等宽皮柑橘和塔罗科血橙(20)、梨橙2号(21)等16个柑橘品种组成最多的一类(E),除甜橙、温州蜜柑、印度酸橘、岑溪酸橘等明显的杂种外,其余宽皮柑橘的遗传组成未表现杂合现象;2个甜橙均显示约3/4的宽皮橘基因以及1/4的其他基因来源,这与甜橙测序的分析结果比较一致[25],但关于另1/4的其他基因来源则有不同;红河大翼橙(26)、元江宜昌橙08-7(27)、
元江宜昌橙08-9(28)、金刀峡宜昌橙(29)、马蜂柑(30)、马来西亚大翼橙(31)构成另一个柑橘属类群(F),除马来西亚大翼橙外,其余大翼橙的遗传组成无杂合现象;金豆(32)、罗纹(33)、金弹(34)、罗浮(35)、四季橘(36)作为金柑属组成一类(G),除四季橘显示出有外缘基因渗入外,其余材料遗传组成无杂合现象;莽山野柑(17)和印度野橘(18)单独成为一类(H),与其他宽皮橘有较大差异,遗传来源较为独特,但二者的遗传组成表现出杂合现象,印度野橘有较少量的枸橼血缘,莽山野柑有较多的宽皮橘血缘,显示二者可能有比较独特的遗传来源,但并非该类群最原始类型;近缘属中的旺苍大叶枳(37)、飞龙枳(38)与澳沙檬(39)、澳圆檬(40)组成一个类群(I),除澳沙檬、澳圆檬显示极少的外缘基因外,遗传组成也未表现杂合现象。
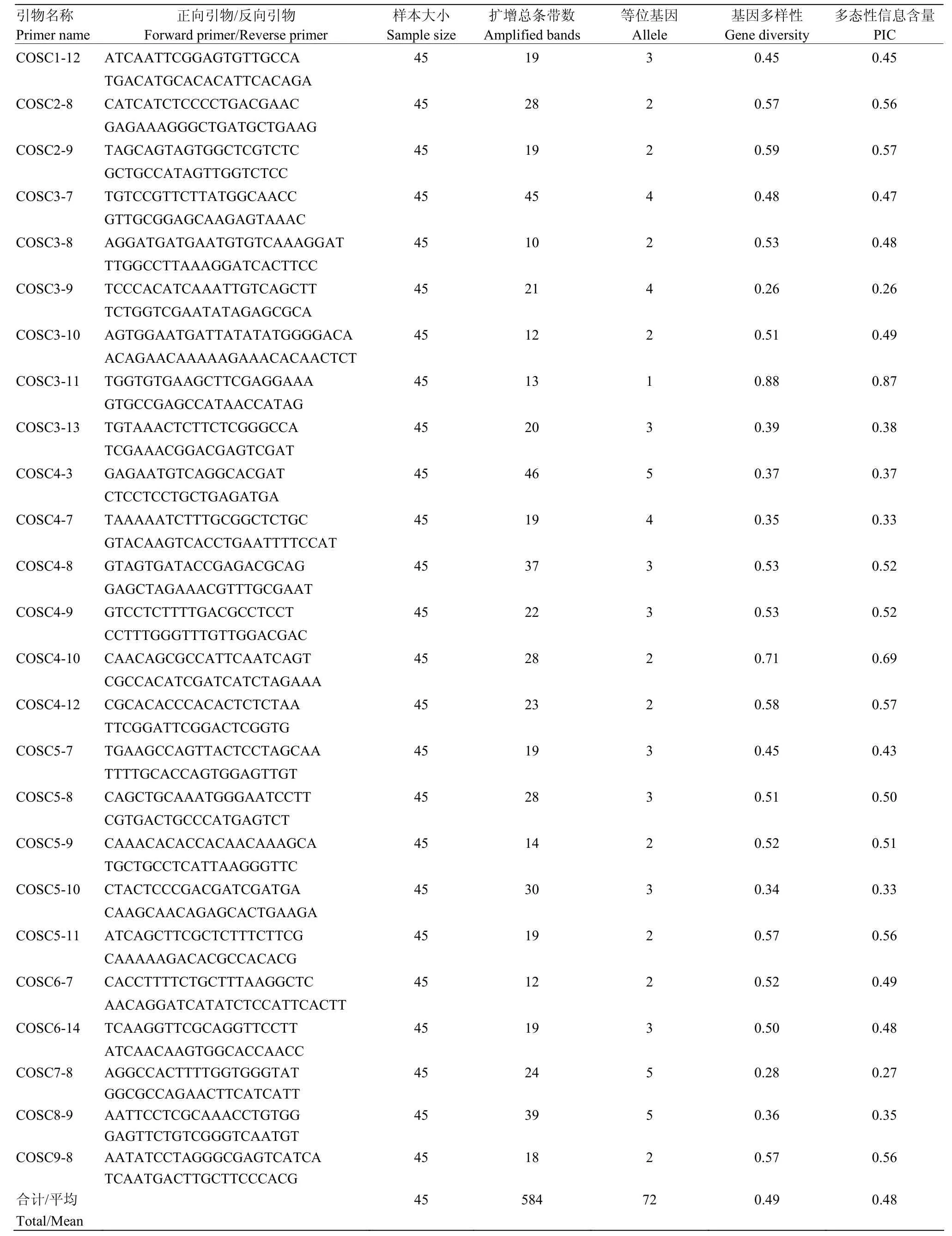
表2 COS引物及其揭示的遗传多样性统计Table 2 Summary of COS Markers and genetic diversity revealed
2.3 聚类分析
运用PowerMarker软件对45份试验材料聚类分析,构建UPGMA聚类树(图4)。第一条分割线将材料分成A、B两个类群:A类群为近远缘属植物;B类群为柑橘属植物。第二条分割线将柑橘属及其近远缘属进一步划分为C、D、E、F、G、H六个类群:C类群为外缘属植物,包括黄皮(41)、酒饼簕(42)、九里香(43)、花椒(44)、野花椒(45);四季橘(36)与其他金柑属植物聚成D类群,但它与其他金柑属植物遗传距离较远,Structure群体遗传结构分析显示四季橘有金柑属血缘并占主导地位,说明四季橘与金柑属植物亲缘关系较近;E类群为枳属(37、38)和澳沙檬(39)、澳圆檬(40)构成的近缘属类群;枸橼类植物和大翼橙类植物构成F类群;G类群包括兴津温州蜜柑(12)、两个甜橙(20、21);H类群为柚类、宽皮柑橘类植物类群。第三条分割线将H类群进一步划分为H1、H2两个类群:莽山野柑(17)、印度野橘(18)、柚类(1、2、3)构成H1类群;其余宽皮橘构成H2类群,在H2类群中,又可细分为以安江红橘(4)、万州少核红橘(5)、砂糖橘(9)、土橘(6)等为代表的栽培种和以旺苍皱皮柑(13)、细皮狗屎柑(14)等为代表的野生、半野生宽皮橘两类。聚类分析结果显示,COS Marker能够明确的区分柑橘属及其近、远缘属种质资源,并对成员众多、关系复杂的宽皮类群进行有效区分。
2.4 群体主坐标轴分析
采用GenAlEx软件基于遗传距离数据对45份材料进行的PCoA分析结果见图5。可以看到在第一主坐标轴区,显示绿色方形部分的宽皮柑橘和蓝色三角形部分甜橙类塔罗科血橙(20)、梨橙2号(21)关系较近;在第二主坐标轴区,柚类和莽山野柑(17)、印度野橘(18)关系较近,与聚类分析结果一致;第三主坐标轴区的紫色菱形部分的大翼橙类和黄色圆形部分的枸橼类关系较近,聚类分析也显示两者有较近的亲缘关系;第四主坐标轴区的浅蓝色部分的金柑属聚在一起,四季橘(36)又远离于其他金柑属,这与 Structure群体遗传结构分析结果、聚类分析结果一致;近远缘属的枳属(37—38)、澳沙檬(39)、澳圆檬(40)、黄皮(41)、酒饼簕(42)、九里香、(43)花椒属(44—45)等与柑橘属距离较远,与聚类分析结果一致。
3 讨论
3.1 柑橘及其近、远缘属植物的分离分化时间
一个比较明确的时间点对认知植物的分类与进化关系非常有价值。地质事件和化石提供了比较准确的分离分化时间估计[26]。在柑橘属及其近、远缘属植物起源和分布的“桶形”区域内[1],发生了古大陆的分离和漂移以及印度板块与亚欧板块碰撞而带来喜马拉雅运动等重大地质事件,对柑橘属及其近、远缘属植物的分离分化和分布产生了重要影响,也为研究其分离分化提供了比较客观的参考时间点。
本研究所采用的材料,按SWINGLE和REECE[1]的分类,包括了亲缘关系很近的5个真正柑橘果树类植物,也包括了关系较近、与真正柑橘类位于相同亚族(subtribe)的酒饼簕,还有与真正柑橘类位于相同亚科但不同族的黄皮和九里香,以及与真正柑橘类不同亚科的花椒和野花椒。这些材料中,采样分析的远缘属材料均来自中国,但花椒属在世界范围内广泛分布,九里香属、黄皮属和酒饼簕局限在中国、东南亚、澳大利亚等地,分布区域与真正柑橘类非常一致;5个近缘属中,中国是柑橘属的主要起源地,是金柑属和枳属的唯一起源地,而澳大利亚和新几内亚地区是澳指檬、澳沙檬和多蕊橘3个属的唯一起源地。由于古大陆的分离和漂移而造成的广阔海洋分隔,澳洲 3属与中国的3属以及九里香等远缘植物间难以完成遗传物质的交流,如果它们是同步由共同祖先分离分化并独立演进形成,反映到分支或聚类关系上,6个柑橘近缘属植物应该首先属内聚类,然后澳洲3属与中国起源3属各自聚类,再在近缘属聚类后与其他远缘属植物聚类。然而在本研究中各近、远缘属却展示了不同的遗传一致性和聚类层次。

图4 基于COS Marker数据构建的UPGMA聚类树(500次bootstrap检验)Fig. 4 An UPGMA tree of 45 different materials derived from bootstrap analysis (500 replications) of COS Marker data
采用COS Marker能够将柑橘属及其近、远缘属植物进行有效区分,但遗传结构分析显示枳属和澳指檬属与澳沙檬属有非常一致的遗传组成,黄皮、酒饼簕和九里香也显示高度一致的遗传组成;UPGMA聚类分析则显示枳属和澳指檬属与澳沙檬属在属一级的分类中关系更近,它们先聚类后再与柑橘属、金柑属及其他属构成较高级分类单元,花椒、九里香、酒饼簕和黄皮等远缘植物彼此在分类上更接近。FEDERICI等[27]也得到类似结果,在其应用RFLP数据进行非杂种来源的材料聚类分析中,显示枳属与多蕊橘属和澳指檬属的M. warburgiana具有较近关系,它们先聚类后再与澳沙檬及澳指檬类型聚类;在应用RFLP数据进行所有材料聚类分析中,显示枳属与澳沙檬及多蕊橘属和澳指檬属的M. warburgiana聚类后,再与其他澳指檬类型聚类。
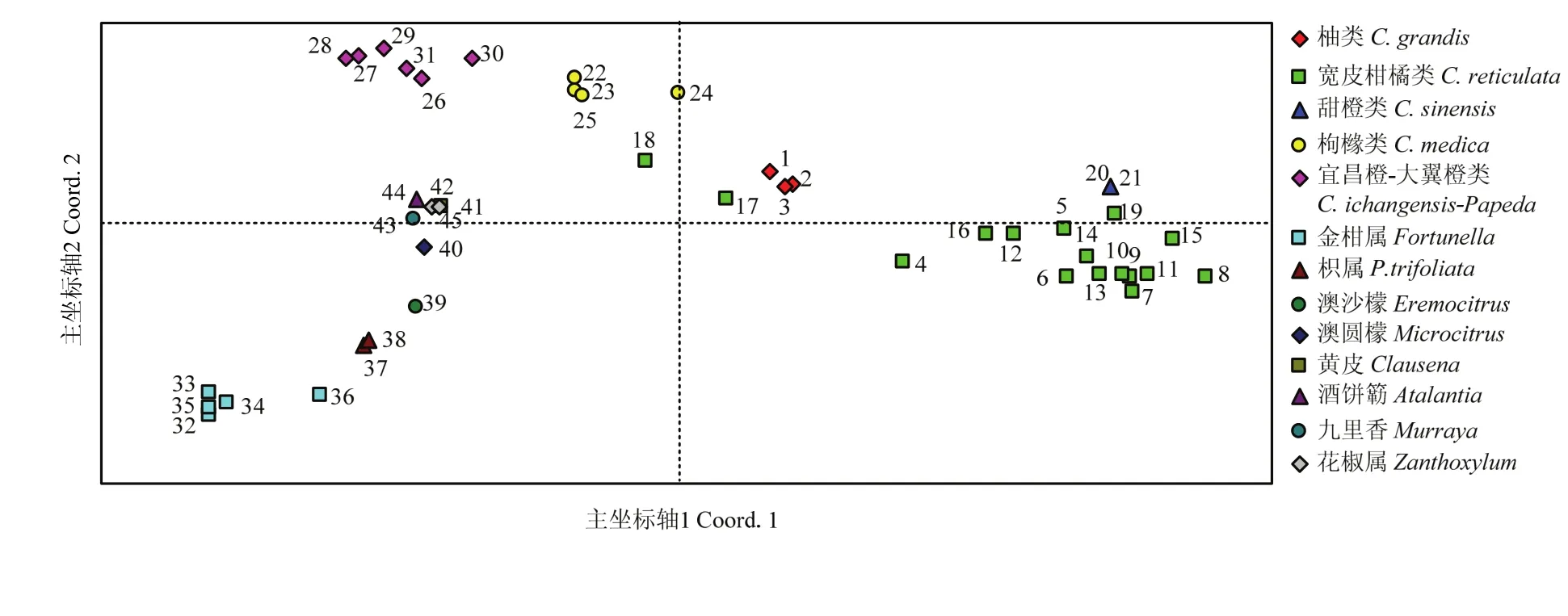
图5 基于COS Marker数据的主坐标轴分析图Fig. 5 PCoA plot constructed with COS Marker data
枳属与3个澳洲近缘属有更近似的遗传组成和亲缘关系,超越与其有长期共同地域分布的柑橘属和金柑属的关系,一个重要的分类学意义就是表明枳属与澳洲3近缘属分离分化的时间应该可能晚于它们与柑橘属和金柑属的分离分化。真正柑橘类的共同祖先可能是先分化形成柑橘属原始类型、金柑属原始类型以及枳属与澳洲3近缘属的共同原始类型,然后在长期的生殖隔离作用下,枳属与澳洲3近缘属的共同原始类型分别演化,形成了形态和适应生境差异甚大的枳、澳沙檬和多蕊橘等类型。真正柑橘类的最近共同祖先在分化形成不同分支类型时,理论上应该仍处于未完全割裂的陆地环境,花粉、种子或果实扩散能够顺利完成,对应的大地质背景应该是中国所在的欧亚古陆与澳大利亚所在的冈瓦纳古陆尚未彻底分离。从古植物学方面看,在欧洲早始新世到晚上新世的第三纪地层中就有大量花椒属种子化石被发现,在北美早中新世也发现有该属种子化石,在中国山东临朐中新世地层曾报道有花椒属的叶片化石。在广东和广西第四纪地层中找到黄檗属及酒饼簕属的叶片化石[28]。SWINGLE和REECE[1]曾认为有充足的理由推测,澳沙檬是由澳指檬经过2千万至3千万年的漫长干旱适应演化形成,结合澳洲大陆分离的重大地质事件和被子植物出现的时期,认为真正柑橘类植物的产生应该在6 500万年前的晚白垩纪。
枳属与澳洲3近缘属的相似遗传组成和相近亲缘关系,以及地理分布特点和区域内的地质事件为估计真正柑橘类植物的分离分化提供了参考,而印度野橘和莽山野柑则为柑橘属内的分离分化提供了依据。印度野橘和莽山野柑是两个地域来源明确、分布区域有限、遗传特性独特的野生原始柑橘种。前者分布在印度东北的阿萨姆邦区域,被认为是栽培柑橘的原始类型[29-30];而后者则分布在中国中部的湖南莽山区域,因其性状的原始,也被认为是宽皮柑橘的原始类型[31]。本研究中COS Marker能够将二者进行有效区分,但遗传结构分析显示二者有较高的相似性,且相似成分非常独特,该成分仅在甜橙、柠檬、来檬等明显杂种来源的其他类型中低比例出现,UPGMA聚类分析也显示印度野橘和莽山野柑亲缘关系比较近,超过与其他宽皮柑橘或枸橼类的相近程度。分布区域遥远且其间横亘了阻断普通生物交流的喜马拉雅山的两个野生柑橘种,有近似的遗传组成和亲缘关系,表明其在起源上可能有独特的关系。从地理上可知,印度东北的阿萨姆邦等区域实际上位于喜马拉雅山南麓,本身是中国所在欧亚板块的一部分,而非印度主体所在的印度板块的组成部分,只是由于印度板块向欧亚板块的伏冲挤压,强烈的“喜马拉雅造山运动”,才被喜马拉雅山分隔开来。印度野橘和莽山野柑可能是在其共同祖先的栖息地被“喜马拉雅造山运动”分隔后,经过第四纪冰川,才在各自现今的区域独自演化为迄今的类型。“喜马拉雅造山运动”达到使普通生物交流困难的时段应该可以作为印度野橘和莽山野柑的分离分化参考时间点,可能在2千万年至3千万年前。
3.2 莽山野柑、印度野橘与柑橘的起源
处于相对隔绝的地域,未受或少受人工选择或栽培柑橘的影响,野生柑橘对柑橘及近缘属的起源和进化研究具有重要作用。莽山野柑和印度野橘是目前尚存但濒危的野生柑橘,被认为是栽培柑橘的原始类型[29-31]。廖振坤等[31]根据汁囊结构、花粉壁的外壁雕纹特征比较及AFLP聚类结果显示,莽山野柑是比道县野橘更原始的类型,并且认为南岭山脉存在莽山野柑→道县野橘→栽培种的宽皮橘的演化路径。JOSE等[32]研究发现宽皮橘多达38个左右的indels,而莽山野柑仅具有4个indels,并且莽山野柑与宜昌橙之间仅有10%的异质一致性,认为莽山野柑与宽皮橘、宜昌橙亲缘关系较远,但其聚类分析显示莽山野柑与宜昌橙有较近的亲缘关系,王福生等[33]也得到两者具有较近的亲缘关系的结果。而刘勇等[34]的SSR-AFLP聚类分析显示莽山野柑与宜昌橙和大翼橙亲缘关系较远。MALIK[29]、LASKAR[35]等认为印度野橘是最原始的柑橘类型,是柑橘栽培种的祖先之一。刘通等[8]的研究涉及莽山野柑与印度野橘,通过SSR-SRAP聚类分析显示,在遗传相似系数为0.725处,它们和柚类聚为一个分支,表明三者有较近的亲缘关系。而FEDERICI[27]认为印度野橘可能是卡西大翼橙或酸橙的杂种,但其RFLP和RAPD标记则显示印度野橘优先与枸橼聚类,NICOLOSI等[36]通过cpDNA聚类分析也显示印度野橘和枸橼优先聚为一类。
本研究中的遗传结构分析显示莽山野柑和印度野橘有较为独特的相似遗传组成,但与其他宽皮橘存在较大差异。莽山野柑有近半的宽皮柑橘遗传组成,且其遗传组成表现杂合现象。而印度野橘则含有较少量的枸橼血缘,这与FEDERICI等[27]、NICOLOSI等[36]结果类似。UPGMA聚类分析也显示印度野橘与莽山野柑优先聚类而远离其他宽皮橘和枸橼等类型,与刘通等[8]SSR-SRAP聚类结果一致。因此笔者认为莽山野柑和印度野橘是比较独特原始的柑橘类型,但不应该是宽皮柑橘的直接祖先。
4 结论
根据甜橙和克里迈丁基因测序信息设计的 COS引物,在柑橘近缘、远缘属植物中均能获得有效扩增,能够有效区分柑橘属及其近、远缘属种质资源。莽山野柑和印度野橘有独特的遗传组成,应该是比较独特的柑橘类型,其遗传组成表现杂合现象,聚类分析中与宽皮柑橘距离比较远,应该不是宽皮柑橘的直接祖先。
[1] SWINGLE W T, REECE P C. The botany of citrus and its wild relatives//REUTHER W, WEBBER H J, BATCHELOR L D. The Citrus Industry. Vol. 1. 2nd ed. Berkley: University of California Press. 1967: 190-422.
[2] BARRETT H C, RHODES A M. A numerical taxonomic study of affinity relationships in cultivated citrus and its close relatives. Systematic Botany, 1976, 1: 105-136.
[3] 谢让金, 周志钦, 邓烈. 真正柑橘果树类植物基于 AFLP 分子标记的分类与进化研究. 植物分类学报, 2008, 46(5): 682-691. XIE R J, ZHOU Z Q, DENG L. Taxonomic and phylogenetic relationships among the genera of the true citrus fruit trees group (Aurantioideae, Rutaceae) based on AFLP markers. Journal of Systematics and Evolution (formerly Acta Phytotaxonomica Sinica), 2008, 46(5): 682-691. (in Chinese)
[4] PANG X M, HU C G, DEND X X. Phylogenetic relationships within citrus and its related genera as inferred from AFLP markers. Genetic Resources and Crop Evolution, 2007, 54: 429-436.
[5] 龚桂芝, 洪棋斌, 彭祝春, 江东, 向素琼. 枳属种质遗传多样性及其近缘属植物亲缘关系的 SSR和 cpSSR分析. 园艺学报, 2008, 38(12): 1742-1750. GONG G Z, HONG Q B, PENG Z C, JIANG D, XIANG S Q. Genetic diversity of poncirus and its phylogenetic relationships with relatives as revealed by nuclear and chloroplast SSR. Acta Horticulturae Sinica, 2008, 38(12): 1742-1750. (in Chinese)
[6] 雷天刚, 何永睿, 彭爱红, 许兰珍, 邹修平, 姚利晓, 李金强, 陈善春. 贵州部分柑橘地方品种遗传背景的 SSR 分析. 西南大学学报(自然科学版), 2015, 37(11): 31-35. LEI T G, HE Y R, PENG A H, XU L Z, ZOU X P, YAO L X, LI J Q, CHEN S C. Genetic background analysis of several Guizhou local citrus varieties based on SSR markers. Journal of Southwest University (Natural Science Edition), 2015, 37(11): 31-35. (in Chinese)
[7] 廖芳蕾, 陈民管, 桑丹, 陈文荣, 郭卫东. 佛手种质资源遗传多样性的ISSR分析. 园艺学报, 2013, 40(11): 2222-2228. LIAO F L, CHEN M G, SANG D, CHEN W R, GUO W D. Genetic diversity analysis of fingered citron germplasm by ISSR. Acta Horticulturae Sinica, 2013, 40(11): 2222-2228. (in Chinese)
[8] 刘通, 邓崇岭, 程玉芳, 李秋景, 陈传武, 刘冰浩, 伊华林. 利用SSR和 SRAP技术分析广西柑橘种质遗传多样性. 华中农业大学学报, 2016, 35(2): 23-29. LIU T, DENG C L, CHENG Y F, LI Q J, CHEN C W, LIU B H, YI H L. Analyzing genetic diversity of citrus germplasm in Guangxi province with SSR and SRAP markers. Journal of Huazhong Agricultural University, 2016, 35(2): 23-29. (in Chinese)
[9] PENJOR T, ANAI T, NAGANO Y, MATSUMOTO R, YAMAMOTO M. Phylogenetic relationships of citrus and its relatives based on rbcL gene sequences. Tree Genet Genomes, 2010, 6: 931-939.
[10] BARKLEY N A, ROOSE M L, KRUEGER R R, FEDRICI C T. Assessing genetic diversity and population structure in a citrus germplasm collection utilizing simple sequence repeat markers (SSRs). Theoretical and Applied Genetics, 2006, 112: 1519-1531.
[11] FULTON T M, HOEVENR V, EANNETTAN T, TANKSLEY S D. Identifiation, analysis, and utilization of conserved ortholog set markers for comparative genomics in higher plants. Plant Cell, 2002, 14: 1457-1467.
[12] KRUTOVSKY K V, TROGGIO M, BROWN G R, JERMSTAD K D, NEALE D B. Comparative mapping in the Pinaceae. Genetics, 2004, 168(1): 447-461.
[13] LIEWLAKSANEEYANAWIN C, ZHUNG J, TANG M, FARZANEH N, LUENG G, CULLIS C, FINDLAY S, RITLAND C E, BOHLMANN J, RITLAND K. Identification of COS markers in the Pinaceae. Tree Genetics & Genomes, 2008, 5(1): 247-255.
[14] CHAPMAN M A, CHANG J, WEISMAN D, KESSELIR V, BURKE J M. Universal markers for comparative mapping and phylogenetic analysis in the Asteraceae (Compositae). Theoretical and Applied Genetics, 2007, 115: 747-755.
[15] QURAISHI U M, ABROUK M, BOLOT S, PONT C, THROUDE M, GUILHOT N, CONFOLENT C, BORTOLINI F, PRAUD S, MURIGEUX A, CHARMET G, SALSE J. Genomics in cereals:from genome-wide conserved orthologous set (COS) sequences to candidate genes for trait dissection. Functional & Integrative Genomics, 2009, 9: 473-484.
[16] WU F N, NANCY T E, XU Y M, PLIESKE J, GANAL M, POZZI C, BAKAHER N, TANKSLEY S D. COSII genetic maps of two diploid nicotiana species provide a detailed picture of synteny with tomato and insights into chromosome evolution in tetraploid N. tabacum. Theoretical and Applied Genetics, 2010, 120: 809-827.
[17] BUSHAKRA J M, SARGENT D J. CABRERA A, CROWHURST R, GIRONA E L, VELASCO R, SYMONDS V V, KNAAP E, TROGGIO M, GARDINER S E. CHAGNE D. Rosaceae conserved orthologous set (RosCOS) markers as a tool to assess genome synteny between Malus and Fragaria. Tree Genetics & Genomes, 2011, 8: 643-658.
[18] JEONG Y M, CHUNG W H, CHUNG H, NAMSHIN K, PARK B S, LIM K B, YU H J, MUN J H. Comparative analysis of the radish genome based on a conserved ortholog set (COS) of Brassica. Theoretical and Applied Genetics, 2014, 127: 1975-1989.
[19] EVANNO G, REGNAUT S, GOUDET J. Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study. Molecular Ecology, 2005, 14(8): 2611-2620.
[20] 强海平, 余国辉, 刘海泉, 高洪文, 刘贵波, 赵海明, 王赞. 基于SSR标记的中美紫花苜蓿品种遗传多样性研究. 中国农业科学, 2014, 47(14): 2853-2862. QIANG H P, YU G H, LIU H Q, GAO H W, LIU G B, ZHAO H M, WANG Z. Genetic diversity and population structure of Chinese and American Alfalfa (Medicago Sativa. L) germplasm assessed by SSR markers. Scientia Agricultura Sinica, 2014, 47(14): 2853-2862.(in Chinese)
[21] 刘志斋, 吴迅, 刘海利, 李永祥, 李清超, 王凤格, 石云素, 宋燕春,宋伟彬, 赵久然, 赖锦盛, 黎裕,王天宇. 基于40个核心SSR标记揭示的 820份中国玉米重要自交系的遗传多样性与群体结构. 中国农业科学, 2012, 45(11): 2107-2138. LIU Z Z, WU X, LIU H L, LI Y X, LI Q C, WANG F G, SHI Y S, SONG Y C, SONG W B, ZHAO J R, LAI J S, LI Y, WANG T Y. Genetic diversity and population structure of important Chinese maize inbred lines revealed by 40 core simple sequence repeats (SSRs). Scientia Agricultura Sinica, 2012, 45(11): 2107-2138. (in Chinese)
[22] GARCIA A L, LURO F, OLLITRAULT P, NAVARRO L. Genetic diversity and population structure analysis of mandarin germplasm by nuclear, chloroplastic and mitochondrial markers. Tree Genetics & Genomes, 2015, 11: 123-137.
[23] 梁国鲁. 部分柑桔属及其近缘属Giemsa C-带带型研究. 遗传学报, 1988, 15(6): 409-415. LIANG G L. Study on the Giemsa C-banding Patterns of Some Citrus and Its Related Genera. Acta Genetica Sinica, 1988, 15(6): 409-415. (in Chinese)
[24] 熊光明. 应用 AFLP分子标记对柑橘属(Cirtus)植物进行鉴别与系统分类研究[D]. 重庆: 西南农业大学, 2002. XIONG G M. Study on citrus Identification and Taxonomy by using AFLP markers [D]. Chongqing: Southwest Agricultural University, 2002. (in Chinese)
[25] XU Q, CHEN L L, RUAN X A, CHEN D J, ZHU A D, CHEN C L, BERTRAND D, JIAO W B, HAO B H, MATTHEW P L, CHEN J J, GAO S, XING F, LAN H, CHANG J W, GE X H, LEI Y, HU Q,MIAO Y, WANG L, XIAO S X, BISWAS M K, ZENG W F, GUO F, CAO H B, YANG X M, XU X W, CHENG Y J, XU J, LIU J H, OSCAR, LUO J H, TANG Z H, GUO W W, KUANG H H, ZHANG H Y, MIKEAL L R, NIRANJAN N, DENG X X, RUAN Y J. The draft genome of sweet orange (Citrus sinensis). Nature Genetics, 2013, 45: 59-66.
[26] DONOGHUE P C J, BENTON M J. Rocks and clocks: Calibrating the tree of life using fossils and molecules. Trends in Ecology & Evolution, 2007, 22: 424-431 .
[27] FEDERICI C T, FANG D Q, SCORA R W, ROOSE M L. Phylogenetic relationships within the genus Citrus (Rutaceae)and related genera as revealed by RFLP and RAPD analysis. Theoretical and Applied Genetics, 1998, 96: 812-822.
[28] 吴征镒, 陈心启. 中国植物志. 北京: 科学出版社, 1997, 43(2): 1. WU Z Y, CHEN X Q. Flora Republicae Popularis Sinicae. Beijing: Science Press, 1997, 43(2): 1. (in Chinese)
[29] MALIK S K, CHAUDHURY R, DHARIWAL O P, RAJWANT K K. Collection and characterization of Citrus indica Tanaka and C. macroptera Montr: wild endangered species of northeastern India. Genetic Resources and Crop Evolution, 2006, 53: 1485-1493.
[30] TANAKA T. Citrologia semi centennial commemoration papers on Citrus studies. Osaka: Citrologia Supporting Foundation, 1961: 114.
[31] 廖振坤, 张秋明, 刘卫国, 丁伟平, 张玲. 南岭山脉宽皮柑橘近缘野生种亲缘关系鉴定. 湖南农业大学学报(自然科学版), 2006, 32(4): 385-388. LIAO Z K, ZHANG Q M, LIU W G, DING W P, ZHANG L. Identification of relative relationships of wild relatives of eucitrus originated from Nanling mountains by AFLP analysis. Journal of Hunan Agricultural University (Natural Sciences Edition), 2006, 32(4): 385-388. (in Chinese)
[32] JOSE C C, ROBERTO A, VICTORIA I, JAVIER T, MANUEL T, JOAQUIN D. A phylogenetic analysis of 34 chloroplast genomes elucidates the relationships between wild and domestic species within the genus citrus. Molecular Biology and Evolution, 2015, 32(8): 2015-2035.
[33] 王福生, 江东. 应用cpSSR和EST-SSR标记进行柑橘特异种质资源遗传背景研究. 园艺学报, 2010, 37(3): 465-474. WANG F S, JIANG D. Studies on genetic background of important germplasm resources among citrus based on cpSSR and ESTSSR marker. Acta Horticulturae Sinica, 2010, 37(3): 465-474. (in Chinese)
[34] 刘勇, 孙中海, 刘德春, 吴波, 陶建军. 柚类种质资源 AFLP与SSR遗传多样性分析. 中国农业科学, 2005, 38(11): 2308-2315. LIU Y, SUN Z H, LIU D C, WU B, TAO J J. Assessment of the genetic diversity of pummelo germplasms using AFLP and SSR markers. Scientia Agricultura Sinica, 2005, 38(11): 2308-2315. (in Chinese)
[35] LASKAR M A, HYNNIEWTA M, RAO C S. In vitro propagation of citrus indica Tanaka—An endangered progenitor species. Indian Journal of Biotechnology, 2009, 8: 311-316.
[36] NICOLOSI E, DENG Z N, GENTILE A, MALFA S L, CONTINELLA G, TRIBULATO E. Citrus phylogeny and genetic origin of important species as investigated by molecular markers. Theoretical and Applied Genetics, 2000, 100: 1155-1166.
(责任编辑 赵伶俐)
Genetic and Phylogenetic Relationships Among Citrus and Its Close and Distant Relatives Based on COS Marker
WANG Jiong, GONG GuiZhi, PENG ZhuChun, LI YiBing, WANG YanJie, HONG QiBin
(Citrus Research Institute, Southwest University/Chinese Academy of Agricultural Sciences, National Citrus Engineering Research Center, Chongqing 400712)
【Objective】To reveal genetic diversity and phylogenetic relationships of Citrus and its close and distant relatives with newly developed conserved ortholog sequence (COS) Marker.【Method】According to the published C. sinensis and C. clementina genomic sequencing information, COS Markers were developed and screened. Polymorphic markers were used to amplify and detect Citrus and its close and distant relatives. Structure, Power Marker and GenAlEx were used to analyze the relevantdata.【Result】A total of 60 COS primers were designed, and 25 primer pairs distributed in different linkage groups were selected based on clear and good DNA polymorphic bands. In 45 samples from Citrus and its relatives, 25 COS markers produced 584 bands, with an average of about 23.36 bands per marker, and genetic diversity ranged from 0.26 to 0.88, with an average of 0.49; PIC value was between 0.26 and 0.87, with an average of 0.48. When K = 9, a clear genetic structure relationship of citrus and its relatives were revealed. They were divided into C. reticulata group, C. grandis group, C. medica group, Papeda group, Fortunella group, P. trifoliate and Eremocitrus and M. australis group, Z. bungeanum group, Clausena lansium and Atalantia buxifolia and Murraya paniculata group as well as C. indica and C. mangshanensis hybrid groups. This result was globally consistent with that in the UPGMA clustering analysis. UPGMA clustering analysis revealed that COS Marker could effectively distinguish Citrus from its close and distant germplasm resources. At higher taxonomy level, Citrus was first separated from P. trifoliate, Eremocitrus, M. australis, Z. bungeanum, Clausena lansium, Atalantia buxifolia and Murraya paniculata; In Citrus, C. medica and Papeda group were first clustered, and then they were clustered with C. reticulata and C. grandis and other plants of Citrus. In C. reticulata group, Daoxian yeju and Cenxi suanju and other half wild C. reticulata were clearly distinguished from ponkan and the rest cultivated C. reticulata, but C. mangshanensis and C. indica were not in the group.【Conclusion】COS primers based on Citrus genomic information could get effective amplification and distinguish Citrus from its close and distant relatives effectively. The genetic makeup of C. mangshanensis and C. indica were heterozygous and different from C. reticulata, so they may not be the most primitive types of C. reticulata.
Citrus; COS marker; phylogenetic relationship; germplasm; genetics and evolution
2016-05-06;接受日期:2016-07-15
国家科技支撑计划(2013BAD02B02)、重庆市基础与前沿研究计划(cstc2014jcyjA80037)
联系方式:王炯,Tel:18375633275;E-mail:18375633275@163.com。通信作者洪棋斌,Tel:023-68349025;E-mail:hongqb@sina.com
