藜科植物藜和灰绿藜实时荧光定量PCR内参基因的选择
2017-01-04刘艳霞兰欣欣张晶华兰海燕
刘艳霞, 兰欣欣, 曹 婧, 张晶华, 兰海燕*
( 新疆大学 生命科学与技术学院/新疆生物资源基因工程重点实验室, 乌鲁木齐 830046 )
藜科植物藜和灰绿藜实时荧光定量PCR内参基因的选择
刘艳霞1, 兰欣欣2, 曹 婧2, 张晶华2, 兰海燕2*
( 新疆大学 生命科学与技术学院/新疆生物资源基因工程重点实验室, 乌鲁木齐 830046 )
选择合适的内参基因是实时荧光定量PCR(qRT-PCR)研究的关键,目前对藜科耐盐(盐生)植物胁迫相关基因的表达分析中所用内参基因的报道较为有限。该研究利用GeNorm、NormFinder和BestKeeper 3个内参基因分析软件,对已选择过的β-TUBULIN、β-ACTIN、GAPDH3个常用候选内参基因进行了比较分析,筛选出在NaCl和PEG胁迫下藜和灰绿藜中表达相对稳定的内参基因。结果表明:GeNorm、NormFinder内参软件分析在NaCl和PEG胁迫下,GAPDH是藜和灰绿藜中均共同稳定表达的内参基因,同时在藜和灰绿藜中也有各自表达较稳定的内参基因,β-ACTIN在藜中稳定表达,β-TUBULIN则在灰绿藜中稳定表达。对相同科不同种的植物内参基因表达差异进行比较,内参基因在相同科中具有相同稳定表达的内参;对相同胁迫下两种不同植物内参基因表达稳定性进行分析,内参基因的选择需根据实际的实验材料和实验条件而定。基于3个分析软件对以上3个常用内参基因的分析结果,初步确定了在藜科植物藜和灰绿藜中相对稳定的内参基因,为藜和灰绿藜胁迫相关基因的定量表达分析提供了参考依据。
藜, 灰绿藜, 实时荧光定量PCR, 内参基因, 胁迫
实时荧光定量PCR (quantitative real time RT-PCR,qRT-PCR) 是在传统聚合酶链式反应(polymerase chain reaction, PCR) 基础上发展起来的一项新的核酸定量技术,该技术操作快速简便,具有特异性、高敏感性、精确性等特点,在农学、微生物学、医学、食品安全检测,特别是分子生物学方面均有重要应用(陈旭等,2010;王彦杰等,2012)。qPCR和传统技术如免疫印迹方法具相似之处,但前者需要稳定的内参基因校正样品总RNA的含量(Radonic et al,2004)。目前,常用的内参基因多为管家基因(house-keeping gene),如甘油醛-3-磷酸脱氢酶基因(GLYCERALDEHYDE-3-PHOSPHATEDEHYDROGENASE,GAPDH) (Zou et al,2011 )、肌动蛋白基因(ACTIN,ACT) (Chen et al,2011)和微管蛋白基因(TUBULIN,TUB) (Zhou et al,2011)、泛素基因(UBIQUITIN) (Parra & Gomez,2011)、28S核糖体RNA基因(28SrRNA)、18S核糖体RNA基因(18SrRNA) (Miao et al,2011)、亲环蛋白基因(CYP)、多聚泛素酶基因(UBQ)、转录延伸因子基因(EF-1α) (Dheda et al,2004)等。由于这些管家基因参与生物体基本生命代谢活动,具组成型稳定表达的特性(Reid et al,2006 )。内参基因不存在通用性,随着物种及实验条件的改变,这些传统的保守基因的表达并非总是稳定的,因此对内参基因的选择应根据具体实验条件和材料方法而定(Gao et al,2012)。目前对qPCR中内参基因的稳定性的分析主要通过GeNorm、NormFinder、BestKeeper 3个常用内参基因分析软件进行稳定指数分析、ΔCt值分析。Ct也称循环阈值,表示在PCR循环过程中,每个反应管内的荧光信号到达阈值时所对应的循环次数,ΔCt值越小起始拷贝数越多(袁伟等,2012)。GeNorm程序由Vandesompele et al(2002)使用Excel编写的稳定性选择程序;NormFinder是Andersen et al(2004)基于方差分析选择合适内参基因的软件;BestKeeper程序是Pfaffl et al(2004)开发的基于对管家基因和目标基因独立分析的基因表达稳定性软件。
灰绿藜(Chenopodiumglaucum)为藜科一年生草本盐生植物,能够有效地降低土壤含盐量,增加土壤有机质,从而改善土壤性质(赵可夫等,1999)。藜(C.album)为藜科一年生耐盐草本植物,具有很强的抗逆性并具有泌盐的盐囊泡结构(邓彦斌等,1998;周三等,2011)。藜科植物已经成为植物耐盐性研究的优良材料(段德玉等,2004)。目前关于藜科植物基因表达分析的内参基因选择的报道还很有限,基于此,本研究以常用的3个内参基因β-ACTIN、β-TUBULIN、GAPDH对藜和灰绿藜两种藜科近源植物胁迫相关基因表达的内参基因进行分析比较,并基于GeNorm、NormFinder、BestKeeper 3个内参基因分析软件选择稳定表达的内参基因,分析在相同胁迫下具有亲缘关系的同科不同种植物内参基因的表达变化,以及在不同胁迫下同一种植物的内参基因表达稳定性的变化,为后续的藜科植物基因表达研究提供参考。
1 材料与方法
1.1 植物材料
选取成熟良好且完整的藜(Chenopodiumalbum)或灰绿藜(C.glaucum)的种子播种于25 cm直径并装有蛭石和珍珠岩(3∶1)基质的花盆中,于光周期16 h 昼/8 h夜,温度23~25℃,光强为150~200 μmol·m-2·s-1的环境中生长,定期浇灌1/2 Hoagland (pH 6.0)营养液(张薇等, 2015)。植株培养生长3个月后进行NaCl和PEG胁迫处理,NaCl浓度分别为50、100、300 mmol·L-1,PEG 6000浓度分别为5%、10%、20%。藜和灰绿藜都经过NaCl 5 h,PEG 48 h的胁迫处理,胁迫处理后选取株型和长势一致的植株,取植株从上向下第2~4片幼嫩叶片,立即置于液氮中,随后保存于-80 ℃冰箱备用。
1.2 方法
1.2.1 植物叶片总RNA的提取和cDNA合成 利用OMEGA植物总RNA样品提取试剂盒(编号:R6827-02 产地:美国)说明书提取样品总RNA,琼脂糖凝胶电泳检测RNA质量。取1 μL总RNA按TAKARA公司的反转录PCR试剂提供的操作步骤进行反转录获得cDNA,保存于-80 ℃备用。
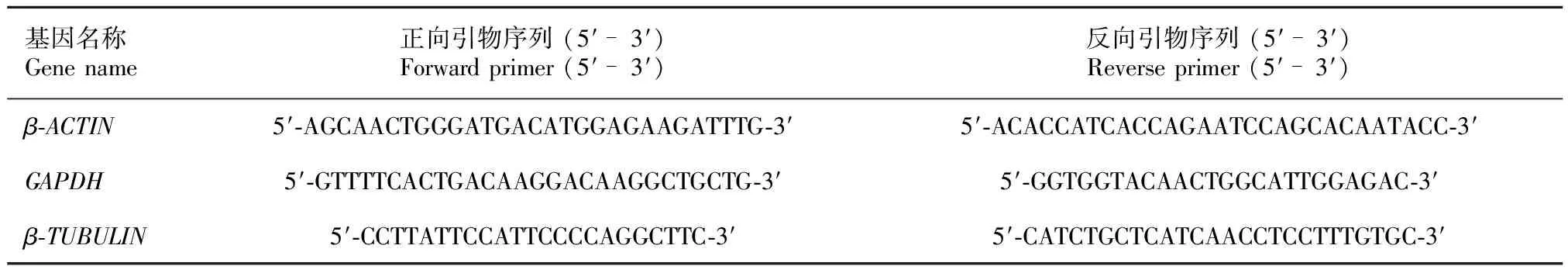
表 1 实时荧光定量PCR检测中所用的3个内参基因的引物序列
1.2.2 引物设计与内参基因的扩增 选用3个常用内参基因-肌动蛋白基因(ACTIN)、微管蛋白基因(TUBULIN)和甘油醛-3-磷酸脱氢酶基因(GAPDH)为目标,利用DNAMAN软件根据其他物种中3个基因的保守序列设计针对两种藜科植物的引物(表1)。3个内参基因的扩增反应条件均为94 ℃预变性5 min,94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s,34个循环,72 ℃延伸10 min。
1.2.3 实时荧光定量PCR分析 利用GeneAmp 7500实时PCR系统及SYBR Green (Qiagen)进行定量PCR分析,参照Qiagen公司荧光定量试剂盒具体操作步骤,将藜和灰绿藜经过胁迫处理后的cDNA为模板,每处理4个生物学重复,每重复两次技术重复,在20 μL体系中反应。具体的反应条件如下:预变性 95 ℃ 2 min;95 ℃ 5 s, 60 ℃ 30 s,40个循环。根据循环Ct值制作3种内参基因的标准曲线。
1.2.4 数据分析 通过琼脂糖凝胶电泳技术对总RNA质量和内参基因目的片段进行检测,且根据PCR循环过程中的Ct(循环阈值)进行比较,荧光定量数据采用4次生物学重复和两次技术重复(共 6 个重复)计算平均值,Ct值越小起始拷贝数越多(袁伟等,2012);并通过三个内参软件GeNorm、NormFinder、BestKeeper对选取的三个内参基因的稳定性进行分析,GeNorm根据M (平均表达稳定值)的大小进行稳定性比较,M值越小稳定性越好(Vandesompele et al,2002);NormFinder根据运算出内参基因S(稳定值)分析内参基因表达稳定性,S值越小表示内参基因稳定性越好(Andersen et al,2004)。BestKeeper是用于比较内参基因表达稳定性和基因表达水平的软件,Geo mean(几何平均数)、SD(标准变异系数)越小稳定性越好(Pfaffl et al,2004)。基于3个软件的分析比较从而筛选藜和灰绿藜在胁迫条件下最合适的内参基因组合。
2 结果与分析
2.1 总RNA质量和内参基因目的片段扩增检测
图1显示,藜和灰绿藜叶片不同处理的总 RNA样品条带清晰,无明显降解,28SrRNA与18SrRNA的亮度比值至少为2∶1。定量检测结果表明,各样品A 260 /A 280值均在 2.0 左右,说明藜和灰绿藜植株叶片总RNA较完整且纯度较高,可进行后续实验。用胁迫下藜和灰绿藜叶片 cDNA分别扩增3个内参基因β-ACTIN、β-TUBULIN、GAPDH的目的DNA片段,琼脂糖凝胶电泳分析显示3个内参基因条带与预期大小一致,且均为单一目的条带(图2),表明各内参基因引物均具有较好的特异性,可用于后续实时荧光定量PCR分析。
2.2 实时荧光定量PCR分析
实时荧光定量PCR分析结果显示,内参基因的标准曲线线性相关系数为R2≥ 0.999,引物的扩增效率为85.6%~92.1% (表2)。以上数据均符合实时荧光定量PCR对扩增效率的要求。各内参基因的熔解曲线只有明显的单一信号峰,样品重复间的扩增重复性较高,阴性对照未检测到荧光信号。这表明实时荧光定量PCR反应中模板与引物结合较好,结果具较高的特异性。
2.3 内参基因的表达稳定性分析
通过GeNorm、NormFinder、BestKeeper 3个内参分析软件对实时荧光定量PCR仪所得到的Ct值进行内参基因稳定性分析。ΔCt值是样品中各基因的循环阈值,ΔCt值最小的基因表达稳定性越高(袁伟等,2012)。图3所示的是藜和灰绿藜中不同内参基因的表达水平(Ct值)。结果显示,藜的β-ACTIN的Ct值的变化范围最小,Ct值变化范围其次是GAPDH,β-TUBULIN;在灰绿藜中β-TUBULIN的Ct值变化范围最小,其次是GAPDH,β-ACTIN。由ΔCt值分析结果可知,在藜中β-ACTIN表达较稳定,在灰绿藜中β-TUBULIN表达较稳定。
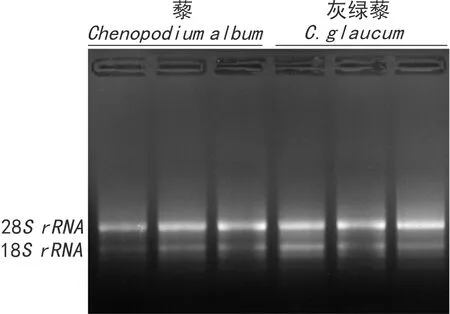
图 1 藜和灰绿藜总RNA琼脂糖凝胶电泳图Fig. 1 Agarose gel electrophoresis of total RNA extracted from Chenopodium album and C. glaucum
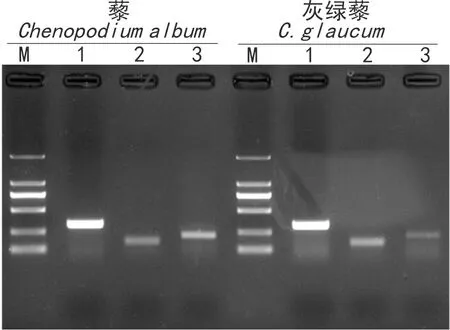
图 2 藜和灰绿藜中3个内参基因的PCR扩增产物1. 肌动蛋白基因; 2. 甘油醛-3-磷酸脱氢酶基因; 3. 微管蛋白基因; M. 分子量标记。Fig. 2 Agarose gel analysis of three candidate reference genes amplification in Chenopodium album and C. glaucum1. β-ACTIN ; 2. GAPDH; 3. β-TUBULIN; M. Marker.
2.3.1 内参基因的GeNorm分析 GeNorm软件通过比较M值确定最稳定内参基因,M值越小表明稳定性越好。通过GeNorm软件分析(图4),藜在NaCl胁迫下β-TUBULIN、β-ACTIN、GAPDH3个内参的平均M值依次为0.726 562、0.552 129、0.552 129,表达稳定度由低到高排列顺序为β-TUBULIN<β-ACTIN=GAPDH,β-ACTIN和GAPDH2个内参的表达均较稳定。在PEG胁迫下3个内参基因表达稳定度由低到高的顺序为GAPDH<β-ACTIN=β-TUBULIN,其中β-ACTIN和β-TUBULIN两个内参的表达均较稳定。灰绿藜在NaCl胁迫下3个内参基因的表达稳定度由低到高顺序为β-ACTIN<β-TUBULIN=GAPDH,其中β-TUBULIN和GAPDH两个内参的表达均较稳定。灰绿藜在PEG胁迫下3个内参基因的表达稳定度由低到高排列顺序为β-ACTIN<β-TUBULIN=GAPDH, 其中β-TUBULIN和GAPDH两个内参的表达均较稳定。由以上结果可知, 藜的β-ACTIN较稳定,β-TUBULIN和GAPDH的稳定性较低;灰绿藜的β-TUBULIN和GAPDH的稳定性最高,β-ACTIN的稳定性最低。
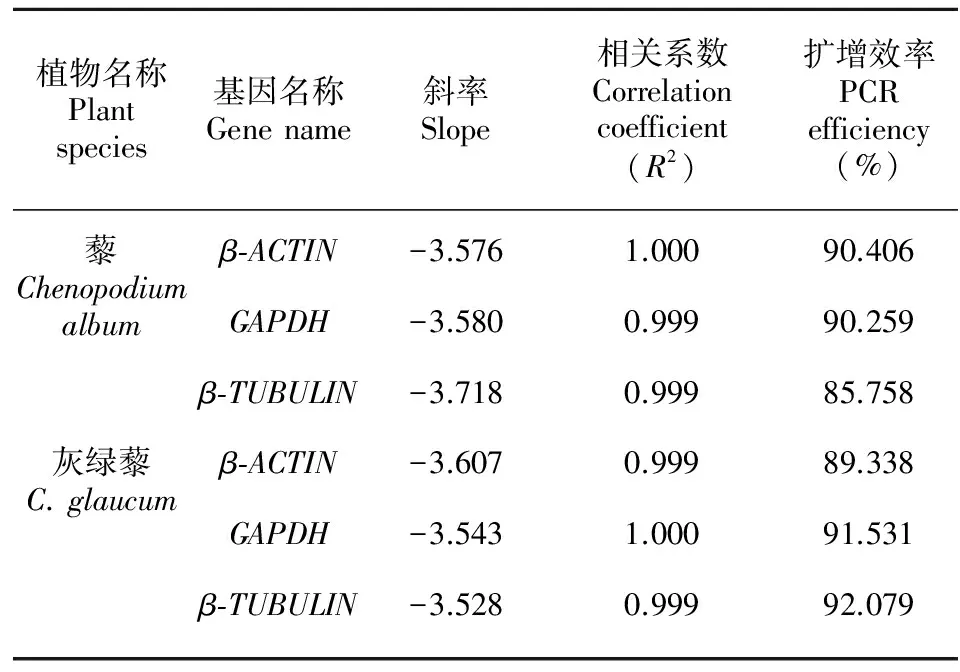
表 2 藜和灰绿藜实时荧光定量PCR分析中3种内参基因的相关系数及扩增效率
注:β-ACTIN. 肌动蛋白基因;β-TUBULIN. 微管蛋白基因;GAPDH. 甘油醛-3-磷酸-脱氢酶基因。
Note:β-ACTIN;β-TUBULIN;GAPDH. Glyceraldehyde-3-phosphatedehydrogenase.
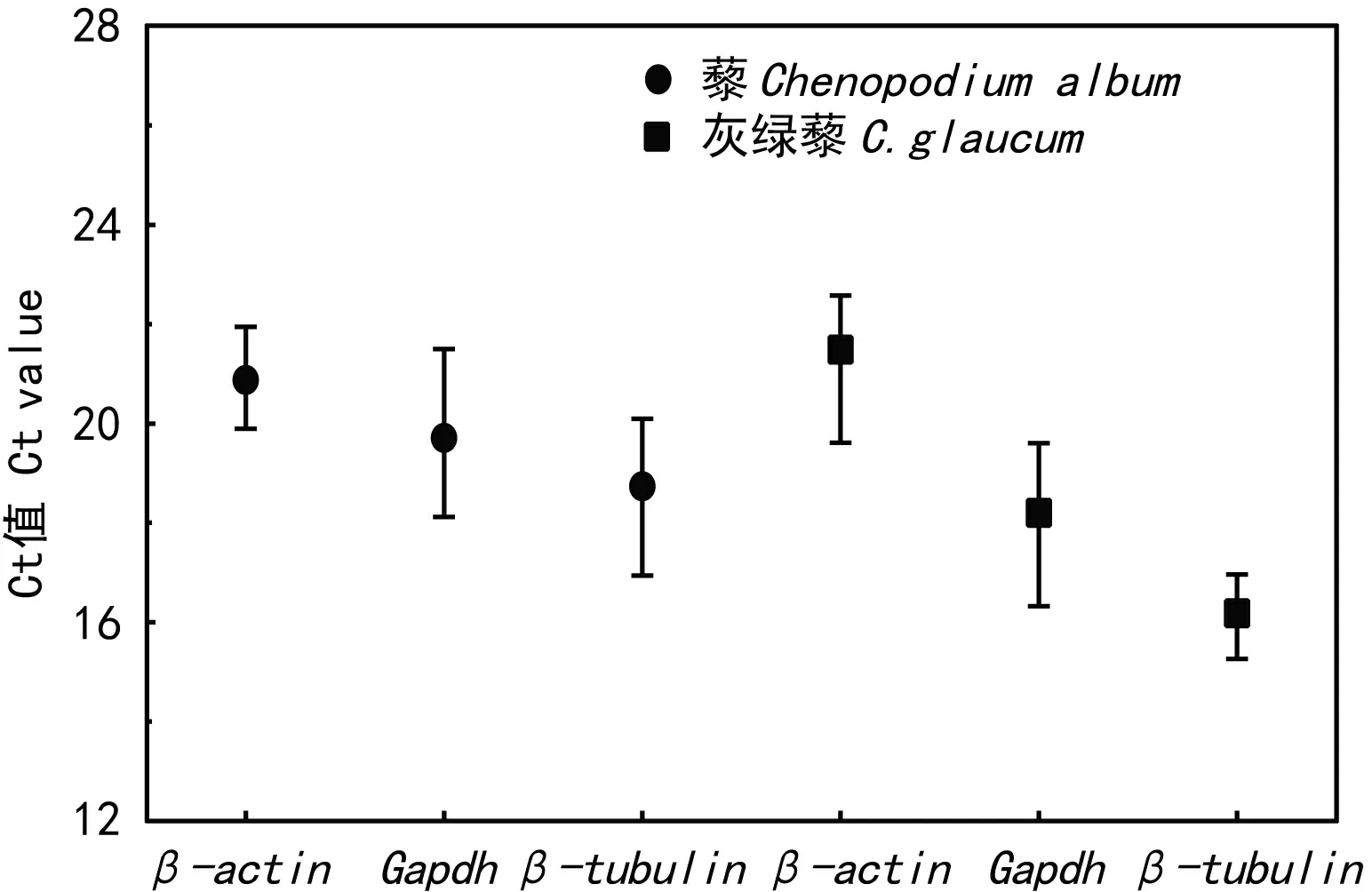
图 3 藜和灰绿藜中内参基因的表达水平(Ct值)Fig. 3 Graphical representation of the expression level of the candidate reference genesin Chenopodium album and C. glaucum
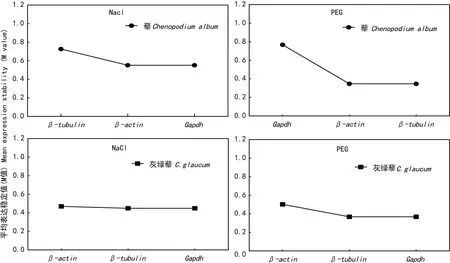
图 4 GeNorm软件分析候选内参基因稳定性Fig. 4 Stability of candidate reference genes analyzed by GeNorm software
2.3.2 内参基因的NormFinder分析 NormFinder软件利用方差分析法对内参基因的稳定性进行分析,所得值越小稳定性越好。与GeNorm软件的分析结果一致,在NaCl和PEG胁迫下,藜的最佳内参基因是β-ACTIN;灰绿藜的最佳内参基因是GAPDH。NormFinder软件计算结果显示(表3),在NaCl胁迫下,3个内参基因在藜中表达稳定度由低到高排列顺序为GAPDH<β-TUBULIN<β-ACTIN,β-ACTIN为最优内参基因。在PEG处理下,3个内参基因在藜中的表达稳定度由低到高排列顺序为GAPDH<β-TUBULIN<β-ACTIN,β-ACTIN是最优内参基因。在NaCl胁迫下,3个内参基因在灰绿藜中的表达稳定度排列顺序为β-ACTIN<β-TUBULIN 2.3.3 内参基因的Bestkeeper分析 BestKeeper软件可以对内参基因和目标基因进行单独分析,是用于分析不同的实验处理下内参基因参数的软件,参数大小反应内参基因稳定性差异,值小的内参基因表达较稳定。BestKeeper软件默认的SD值大于1时,说明该内参基因的稳定性较差。 表4显示,对藜而言,在NaCl胁迫下内参基因β-TUBULIN的SD值为0.48,Geo mean为18.16,Min、Max分别为16.94,19.00,在3个内参基因中值最小,故β-TUBULIN为表达最稳定的内参基因;在PEG胁迫下内参基因β-TUBULIN的SD值为0.32,Geo mean为19.32,Min,Max值分别为18.69、20.10,在3个内参基因中值也最小,故在PEG处理下β-TUBULIN也是表达最稳定的内参基因。对灰绿藜而言,在NaCl胁迫下内参基因β-TUBULIN的SD值为0.29,Geo mean为16.17,Min、Max值分别为15.58、16.93,在3个内参基因中值最小,因此β-TUBULIN为表达最稳定的内参基因;在NaCl胁迫下内参基因β-ACTIN的SD值为0.61,Geo mean(几何平均数)为21.43,Min、Max值分别为19.61、22.58,在3个内参基因中值最小, 因此β-ACTIN为表达最稳定的内参基因。 表 3 NormFinder软件分析候选内参基因稳定性 注: S. 稳定值。 Note: S. Stable value. 表 4 Bestkeeper软件分析候选内参基因稳定性 注: [Ct]. 循环数; Geo Mean. 几何平均数; Min, max [Ct]. 极端值 [Ct]; Min, max [x-fold]. 表达水平的极端值; Std dev [± Ct]. 标准变异系数 [± Ct]。 Note: Ct. Cycle threshold; Geo Mean. Geometric mean; Min, max [Ct]. Extreme values of Ct; Min, max [x-fold]. Extreme values of expression levels expressed as absolute x-fold over or under coefficient; Std dev [± Ct]. Standard deviation [Ct]. 实时荧光定量PCR技术具有特异性、准确性高、灵敏度高等特点,为了实验获得更好的结果,这项技术必须严格遵循相关的实验规范(Udvardi et al,2008;Bustin et al,2009)。内参基因表达随着实验条件的变化而变化(Czechowski et al,2005;Paolacci et al,2009;Nicot et al,2005),目的基因最终计算结果需要用内参基因进行校正,所以针对不同的实验条件分析和筛选稳定的内参基因很重要。在筛选相关内参基因时可首先参考前人研究的报道,若无相关内参基因信息,应选择常用的且稳定表达的内参基因再进行后续实验(魏毅东等,2013)。本研究中,GAPDH在藜和灰绿藜中均稳定表达,说明属于同种的植物具有相似性,在内参基因的稳定性表达上有相同的情况,然而相同的内参基因在同一种植物中表达不一定相同,即β-ACTIN在藜中表达较稳定,β-TUBULIN则在灰绿藜中表达较稳定。GAPDH及β-ACTIN作为内参基因被广泛应用(孙美莲等,2010;Lovdal & Lillo,2009)。理想的内参基因表达稳定性不随光周期、温度等外界环境的变化而改变,内参基因在所有细胞中均可稳定表达(Paolacci et al, 2009)。在不同组织器官和生理状态下均稳定表达的内参基因是不存在的,内参基因的稳定性具有相对性,相同生理条件下不同内参基因的表达量通常不同,一种实验条件下表现稳定的内参基因在另一种条件下表达可能是不稳定的(Nicot et al,2005)。Bustin et al (2009)证明实验中使用不稳定的内参基因会影响结果的可靠性。因此,在利用实时荧光定量PCR进行基因表达分析时,为了得到更可靠的实验结果,在实验过程中需要根据研究对象和实验条件选择合适的内参基因(Ohl et al,2005)。 GeNorm、NormFinder、BestKeeper是基于统计学的3个内参分析软件。GeNorm内参软件是Vandesompele et al (2002)发明对内参基因稳定性进行分析的软件。通过软件运算出基因表达稳定性的M值分析内参基因表达稳定性,M值越小稳定性越好。GeNorm软件还可确定在不同生理条件下最适的内参基因数目具有可靠的表达分析结果。NormFinder内参软件是Andersen et al (2004)发明的基于方差分析判断在不同条件下内参基因表达稳定性的程序,NormFinder运算出内参基因的表达稳定值,按稳定值的大小排序,值越小稳定性越好,进而选择出最稳定表达的内参基因。BestKeeper内参软件是Pfaffl et al(2004)开发的用于比较内参基因表达稳定性和基因表达水平的软件,Ct值标准偏差和变异系数越小稳定性越好。 本研究选用GeNorm、NormFinder、BestKeeper 3个常用内参分析软件对ΔCt(qRT-PCR过程中循环数)、M(平均表达稳定性值)、S(稳定值)、Geo mean(几何平均数)、SD(标准变异系数)等数值对内参基因的稳定性进行分析。GeNorm软件对3个内参基因分析显示:藜在NaCl和PEG胁迫处理下β-ACTIN和GAPDH是表达较稳定的内参基因,灰绿藜相对表达稳定的内参基因是β-TUBULIN和GAPDH,NormFinder的计算结果和GeNorm一致。BestKeeper内参软件的分析结果显示,β-TUBULIN在经过NaCl和PEG胁迫的藜中和NaCl处理下的灰绿藜中是表达较稳定的内参基因,β-ACTIN为PEG胁迫下灰绿藜中表达稳定的基因。BestKeeper和NormFinder、GeNorm软件分析结果不完全一致。ΔCt值是样品中各基因的循环阈值,ΔCt值最小的基因表达稳定性越高,通过对2个内参基因的ΔCt(RT-PCR过程中循环数)值比较可助筛选表达最稳定的管家内参基因(胡瑞波等,2009)。通过对ΔCt进行分析,结果显示在藜中β-ACTIN内参基因的Ct值变化范围最小,其次是GAPDH,β-TUBULIN的Ct值最小;在灰绿藜中β-TUBULIN的Ct值变化范围最小,与NormFinder、GeNorm软件分析一致。 3个内参软件对3个内参基因进行分析时,GeNorm和NormFinder分析的结果完全一致,而BestKeeper在藜的内参基因分析时结果和GeNorm和NormFinder分析结果部分不一致,造成这种差异的根本原因可能是软件的统计学原理不同造成的(Hu et al,2009)。大多发表的文章均已使用GeNorm和NormFinder内参软件进行内参稳定性分析并获得合适的内参基因(Kim S & Kim T,2003;Savli et al,2003;Aandersen et al,2004;Hong et al,2008;陈孝仁等,2013)。本研究通过GeNorm、NormFinder两个内参软件以及对实时荧光定量PCR的Ct值进行分析显示,没有一个固定的基因在任何条件下均稳定表达。本研究由NormFinder和GeNorm软件分析结果得出一致结论:藜中β-ACTIN在NaCl和PEG胁迫下均稳定表达;在灰绿藜中NaCl和PEG胁迫下β-TUBULIN均稳定表达,而GAPDH是两种藜科植株中共同稳定表达的内参基因。由此暗示,相同科不同种的植物在内参基因表达上有共性也有差异。在相同胁迫下的内参基因表达稳定性是随机的,内参基因的表达稳定性与胁迫条件无关,因此内参的选择还要根据实验材料和实验条件进行选择(Warrington et al,2000;Suzuki et al,2000)。 ANDERSEN CL, JENSEN JL, ØMTOFT TF, 2004. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets [J]. Canc Res, 64(15): 5245-5250.BUSTIN SA, BENES V, GARSON JA, et al,2009. The MIQE guidelines: Minimum information for publication of quantitative Real-time PCR experiments [J]. Clin Chem, 55(4): 611-622. CHEN J H, SUN Y, SUN F, et al, 2011. Tobacco plants ectopically expressing theAmmopiptanthusmongolicusAmCBL1 gene display enhanced tolerance to multiple abiotic stresses [J]. Plant Growth Regul, 63(3): 259-269. CHEN X, QI FK, KANG LG, et al, 2010. Advance and application of real-time fluorescent quantitative PCR [J]. J NE Agric Univ, 41(8): 148-155. [陈旭, 齐凤坤, 康立功, 等, 2010. 实时荧光定量PCR技术研究进展及其应用 [J]. 东北农业大学学报, 41(8): 148-155.]CHEN XR, ZHANG BY, XING YP, et al, 2013. Selection of internal control genes for real-time RT-PCR data normalization inPhytophthoracactorum[J]. Chin Agric Sci Bull, 28(30): 201-207. [陈孝仁, 张博月, 邢玉平, 等, 2013. 恶疫霉实时定量RT-PCR分析中内参基因的选择 [J]. 中国农学通报, 28(30): 201-207.] CZECHOWSKI T, STITT M, ALMANN T, et al, 2005. Genome wide identification and testing of superior reference genes for transcript normalization inArabidopsis[J]. Plant Physiol, 139(1): 5-17. DENG YB, JIANG YC, LIU J, 1998. The xeromorphic and saline morphic structure of leaves and assimilating branches in ten chenopodiacea species in xinjiang [J]. Acta Phytoecol Sin, 22(2): 164-170. [邓彦斌, 姜彦成, 刘健, 1998. 新疆10种藜科植物叶片和同化枝的旱生和盐生结构的研究 [J]. 植物生态学报, 22(02): 164-170.] DHEDA K, HUGGETT JF, BUSTIN SA, et al, 2004. Validation of housekeeping genes for normalizing RNA expression in real-time PCR [J]. Biotechniques, 37(1): 112-119. DUAN DY, LIU XJ, FENG FL, et al, 2004. Effects of salt and water stress on seed germination of halophyteChenopodiumglaucumL. [J]. J Plant Res Environ, 13(1): 7-11. [段德玉, 刘小京, 冯凤莲, 等, 2004. 盐分和水分胁迫对盐生植物灰绿藜种子萌发的影响 [J]. 植物资源与环境学报, 13(1): 7-11.] GAO ZH, WEI JH, YANG Y, et al, 2012. Selection and validation of reference genes for studying stress-related agarwood formation ofAquilariasinensis[J]. Plant Cell Rep, 31(9): 1759-1768. HAKAN S, AYNUR K, FETIVE K, et al, 2003. Expression stability of six housekeeping genes: A proposal for resistance gene quantification studies ofPseudomonasaeruginosaby real-time quantitative RT-PCR [J]. J Med Microl, 52(5): 403-408. HONG SY, SEO PJ, YANG MS, et al, 2008. Exploring valid reference genes for gene expression studies inBrachypodiumdistachyonby real-time PCR [J]. BMC Plant Biol, 8(1): 112-123. HU RB, FAN CM, FU YF, 2009. Reference gene selection in plant real-time quantitative reverse transcription PCR (qRT-PCR) [J]. J Agric Sci Technol, 11(6): 30-36. [胡瑞波, 范成明, 傅永福, 2009. 植物实时荧光定量PCR内参基因的选择 [J]. 中国农业科技导报, 11(6): 30-36.] JAIN M, NIJHAWAN A, TYAGI AK, et al, 2006. Validation of housekeeping genes as internal control for studying gene expression in rice by quantitative real-time PCR [J]. Biochem Bioph Res Co, 345(2): 646-651. KIM S, KIM T, 2003. Selection of optimal internal controls for gene expression profiling of liver disease [J]. Biotechniques, 35(3): 456-460. LOVDAL T, LILLO C, 2009. Reference gene selection for quantitative real-time PCR normalization in tomato subjected to nitrogen, cold, and light stress [J]. Anal Biochem, 387(2): 238-242. MIAO M, YANG X, HAN X, et al, 2011. Sugar signalling is involved in the sex expression response of monoecious cucumber to low temperature [J]. J Exp Bot, 62(2): 797-804. NICOT N, HAUSMAN JF, HOFFMANN, et al, 2005. Housekeeping gene selection for Real-time RT-PCR normalization in potato during biotic and abiotic stress [J]. J Exp Bot, 56(421): 2907-2914. OHL F, JUNG M, XU C, et al, 2005. Gene expression studies in prostate cancer tissue: which reference gene should be selected for normalization [J]. J Mol Med, 83(12): 1014-1024. PAOLACCI AR, TANZARELLA OA, PORCEDDU, et al, 2005. Identification and validation of reference genes for quantitative RT-PCR normalization in wheat [J]. BMC Mol Biol, 10 (11): 1-27. PARRA MC, GOMEZ MC, 2011. Polyamine-induced modulation of genes involved in ethylene biosynthesis and signalling pathways and nitric oxide production during olive mature fruit abscission [J]. J Exp Bot, 62(13): 4447-4465. PFAFFL MW, TICHOPAD A, PRGOMET C, et al, 2004. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: Bestkeeper-excel-based tool using pair-wise correlations [J]. Biotechnol Lett, 26(6): 509-515. RADONIC A, THULKE S, MACKAY IM, et al, 2004. Guideline to reference gene selection for quantitative Real-time PCR [J]. Biochem Bioph Res Co, 313(4): 856-862. REID KE, OLSSON N, SCHLOSSER J, et al, 2006. An optimized grapevine RNA isolation procedure and statistical determination of reference genes for real-time RT-PCR during berry development [J]. BMC Plant Biol, 6 (27): 1-11. SUN ML, WANG YS, YANG DQ, et al, 2010. Reference genes for real-time fluorescence quantitative PCR inCamelliasinensis[J]. Chin Bull Bot, 45(5): 579-587. [孙美莲, 王云生, 杨冬青, 等, 2010. 茶树实时荧光定量PCR分析中内参基因的选择 [J]. 植物学报, 45(5): 579-587.] SUZUKI T, HIGGINS P, CRAWFORD D, 2000. Control selection (Continueonpage1459)(Continuefrompage1518) for RNA quantitation [J]. Biotechniques, 29(2): 332-337. UDVARDI MK, CZECHOWSKI T, SCHEIBLE WR, 2008. Eleven golden rules of quantitative RT-PCR [J]. Plant Cell, 20(7): 1736-1737. VANDESOMPELE J, PRETER KD, PATTYN F, et al, 2002. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes [J]. Genome Biol, 3(7): research0034. 1-research0034.11. WARRINGTON JA, NAIR A, MAHADEVEPPA M, et al, 2000. Comparison of human adult and fetal expression and identification of 535 housekeeping/ maintenance genes [J]. Physiol Genomics, 2(3): 143-147. WANG YJ, DONG L, ZHANG C, et al, 2012. Reference gene selection for real-time quantitative PCR normalization in tree peony (PaeoniasuffruticosaAndr. ) [J]. J Agric Biol, 20(5): 521-528. [王彦杰, 董丽, 张超, 等, 2012. 牡丹实时定量PCR分析中内参基因的选择 [J]. 农业生物技术学报, 20(5): 521-528.] WEI YD, CHEN Y, GUO HP, et al, 2013. Selection of reference genes for real-time quantitative RT-PCR in rice (OryzasativaL. ssp.japonica) under nutrient deficiency [J]. J Agric Biol, 21(11): 1302-1312. [魏毅东, 陈玉, 郭海萍, 等, 2013. 水稻缺素胁迫下实时荧光定量RT-PCR的内参基因的选择 [J]. 农业生物技术学报, 21(11): 1302-1312.]YUAN W, WAN HJ, YANG YJ, 2012. Characterization and selection of renference genes for real-time quantitative RT-PCR of plants [J]. Chin Bull Bot, 47 (4): 427-436. [袁伟, 万红建, 杨悦俭, 2012. 植物实时荧光定量PCR内参基因的特点及选择 [J]. 植物学报, 47(4): 427-436.]ZHANG W, CUI WT, DUAN XL, et al, 2015. Reference gene selection for qRT-PCR normalization in alfalfa under UV-B irradiation [J]. J Nanjing Agric Univ, 2015, 38(2): 248-254. [张薇, 崔为体, 段星亮, 等, 2015. UV-B胁迫下紫花苜蓿qRT-PCR内参基因的筛选 [J]. 南京农业大学学报, 38(2): 248-254.]ZHAO KF, LI FZ, FAN SJ, et al, 1999. Halophytes in China [J]. Chin Bull Bot,16(3): 201-207. [赵可夫, 李法曾, 樊守金, 等, 1999. 中国的盐生植物 [J]. 植物学报, 16(3): 201-207.] ZHOU L, WANG Y, PENG ZH, 2011. Molecular characterization and expression analysis of chalcone synthase gene during flower development in tree peony (Paeoniasuffruticosa) [J]. Afr J Biotechnol, 10(8): 1275-1284. ZHOU S, HAN JL, ZHAO KF, 2011. Advance of study on recretohalophytes [J]. Chin J Appl Environ Biol, 7(5): 496-501. [周三, 韩军丽, 赵可夫, 2011. 泌盐盐生植物研究进展 [J]. 应用与环境生物学报, 7(5): 496-501.] ZOU AL, ZHANG WJ, PAN QY, et al, 2011. Cloning characterization and expression of LeEIL-1, anArabidopsisEIN3 homolog, inLithospermumerythrorhizon[J]. Plant Cell Tiss Org, 106(1): 71-79. Screening of qRT-PCR reference genes for ChenopodiumalbumandC.glaucumof Chenopodiaceae LIU Yan-Xia1, LAN Xin-Xin2, CAO Jing2, ZHANG Jing-Hua2, LAN Hai-Yan2* ( College of Life Sciences and Technology, Xinjiang University, Xinjiang Key Laboratory ofBiologicalResourcesandGeneticEngineering, Urumqi 830046, China ) Selection of suitable reference gene is a critical step in real-time quantitative PCR (qRT-PCR) analysis. So far, reports on reference gene screening on stress-tolerant gene expression of Chenopodiaceae species are limited. In the present study, by using three reference-gene-analysis softwares-GeNorm, NormFinder, BestKeeper, the stability of three commonly used candidate reference genesβ-ACTIN,β-TUBULINandGAPDHofChenopodiumalbumandC.glaucumunder NaCl and PEG treatments were compared. The results showed that under NaCl and PEG stresses,GAPDHshowed stable both inC.albumandC.glaucum. The results also demonstated that the same reference genes expressed stable in the same family.β-ACTINexpressed stable inC.album, whileβ-TUBULINexpressed stable inC.glaucum. We also studied the plants of different gera in the same family and the same stress of the stability of the expression of reference genes. The results suggested that same reference gene expressed stable in the same species. For the same stress in these two species, the results showed that selection of the most stable reference gene depended on the experimental materials and the conditions. The analysis results of the three candidate reference genes were based on the three analysis softwares. In our study, we determined the ralatively stable reference genes inC.albumandC.glaucum. This study provides the reference for the further study on qRT-PCR analysis of stress-relevant gene expression in Chenopodiaceae species. Chenopodiumalbum,C.glaucum, quantitative real-time PCR (qRT-PCR), reference gene, stress 10.11931/guihaia.gxzw201601034 2016-03-22 2016-06-20 国家自然科学基金(31260037, 31460043, 31660068);新疆自治区优秀青年科技人才培养项目(2013721013) [Supported by the National Natural Science Foundation of China (31260037, 31460043, 31660068);Program for Training Young Talents of Xinjiang Uygur Autonomous Region (2013721013)]。 刘艳霞(1994-),女,甘肃民勤人,硕士研究生,研究方向为生物化学与分子生物学,(E-mail)15292852327@163.com。 *通讯作者: 兰海燕,博士,教授,研究方向为植物抗逆分子生物学,(E-mail)lanhaiyan@xju.edu.cn。 Q945.11 A 1000-3142(2016)12-1511-09 刘艳霞, 兰欣欣, 曹婧, 等. 藜科植物藜和灰绿藜实时荧光定量PCR内参基因的选择 [J]. 广西植物, 2016, 36(12):1511-1518 LIU YX, LAN XX, CAO J, et al. Screening of qRT-PCR reference genes forChenopodiumalbumandC.glaucumof Chenopodiaceae [J]. Guihaia, 2016, 36(12):1511-1518
3 讨论与结论
