一个受黄萎病菌诱导的海岛棉功能基因GbVWR的克隆与表达
2016-12-16张力佳张艳荣张桂寅吴立强李志坤吴金华马峙英王省芬
张力佳张 艳荣 伟 杨 君 张桂寅 吴立强 李志坤吴金华 马峙英 王省芬
华北作物种质资源研究与利用重点实验室 / 河北省作物种质资源重点实验室 / 河北农业大学, 河北保定 071001
一个受黄萎病菌诱导的海岛棉功能基因GbVWR的克隆与表达
张力佳**张 艳**荣 伟 杨 君 张桂寅 吴立强 李志坤吴金华 马峙英 王省芬*
华北作物种质资源研究与利用重点实验室 / 河北省作物种质资源重点实验室 / 河北农业大学, 河北保定 071001
黄萎病是一种土传真菌维管束病害, 严重影响棉花产量和品质。挖掘抗黄萎病相关基因对于棉花抗黄萎病遗传改良具有重要意义。本研究通过筛选黄萎病菌胁迫下的海岛棉全长cDNA文库和陆地棉SSH文库, 获得一个与黄萎病菌胁迫相关的基因, 命名为GbVWR。生物信息学特征和基因表达分析表明, GbVWR基因全长520 bp, 开放读码框198 bp, 编码65个氨基酸残基组成的蛋白, 理论等电点为5.32, 包含一个信号肽和一个跨膜区, 是一种分泌蛋白。GbVWR基因可以诱导表达7 kD的蛋白。在GbVWR基因起始密码子上游1500 bp的核苷酸序列区间预测到真菌激发子响应、激素响应、伤口响应及黄酮生物合成基因调节等应答元件。GbVWR基因在海岛棉根中表达最高, 茎中其次, 叶中最低。受黄萎病菌诱导后, GbVWR基因在接菌后2 h可对黄萎病菌作出应答反应。此外SA、ET和GA均可显著诱导GbVWR基因的表达。初步推断GbVWR是海岛棉抵御黄萎病菌过程中一个新的功能基因, 通过参与多种激素信号途径发挥功能。
海岛棉; 抗黄萎病; 未知基因(GbVWR); 克隆; 基因表达
陆地棉(Gossypium hirsutum)和海岛棉(G. barbadense)是世界上两大主要栽培棉种。陆地棉以产量高、适应性强等特点而成为第一大栽培棉种, 年产量超过总产量的 95%。黄萎病是棉花的重要病害之一[1], 常带来较大产量损失和品质下降, 是当前影响棉花生产的重要因素[2-3]。黄萎病致病菌从棉花根部侵染, 通过维管束进入植物体进一步繁殖扩散,最终引起棉株系统发病[4]。目前, 防治黄萎病尚无高效杀菌剂和有效栽培耕作方法[6-7], 种植抗病品种被认为是一种最有效、最经济和环保的策略[8-9]。
陆地棉和海岛棉起源于共同的祖先, 但二者在农艺性状上具有显著的差别[11]。陆地棉中缺乏免疫或高抗黄萎病的抗源材料, 而栽培海岛棉具有免疫或高抗黄萎病、且品质优良的特点[7,11-12]。将海岛棉的黄萎病抗性转育到陆地棉或利用基因工程手段将海岛棉的抗病基因转化到陆地棉中来改良陆地棉的抗病性是现代棉花抗病育种的重要策略之一。因此研究海岛棉抗黄萎病机制及分离鉴定抗病相关基因对于棉花抗黄萎病分子育种具有重要意义。
棉花的抗病分子机制极为复杂[3,10]。目前, 通过海陆杂交将海岛棉的黄萎病抗性转育到陆地棉存在很多困难, 杂交后代往往由于种间杂交不亲和出现杂交衰败[12-13]。因此, 更好地了解棉花抗黄萎病的分子机制尤为重要。关于棉花抗黄萎病相关基因的挖掘与克隆已取得一定进展。例如, Zhang等[14]利用同源法克隆了海岛棉GbVe基因, 该基因与番茄Ve1基因同源, 并进一步证明转基因拟南芥增强了对黄萎病的抗病性。随后, 有研究者也从海岛棉中克隆了Gbve1基因, 且证明Gbve1对落叶型和非落叶型大丽轮枝菌具有抗性[15]。还有研究利用VIGS技术证明棉花抗黄萎病需要 GhNDR1和 GhMKK2的参与[16], GbSTK、GhPAO、GbHyPRP1等基因对棉花抗黄萎病具有作用[17-19]。近年来, 许多研究利用转录组测序、蛋白质组学及芯片技术鉴定了大量的参与棉花与大丽轮枝菌互作过程的差异表达基因[20-21], 然而这些基因与抗病遗传的关系及对抗性的贡献仍不清楚, 因此需要开展深入研究, 以明确基因的功能,为进一步解析棉花抗黄萎病的分子机制及抗病分子育种奠定重要基础。
随着二倍体、四倍体棉种基因组测序的完成,基因组序列信息为深入挖掘与利用棉花重要功能基因奠定基础[22-26]。棉花抗黄萎病基因的研究已有许多报道, 然而, 仍有部分序列信息在基因组对应区段中没有任何解读, 可能属于未知基因。课题组前期构建的黄萎病菌诱导下海岛棉全长cDNA文库陆地棉SSH文库及转录组文库测序中均发现多条受黄萎病菌诱导而功能信息完全未知的基因序列, 对这些基因开展相关研究, 将为解析棉花抗黄萎病机制奠定基础, 为棉花抗黄萎病增添新的功能基因。通过筛选黄萎病菌诱导的抗病海岛棉全长cDNA文库[21]和耐病陆地棉 SSH 文库[27], 发现一个受黄萎病菌快速、强烈诱导表达的基因, 经NCBI比对, 该基因目前没有任何注释信息, 将其命名为GbVWR。为了解GbVWR在棉花抗黄萎病中的作用, 本研究对该基因进行生物信息学、组织表达特异性、黄萎病菌胁迫及激素诱导的表达模式分析。研究结果将为进一步研究该基因的功能提供理论参考。
1 材料与方法
1.1 试验材料
以抗黄萎病海岛棉品系Pima 90-53为材料, 选取饱满、一致的种子, 经干净自来水浸泡直至露白,放在发芽盘中, 28℃恒温暗培养 8~12 h, 待长出0.5~1.0 cm 胚根时, 挑选发芽一致的种于浇透营养液的蛭石中, 上面盖一层蛭石及保鲜膜, 在温光湿可控的人工气候室培养, 待棉苗生长至二叶一心时用于接菌试验或激素处理。
1.1.1 黄萎病菌诱导处理 强致病力落叶型菌系(Verticillium dahliae)临西 2-1由本室分离鉴定并继代保存于PDA培养基[28]。挑选生长状况健康一致的棉苗, 参照 Zhang等[21]的方法接菌, 根据大丽轮枝菌侵染植物根部的定殖过程[29], 参照囊括大丽轮枝菌孢子附着、萌发, 形成菌丝及进一步扩展等各时间段, 分别在接菌后2、4、6、12、24和36 h取植株根部组织。每个时间点设3个生物学重复。对照组用蒸馏水代替黄萎病菌液对棉苗进行处理。取样时, 先用灭菌水将根清洗干净, 再将根、茎、叶分别用液氮速冻并保存在-80℃。提取上述各样本总RNA用于GbVWR基因表达分析
1.1.2 乙烯(ET)和水杨酸(SA)处理 挑选生长健康一致的棉苗, 叶面喷施ET (5 mmol L-1)和SA (10 mmol L-1)溶液, 对照组喷施无菌水。分别在处理后6、12、24、36和48 h取植株根组织, 每个时间点取3个生物学重复; 分别提取各样本总RNA, 分析GbVWR基因受激素诱导的表达规律。
1.1.3 赤霉素(GA)处理 参照 Peng等[30]的方法水培海岛棉幼苗, 待幼苗长至二叶一心时选取生长健壮、整齐一致的幼苗转移至含有100 mg L-1[31-32]赤霉素水溶液中生长, 对照组采用无菌水处理。分别取处理后6、12、24、36和48 h的根, 每个时间点取3个生物学重复。样品经液氮速冻并保存在-80℃备用
1.2 棉花总RNA的提取及cDNA的合成
将-80℃保存的样本用液氮研磨成粉状, 使用EASYspin植物RNA提取试剂盒(重庆波尔生物科技有限公司), 按照说明书提取样本总RNA。通过琼脂糖电泳和NanoDrop2000超微量分光光度计(Thermo Scientific)检测RNA的完整度和浓度。采用ReverTra Are qPCR RT Master Mix with gDNA Remover (TOYOBO)试剂盒, 参照说明书合成第1链cDNA。
1.3 GbVWR基因gDNA和cDNA序列的克隆
黄萎病菌胁迫处理的海岛棉Pima 90-53根组织全长 cDNA文库由本课题组构建并保存[25]。利用cDNA文库中EST 序列在棉花D基因组进行比对,根据比对到的序列设计引物ORF-F和ORF-R (表1),分别以Pima 90-53根基因组DNA和cDNA为模板进行PCR扩增。PCR产物经0.8%琼脂糖凝胶电泳检测后回收目的条带。将目的片段连接到PMD19-T (TaKaRa)载体, 再通过转化大肠杆菌TOP10及菌落PCR检测, 最终测序分析获得目的基因序列。采用EasyPure Plant Genomic DNA Kit (TransGen Biotech)提取棉苗根基因组 DNA; 分别采用 EasySpin植物RNA提取试剂盒(重庆波尔生物科技有限公司)和ReverTra Are qPCR RT Master Mix with gDNA Remover (TOYOBO)按照相应说明书提取RNA及合成cDNA。
1.4 GbVWR基因的生物信息学分析
通过NCBI的Blast和CDD数据库分析GbVWR的氨基酸序列和结构域; 利用ProtParam (http://web. expasy.org/protparam/)在线分析蛋白质的氨基酸组成、等电点、相对分子量等理化性质; 利用SignalP3.0 (http://www.cbs.dtu.dk/services/SignalP-3.0/)进行信号肽分析; 通过在线软件 ProtScale (http://cn.expasy.org/ cgi-bin/protscale.pl)进行亲/疏水性分析; 利用TMHMM v2.0在线软件(http://www.cbs.dtu.dk/services/ TMHMM-2.0/)分析蛋白跨膜域。在 NCBI数据库(http://www.ncbi.nlm.nih.gov/)和中国农业科学院棉花研究所棉花基因组数据库(http://cgp.genomics. org.cn/page/cn/index.jsp)中 Blast GbVWR同源基因,通过 DNAMAN7.0进行多重序列比对分析。通过PlantCARE (http://bioinformatics.psb.ugent.be/webtools/ plantcare/html/)分析启动子的顺式作用元件。
1.5 GbVWR基因原核表达载体的构建及在大肠杆菌中的表达
将 GbVWR基因的 ORF片段连接到表达载体pET32a(+)中构建融合表达载体。具体步骤为, 根据所克隆的 GbVWR基因的序列及原核表达载体pET32a(+)多克隆位点序列, 设计原核表达载体引物GbVWR-EF1/VWR-ER1 (表1), 用BamH I和EcoR I双酶切含有 GbVWR基因的质粒和 pET32a(+)质粒,分别回收目的片段, 经T4 DNA连接酶连接后转化大肠杆菌Top10感受态细胞, 经PCR检测后挑取阳性克隆送至上海生工生物科技有限公司测序, 将测序正确的重组表达载体命名为 pET32a-GbVWR。GbVWR蛋白的表达与检测参照 Mo等[18]的方法。为了区分目标蛋白, 在试验设计中设置BL21 (DE3)空菌株和含有空载体质粒 pET32a(+)的 BL21(DE3)菌株 2个对照。取样时间、数量和加入的诱导物浓度均与转化菌株一致。
1.6 GbVWR基因的荧光定量PCR表达分析
参照TOYOBO的荧光定量试剂盒THUNDERBIRD SYBR qPCR Mix的操作说明进行荧光定量PCR分析。根据qRT-PCR引物的设计要求, 采用Primer 5.0软件设计引物, 引物的退火温度为58~62℃, 长度为19~26 bp, GC含量为50%~60%, 扩增长度为90~150 bp。采用20 μL扩增体系, 包括2×SYBR mix 10 μL, cDNA 1 μL ( <100 ng), 上下游引物各1 μL (引物浓度为5 μmol L-1), 灭菌ddH2O 7 μL。基因扩增上游引物为 GbVWR-RT-F、下游引物的 GbVWR-RT-R,以棉花PP2A和UBQ14基因作为内参, 内参基因扩增中所用上下游引物为 PP2A-F、PP2A-R和UBQ14-F、UBQ14-R (表1)。反应条件为95℃ 10 s; 56℃ 15 s; 72℃ 15 s, 40个循环, 然后50℃ 2 min; 65℃ 5 s; 95℃ 5 s。反应在Bio-Rad CFX96 Real-time System荧光定量PCR仪上运行。每个反应设置3个重复, 采用2-ΔΔCt法进行基因相对定量分析[33]。利用GraphPad Prism 6软件进行统计分析并作图。

表1 本试验使用的引物Table 1 Primer sequences used in this study
2 结果与分析
2.1 GbVWR基因的克隆及生物信息学分析
2.1.1 GbVWR全长cDNA和gDNA的克隆与序列分析 分别以海岛棉 Pima90-53根组织的 cDNA和gDNA为模板, 克隆得到的海岛棉GbVWR序列与cDNA全长文库中的301-G12克隆所包含的CDS序列完全一致; GbVWR基因全长520 bp, 包含198 bp ORF (Open Reading Frame), 其中A、T、C、G的含量分别为27.27%、27.27%、28.28%和17.17%, 编码65个氨基酸残基, 预测分子量为7.0 kD。通过在线ProtParam 分析, GbVWR 蛋白的分子式为C614H1032N198O253S56, 相对分子量为7 kD, 理论等电点(pI)为5.32, 该蛋白中氨基酸相对含量较高的有丙氨酸Ala (27.3%, 54个)、半胱氨酸Cys (28.3%, 56个)、甘氨酸Gly (17.2%, 34个)、苏氨酸Thr (27.3%, 54个)。将GbVWR基因序列与中国农业科学院棉花研究所雷蒙德氏棉(D基因组)、亚洲棉(A基因组)以及陆地棉AD基因组比对, 发现GbVWR在A和D基因组分别存在1个拷贝, 在AD基因组存在2个拷贝。
2.1.2 GbVWR基因的生物信息学分析 利用TMpred在线预测蛋白跨膜域, 显示GbVWR基因包含一个跨膜域, 位于第4至第24个氨基酸, 方向为由膜内向膜外(图1)。利用ProtScale在线分析表明,该基因是疏水性不可溶蛋白, 属于脂溶性蛋白, 且有一个信号肽。亚细胞定位预测表明该蛋白可能在细胞外。利用NCBI的CDD数据库分析表明, 该基因不存在已知的蛋白结构域, 没有相关的蛋白功能报道, 没有同源基因和其他相似序列, 说明是一个未知的新基因。
2.1.3 GbVWR基因的上游启动子分析 对位于GbVWR基因起始密码子ATG的5′区上游1500 bp的启动子核苷酸序列, 利用 PlantCARE启动子分析软件预测到多种顺式作用元件(表 2), 包括与光响应、真菌激发子响应、热响应、干旱响应、激素响应、伤口响应、黄酮生物合成基因调节、代谢调节及器官发育等相关元件。
2.2 GbVWR原核表达重组子的鉴定及在大肠杆菌BL21中的表达
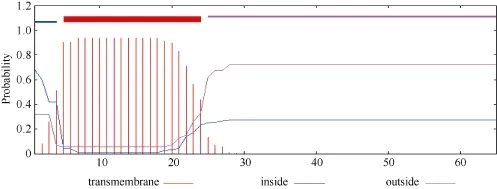
图1 GbVWR蛋白跨膜域与方向预测Fig. 1 Prediction of transmembrane region and orientation for GbVWR
将PCR鉴定和测序后pET32a-GbVWR转化大肠杆菌BL21 (DE3), 经1.0 mmol L-1的IPTG诱导表达后进行 SDS-PAGE分析(图 2)。融合表达蛋白在37℃条件下诱导出1条约25 kD的特异条带, 与预期目标表达蛋白大小基本一致。其中pET32a(+)空载体表达约18 kD的蛋白, GbVWR理论上表达7 kD的蛋白, 二者融合蛋白为25 kD。37℃、200转 min-1的诱导条件下, 目的蛋白9 h即可高效表达, 且24 h后依然保持较高的表达水平(图2)。
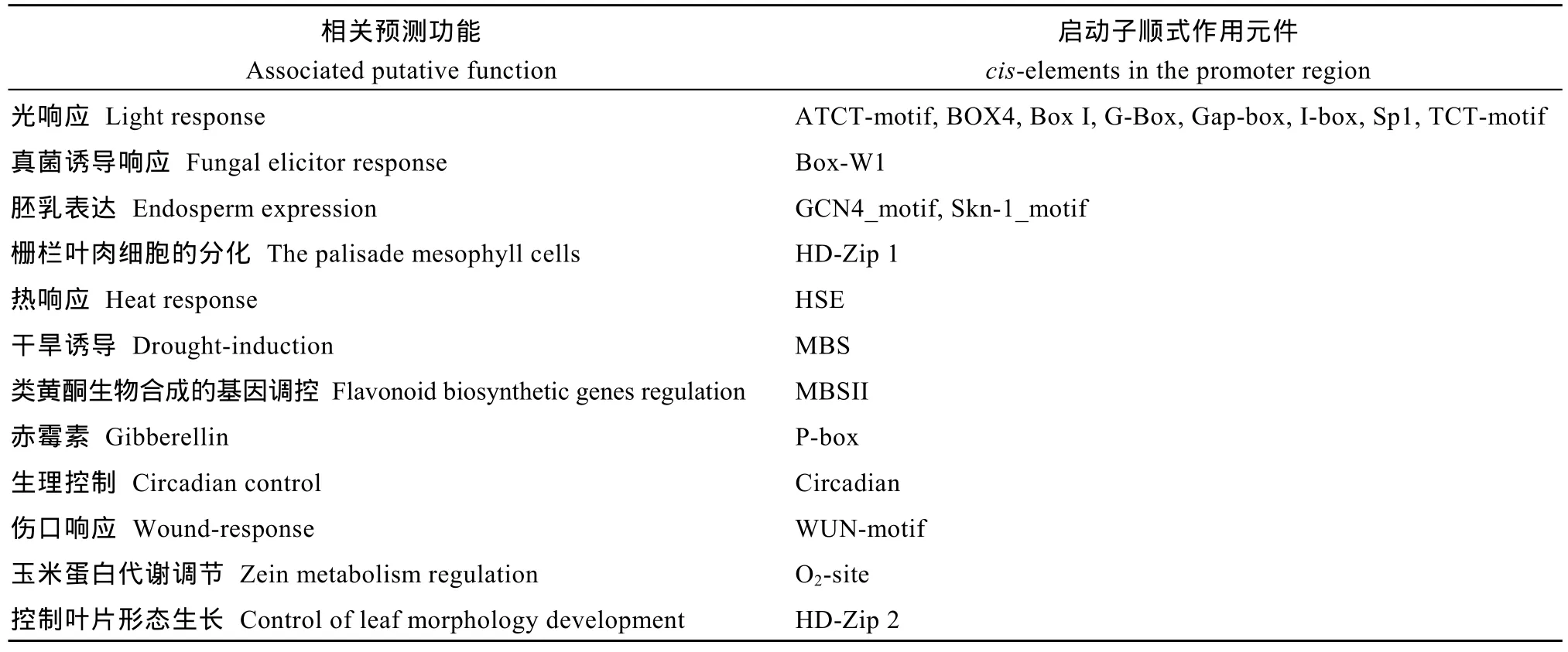
表2 GbVWR基因上游调控区顺式作用元件Table 2 cis-elements in the upstream regulation region of GbVWR gene
2.3 GbVWR基因的组织表达特性分析
以海岛棉品系Pima 90-53根、茎、叶组织的总RNA反转录的 cDNA为模板, 利用 qRT-PCR分析GbVWR基因的组织表达特异性。荧光定量 PCR结果表明 GbVWR基因在海岛棉的根、茎、叶中均有表达, 但表达量存在差异, GbVWR基因在根中的表达量最高, 茎次之, 叶中表达量最低(图3)。
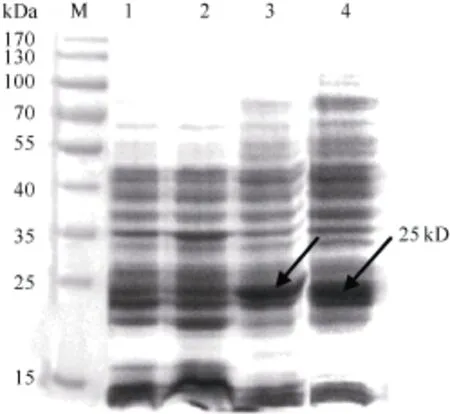
图2 重组蛋白pET32a-GbVWR表达产物SDS-PAGE分析Fig. 2 SDS-PAGE analysis of the expressed product of recombinant pET32a-GbVWR箭头所指为目的蛋白条带; M: 蛋白分子量标准; 1: pET32a(+)空载体37℃下诱导9 h; 2: BL21(DE3)空菌株37℃下诱导9 h; 3~4:pET32a-GbVWR 37℃下分别诱导9 h和24 h。Arrows indicate the target protein bands; M: protein molecular weight standard; 1: strains harbouring pET-32a(+) after induction for 9 hours at 37℃; 2: strains harbouring BL21 (DE3) after induction for 9 hours at 37℃; 3-4: total proteins of engineering bacteria strains after induction for 9 and 24 hours at 37℃, respectively.
2.4 GbVWR基因在黄萎病菌和激素处理下的荧光定量分析
2.4.1 黄萎病菌诱导下GbVWR基因的表达规律
GbVWR基因在黄萎病菌胁迫下的表达模式如图 4所示, 分别以各时间点的水处理样品为对照, GbVWR基因受黄萎病菌快速、强烈诱导表达, 在接菌后2 h的表达量为对照的2倍多; 随着病菌胁迫时间的延长, GbVWR基因的表达量在6 h和12 h较对照显著降低; 而在24 h和36 h, GbVWR基因的表达量又显著高于同一时期的对照。这表明GbVWR基因参与了棉花与黄萎病菌的互作, 且在病原菌侵染棉花的早期阶段发挥作用。
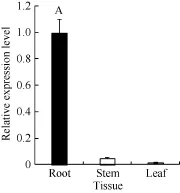
图3 GbVWR在海岛棉品系Pima 90-53不同组织中的RT-PCR分析Fig. 3 Tissue-specific expression analysis pattern of GbVWR by qRT-PCR in the G. barbadense Pima 90-53同一处理时间内, “A”表示在0.01水平上存在极显著差异。“A” means the significant difference at the 0.01 probability level at the same treatment.
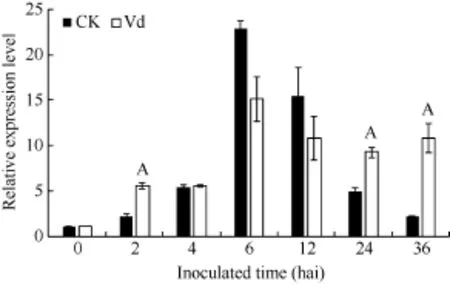
图4 GbVWR在棉花黄萎病菌胁迫下的RT-PCR分析Fig. 4 Expression analysis of GbVWR under Verticillium dahliae stress同一处理时间内, “A”表示在0.01水平上存在极显著差异。“A” means the significant difference at the 0.01 probability level at the same treatment.
2.4.2 外施激素处理下 GbVWR基因的模式表达
GbVWR基因的表达受到水杨酸(SA)明显的诱导。在处理后6 h, GbVWR基因的表达量显著增加, 在24 h出现峰值(处理后的基因表达量约是对照的 20倍),随后表达量降低, 在 48 h基本恢复至处理前的水平。受乙烯处理后, GbVWR基因的表达出现明显被诱导表达的趋势。在处理后的48 h内, GbVWR基因的表达量显著高于对照(12 h除外), 并在6 h出现表达峰值。基于GbVWR基因启动子序列中存在参与赤霉素响应的元件, 为进一步确定 GbVWR基因是否能够响应赤霉素(GA)处理, 进行了该基因在赤霉素处理条件下的基因表达分析。荧光定量结果显示, 用 100 mg L-1的 GA水培处理棉苗6 h后, GbVWR基因表达极显著高于对照, 随着处理时间的推移直至24 h, 基因的表达持续升高, 并在12 h仍显著高于对照; 在36 h基因的表达开始迅速下降,但表达量依然极显著高于对照(图 5)。上述研究结果表明, GbVWR基因受SA、ET和GA激素的诱导表达。

图5 外源激素处理下GbVWR的表达模式分析Fig. 5 Expression analysis of VWR after treatment by hormones同一处理时间内, “A”表示在0.01水平上存在极显著差异,“a”表示在0.05水平上存在显著差异。“A” means the significant difference at the 0.01 probability level at the same treatment, “a” means the significant difference at the 0.05 probability level.
3 讨论
本研究基于课题组已构建的黄萎病菌胁迫海岛棉品系Pima90-53cDNA文库[25], 筛选出一个未知的基因序列(GbVWR), 其与二倍体棉花和四倍体棉花均具很高同源性的区段, 表明 GbVWR序列的确为棉花中存在的基因。NCBI中Blast分析发现, GbVWR基因与NCBI中已经注册的XR_001125651.1为同一基因, 而基因注册者对该序列的注释为 ncRNA, 本研究结果显示GbVWR基因可以在37℃条件下被诱导表达7 kD的蛋白, 表明GbVWR基因是一个有编码产物的新基因, 而非ncRNA。
生物信息学分析显示, GbVWR基因的启动子区域含有响应真菌激发子和赤霉素的调控元件, 同时含有调控黄酮生物合成基因的元件, 暗示 GbVWR基因可能通过响应赤霉素信号及调节植株次生代谢物的合成参与棉花对大丽轮枝菌的抵御过程。基于此, 本研究检测了在黄萎病菌诱导及激素信号处理下 GbVWR基因的表达。黄萎病菌胁迫下, GbVWR基因在2 h被快速诱导, 此时可能是GbVWR基因对黄萎病菌激发子作出的响应; 随着接菌的推迟, 与对照相比该基因的表达量在6 h和12 h降低; 而到24 h和36 h再次升高。推测GbVWR基因在棉花与黄萎病菌互作过程的初期和后期均有重要作用。此外, GbVWR基因的表达具有很强的组织特异性, 主要在根中表达, 这与黄萎病菌在棉花根部侵染的特点也是相吻合的[4]。基于上述研究结果推测GbVWR基因为棉花抗黄萎病反应中一个具有重要功能的新基因。
大量研究表明, 激素信号在植物抗病反应中具有重要的作用, 一些抗病基因通过参与一个或几个激素信号调节通路发挥其重要功能[34-35]。本研究结果显示, GbVWR基因在处理后6 h即可响应SA信号,并且其表达量在24 h达到最高峰, 较对照高出约18倍。ET处理下, GbVWR基因的表达量整体呈上升趋势。外施GA处理下, GbVWR基因同样被显著诱导表达。上述结果证明 GbVWR基因可以参与不同植物激素信号分子调控, 推测 GbVWR基因可能位于复杂信号传导网络的节点位置, 通过响应多种激素信号分子调控网络在棉花抗黄萎病反应中发挥重要作用。在今后的研究中, 我们将通过构建植物超表达载体和基因沉默载体分别转化棉花, 进一步验证该基因的功能及其作用机制。
4 结论
在抗病海岛棉中克隆了参与抗黄萎病反应的新基因GbVWR, 该基因ORF全长198 bp, 编码65个氨基酸, 预测分子量为 7 kD, 理论等电点为 5.32,是一个分泌型蛋白。GbVWR可以表达一个约 7 kD的蛋白。GbVWR启动子含有响应真菌激发子、激素、干旱、及调节黄酮生物合成等相关元件。GbVWR基因在棉花根、茎、叶中均有表达, 但在根中的表达量最高; 此外, 该基因受黄萎病菌、SA、ET和GA诱导表达, 初步推断 GbVWR是海岛棉抵御黄萎病菌过程中的一个新的功能基因, 通过参与多种激素信号途径发挥功能。
[1] Zhang J F, Fang H, Zhou H P, Sanogo S, Ma Z Y. Genetics, breeding, and marker-assisted selection for Verticillium wilt resistance in cotton. Crop Sci, 2014, 54: 1289-1303
[2] Blasingame D, Patel M V. Cotton disease loss estimate committee report. In: Proc, Beltwide Cotton Conf. National Cotton Council, Memphis, TN. 2011. pp 306-308
[3] Zhang J F, Sanogo S, Flynn R, Baral J B, Bajaj S, Hughs S E, Percy R G. Germplasm evaluation and transfer of Verticillium wilt resistance from Pima (Gossypium barbadense) to upland cotton (G. hirsutum). Euphytica, 2012, 187: 147-160
[4] 徐理, 朱龙付, 张献龙. 棉花抗黄萎病机制研究进展. 作物学报, 2012, 38: 1553-1560
Xu L, Zhu L F, Zhang X L. Research on resistance mechanism of cotton to Verticillium wilt. Acta Agron Sin, 2012, 38: 1553-1560 (in Chinese with English abstract)
[5] Sanogo S, Zhang J F. Resistance sources, resistance screening techniques and disease management for Fusarium wilt in cotton. Euphytica, 2016, 207: 255-271
[6] Wilhelm S, Sagen J E, Tietz H. Resistance to Verticillium wilt in cotton: sources, techniques of identification, inheritance trends, and the resistance potential of multilinecultivars. Phytopathology, 1974, 64: 924-931
[7] Bell A A. Verticillium wilt. In: Hillocks R J ed. Cotton Diseases. Wallingford, UK: CAB Intern, 1992. pp 87-126
[8] Bell AA. Verticillium wilt. In: Kirkpatrick T L, Rothrock C S, eds. Compendium of Cotton Diseases. Amer. St. Paul, MN: Phytopathol. Soc, 2001. pp 28-31
[9] Cai Y F, He X H, Mo J C, Sun Q, Yang J P, Liu J G. Molecular research and genetic engineering of resistance to Verticillium wilt in cotton. Afr J Biotechnol, 2009, 8: 7363-7372
[10] Wendel J F, Cronn R C. Polyploidy and the evolutionary history of cotton. Adv Agron, 2003, 78: 139-186
[11] Zhang J F, Sanogo S, Flynn R, Baral J B, Bajaj S, Hughs S E. Germplasm evaluation and transfer of Verticillium wilt resistance from Pima (Gossypium barbadense) to Upland cotton (G. hirsutum). Euphytica, 2012, 187: 147-160
[12] Zhou H P, Fang H, Sanogo S, Hughs S E, Jones D C, Zhang J F. Evaluation of Verticillium wilt resistance in commercial cultivars and advanced breeding lines of cotton. Euphytica, 2014, 196:437-448
[13] Zhang J F, Percy R G, McCarty J J. Introgression genetics and breeding between Upland and Pima cotton: a review. Euphytica, 2014, 198: 1-12
[14] Zhang Y, Wang X F, Yang S, Chi J N, Zhang G Y, Ma Z Y. Cloning and characterization of a Verticillium wilt resistance gene from Gossypium barbadense and functional analysis in Arabidopsis thaliana. Plant Cell Rep, 2011, 30: 2085-2096
[15] Zhang B L, Yang Y W, Chen T Z, Yu W G, Liu T L, Li H J, Fan X H, Ren Y Z, Shen D Y, Liu L, Duo D L, Chang Y H. Island cotton Gbve1 gene encoding a receptor-like protein confers resistance to both defoliating and non-defoliating isolates of Verticillium dahliae. PLoS One, 2012, 7(12): e51091
[16] Gao X Q, Wheeler T, Li Z H, Kenerley C M, He P, Shan L B. Silencing GhNDR1 and GhMKK2 compromises cotton resistance to Verticillium wilt. Plant J, 2011, 66: 293-305
[17] Zhang Y, Wang X F, Li Y Y, Wu L Z, Zhou H M, Zhang G Y, Ma Z Y. Ectopic expression of a novel Ser/Thr protein kinase from cotton (Gossypium barbadense), enhances resistance to Verticillium dahliae infection and oxidative stress in Arabidopsis. Plant Cell Rep, 2013, 32: 1703-1713
[18] Mo H J, Wang X F, Zhang Y, Zhang G Y, Zhang J F, Ma Z Y. Cotton polyamine oxidase is required for spermine and camalexin signalling in the defence response to Verticillium dahliae. Plant J, 2015, 83: 962-975
[19] 杨君, 张艳, 王伟巧, 吴金华, 王国宁, 马峙英, 王省芬. 海岛棉 GbHyPRP1克隆及其转基因拟南芥抗黄萎病验证. 植物资源学报, 2015, 16: 594-602
Yang J, Zhang Y, Wang W Q, Wu J H, Wang G N, Ma Z Y, Wang X F. Cloning of GbHyPRP1 from Gossypium barbadense and validation of Verticillium wilt resistance in transgenic Arabidopsis. J Plant Genet Resour, 2015, 16: 594-602 (in Chinese with English abstract)
[20] Xu L, Zhu L F, Tu L L, Yuan D J, Jin L, Long L, Zhang X L. Lignin metabolism has a central role in the resistance of cotton to the wilt fungus Verticillium dahliae as revealed by RNA-Seq-dependent transcriptional analysis and histochemistry. J Exp Bot, 2011, 62: 5607-5621
[21] Zhang Y, Wang X F, Ding Z G, Ma Q, Zhang G R, Zhang S L, Li Z K, Wu L Q, Zhang G Y, Ma Z Y. Transcriptome profiling of Gossypium barbadense inoculated with Verticillium dahliae provides a resource for cotton improvement. BMC Genom, 2013, 14:637
[22] Wang K, Wang Z, Li F, Ye W, Wang J, Song G, Yue Z, Cong L, Shang H, Zhu S, Zou C, Li Q, Yuan Y, Lu C, Wei H, Gou C, Zheng Z, Yin Y, Zhang X, Liu K, Wang B, Song C, Shi N, Kohel R J, Percy R G, Yu J Z, Zhu Y X, Wang J, Yu S. The draft genome of a diploid cotton Gossypium raimondii. Nat Genet, 2012, 44:1098-1103
[23] Li F, Fan G, Wang K, Sun F, Yuan Y, Song G, Li Q, Ma Z, Lu C, Zou C, Chen W, Liang X, Shang H, Liu W, Shi C, Xiao G, Gou C, Ye W, Xu X, Zhang X, Wei H, Li Z, Zhang G, Wang J, Liu K, Kohel R J, Percy R G, Yu J Z, Zhu Y X, Wang J, Yu S. Genome sequence of the cultivated cotton Gossypium arboreum. Nat Genet, 2014, 46: 567-572
[24] Liu X, Zhao B, Zheng H J, Hu Y, Lu G, Yang C Q, Chen J D, Chen J J, Chen D Y, Zhang L, Zhou Y, Wang L J, Guo W Z, Bai Y L, Ruan J X, Shang-Guan X X, Mao Y B, Shan C M, Jiang J P, Zhu Y Q, Jin L, Kang H, Chen S T, He X L, Wang R, Wang Y Z, Chen J, Wang L J, Yu S T, Wang B Y, Wei J, Song S C, Lu X Y, Gao Z C, Gu W Y, Deng X, Ma D, Wang S, Liang W H, Fang L, Cai C P, Zhu X F, Zhou B L, Chen Z J, Xu S H, Zhang Y G, Wang S Y, Zhang T Z, Zhao G P, Chen X Y. Gossypium barbadense genome sequence provides insight into the evolution of extralong staple fiber and specialized metabolites. Sci Rep, 2015, 5:14139
[25] Li F, Fan G, Lu C, Xiao G, Zou C, Kohel R J, Ma Z, Shang H, Ma X, Wu J, Liang X, Huang G, Percy R G, Liu K, Yang W, Chen W, Du X, Shi C, Yuan Y, Ye W, Liu X, Zhang X, Liu W, Wei H, Wei S, Huang G, Zhang X, Zhu S, Zhang H, Sun F, Wang X, Liang J, Wang J, He Q, Huang L, Wang J, Cui J, Song G, Wang K, Xu X, Yu J Z, Zhu Y, Yu S. Genome sequence of cultivated Upland cotton (Gossypium hirsutum TM-1) provides insights into genome evolution. Nat Biotechnol, 2015, 33: 524-530
[26] Zhang T, Hu Y, Jiang W, Fang L, Guan X, Chen J, Zhang J, Saski C A, Scheffler B E, Stelly D M, Hulse-Kemp A M, Wan Q, Liu B, Liu C, Wang S, Pan M, Wang Y, Wang D, Ye W, Chang L, Zhang W, Song Q, Kirkbride R C, Chen X, Dennis E, Llewellyn D J, Peterson D G, Thaxton P, Jones D C, Wang Q, Xu X, Zhang H, Wu H, Zhou L, Mei G, Chen S, Tian Y, Xiang D, Li X, Ding J, Zuo Q, Tao L, Liu Y, Li J, Lin Y, Hui Y, Cao Z, Cai C, Zhu X, Jiang Z, Zhou B, Guo W, Li R, Chen Z J. Sequencing of allotetraploid cotton (Gossypium hirsutum L. acc. TM-1) provides a resource for fiber improvement. Nat Biotechnol, 2015, 33:531-537
[27] 张纯颖, 王省芬, 张桂寅, 吴立强, 迟吉娜, 李志坤, 马峙英.黄萎病菌诱导下陆地棉抗病品种SSH文库的EST分析. 棉花学报, 2010, 22: 17-22
Zhang C Y, Wang X F, Zhang G Y, Wu L Q, Chi J N, Li Z K, Ma Z Y. EST analysis of suppression subtractive hybridization library from upland cotton resistant cultivar infection by Verticillium dahliae. Cotton Sci, 2010, 22: 17-22 (in Chinese with English abstract)
[28] 王国宁, 赵贵元, 岳晓伟, 李志坤, 张艳, 张桂寅, 吴立强, 王省芬, 马峙英. 河北省棉花黄萎病菌致病性 ISSR遗传分化.棉花学报, 2012, 24: 348-357
Wang G N, Zhao G Y, Yue X W, Li Z K, Zhang Y, Wu L Q, Wang X F, Ma Z Y. Pathogenicity and ISSR genetic differentiation of Verticillium dahliae isolates from cotton growing areas of Hebei province. Cotton Sci, 2012, 24: 348-357 (in Chinese with English abstract)
[29] Zhao P, Zhao Y L, Jin Y, Zhang T, Guo H S. Colonization process of Arabidopsis thaliana roots by a green fluorescent protein-tagged isolate of Verticillium dahliae. Protein Cell, 2014, 5:94-98
[30] 彭姗, 吕学莲, 高峰, 李国英, 李晖. 一种新的棉花黄、枯萎病快速接种方法的研究. 棉花学报, 2008, 20: 174-178
Peng S, Lyu X L, Gao F, Li G Y, Li H. Study on a new rapid inoculation method for Verticillium wilt and Fusarium wilt of cotton. Cotton Sci, 2008, 20: 174-178 (in Chinese with English abstract)
[31] Cheng C X, Jiao C, Singer S D, Gao M, Xu X Z, Zhou Y M, Li Z, Fei Z G, Wang Y J, Wang X P. Gibberellin-induced changes in the transcriptome of grapevine (Vitis labrusca × V. vinifera ) cv. Kyoho flowers. BMC Genom, 2015, 16: 128
[32] 陈士林, 王春虎. 钙和赤霉素对棉花种子发芽力及活力的影响. 中国农学通报, 2004, 20(3): 112-113
Chen S L, Wang C H. Effects of Ca2+and GA on the germination and the seed vigour in cotton. Chin Agric Sci Bull, 2004, 20(3):112-113 (in Chinese with English abstract)
[33] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCtmethod. Methods, 2001, 25: 402-408
[34] Bari R, Jones J D G. Role of plant hormones in plant defence responses. Plant Mol Biol, 2009 69: 473-488
[35] Clarke J D, Volko S M, Ledford H, Ausubel F M, Dong X. Roles of salicylic acid, jasmonic acid, and ethylene in cpr-induced resistance in Arabidopsis. Plant Cell, 2000, 12: 2175-2190
Cloning and Expression Analysis of a Functional Gene GbVWR Induced by Verticillium dahliae in Gossypium barbadense
ZHANG Li-Jia**, ZHANG Yan**, RONG Wei, YANG Jun, ZHANG Gui-Yin, WU Li-Qiang, LI Zhi-Kun, WU Jin-Hua, MA Zhi-Ying, and WANG Xing-Fen*
North China Key Laboratory for Crop Germplasm Resources of Education Ministry / Key Laboratory for Crop Germplasm Resources of Hebei / Agricultural University of Hebei, Baoding 071001, China
Verticillium dahliae is a destructive, soil-borne fungal pathogen that causes severe losses in cotton yield and fiber quality. Mining functional genes related to resistance against V. dahliae will benefit efforts to genetically improve crop plants. In this study, we identified a gene that involved in cotton defense against V. dahliae based on screening the full-length cDNA library and suppression subtractive hybridization library (SSH) induced by V. dahliae in Gossypium barbadense and Gossypium. hirsutum, respectively. Sequence analysis indicated that there was no any annotation in NCBI database, and we named the sequence from G. barbadense as GbVWR. We characterized GbVWR gene and analyzed its expression. The full length cDNA of GbVWR was 520 bp including a 198 bp open reading frame (ORF), encoding 65 amino acid residues. Bioinformatic analyses suggested that GbVWR belonged to secretory protein and tis theoretical isoelectric point was 5.32. Using pET-32a(+) as a fused expression vector, a recombinant plasmid pET32a-GbVWR was constructed. The recombinant protein was induced in Escherichia coli BL21 (DE3) with 1.0 mmol L-1IPTG then GbVWR could express about 7 kD protein in E. coli BL21 (DE3). In addition, diverse cis-acting promoter elements involved in fungal elicitor response, hormone response, wound-response, and flavonoid biosynthetic gene regulation were discovered in the promoter region of GbVWR. qPCR analysis showed that expression level of GbVWR was the highest in roots, and was significantly induced by V. dahliae. Besides, GbVWR could also be induced by SA, ET, and GA treatments, respec-tively. In conclusion, GbVWR is a new functional gene, which involved in multiple signal pathways in cotton defense response to Verticillium wilt.
Gossypium barbadense; Verticillium wilt resistance; GbVWR; Clone; Gene expression
10.3724/SP.J.1006.2016.01779
本研究由河北省自然科学基金项目(C2013204141), 高等学校博士学科点专项科研基金(20131302120002)和河北省百名优秀创新人才支持计划(14226308D)资助。
This study was supported by the Natural Science Foundation of Hebei Province (C2013204141), the Special Research Found for the Doctoral Program of Higher Education (20131302120002), and the Science and Technology Support Project of Hebei Province (14226308D).
*通讯作者(Corresponding author): 王省芬, E-mail: cotton@hebau.edu.cn
联系方式: E-mail: 646560324@qq.com**同等贡献(Contributed equally to this work)
稿日期): 2016-03-14; Accepted(接受日期): 2016-06-20; Published online(
日期): 2016-06-27.
URL: http://www.cnki.net/kcms/detail/11.1809.S.20160627.0839.018.html
