无乳链球菌sodA基因的克隆、序列分析及原核表达载体构建
2016-11-11王松艳蔡双虎鲁义善简纪常吴灶和
王松艳,蔡双虎,鲁义善,简纪常,吴灶和
(1.广东海洋大学水产学院 // 2.广东省水产经济动物病原生物学及流行病学重点实验室暨水产经济动物病害控制广东省普通高等学校重点实验室,广东 湛江 524088)
无乳链球菌sodA基因的克隆、序列分析及原核表达载体构建
王松艳1,2,蔡双虎1,2,鲁义善1,2,简纪常1,2,吴灶和2
(1.广东海洋大学水产学院 // 2.广东省水产经济动物病原生物学及流行病学重点实验室暨水产经济动物病害控制广东省普通高等学校重点实验室,广东 湛江 524088)
无乳链球菌(Streptococcus agalactiae)是引起我国南方罗非鱼链球菌病的主要病原之一,SodA是无乳链球菌重要的毒力因子之一。根据链球菌sodA基因保守区设计引物,采用PCR扩增sodA基因部分序列,反向PCR和巢式PCR扩增其侧翼序列,成功获得无乳链球菌sodA基因。无乳链球菌sodA基因序列全长为699 bp,含开放阅读框609 bp,可编码202个氨基酸,演绎的SodA蛋白主要由α螺旋和不规则卷曲构成。BLAST 分析表明,该蛋白氨基酸序列与犬链球菌(S.canis)及副乳房链球菌(S.parauberis)相应蛋白的氨基酸序列相似性分别高达 80%和 79%。将 sodA基因克隆至原核表达载体 pET28a上成功构建了重组质粒 pET28a-sodA,导入Escherichia coli BL21(DE3),诱导表达的重组蛋白质分子质量约为24 ku,主要以包涵体形式存在于表达菌中。
无乳链球菌;sodA基因;克隆;原核表达
无乳链球菌(Streptococcus agalactiae)是一种革兰阳性菌,属于B族链球菌,该菌可引起新生儿患脑膜炎、肺炎和败血症等疾病,也是近年来我国南方地区罗非鱼(Oreochromis)链球菌病的主要病原菌之一[1-2],给我国罗非鱼产业造成巨大经济损失。
超氧化物歧化酶(Superoxide Dismutase,SOD)主要功能是催化超氧阴离子发生歧化反应生成H2O2和 O2,可消除超氧阴离子自由基对细胞膜的损伤,是生物体抗氧防御系统的第一道防线[3]。研究表明,链球菌含有 sodA基因编码蛋白,该蛋白具有结合锰的超氧化物歧化酶(MnSOD)的特性,可迅速清除吞噬细胞释放的超氧化物,中和超氧化物阴离子自由基O2-和NO自由基[5],达到提升链球菌在宿主吞噬细胞内的存活作用[4-5]。
目前,对sodA基因的研究报道主要集中在与人类或兽医疾病相关链球菌上,而对罗非鱼链球菌病主要病原无乳链球菌中该基因序列、功能等详细信息知之甚少,不利于揭开罗非鱼链球菌病的致病机理。本研究以sodA基因在链球菌属中的保守序列设计引物,以普通PCR、反向PCR和巢式PCR技术克隆无乳链球菌sodA基因,并构建sodA基因原核表达载体,为该基因功能研究提供基础资料。
1 材料与方法
1.1 菌株、质粒及主要试剂
无乳链球菌 ZQ0910,分离自患病罗非鱼(O.niloticus)。原核表达载体 pET28a和大肠杆菌Escherichia coli BL21(DE3)、DH5α保存于广东省水产经济动物病原生物学及流行病学重点实验室。BHI培养基和LB培养基购自广东环凯微生物科技有限公司。质粒pMD18-T、限制性内切酶、PCR相关试剂、MiniBEST细菌基因组提取试剂盒及MiniBEST切胶回收试剂盒购自宝生物工程(大连)有限公司。
1.2 无乳链球菌基因组制备
将无乳链球菌ZQ0910接种于BHI 培养基中活化,用MiniBEST细菌基因组提取试剂盒提取无乳链球菌基因组DNA,于- 20℃冰箱保存,备用。
1.3 sodA基因部分序列扩增
根据Genbank 中链球菌属细菌sodA基因序列保守区设计引物S1:5′ -TGATGCGCTTGAGCCACA TA-3′,S2:5′ -AACAACAAGCCAAGCCCAAC-3′,用PCR扩增sodA基因部分序列。反应体系为25 μL,反应条件为:94℃ 5 min ;94℃ 1 min ,55℃ 1 min,72℃ 1.5 min ,30个循环;72℃ 10 min。用MiniBEST切胶回收试剂盒回收产物后,克隆至pMD18-T中,然后转入E.coli DH5α感受态细胞,采用氨苄青霉素(Amp)与β-半乳糖苷酶实验筛选含重组质粒PMD18T-sodA阳性菌落,在LB液体培养基中扩大培养后送生工生物工程(上海)股份有限公司(下称“上海生工”)测序。
1.4 sodA基因侧翼序列扩增
将无乳链球菌ZQ0910基因组DNA用限制性内切酶HphI进行完全酶切,灭活内切酶活性后用酚-氯仿抽提,再用无水乙醇沉淀,将沉淀物重溶于双蒸水中,T4 DNA连接酶连接后作为反向PCR的模板保存于- 20℃冰箱中备用。根据sodA基因部分序列测序结果,设计扩增sodA基因侧翼序列反向引物P1:5′-CAGCAGCAACAGGACGTTTT-3′ ,P2:5′-GGTTAAGATGTCCACCACCG-3′巢式 PCR引物T1:5′-ACAGGACGTTTTGGTTCAGGT-3′,T2:5′-GAAACATCAGCTAAGAGCGCC-3′。反向PCR反应体系为25 μL,具体条件为:94℃ 5 min;94℃ 1 min,54℃ 1 min ,72℃1.5 min,35个循环;72℃10 min。取1 μL反向PCR产物,稀释50倍后作为随后的巢式PCR反应模板。巢式PCR扩增条件:94℃ 5 min;94℃ 1 min ,55℃ 1 min ,72℃ 1.5 min,共30个循环;72℃ 10 min。回收巢式PCR产物,克隆至pMD18-T后送上海生工测序。
1.5 sodA基因序列的拼接与生物信息学分析
应用DNAStar软件的Seqman模块对sodA基因部分序列和侧翼序列进行拼接。采用ORF Finder( http://www.ncbi.nlm.nih.gov/gorf/gorf.html) 和ProtParam(http://web.expasy.org/protparam/)预测序列开放阅读框(ORF)、演绎氨基酸序列、分子质量计算值和理论等电点等。应用Blast和ClustalX进行同源序列比对。通过在线分析软件SignalP 4.0 Server(http://www.cbs.dtu.dk/services/SignalP)预测信号肽序列。
1.6 sodA原核表达载体构建
根据 sodA基因完整序列设计引物(F1:5′-CATGCCATGGCAATTATTTTACCAG-3′,F2:5′-CCGCTCGAGAGCTTTAGCAGCTTGGT-3′),其中引物F1和F2分别包含NcoI和XhoI酶切位点,PCR扩增sodA基因完整ORF。PCR反应体系为25 μL,反应条件为:94℃ 5 min ;94℃ 1 min,55℃ 1 min,72℃ 1.5 min,30个循环;72℃ 10 min。PCR产物回收后克隆至pMD18-T,用NcoI和XhoI双酶切,经8 mg/mL琼脂糖凝胶电泳,切胶回收。同样将质粒pET28a用NcoI和XhoI双酶切,经琼脂糖凝胶电泳,切胶回收。应用T4 DNA连接酶将已双酶切的质粒pET28a与已双酶切的 sodA基因完整ORF连接起来。连接产物转入E.coli DH5α感受态细胞,采用Amp与β-半乳糖苷酶实验筛选含重组质粒的阳性菌落,然后用菌落PCR进一步筛选。扩大培养含重组质粒pET28a-sodA的E.coli DH5α,提取重组质粒pET28a-sodA,用通用引物M13测序,以检测重组质粒中sodA基因读码框是否正确。
1.7 重组质粒的诱导表达
将重组质粒 pET28a-sodA转入表达菌 E.coli BL21(DE3)后,接种于含有Amp的LB固体平板,37℃下培养 12~16 h,从平板上挑取单菌落于含Amp的LB液体培养基至D(600nm)为0.6~1.0,取2mL接种至50mL含Amp的LB液体培养基中,于37℃摇床培养3 h,至D(600nm)为0.6,加入100mmol/L IPTG储液至IPTG终浓度为1mmol/L,于37℃下诱导培养4 h。诱导培养结束后,取菌液1mL离心,收集沉淀的菌体,在沉淀中加入PBS,用超声破碎仪破碎细菌,取破碎后的菌液全细胞离心后的上清液和沉淀进行SDS-PAGE电泳检测。
2 结 果
2.1 sodA基因克隆
用引物 S1和S2成功扩增出大小约为370 bp片段(图1,泳道1)。测序后Blast分析发现,与其他链球菌sodA相似性高,由此判定该扩增产物为无乳链球菌sodA基因片段。反向PCR扩增出1条清晰的1.6 kb DNA片段(图1,泳道2);以反向PCR产物为模板的巢式PCR扩增出一条约1.34 kb的DNA片段(图1,泳道3)。巢式PCR 产物经测序后与前面测序的sodA基因部分序列拼接,获得699 bp包含 sodA基因完整 ORF的序列,提交GenBank,登录号为KU598928。
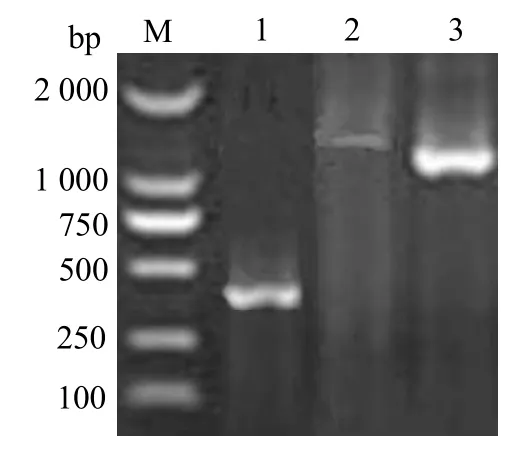
图1 无乳链球菌sodA基因PCR扩增电泳结果Fig.1 Electrophoretic results of cloning sodA gene sequence from S.agalactiae
2.2 sodA基因序列分析
经ORF Finder分析发现,无乳链球菌sodA基因ORF由609 bp组成,编码202个氨基酸(图2),该基因5′ 上游未发现原核生物基因,应具备-35区和-10区序列。用Blast对sodA基因进行同源性分析发现,无乳链球菌 sodA基因与犬链球菌(S.canis)ATCC 43496及副乳房链球菌(S.parauberis)KCTC 11537相关基因相似性分别高达80%和79%。

图2 无乳链球菌sodA基因序列及其演绎的氨基酸序列Fig.2 Complete gene sequence and deduced amino acids of sodA gene from S.agalactiae
分析表明,SodA蛋白分子质量为22.7 ku,等电点为4.73,为酸性蛋白;其N端未见信号肽序列,表明该蛋白可能仅在胞内执行功能。该蛋白有4个活性位点,分别是His27,81,167和Asp163,这些位点可能是该蛋白折叠成的关键氨基酸残基;活性位点中His27,81,167被磷酸化,可能对维持 SodA 蛋白活性至关重要。Swiss-Model预测表明,SodA 蛋白三级结构中含约41.47%的α螺旋、14.94%的β转角、8.38%的延伸串和35.21%的不规则卷曲(图3)。
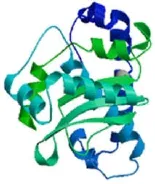
图3 无乳链球菌SodA蛋白三级结构的预测。Fig.3 Predicting results of tertiary structure of SodA from S.agalactiae
2.3 无乳链球菌SodA蛋白同源性分析Blast比对分析表明,无乳链球菌SodA蛋白与其他链球菌有较高相似性;与猪链球菌(S.suis)、野生链球菌(S.ferus)、表兄链球菌(S.sobrinus)及乳房链球菌(S.uberis)相关蛋白相似性分别为88%、87%、87%、85%。对上述各链球菌SodA蛋白进行多重序列同源比对,发现该蛋白N端的氨基酸序列保守性较强,12个氨基酸残基中有11个完全保守;而在C末端氨基酸序列差异较大,12个氨基酸残基中仅7个完全保守(图4)。

图4 无乳链球菌SodA蛋白的氨基酸序列相似性分析Fig.4 Putative amino sequence alignment of SodA among S.agalactiae and other Streptococcus
2.4 重组质粒诱导表达
重组质粒pET28a-sodA经测序后,翻译表明,sodA基因在重组质粒pET28a-sodA中读码框正确,表明它可在质粒pET28a-sodA中表达出正确的肽链及C末端6×His标签。重组质粒pET28a-sodA转入表达菌 E.coli BL21(DE3),经 IPTG诱导后,SDS-PAGE电泳可见,在破碎后菌液全细胞的沉淀中观察到一条分子质量约为24 ku的诱导表达蛋白条带,与重组SodA蛋白的理论分子质量相符(图5,泳道3),而经IPTG诱导包含空载体pET28a和未经诱导的包含重组质粒pET28a-sodA的表达菌的全细胞液中均未发现该蛋白条带(图5),表明重组质粒pET28a-sodA诱导后表达为重组SodA蛋白。
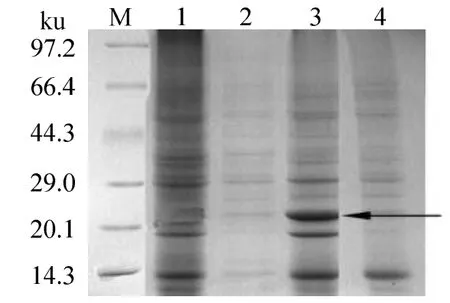
图5 表达的SodA的SDS-PAGE图谱Fig.5 SDS-PAGE analysis of the expressed SodA
3 讨 论
本研究采用反向 PCR和巢式PCR扩增无乳链球菌的sodA基因片段侧翼序列,然后与sodA基因部分序列拼接后获得该基因全长,与程海燕等[6]用反向 PCR获得溶藻弧菌的acfA基因全序列相比,本研究中sodA在链球菌中保守性更高,避免了可能因设计简并性引物或应用Touchdown-PCR而增加的实验失败风险,方法更实用和简单。当然,反向PCR还存在环化反应难以控制、线状串联体成为副产物甚至是主要产物,导致非特异性扩增及酶切片段太长而降低扩增效率等缺陷,使该扩增方法的应用受到一定限制[7]。本研究中,无乳链球菌sodA基因序列5′ 端上游无典型的-35区和-10区序列,表明该基因可能与其他基因在基因组上串联成一个基因簇,在转录时共用一个启动子转录成一个长的mRNA,翻译的多肽经剪切后形成各独立的蛋白。SignalP分析未发现演绎的SodA蛋白在N末端有信号肽序列,表明该蛋白不能通过细胞膜转运至细胞外[8]。
pET载体系列由于其高效的表达和纯化工艺的简便而受到科研人员的青睐,本研究选择该系列的pET28a载体。重组蛋白SodA在37℃时表达效果较好,表达的SodA蛋白仅存在于破碎的表达菌沉淀中,表明该表达蛋白以不溶性的包涵体形式存在。包涵体的形成是由多种因素决定的。或由于重组蛋白在折叠中缺乏某些辅助因子,或由于环境因素的不适:如大量外源表达蛋白的出现对宿主细胞产生毒性,宿主为保护自身细胞而产生一种机制包裹外源蛋白,致使外源蛋白形成包涵体,从而减少外源蛋白对其产生的毒性[9-10]。但是重组蛋白以包涵体形式存在有利于蛋白的分离纯化,不易为蛋白酶水解破坏,进而可获得较高含量的目的蛋白。
细菌的黏附、侵袭和抗吞噬功能在致病过程中起关键作用。B族链球菌的SodA主要作用是催化在吞噬细胞中由呼吸爆发所释放的超氧化离子,从而保护链球菌不被吞噬细胞损失和吞噬[11-13]。本研究克隆和原核表达无乳链球菌 sodA基因,将为研究其保守区域、抗原决定簇组成、金属Mn2+受体结合部位及催化释放超氧化离子的活性部位等,最终为揭示无乳链球菌对罗非鱼的致病机制奠定基础。
[1]王蓓,李桂欢,王培,等.罗非鱼源无乳链球菌溶血素基因体外表达及其免疫原性[J].渔业科学进展,2014,35(6): 60-67.
[2]CHEN M,WANG R,LUO F G,et al.Streptococcus agalactiae isolates of serotypes Ⅰa,ⅠⅠⅠ and V from human and cow are able to infect tilapia [J].Veterinary Microbiology,2015,180(1/2): 129-135.
[3]GAⅠLLOT O,POYART C,BERCHE P,et al.Molecular characterization and expression analysis of the superoxide dismutase gene from Streptococcus agalactiae [J].Gene,1997,204(1/2): 213-218.
[4]BRUNO-BÁRCENA J M,AZCÁRATE-PERⅠL M A,KLAENHAMMER T R,et al.Marker-free chromosomal integration of the manganese superoxide dismutase gene(sodA)from Streptococcus thermophilus into Lactobacillus gasseri [J].FEMS Microbiology Letter,2005,246(1): 91-101.
[5]TANG Y,ZHANG X Y,WU W,et al.Ⅰnactivation of the sodA gene of Streptococcus suis type 2 encoding superoxide dismutase leads to reduced virulence to mice [J].Veterinary Microbiology,2012,158(3/4): 360-366.
[6]程海燕,庞欢瑛,鲁义善,等.溶藻弧菌acfA基因克隆与生物信息学分析[J].广东海洋大学学报,2014,34(1):9-14.
[7]GARCⅠA-SANCHEZ A,CERRATO R,LARRASA J,et al.Characterisation of an extracellular serine protease gene(nasp gene) from Dermatophilus congolensis [J].FEMS Microbiology Letter,2004,231(1): 53-57.
[8]EGNELL P,FLOCK J Ⅰ.The subtilisin Carlsberg pro-region is a membrane anchorage for two fusion proteins produced in Bacillus subtilis [J].Gene,1991,97(1): 49-54.
[9]DEMONTE D,DUNDAS C M,PARK S.Expression and purification of soluble monomeric streptavidin in Escherichia coli[J].Applied Microbiology and Biotechnology,2014,98(14): 6 285-6 295.
[10]YUASA N,KOYAMA T,FUJⅠTA-YAMAGUCHⅠ Y.Purification and refolding of anti-T-antigen single chain antibodies(scFvs) expressed in Escherichia coli as inclusion bodies [J].Bioscience Trends,2014,8(1): 24-31.
[11]YESⅠLKAYA H,KADⅠOGLU A,GⅠNGLES N,et al.Role of manganese-containing superoxide dismutase in oxidative stress and virulence of Streptococcus pneumoniae [J].Ⅰnfection and Ⅰmmunity,2000,68(5): 2819-2826.
[12]POYART C,PELLEGRⅠNⅠ E,GAⅠLLOT O,et al.Contribution of Mn-cofactored superoxide dismutase(SodA) to the virulence of Streptococcus agalactiae [J].Ⅰnfection and Ⅰmmunity,2001,69(8): 5 098-5 106.
[13]ANDRUS J M,BOWEN S W,KLAENHAMMER T R,et al.Molecular characterization and functional analysis of the manganese-containing superoxide dismutase gene(sodA)from Streptococcus thermophilus AO54 [J].Archives of Biochemistry and Biophysics,2003,420(1): 103-113.
(责任编辑:刘庆颖)
Cloning and Bioinformatics Analysis of sodA Gene from Streptococcus agalactiae and Constructing pET28a-sodA Plasmid
WANG Song-yan1,2,CAI Shuang-hu1,2,LU Yi-shan1,2,JIAN Ji-chang1,2,WU Zao-he2
(1.Fisheries College of Guangdong Ocean University // 2.Guangdong Provincial Key Laboratory of Pathogenic Biology and Epidemiology for Aquatic Economic Animals & Key Laboratory of Control for Diseases of Aquatic Economic Animals of Guangdong Higher Education Institutes,Zhanjiang 524088,China)
Streptococcus agalactiae is one of the most important pathogens with a virulence factor SodA causing Streptococcal diseases of cultured Tilapia in South China.Based on the sequences of the highly conserved sequence of the sodA gene among Streptococcus sp,primers for PCR were designed.The partial sequence of the gene was amplified by PCR,and the flanking sequence was amplified by inverse PCR and nested PCR.The complete cDNA sequence of sodA gene was 699 bp with an ORF of 609 bp encoding a 202-peptide.Analysing of the structure SodA protein showed it mainly consisted of α-helix and random coil.Blast analysis indicated the deduced amino acid sequence of SodA from S.agalactiae showing 80% and 79% high similarity with S.canis and S.parauberis respectively.The sodA was cloned into the plasmid pET28a to express in Escherichia coli.The pET28a-sodA were efficiently expressed in host bacterium E.coli BL21(DE3) following induction with IPTG.SDS-PAGE analysis showed the molecular weight of the recombinant protein was 24 ku and the expressed SodA was expressed as inclusion bodies in E.coli.
Streptococcus agalactiae; sodA gene;Cloning;Prokaryotic expression
Q78;Q939.1
A
1673-9159(2016)03-0015-05
10.3969/j.issn.1673-9159.2016.03.003
2016-01-09
国家自然科学基金(31302226)
王松艳(1989-),女,硕士研究生,从事水产动物病害防控研究。E-mail: 1184015027@qq.com
蔡双虎,男,教授。Tel:0759-2383509,E-mail:cshcai@163.com
