藤黄微球菌rpf基因的克隆表达及其结构预测
2016-11-07张秀敏景凤霞王海强赵若玮张艳芬
张秀敏,景凤霞,王海强,赵若玮,张艳芬
(1.河北大学 生命科学学院,河北 保定 071002;2.河北大学 科学技术处,河北 保定 071002)
藤黄微球菌rpf基因的克隆表达及其结构预测
张秀敏1,景凤霞1,王海强1,赵若玮1,张艳芬2
(1.河北大学 生命科学学院,河北 保定071002;2.河北大学 科学技术处,河北 保定071002)
为了了解藤黄微球菌(Micrococcusluteus)复苏促进因子(resuscitation-promoting factor,Rpf)的性质,用自行设计的引物,以藤黄微球菌ACCC41016基因组DNA为模板,扩增rpf基因.随后,构建pET-28a(+)-rpf表达载体,在EscherichiacoliBL21(DE3)中进行IPTG诱导表达,并以克隆测得的rpf基因核酸序列为基础,对Rpf蛋白的氨基酸序列、疏水性、信号肽、磷酸化位点、三级结构以及结构功能域进行生物信息学预测分析.结果表明:该rpf基因与文献报道的藤黄微球菌IAM 14879rpf基因核苷酸序列一致性为69.72%;该rpf基因所编码蛋白的氨基酸序列与文献报道的Rpf蛋白氨基酸序列一致性为63.44%.在E.coliBL21(DE3)中成功表达了Rpf目的蛋白.初步了解了Rpf蛋白的理化性质及生物学功能.解释了Rpf蛋白能够使细菌复苏并促进其生长的理论,为进一步探讨Rpf蛋白的作用机理奠定了基础.
藤黄微球菌;复苏促进因子(Rpf);克隆;表达;结构预测
1998 年,Mukamolova等[1]从藤黄微球菌(Micrococcusluteus)的培养液中发现一种能够促进细菌复苏的物质.他们将培养液的上清液灭菌处理后,加入到处于活的非可培养状态(VBNC)的细菌培养液中,发现这些细菌能够快速复苏和生长,将培养液纯化后发现了复苏促进因子(resuscitation-promoting factor,Rpf),命名为Rpf[2].
Rpf是首次被发现的能促进休眠态的细菌复活同时促进其生长的因子,该因子是一种分泌蛋白,加热情况下或经胰蛋白酶水解后会失去活性.Rpf能够对结核分枝杆菌和处于休眠期的多种革兰氏阳性细菌的复苏和生长起到促进作用[3-7].到目前为止,Rpf的作用机理尚待探索.
本实验通过扩增得到藤黄微球菌rpf基因,并构建表达载体(pET-28a(+)-rpf);利用IPTG诱导目的基因在EscherichiacoliBL21(DE3)中表达,SDS-PAGE检验蛋白表达情况;将克隆得到的rpf基因与丁林贤等[8]报道的rpf基因进行同源性比对,并通过生物信息学技术对2段基因进行翻译,将得到的氨基酸序列进行结构、性质、功能等预测分析比较,得到了有价值的数据信息,为以后的研究奠定了基础.
1 材料和方法
1.1材料
藤黄微球菌(M.luteus)ACCC41016,来自中国农业微生物菌种保藏管理中心(ACCC).
1.2方法
1.2.1藤黄微球菌基因组DNA的提取
采用细菌基因组DNA提取试剂盒(北京康为世纪生物科技有限公司)提取.
1.2.2藤黄微球菌rpf基因的扩增、克隆及测序
根据藤黄微球菌rpf基因序列(Z96935)自行设计5′端引入NdeI和BamHI限制性内切酶酶切位点的引物,上游引物rpfF:5’-CGAcatatgACCATGGACACCATGACTCTCTTCAC-3’,下游引物rpfR:5’-TCAggatccTCAGGCCTGCGGCAGGACGAGCTCCT-3’. 以藤黄微球菌基因组DNA为模板,扩增rpf基因.扩增条件为:94 ℃预变性5 min;94 ℃变性1 min、45 ℃退火1 min、72 ℃延伸1 min,10个循环;94 ℃变性1 min、60 ℃退火1 min、72 ℃延伸1 min,25个循环;72 ℃延伸10 min.PCR产物经质量分数为1.0%的琼脂糖凝胶电泳,切胶回收目的片段,进行测序.经测序确定的rpf基因片段与pMD18-T载体连接,转化E.coliDH5α感受态细胞,然后涂布于LB平板(含质量浓度为100 mg/L的氨苄青霉素),37 ℃培养14~16 h,随机筛选2个白色菌落,分别接种于5 mL含氨苄青霉素的LB培养液中,37 ℃振荡培养过夜,提取质粒DNA,酶切进行验证.经验证正确的阳性克隆命名为pMD18-T-rpf.
1.2.3Rpf蛋白结构及性质预测
1)rpf基因核酸序列基本分析:使用核酸序列分析软件BioEdit(版本号7.0.9.1)对已经过测序获得的rpf基因核酸序列的碱基组成及碱基分布进行基本分析,并翻译得到rpf全长基因的氨基酸序列;2)Rpf蛋白氨基酸序列的基本分析:使用瑞士生物信息学研究所(SIB)的ExPASy服务器 (http://www.expasy.org)上的ProtParam程序对Rpf蛋白的氨基酸数量、氨基酸组成等一些基本性质进行分析;3)Rpf蛋白疏水性的基本分析:使用瑞士生物信息学研究所(SIB)的ExPASy服务器 (http://www.expasy.org)上的ProtScale程序对Rpf蛋白肽链的疏水性进行基本分析;4)Rpf蛋白信号肽的预测分析:使用丹麦科技大学(DTU)CBS服务器(http://www.cbs.dtu.dk)上的SignalP 4.1程序对Rpf蛋白序列中信号肽的存在位点进行预测分析;5)Rpf蛋白亚细胞定位的预测分析:使用PSORT服务器(http://www.psort.org)上的PSORTb v.3.0程序对Rpf蛋白做亚细胞定位分析;6)Rpf蛋白三级结构的预测分析:使用瑞士生物信息学研究所(SIB)的ExPASy服务器(http://www.expasy.org)上的SWISS-MODEL Workspace程序对Rpf蛋白做同源建模分析;7)Rpf蛋白结构功能域的预测分析:使用NCBI上的Blast服务器(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)对Rpf蛋白氨基酸序列的保守区域进行保守区检索,通过结构功能域预测分析Rpf蛋白结构功能组成;8)Rpf蛋白同源性分析对比:从NCBI基因数据库检索丁林贤[8]等人克隆得到的rpf基因(GenBank accession no:Z96935.2)序列,使用软件BioEdit(版本号7.0.9.1)预测得到Rpf蛋白氨基酸序列信息,并利用DNAMAN软件将其与本实验预测得到的Rpf蛋白氨基酸序列进行比对分析.
1.2.4pET-28a(+)-rpf 融合蛋白表达载体的构建及融合蛋白的诱导表达
提取阳性克隆pMD18-T-rpf的质粒,经NdeI和BamHI双酶切后,回收目的基因片段,取目的基因片段和同样用NdeI和BamHI双酶切的pET-28a(+)质粒大片段,用T4连接酶连接,转化E.coliBL21(DE3)感受态细胞,然后涂布于LB平板(含质量浓度为100 mg/L的卡那霉素),37 ℃培养14~16 h,随机筛选2个菌落,分别接种于5 mL含卡那霉素的LB培养液中,37 ℃振荡培养过夜,提取质粒DNA,酶切进行验证.经验证,rpf基因插入正确的阳性克隆,命名为pET-28a(+)-rpf.将pET-28a(+)-rpf阳性克隆子和带有pET-28a(+)空载体的E.coliBL21(DE3)分别接种于10 mL LB液体培养基(含质量浓度为100 mg/L的卡那霉素)中,37 ℃振荡培养至OD600值达到0.6~0.8时,以终浓度为0.8 mmol/L的IPTG分别于20 ℃和37 ℃下诱导过夜,培养后的菌液倒入50 mL离心管中,12 000 r/min离心2 min,用去离子水重新悬浮后再次离心,收集菌体.取适量菌体加入300 μL的去离子水和等体积的2×SDS-PAGE Loading Buffer,用涡旋混合器悬浮,做好标记,煮沸5 min,12 000 r/min离心10 min,取20 μL上清液进行SDS-PAGE检查,电泳条件为200 V恒压.电泳结束后,用质量分数为0.1%的考马斯亮蓝R-250染液(含体积分数分别为45%、45%、10%的甲醇、水、冰醋酸,染色3 h,在脱色液(含体积分数分别为45%、45%、10%的甲醇、水、冰醋酸)中脱色2 h后观察分析结果.
1.2.5Rpf蛋白的纯化
将含有pET-28a(+)-rpf表达载体的E.coliBL21(DE3)20 ℃诱导后,获取重组蛋白培养菌液,10 000 r/min离心10 min,收集菌体,加入15 mL细胞裂解液和100 μL 浓度为100 mmol/L的PMSF(蛋白酶抑制剂),悬浮混匀置于冰浴中.使用超声波400 W功率进行破碎,每次工作5 s,间隔5 s,循环60次,直至菌体溶液变清澈为止.将破碎好的菌体4 ℃,10 000 r/min离心15 min,分离上清液和沉淀,分别取少量用300 μL 超纯水和等体积的2×SDS-PAGE Loading Buffer悬浮,做好标记,煮沸5 min,用作SDS-PAGE电泳的检测样品;将离心得到的大部分沉淀重悬于His标签蛋白纯化试剂盒(包涵体蛋白)(康为世纪,CW0893S)的Binding Buffer中,尽量混匀使包涵体充分溶解,10 000 r/min离心20 min,收集上清液,经Ni-Argarose His标签蛋白纯化试剂盒洗脱纯化并用SDS-PAGE检测.
2 结果
2.1rpf基因的扩增、克隆和测序
应用上述合成的1对引物,以藤黄微球菌基因组为模板,扩增藤黄微球菌rpf基因序列,琼脂糖凝胶电泳结果见图1,PCR扩增产物为800 bp左右的片段.将PCR扩增产物克隆于pMD18-T载体中,转化E.coliDH5α感受态细胞,挑选经酶切鉴定正确的克隆pMD18-T-rpf(图2),送北京三博远志生物技术有限公司进行测序.测序结果表明所得到的rpf目的片段为813 pb.将该基因序列提交到GenBank,获得基因注册号(accession number) 为KU504338.

1.rpf基因;M.Marker(bp).图1 藤黄微球菌rpf基因的PCR扩增电泳Fig.1 PCR amplification electrophoresis figure of the rpf gene of Micrococcus luteus

1.pMD18-T-rpf 载体酶切后的片段;M.Marker(bp).图2 pMD18-T-rpf质粒酶切鉴定图谱Fig.2 Electrophoresis figure of pMD18-T-rpf vector digested
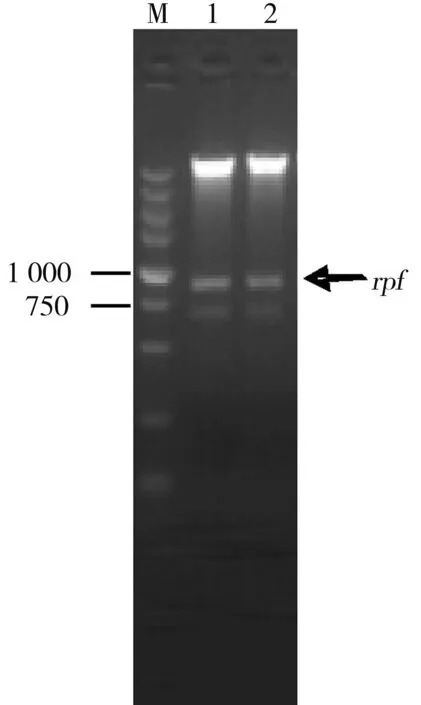
M.Marker(bp);1.1#阳性克隆; 2.2#阳性克隆(bp).图3 pET-28a(+)-rpf融合蛋白表达载体酶切电泳Fig.3 Electrophoresis figure of pET-28 a (+)-rpf fusion protein expression vector digested by restriction enzymes
2.2pET-28a(+)-rpf 融合蛋白表达载体的鉴定
将纯化的重组质粒pMD18-T-rpf双酶切后回收,与用相同酶切的pET-28a(+)质粒连接、转化,随机挑选2个克隆,提取质粒进行酶切验证,结果表明,2个质粒均能酶切得到813 bp的片段(图3),将2#阳性克隆命名为pET-28a(+)-rpf.
2.3Rpf蛋白性质预测
2.3.1rpf基因核酸序列的基本分析结果
本实验克隆得到的藤黄微球菌rpf基因序列长度为813 bp,“G+C”物质的量分数为73.40%,“A+T” 物质的量分数为26.60%.
2.3.2Rpf蛋白氨基酸序列的基本分析结果
本研究克隆得到的藤黄微球菌rpf基因中包括完整的编码区序列807 bp,经过对其氨基酸序列的基本分析预测,翻译得到的藤黄微球菌rpf基因氨基酸序列如图4所示.
2.3.3Rpf蛋白疏水性的基本分析结果
从表1中可以看出,藤黄微球菌Rpf蛋白在18~37个氨基酸具有很强的疏水性,最高达1.922,推测这段序列可能是Rpf蛋白的信号肽序列;而在之后的序列中也陆续出现了几个疏水性较强的氨基酸区域.

图4 Rpf蛋白氨基酸序列Fig.4 Amino acid sequence of Rpf protein

氨基酸区域疏水性18~371.922125~1311.089211~2261.444255~2651.422
2.3.4Rpf蛋白信号肽的预测分析结果
通过对藤黄微球菌Rpf蛋白的预测分析,初步确定前40个氨基酸为信号肽(图5).
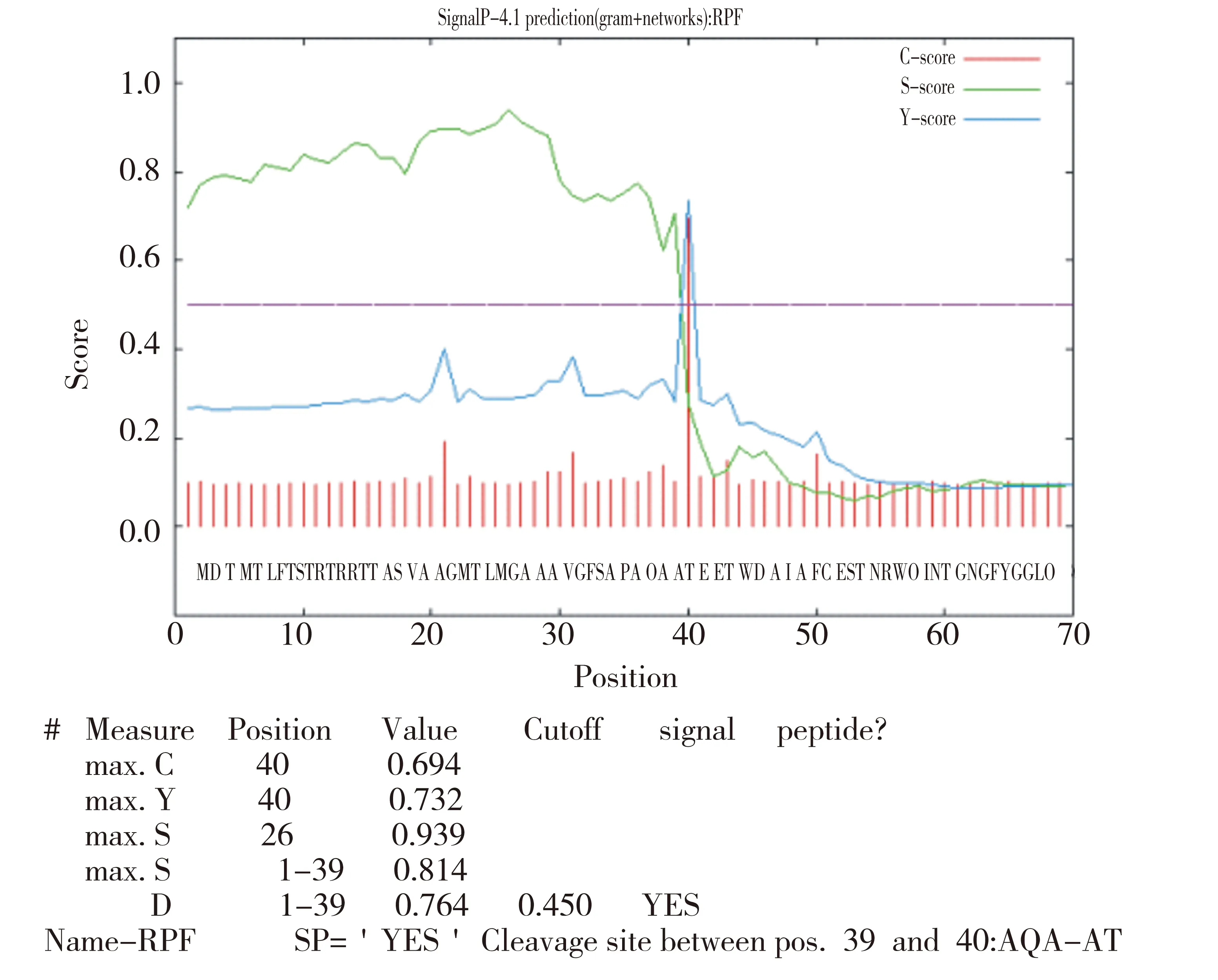
图5 Rpf蛋白信号肽预测分析Fig.5 Signal peptide prediction analysis of Rpf protein
2.3.5Rpf蛋白亚细胞定位的预测分析结果
蛋白质正常行使功能的一个重要因素是蛋白质的亚细胞定位,因此确定藤黄微球菌Rpf蛋白的亚细胞定位对于Rpf蛋白的功能研究十分重要.根据预测分析结果(图6),藤黄微球菌Rpf蛋白在细胞质膜上的定位得分为9.87,在细胞壁上的定位得分为0.12,在细胞外的定位得分为0.01,在细胞质中的定位得分为0,由此可预测藤黄微球菌Rpf蛋白极大可能存在于细胞质膜上.

图6 Rpf蛋白亚细胞定位预测分析Fig.6 Subcellular localization prediction analysis of Rpf protein
2.3.6Rpf蛋白结构功能域的预测分析结果
从图7中可以看出,藤黄微球菌Rpf蛋白由2个结构功能域组成,分别是属于溶菌酶超家族的转糖基酶结构域(aa39~112)和属于LysM结构域家族的LysM结构域(aa220~267).LysM结构域为小球状,有大约40个氨基酸,是一种广泛存在的肽聚糖黏合蛋白功能域,该结构域最初发现在细菌细胞壁降解酶中,由此证明所克隆的目的基因可能编码一种细菌细胞壁降解酶.

图7 Rpf蛋白结构功能域预测分析Fig.7 Structure function domain forecast analysis of Rpf protein
2.3.7Rpf 蛋白的数据库分析对比结果
利用DNAMAN软件将丁林贤等[8]人报道的rpf基因(GenBank accession no.Z96935.2)和本研究克隆得到的藤黄微球菌rpf基因进行序列同源性分析,结果见图8,经分析得知2段序列相似性为69.72%.
利用BioEdit(版本号7.0.9.1)对已报道的rpf基因(GenBank accession no.Z96935.2)进行翻译得到其氨基酸序列,共有223个氨基酸,见图9.将该氨基酸序列与本研究得到的氨基酸序列进行同源性对比,结果见图10,经分析得知氨基酸序列相似性为63.44%.

图8 已报道的rpf基因与本研究克隆得到的rpf基因对比分析结果Fig.8 Comparing results of nucleotide sequences between reported rpf gene and cloning rpf gene in this study

图9 已报道的rpf基因氨基酸序列Fig.9 Reported rpf gene amino acid sequence

图10 已报道的Rpf蛋白与本研究得到的Rpf蛋白氨基酸序列对比分析结果Fig.10 Comparing results of amino acid sequences between reported Rpf protein and Rpf fusion protein in this study
用DNAMAN软件对已报道的rpf基因氨基酸序列和本研究得到的氨基酸序列进行疏水性比对,结果见表2,从表2可知,已报道的Rpf蛋白与本研究得到的Rpf蛋白的氨基酸序列均在18~37个氨基酸,具有很强的疏水性,推测这段序列可能是信号肽序列;而在之后的序列中也陆续出现了几个疏水性较强的氨基酸区域.

表2 已报道的Rpf蛋白与本研究得到的Rpf蛋白氨基酸疏水性统计
利用SWISS-MODEL对已报道的Rpf蛋白氨基酸序列进行同源性建模,得到相似度最高的三维模型(图11),与本研究的Rpf蛋白预测的三维结构相似度极高,两者都具有较多的α-helix结构,这可能与两者的生物学功能相关,具有类似的生物学功能,但两者也存在一定差异,报道的Rpf蛋白三维结构比本研究得到的Rpf蛋白三维结构多出2股α-helix,后者比前者在模型中心区域多出一部分无规则卷曲结构,这与两者序列上的差异是分不开的.

a.文献报道的Rpf 蛋白;b.本研究中的Rpf 蛋白.图11 已报道的rpf基因编码蛋白与本研究Rpf融合蛋白三级结构模型对比Fig.11 Comparison of tertiary structure model between reported Rpf protein and protein encoded by rpf in this study
利用NCBI上的Blast服务器对报道的rpf基因分析得到的氨基酸序列保守区域进行检索(图12),并与本研究得到的蛋白进行结构功能域预测,比对发现,已报道的rpf基因编码的蛋白也由2个结构功能域组成,分别是属于溶菌酶超家族的转糖基酶结构域(aa41~114)和属于LysM结构域家族的LysM结构域(aa173~220),与预测的本实验的Rpf融合蛋白功能结构域完全吻合,只是所在氨基酸区域存在差别,见表3.

图12 报道的rpf基因编码蛋白的结构功能域预测Fig.12 Prediction of structure function domain of reported Rpf protein

功能域文献报道的rpf基因(Z96935.2)编码的蛋白的氨基酸位点本研究中的Rpf蛋白的氨基酸位点溶菌酶超家族转糖基酶结构域41~11439~112LysM超家族LysM结构域173~220220~267
根据比对结果,发现本研究克隆得到的rpf基因与文献中报道的基因序列(登录号Z96935.2)在核酸序列和氨基酸序列同源性上相似性较高,分别为69.72%和63.44%,但仍然存在一定差别,本研究克隆得到的rpf基因在核酸序列和氨基酸序列水平上比文献中报道的基因序列多出一部分序列,这部分序列存在于序列中部;但从比对结果来看,两者在理化性质上不存在较大差异,三维结构也大致相同.
2.4Rpf蛋白的诱导表达及检测
将携带有pET-28a(+)-rpf表达载体的E.coliBL21(DE3)和携带有pET-28a(+)空载体的E.coliBL21(DE3)分别在37 ℃和20 ℃经IPTG诱导后,用SDS-PAGE检测分析,发现经20 ℃诱导的菌体裂解液的沉淀在35 ku处都有1条明显的蛋白条带(图13),同预计大小吻合.
2.5Rpf蛋白的诱导表达及检测
经过Ni-Argarose His标签蛋白纯化试剂盒洗脱纯化并用SDS-PAGE检测后,得到了如图14的结果.从图14中,可以清楚地看出,蛋白洗脱液中含有清晰的目的条带,且杂带数量明显减少,说明经过Ni-Argarose层析柱洗脱后得到了纯度较高的Rpf蛋白,表达的重组Rpf蛋白为包涵体蛋白.

M.Marker(ku);1. 20 ℃下诱导的含有pET-28a(+)空载体的E.coliBL21(DE3)菌体;2. 37 ℃下诱导的含有pET-28a(+)空载体的E.coliBL21(DE3)菌体; 3. 20 ℃下诱导的含有pET-28a(+)-rpf表达载体的E.coliBL21(DE3)菌体; 4. 37 ℃下诱导的含有pET-28a(+)-rpf表达载体的E.coliBL21(DE3)菌体.
图13Rpf蛋白诱导表达的SDS-PAGE
Fig.13SDS-PAGE of Rpf protein induced expression

M.Marker(ku);1. 菌体;2. 细胞裂解液的上清液;3. 细胞裂解液沉淀;4. 流穿液;5、6. 蛋白洗脱液.图14 纯化的Rpf蛋白SDS-PAGEFig.14 SDS-PAGE of purified Rpf protein
3 讨论
Rpf蛋白作用机理虽尚不明确,但Rpf蛋白具有细菌细胞壁溶解活性已被证实[9-10].从比对的蛋白质结构功能域中发现,虽然两者的2种功能结构域处于各自序列的不同位点,但它们所属的结构域家族相同,都拥有溶菌酶超家族和LysM 超家族.溶菌酶超家族具有溶菌酶活性;LysM超家族LysM结构域最早是在芽孢杆菌噬菌体溶菌酶中被发现的,该酶通过降解N-乙酰胞壁酸和N-乙酰葡萄糖胺之间的糖苷键来降解细胞壁[11],这表明2种蛋白质的理论生物学功能相似,均具有细胞壁溶解活性,这与已报道内容相同,证实了本实验中克隆的rpf基因的正确性.
亚细胞定位结果显示Rpf蛋白存在于细胞质膜上,因此它可以在细胞处于休眠状态,细胞壁增厚时不断溶解细胞壁,当延长培养时间后,Rpf蛋白将细胞壁完全溶解,释放到培养液中.这一分析也解释了Rpf蛋白能够使细菌复苏并促进其生长的理论.
[1]MUKAMOLOVA G V,KAPRELYANTS A S,YOUNG D I,et al.A bacterial cytokine[J].Proceedings of the National Academy of Sciences,1998,95(15):8916-8921.
[2]丛延广,胡福泉.细菌复苏促进因子Rpf[J].生命的化学,2005,25(5):482-484.
CONG Y G,HU F Q.Resuscitation-promoting factors(RPF)of bacteria[J].Chemistry of Life,2005,25(5):482-484.
[3]XU H S,ROBERTS N,SINGLETON F L,et al.Survival and viability of nonculturableEscherichiacoliandVibriocholeraein the estuarine and marine environment[J].Microbial Ecology,1982,8(4):313-323. DOI:10.1007/BF02010671.
[4]RAVAGNANI A,FINAN C L,YOUNG M.A novel firmicute protein family related to the actinobacterial resuscitation-promoting factors by non-orthologous domain displacement[J].BMC Genomics,2005,6:39.DOI:10.1186/1471-2164-6-39.
[5]DEWI P I,UEHARA M,KATAYAMA,et al.Resuscitation promoting factor(Rpf)from Tomitella biformata AHU 1821(T)promotes growth and resuscitates non-dividing cells[J].Microbes and Environments,2013,28(1):58-64.DOI:10.1264/jsme2.ME12122.
[6]HUANG W,QI Y,DIAO Y,et al.Use of resuscitation-promoting factor proteins improves the sensitivity of culture-based tuberculosis testing in special samples[J].Am J Respir Crit Care Med,2014,189(5):612-614. DOI:10.1164/rccm.201310-1899LE.
[7]魏忠彬,李影,钱爱东,等.VBNC 乳酸杆菌的复苏[J].中国饲料,2006(18):27-28.
WEI Z B,LI Y,QIAN A D,et al. Recovery of VBNC Lactobacill[J].China’s Feed,2006(18):27-28.
[8]丁林贤,张萍华,洪华嫦,等.藤黄微球菌Rpf活性蛋白的制取及其对红球菌VBNC菌体的复苏作用[J]. 微生物学报,2012,52(1):77-89.
DING L X,ZHANG P H,HONG H C,et al.Cloning and expression ofMicrococcusluteusIAM 14879 Rpf and its role in the recovery of the VBNC state inRhodococcussp.DS471[J].Acta Microbiologica Sinica,2012,52(1):77-89.
[9]COHEN G M,BARTHE P,BAGNERIS C,et al.The structure of a resuscitation-promoting factor domain fromMycobacteriumtuberculosisshows homology to lysozymes[J].Nature Structural and Molecular Biology,2005,12(3):270-273.DOI:10.1038/nsmb905.
[10]李伦,李元,白婧,等.细菌复苏促进因子[J].动物医学进展,2010,31(4):103-106.
LI L,LI Y,BAI J,et al.Progress on resuscitation-promoting factor[J].Progress in Veterinary Medicine,2010,31(4):103-106.
[11]GARVEY K J,SAEDI M S,ITO J.Nucleotide sequence ofBacillusphage phi 29 genes 14 and 15:homology of gene 15 with other phage lysozymes[J].Nucleic Acid Res,1986,14:10001-10008.
(责任编辑:赵藏赏)
Cloning and expression of Micrococcus luteus rpf gene and structure prediction of Rpf protein
ZHANG Xiumin1,JING Fengxia1,WANG Haiqiang1,ZHAO Ruowei1,ZHANG Yanfen2
(1.College of Life Scineces,Hebei University,Baoding 071002,China; 2.Office of Science and Technology,Hebei University,Baoding 071002,China)
In order to study the properties of resuscitation-promoting factor (Rpf) ofMicrococcusluteus,rpfgene (813 pb)was amplified by using genome DNA ofM.luteusACCC41016 as templates.pET-28a (+)-rpfexpression vector was constructed and Rpf protein was induced using IPTG to express inEscherichiacoliBL21(DE3).Based onrpfnucleic acid sequence,the bioinformation of Rpf protein,such as protein amino acid sequence,hydrophobic,signal peptide,phosphorylation sites,tertiary structure,structure and functional domains,were predicted by using bioinformatics method.The results showed that the identity of nucleotide sequences between the cloningrpfgene and the reportorialrpfgene was 69.72%.And the identity of amino acids sequences between the protein encoded by the cloningrpfgene and the reportorial Rpf protein was 63.44%.SDS-PAGE results confirmed that the expression ofrpfgene in theE.coliBL21 (DE3)was successful.According to the bioinformatics and homology analysis of Rpf protein,the physical and chemical properties and biological function of Rpf protein were preliminary elucidated.The study explained the theory of the Rpf protein can make the bacteria recovery and promote its growth.This laid the groundwork for further mechanistic studies of Rpf protein.
MicrococcusLuteus;resuscitation-promoting factor(Rpf);clone;express; structure prediction
10.3969/j.issn.1000-1565.2016.04.012
2016-04-13
国家自然科学基金资助项目(31270053);河北省自然科学基金资助项目(C2014201141);河北省生物工程重点学科建设经费资助项目(1050-5030023);河北省高等学校科学技术研究资助项目(Z2015013)
张秀敏(1970—),女,河北涞水人,河北大学教授,博士,主要从事微生物系统性及多样性研究.
E-mail:zhxiumin1106@126.com
张艳芬(1979—),女,河北邯郸人,河北大学讲师,主要从事分子生物学研究.
E-mail:zhangjing@hbu.edu.cn
Q939.13;
A
1000-1565(2016)04-0402-10
