MITE类转座子mPing在水稻不同亚种间的差异分析
2016-11-01张宁1阮亚男1王姗姗1刘洋1赵宸1王晶晶1王凯玺3王艳丽1王红艳1
张宁1*,阮亚男1*,王姗姗1,2,刘洋1,赵宸1,王晶晶1,王凯玺3,王艳丽1,王红艳1
MITE类转座子m在水稻不同亚种间的差异分析
张宁1*,阮亚男1*,王姗姗1,2,刘洋1,赵宸1,王晶晶1,王凯玺3,王艳丽1,王红艳1
1 辽宁大学生命科学院植物表观遗传与进化实验室,辽宁沈阳110036 2 西藏自治区农牧科学院农业研究所品种资源研究室,西藏拉萨850000 3 辽宁省水土保持研究所,辽宁朝阳 122000
m是水稻中第一个被鉴定出的有活性的MITE类转座子,为了探索m在水稻粳稻品种日本晴和籼稻品种93-11基因组中的分布差异,本研究首先运用Southern杂交的方法初步检测m在两个亚种中拷贝数的差异,然后通过同源性探寻方法发现,m在水稻亚种日本晴和93-11基因组中拷贝数分别为52和14,并且日本晴基因组中的m均为m-1,93-11中m-1的拷贝数为3,m-2的拷贝数为11。通过分析m上下游5 kb侧翼序列发现m在日本晴和93-11中分别与23和3个已知基因相关联。本研究为阐明以m的分布多样性为主要原因的粳稻和籼稻之间的遗传差异提供初步理论基础。
粳稻,籼稻,转座子,m,水稻
转座子(Transpososable elements或transposons)是指基因组中可移动或复制自身DNA并整合到新位点的DNA片段[1]。转座子在基因组中常常具有很多拷贝,是真核生物基因组重要的组成部分[2]。人类基因组中转座子约占整个基因组的45%,植物中约为50%−90%[3]。由于转座子激活可造成基因的失活或缺失、基因组重排、甚至是着丝粒区序列的进化和分 化[4-7],因此对于研究基因组结构、侧翼基因表达调控以及物种进化等都起到重要作用[3]。
根据转座方式可将转座子分为两类:Class I称为反转录转座子(Retrotransposon),是利用RNA介导的转座[8]。根据其是否包含有长末端重复序列(LTR),分为LTR类反转座子(LTR retrotransposon) 和非LTR类反转座子(Non-LTR retrotransposon),由于其利用复制/插入方式实现转座,因此可改变基因组的大小[3]。Class II称为转座子(Transposons),是利用DNA介导转座。根据其转座的自主性,可分为自主性转座子(Autonomous element)、非自主性转座子(Non-autonomous element) 及微型反向重复转座子(Miniature inverted-repeat transposable element, MITE),由于其利用切除/修复方式实现转座,因此基因组大小一般不会发生改变[9-10]。
MITE类转座子是一类在植物中分布广泛的非自主性转座子,自身不具编码转座酶的基因,序列只有100−500bp[11],常常位于基因组的常染色质区[12-13]。水稻中第一个被鉴定的有活性的MITE类转座子是m(GenBank AccessionNo. BK000588),序列长430 bp[14],m的结构中包括15 bp的TIRs (Terminal inverted repeats),但不带有ORF (Open reading frame),它的转座依靠自主类转座子或的ORF2编码的转座酶进行转座[15-17]。m在水稻中的突变体有两种类型:m-1和m-2,其中m-1序列长430 bp,m-2序列长419 bp[18]。
水稻作为世界一半人口的食物来源,对人类的生存与发展有着重要的意义,在其9 000多年的驯化历史中[18-19],有20多个稻属物种,其主要分化为籼稻和粳稻两种生态型[20]。籼稻和粳稻有着形态、生理、遗传等方面的差异。比如籼稻植株一般高于粳稻植株,抗倒伏能力较强,发芽较快,分蘖数较强,但籼稻比粳稻有更高的净光合速率[21];蔡星星 等[20]利用籼稻93-11和粳稻日本晴基因组序列的差异片段进行研究,结果显示这两种生态型全基因序列具有很大的差异性。m的拷贝数差异就是其中的一个原因,为了更加完整地探寻m在水稻亚种分化以及基因组形成中的可能作用,本研究主要运用Southern杂交分析并结合生物信息学的方法,利用日本晴和93-11全基因组信息,对m在水稻两个亚种基因组中的结构特点、分布特征、以及m的插入对侧翼基因的影响进行研究,探寻m在水稻亚种形成过程中的可能作用。本研究为阐明粳稻和籼稻之间的遗传差异、探索粳稻和籼稻的进化历史提供初步理论基础。
1 材料与方法
1.1 材料来源
本研究采用的实验材料为粳稻品种日本晴(cv.) 和籼稻品种93-11 (cv.)。
1.2 Southern杂交
利用dIII (购自New England Biolabs公司) 分别对日本晴和93-11的DNA样品进行酶切,选取这个酶的目的在于m中不存在d III的酶切位点,因此我们可以估测m在这两个水稻亚种中的拷贝数。根据Shan等[5]提供的方法进行Southern杂交分析,以m-1 (AB087615) 全长设计探针引物(由上海生工生物技术有限公司合成),具体序列为m-1 (positions 6–430):forward,5’-GTCACAATGGG GGTTTCACT-3’,reverse,5’-GGCCAGTCACA ATGGCTAGT-3’。
1.3 生物信息学及比较基因组学
m-1 (AB087615) 的基因序列在NCBI网站(http://archive-dtd.ncbi.nlm.nih.gov/) 中下载,根据m-1的序列,可得到m-2的序列[18]。根据GRAMENE (http://www.gram ene.org) 网站,做Blast分析获得m在日本晴和93-11中的拷贝信息,根据RICE-MAP (http://www.ricemap.org/) 数据库网站,可得到该m拷贝所在染色体的上下游5 kb的碱基序列信息,再根据KOME数据库(http://cdna01. dna.affrc.go.jp/cDNA/),E值设为0,即只有100%同源序列才被挑选出来,以此方法进行同源性探寻可得到侧翼序列的外显子信息及表达蛋白的功能信息。
2 结果与分析
2.1 日本晴和93-11的Southern杂交分析
通过dIII酶切的Southern杂交分析可保守估测m在日本晴和93-11中的拷贝数。由结果可知(图1),m在日本晴中和93-11中均存在,且m在两亚种中的数量是不同的,在日本晴中的拷贝数明显多于93-11,说明m在水稻亚种的分布有明显的差异,暗示其在物种分化中可能存在某些作用。

图1 mPing在日本晴和93-11中的Southern杂交分析
2.2 m不同拷贝在籼稻品种93-11和粳稻品种日本晴基因组中的定位情况
为了进一步确定m在两个亚种中的差别信息,通过同源性探寻方法,对m进行定位分析。结果表明,m在水稻亚种中分布数量和位置均不相同(表1,表2),m在93-11中的拷贝数是14 (图2),在日本晴中的拷贝数是52 (图3)。其中93-11中m-1的拷贝数是3,m-2的拷贝数是11,日本晴中的m均为m-1,m在日本晴中的拷贝数是93-11的3.7倍。此外,日本晴每条染色体均有m的分布,拷贝数最多的是10号染色体,最少的是1号染色体,93-11中7、8、10号染色体没有m的拷贝,1号染色体上的拷贝数最多,为3个拷贝。m在日本晴和93-11基因组中结构特点和分布特征的多样性,进一步说明它们是导致水稻不同亚种间基因组差异的原因。
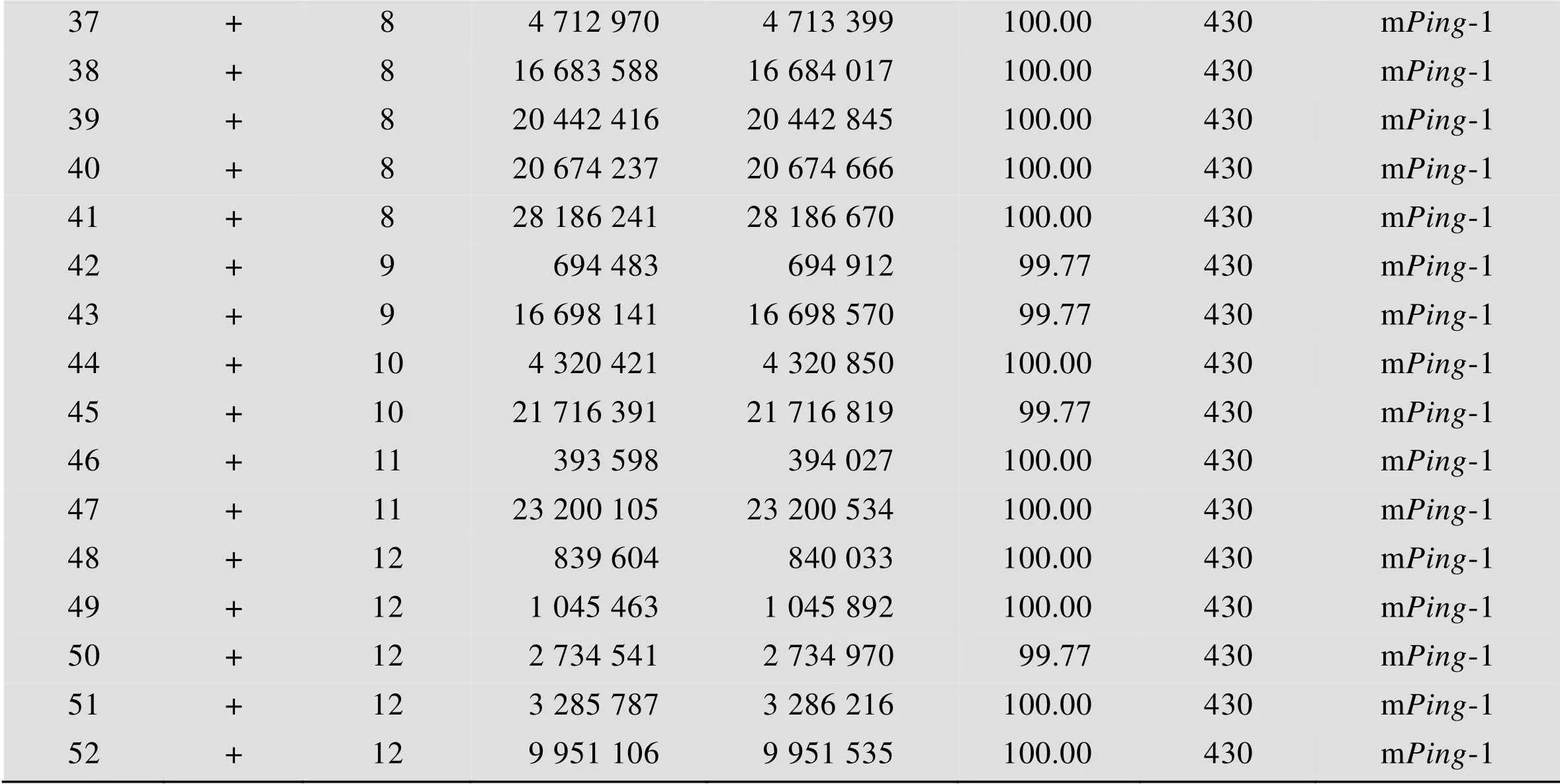
表1 mPing在日本晴基因组中的定位信息

表2 mPing在93-11基因组中的定位信息

图2 mPing在93-11基因组染色体上的定位

图3 mPing在日本晴基因组染色体上的定位
2.3 日本晴与93-11的m插入位点侧翼序列分析
转座子的转座会影响其侧翼基因的表达,在基因的结构和进化过程中具有重要的作用[8]。因此对m所有拷贝的侧翼序列上下游5 kb进行功能基因的同源性探寻。研究发现,m插入到了日本晴基因组中的某些基因的上游、下游以及内部,93-11基因组中的某些基因的上游和下游。日本晴中有23个m侧翼序列与已知功能基因有100%的同源性,这些已知基因中有与拟南芥相关的转座酶蛋白基因、与抗性相关的蛋白基因等(表3)。同时,m插入到了1号染色体中的AK073838,11号染色体中的AK109523,12号染色体中的AK243131基因的内部。93-11中有3个m侧翼序列与已知的功能基因有100%的同源性,且m均插入到了基因的上游区域(表4)。

表3 日本晴中mPing的侧翼序列分析

表4 93-11中mPing的侧翼序列分析
3 讨论
转座子的移动会影响宿主基因组的结构、功能和进化[2-5,22-26]。在本研究中,我们以MITE类转座子为研究对象,探讨了以其多样性为主要原因的水稻亚种间的基因组差异。结果表明,在日本晴和93-11基因组中,m的分布和类型均有较大差异。日本晴基因组中只存在m-1一种类型,共有52个拷贝;93-11基因组中有m14个,包括3个m-1和11个m-2。这与Hu等的研究结果,粳稻品种中m-1所占比例高于m-2,而籼稻品种m2所占比例高于m-1一致[18]。m在水稻亚种基因组中分布比例和位置的差异,暗示m在水稻驯化历史中有着不可忽视的作用;同时,m有望作为一种分子标签来快速鉴别水稻亚种。另一方面,m往往会插入到基因中或者基因附近[14],为了研究m与侧翼基因的关系,本研究对m所在位置的上下游延伸5 kb序列进行功能基因的同源性探寻,发现日本晴中有24个序列与已知基因序列100%同源,93-11中有3个序列与已知基因100%同源,并且日本晴中有3个基因内部插入了m,这3个基因与抗性基因、漆酶基因和细胞分裂基因有关,暗示着m在调控这3类基因表达的过程中存在一定的潜在影响。同时,日本晴和93-11中除了1个已知基因相同,剩余基因不相同,说明这些原因可能是导致日本晴和93-11基因组差异性的原因。然而m的插入机制、它在宿主基因组中对侧翼基因表达的影响、及其在水稻进化历史中的作用,有待进一步探索和研究。
REFERENCES
[1] McClintock B. Chromosome organization and genic expression. Cold Spring Harb Symp Quant Biol, 1951, 16: 13–47.
[2] Roffler S, Wicker T. Genome-wide comparison of Asian and African rice reveals high recent activity of DNA transposons. Mob DNA, 2015, 6: 8.
[3] Feschotte C, Jiang N, Wessler SR. Plant transposable elements: where genetics meets genomics. Nat Rev Genet, 2002, 3(5): 329–341.
[4] McClintock B. The significance of responses of the genome to challenge. Science, 1984, 226(4676): 792–801.
[5] Shan XH, Liu ZL, Dong ZY, et al. Mobilization of the active MITE transposons mandin rice by introgression from wild rice (Griseb.). Mol Biol Evol, 2005, 22(4): 976–990.
[6] Michalak P. Epigenetic, transposon and small RNA determinants of hybrid dysfunctions. Heredity, 2009, 102(1): 45–50.
[7] Gao DY, Jiang N, Wing RA, et al. Transposons play an important role in the evolution and diversification of centromeres among closely related species. Front Plant Sci, 2015, 6: 216.
[8] Lisch D. How important are transposons for plant evolution?. Nat Rev Genet, 2013, 14(1): 49–61.
[9] Shan XH, Lin XY, Long LK, et al.mobilization of mand its putative autonomous elementin rice by hydrostatic pressurization. J Exp Bot, 2006, 57(10): 2313–2323.
[10] WangHY, Chai Y, Chu XC, et al. Molecular characterization of a rice mutator-phenotype derived from an incompatible cross-pollination reveals transgenerational mobilization of multiple transposable elements and extensive epigenetic instability. BMC Plant Biol, 2009, 9: 63.
[11] Jiang N, Feschotte C, Zhang XY, et al. Using rice to understand the origin and amplification of miniature inverted repeat transposable elements (MITEs). Curr Opin Plant Biol, 2004, 7(2): 115–119.
[12] Zhang Q, Arbuckle J, Wessler SR. Recent, extensive, and preferential insertion of members of the miniature inverted-repeat transposable element familyinto genic regions of maize. Proc Natl Acad Sci USA, 2000, 97(3): 1160–1165.
[13] Feng Q, Zhang YJ, Hao P, et al. Sequence and analysis of rice chromosome 4. Nature, 2002, 420(6913): 316–320.
[14] Yasuda K, Tsukiyama T, Karki S, et al. Mobilization of the active transposon min interspecific hybrid rice betweenand. Euphytica, 2013, 192(1): 17–24.
[15] Jiang N, Bao ZR, Zhang XY, et al. An active DNA transposon family in rice. Nature, 2003, 421(6919): 163–167.
[16] Kikuchi K, Terauchi K, Wada M, et al. The plant MITE mis mobilized in anther culture. Nature, 2003, 421(6919): 167–170.
[17] Yang GJ, Zhang F, Hancock CN, et al. Transposition of the rice miniature inverted repeat transposable element min. Proc Natl Acad Sci USA, 2007, 104(26): 10962–10967.
[18] Hu H, Mu J, Zhang HJ, et al. Differentiation of a miniature inverted transposable element (MITE) system in Asian rice cultivars and its inference for a diphyletic origin of two subspecies of Asian cultivated rice. J Integr Plant Biol, 2006, 48(3): 260−267.
[19] Khush GS. Origin, dispersal, cultivation and variation of rice. Plant Mol Biol, 1997, 35(1/2): 25–34.
[20] Cai XX, Liu J, Chou YQ, et al. Differentiation ofrice revealed by insertion/deletion fragments obtained from comparative genomic study of DNA sequences between 93-11 () and(). J Fudan Univ: Nat Sci, 2006, 45(3): 309–315 (in Chinese). 蔡星星, 刘晶, 仇吟秋, 等. 籼稻93-11和粳稻日本晴DNA插入缺失差异片段揭示的水稻籼-粳分化. 复旦学报: 自然科学版, 2006, 45(3): 309–315.
[21] Zhang Z, Mao T, Li X, et al. Research on difference and classification of Indica-japonica subspecies at Asian cultivated rice. North Rice, 2013, 43(2): 66–69 (in Chinese). 张战, 毛艇, 李鑫, 等. 亚洲栽培稻籼粳亚种差异及主要分类方法研究进展. 北方水稻, 2013, 43(2): 66–69.
[22] Marillonnet S, Wessler SR. Retrotransposon insertion into the maizegene results in tissure-specific RNA processing. Plant Cell, 1997, 9(6): 967–978.
[23] Wang HY, Tian Q, Ma YQ, et al. Transpositional reactivation of two LTR retrotransposons in rice-Zizania recombinant inbred lines (RILs). Hereditas, 2010, 147: 264–277.
[24] asacuberta E, Gonzalez J. The impact of transposable elements in environmental adaptation. Mol Ecol, 2013, 22: 1503–1517.
[25] Bennetzen JL, Wang H. The contributions of transposable elements to the structure, function, and evolution of plant genomes. Annu Rev Plant Biol, 2014, 65: 505–530.
[26] Lisch D, Bennetzen JL. Transposable element origins of epigenetic gene regulation. Curr Opin Plant Biol, 2011, 14: 156–161.
(本文责编郝丽芳)
Comparison of MITE transposons min different rice subspecies
Ning Zhang1*, Yanan Ruan1*, Shanshan Wang1,2, Yang Liu1, Chen Zhao1, Jingjing Wang1, Kaixi Wang3, Yanli Wang1, and Hongyan Wang1
1,,110036,,2,850000,,Liaoning Institute of Soil and Water ConservationChaoyangLiaoningChina
The mfamily is the first active MITE TE family identified in rice genome. In order to compare the compositions and distributions of mfamily in the genomes of two rice subspecies(cv. Nipponbare) and(cv. 93-11), we initially estimated the copy numbers of mfamily in those two subspecies using Southern blot and then confirmed the results by searching homologous copies in each reference genome using Blastn program, which turned out to have 52 and 14 mcopies in corresponding reference genome, respectively. All mmembers in Nipponbare genome belong to m-1, while there are 3 m-1 and 11 m-2 copies in 93-11 genome. By further investigating the 5-kb flanking sequences of those mcopies, it was found that 23 and 3 protein-coding genes in Nipponbare and 93-11 genome are residing adjacent to those mcopies respectively. These results establish the preliminary theoretical foundation for further dissecting the genetic differences of japonica and indica rice in terms of the diversities and distributions of their component m.
,, transposon, m, rice
December 25, 2015; Accepted:April 12, 2016
Hongyan Wang. Tel: +86-24-62202232; E-mail: hongyan2003@126.com *These authors contributed equally to this work.
Supported by:Natural Science Foundation of China (No. 31100172), Program for Liaoning Excellent Talents in University (No. LJQ2013003), Youth Foundation of Liaoning University (No. 2010LDQN04).
国家自然科学基金 (No. 31100172),辽宁省高等学校优秀人才支持计划(No. LJQ2013003),辽宁大学青年科研基金 (No. 2010LDQN04) 资助。
张宁, 阮亚男, 王姗姗, 等. MITE类转座子m在水稻不同亚种间的差异分析. 生物工程学报, 2016, 32(9): 1264–1272.
Zhang N, Ruan YN, Wang SS, et al. Comparison of MITE transposons min different rice subspecies. Chin J Biotech, 2016, 32(9): 1264–1272.
