珍稀濒危植物黄檗种群遗传多样性ISSR分析
2016-10-26杨洪升李富恒王长宝戚晓利罗志文李修平张卫东东北农业大学生命科学学院哈尔滨150030佳木斯大学生命科学学院黑龙江佳木斯154007
杨洪升,李富恒,王长宝,戚晓利,罗志文,李修平,张卫东(1.东北农业大学生命科学学院,哈尔滨150030;.佳木斯大学生命科学学院,黑龙江佳木斯154007)
珍稀濒危植物黄檗种群遗传多样性ISSR分析
杨洪升1,2,李富恒1*,王长宝2,戚晓利2,罗志文2,李修平2,张卫东2
(1.东北农业大学生命科学学院,哈尔滨150030;2.佳木斯大学生命科学学院,黑龙江佳木斯154007)
利用ISSR分子标记对黄檗17个自然种群作遗传多样性分析,8条引物共扩增出81条带,其中61条具有多态性。结果表明,在物种水平上多态位点百分率(PPL)为75.31%,Shannon多样性指数(I)为0.3045,Nei's基因多样性指数(H)为0.2788。在种群水平上PPL为43.87%,I为0.2243,H为0.2628,种群间遗传分化系数Gst为0.2065,基因流Nm为1.9213。AMOVA分析表明,种群间遗传分化水平Fst为0.2235,黄檗遗传变异主要存在于种群内(77.65%)。Mantel检验表明,黄檗种群间地理距离与遗传距离间存在极显著正相关关系(R=0.789,P<0.01)。UPGMA聚类分析将17个黄檗种群分为两大类:华北种群组和东北种群组,地理较近种群有聚集趋势。推测长白山地区为黄檗现代遗传多样性分布中心。
黄檗;遗传多样性;遗传分化;ISSR;保护策略
网络出版时间2016-6-17 15:21:46[URL]http://www.cnki.net/kcms/detail/23.1391.S.20160617.1521.004.htm l
杨洪升,李富恒,王长宝,等.珍稀濒危植物黄檗种群遗传多样性ISSR分析[J].东北农业大学学报,2016,47(6):26-32.
Yang Hongsheng,Li Fuheng,Wang Changbao,et al.ISSR analysis on genetic diversity in natural populations of rare and endangered species Phellodendron amurense[J].Journal of Northeast Agricultural University,2016,47(6):26-32.(in Chinese with English abstract)
黄檗(Phellodendron amurense Rupr.)又称黄菠萝、黄柏,是芸香科(Rutaceae)黄檗属(Phelloden⁃dron)第三纪古热带植物区系孑遗植物。国内主要分布在东北小兴安岭、长白山、张广才岭、完达山、老爷岭及华北燕山等地,国外分布于俄罗斯远东地区、朝鲜和日本[1]。
黄檗作为我国东北阔叶红松林重要伴生乔木,三大硬阔树种之一,既是珍贵用材树种,也是我国传统中药黄柏药源植物。由于长期不合理药材采收及过量林木采伐,野生黄檗资源急剧减少,已被列入国家二级保护植物名录[2]。目前对黄檗研究主要集中在种群结构、化学成分含量、药理作用和更新繁殖等方面[3-6]。在黄檗遗传多样性研究方面,闫志峰等对10个野生黄檗种群作AFLP分析,认为黄檗物种水平遗传多样性高于其种群水平遗传变异[7]。李绍臣等利用ISSR分子标记吉林省10个黄檗种群作遗传多样性分析,结果表明黄檗种群间存在明显分化[8],由于所用分子标记及采样策略局限,未能全面揭示黄檗物种水平上遗传多样性和分布格局。
简单序列重复区间多态性(ISSR)是新型分子标记技术,综合其他分子标记技术特点[9],具有操作简单、所需DNA量少、多态性高、重复性好、不需预知研究对象的基因组序列等优点,已被成功应用于多种植物遗传多样性研究,并取得较好效果[10-13]。本研究采用ISSR分子标记,在分布区代表性山脉采样基础上,从核基因角度揭示野生黄檗种群遗传多样性,为合理开发利用黄檗资源及制定保护策略提供理论依据。
1 材料与方法
1.1材料
选取小兴安岭、长白山、张广才岭、完达山、老爷岭以及燕山等山脉17个野生种群,每个种群采集20~24个植株,为避免重复取样,植株间至少相隔50m,对每株样树胸径、树高、生境记录、拍照,并做好标记,每个植株采摘其健康幼嫩叶片8~10片,置于装有硅胶塑料自封袋迅速干燥,带回实验室于-20℃冰箱保存,用于DNA提取。采样地点种群编号、经纬度、海拔高度及每个种群所采集样本数量,采样种群地理位置分布见图1。
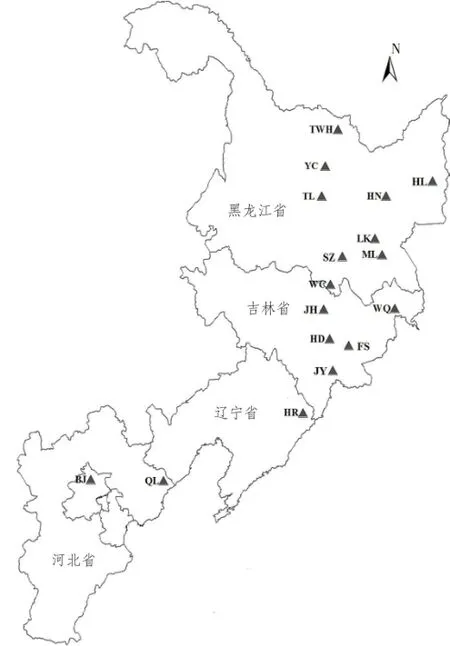
图1 黄檗样品采集分布Fig.1 Sampled populations distribution of Phellodendron amurense
1.2DNA提取和PCR扩增
采用改良CTAB法[14],从硅胶干燥后黄檗叶片中提取总DNA。分光光度计测定OD260和OD280,确定基因组DNA浓度和纯度后,稀释至40 ng·μL-1于-20℃冰箱保存备用。扩增反应在Eppendorf梯度PCR仪上进行,通过优化设计适合黄檗ISSRPCR最佳反应体系,最终确定为20μL反应体系,包括:ddH2O 13μL,10×Buffer 2μL,25mmol·L-1MgCl21.6μL,10 mmol·L-1dNTPs 0.4μL,10 μmol·L-1引物0.8μL,5 U·μL-1Taq DNA聚合酶0.2μL,40 ng·μL-1模板DNA 2μL。PCR扩增程序为:94℃预变性4min;35个循环:94℃变性30 s,退火45 s,72℃延伸2min;最后72℃延伸8min,4℃保存。PCR产物1.5%琼脂糖胶(含有0.5μg·mL-1EB)中电泳,电泳缓冲液为0.5× TBE,以DL2000Marker(TaKaRa,宝生物工程大连有限公司)为分子质量标准,5 V·cm-1稳定电压条件下电泳2 h,电泳后用凝胶成像系统拍照。
1.3引物筛选
本研究所用ISSR引物根据加拿大哥伦比亚大学(UBC)公布第9套ISSR引物序列(100条)进行特异性筛选,由上海生工生物工程股份有限公司合成。随机从12个种群中各选取1个个体,20μL反应体系扩增筛选。最终筛选出8条重复性好、扩增条带清晰且具有多态位点引物用于个体扩增。
1.4数据统计分析
由于ISSR是显性标记,在相同电泳迁移位置条带具有同源性。ISSR扩增结果电泳图谱中,有带记作“1”,无带或者弱带记作“0”,建立“0,1”二元数据矩阵,将二元数据输入计算机。采用软件POPGENE 1.32在假定这些ISSR标记处于Hardy-Weinberg平衡条件下作遗传参数分析。分别计算:多态位点百分率PPL(Proportion of polymor⁃phic loci)、有效等位基因数Ne(Effective number of alleles)、Nei's基因多样性指数H(Nei's gene diver⁃sity)、Shannon信息指数I(Shannon information in⁃dex)、群体总基因多样度Ht(Total gene diversity)、群体内基因多样度HS(Geniediversity within popula⁃tion)以及Nei's遗传距离D。群体间Nei's遗传分化系数Gst(Coefficient of genetic differentiation)由公式1-HS/Ht算得,基因流Nm(Gene flow)由公式(1-Gst)/ 4Gst算得。AMOVA 1.55软件作分子方差分析,计算种群内、种群间变异方差分布。采用GenALEX 6.5进行Mantel检测,检测种群间地理距离与遗传距离相关关系。用MEGA 5.0软件,采用Nei's(1972)计算遗传距离,作种群间UPGMA(非加权配对算数平均法)聚类分析。
2 结果与分析
2.1遗传多样性
利用8个ISSR引物对17个黄檗自然种群共398份样品作遗传多样性分析,每个引物均能扩增出清晰、稳定条带,其中引物UBC 827对桓仁(HR)种群24个个体DNA样品扩增结果见图2。8个引物共扩增出81条
清晰可重复条带,其中多态性条带61条,每条引物扩增出总带数范围从9(UBC827和UBC836)到12条(UBC818)不等,平均每个引物扩增条带数为10.1(见表1),所得片段大小分布在200~2 500 bp。
分析结果表明(见表2),在物种水平上,黄檗多态位点百分率为75.31%,显示较高遗传多样性水平,平均每个位点有效等位基因数为1.7382,Nei's基因多样性指数为0.2788,Shannon多样性指数为0.3045。
图2 UBC827引物对桓仁种群扩增结果Fig.2 ISSR bands amplified with primer UBC827 from Huanren population
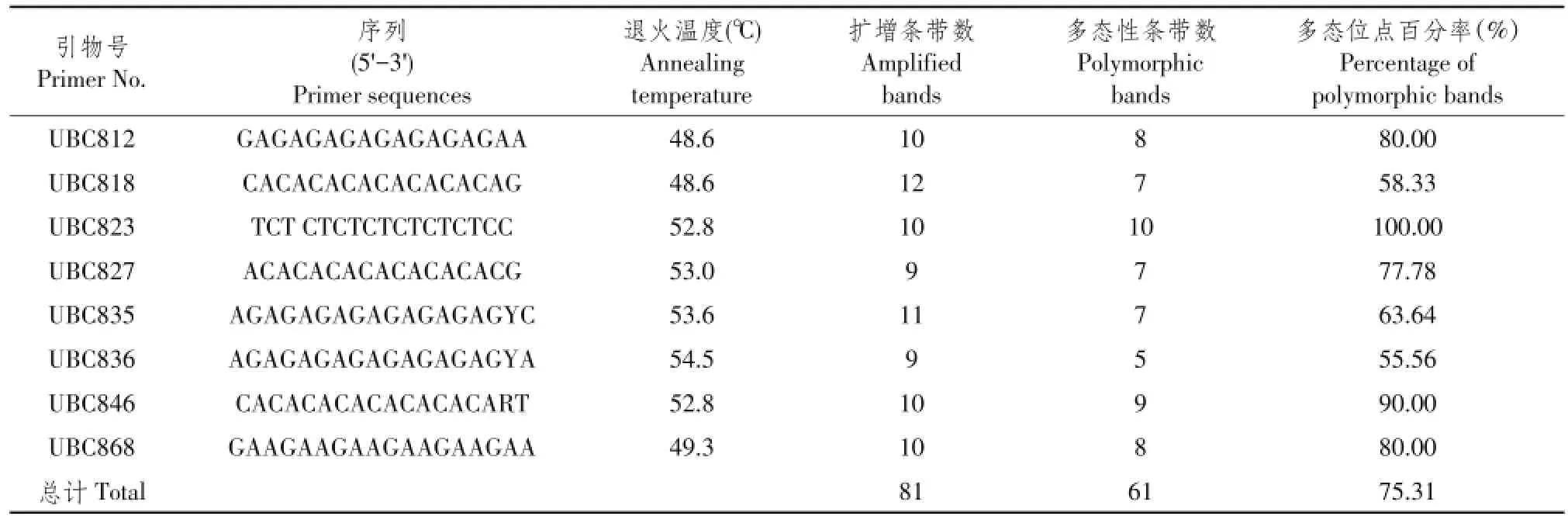
表1 ISSR引物及其扩增结果Table1 ISSR primers and their polymorphisms of amplified bands
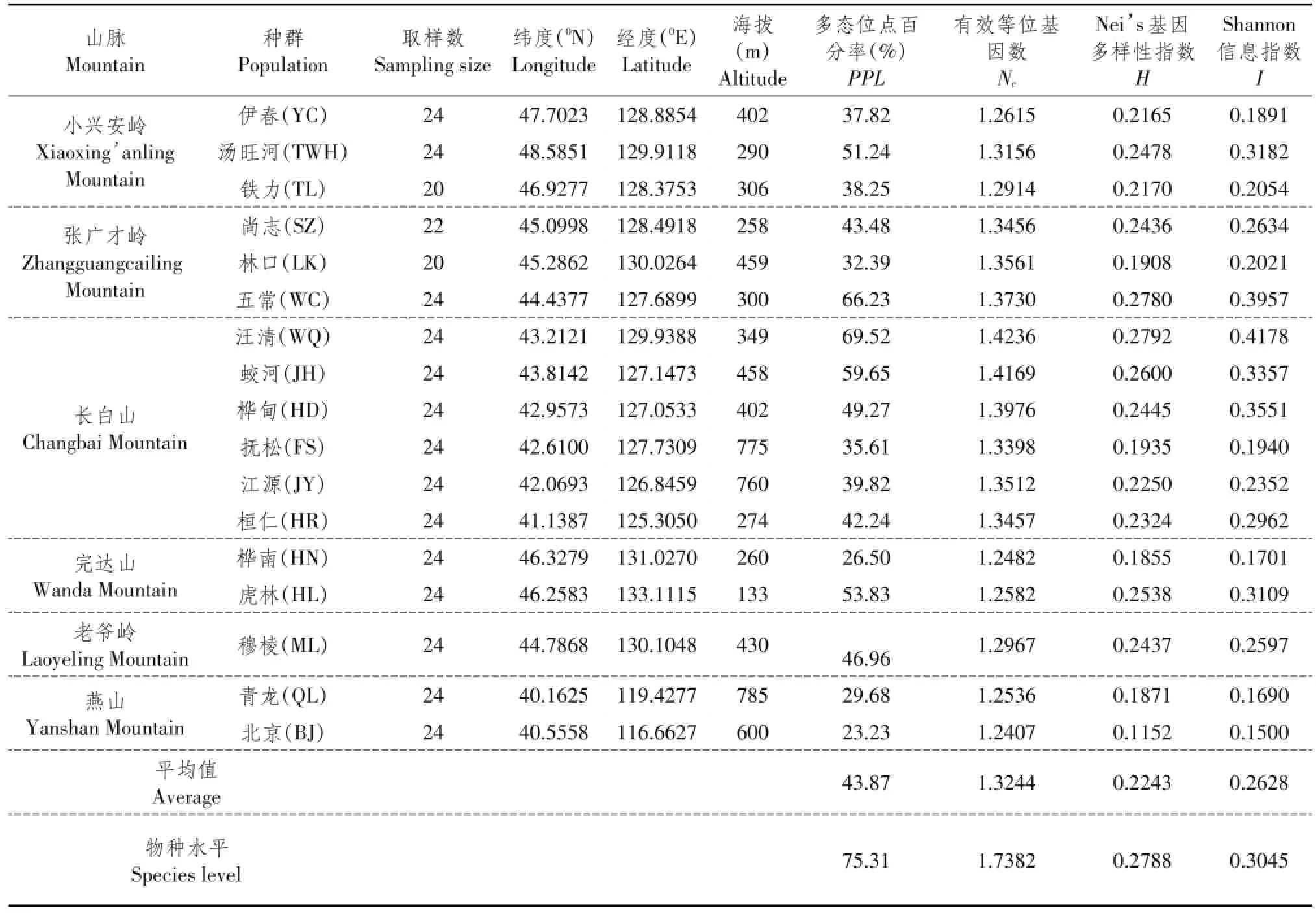
表2 黄檗取样种群信息及基于ISSR标记遗传多样性指数Table 2 Sampling details of P.amurense and genetic diversity based on ISSR marker
由表2可知,黄檗种群内多态性位点百分率23.23%~69.52%,平均43.87%,各种群多态性百分率差异较大,其中WQ种群多态位点百分率最高69.52%;BJ种群多态位点百分率最低23.23%。每个位点平均有效等位基因数(Ne)为1.3244,种群Nei's基因多样性指数在0.1152~0.2792,平均值0.2243;Shannon多样性指数0.1500~0.4178,平均值0.2628,且H和I较高值均出现在长白山WQ、WC和JH种群,较低值出现在BJ、HN和QL种群,两者大小与种群多态位点百分率趋势基本一致。
2.2黄檗种群间遗传变异
种群遗传变异AMOVA分析显示(见表3),黄檗种群间遗传分化系数Fst为0.2235,即在总遗传变异中有77.65%变异存在于种群内,种群内遗传变异比种群间遗传变异高,且种群内和种群间差异均极显著(P<0.001)。通过POPGENE分析,在物种水平上黄檗17个群体基因多样度(Ht)为0.2788,种群内基因多样度(Hs)为0.2212,种群间基因多样度Dst为0.0576,种群间遗传分化系数Gst为0.2065,显示总遗传变异中有79.35%来自种群内,20.65%来自种群间,遗传变异主要存在于种群内。由此可见,AMOVA和POPGENE分析结果基本一致,均表明种群内遗传分化大于种群间遗传分化。种群间基因流(Nm)估测值为1.9213,表明种群间基因流处于较高水平,有利于种群间基因交流。
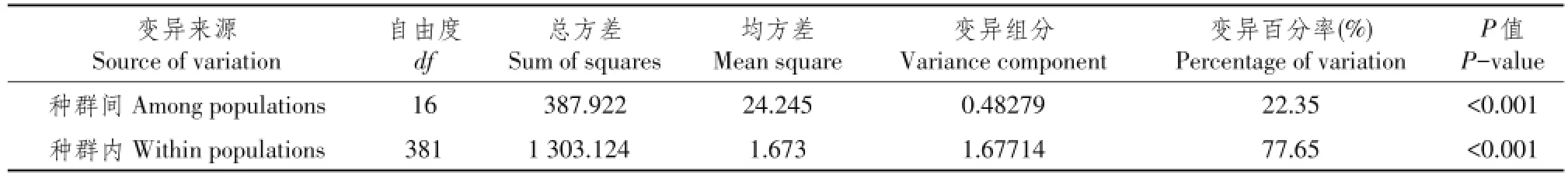
表3 17个黄檗种群分子方差分析Tab le3 AMOVA analyses of P.amurense from 17 populations
2.3种群间遗传距离及其聚类
POPGENE软件计算黄檗17个种群间Nei's遗传距离介于0.036与0.304之间。其中伊春(YC)种群与汤旺河(TWH)种群遗传距离最小,为0.036;林口(LK)种群与北京(BJ)种群遗传距离最大,为0.304。根据黄檗种群间遗传距离,运用MEGA软件采用算术平均数非加权成组配对法(UPGMA),对17个种群聚类分析,UPGMA聚类分析结果表明(见图3)。
17个黄檗种群主要分为两大类群:华北种群组和东北种群组。华北种群组包括北京(BJ)种群和青龙(QL)种群;东北种群组包括其余15个种群,其中长白山6个种群与老爷岭1个种群聚成1组,小兴安岭3个种群与张广才岭2个种群聚成1组,完达山2个种群单独聚成1组,3组聚成1个大类群,与其地理分布格局大致吻合。Mantel检验表明,黄檗各种群间遗传距离与地理距离存在正相关关系,且相关性极显著(R=0.789,P<0.01),表明地理距离越近种群,其遗传距离也越近;反之越远。
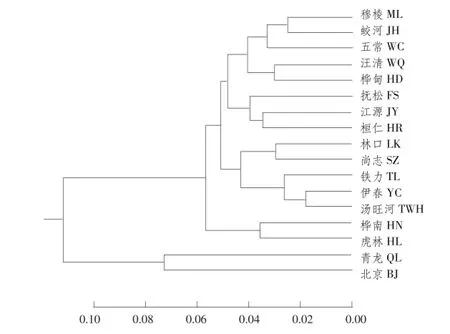
图3 17个黄檗种群间基于Nei's遗传距离UPGMA聚类结果Fig.3 UPGMA result for 17 sampled populations of P.amurense based on Nei'sgenetic distance
3 讨论与结论
3.1遗传多样性
遗传多样性是生物携带遗传信息总和,是长期进化产物,也是其生存、发展和进化基础[15]。在评价种群遗传多样性参数中,多态位点百分率、Shannon信息指数和Nei's基因多样性指数是衡量种群遗传多样性主要指标。本研究所用ISSR反应体系具有良好稳定性和可重复性,应用该分子标记技术对筛选出的8个引物PCR扩增,得出平均多态位点数7.6个,多态位点百分率75.31%,Shannon多样性指数0.3045,Nei's基因多样性指数0.2788。与其他采用ISSR分子标记检测的东北地区常见温带阔叶落叶树种蒙古栎[16]、胡桃楸[17]和紫椴[18]一致,表现出较高水平遗传多样性。本研究结果与闫志峰等采用AFLP分子标记对10个黄檗代表种群遗传多致性研究结果基本一致(PPL=92.77%;H= 0.2316;I=0.4275)[7],但比李绍臣等采用ISSR分子标记对局部区域黄檗遗传多样性要高[8],原因是本研究从黄檗自然分布大范围取样,基本覆盖黄檗在我国的自然分布区域。
黄檗具有较高遗传多样性可能与其繁育系统和生活史有关。黄檗属多年生落叶乔木,雌雄异株、虫媒传粉,这种异交繁育系统被认为是物种维持较高遗传多样性水平重要因素[19]。此外,黄檗具有较大分布范围、长命生活史及良好生态适应性[20],也有利于维持一定遗传多样性水平。不同黄檗种群遗传多样性差异显著(见表1),长白山地区野生资源分布集中,遗传多样性水平相对较高,由此推测长白山地区是黄檗遗传多样性分布中心。
3.2遗传分化
黄檗种群间Nei's基因分化系数Gst=0.2065,表明其遗传变异主要存在于种群内,即种群间遗传分化较小。AMOVA分析也显示,黄檗遗传变异中22.35%发生在种群间,77.65%发生在种群内。如前所述,黄檗生殖方式是有性生殖,主要传粉方式是虫媒,主要靠食果鸟类进行较远距离种子传播[6],这种传粉方式及种子传播方式有利于种群间基因流发生,可防止种群间遗传分化。根据Gst值,推算黄檗种群间基因流Nm=1.9213,表明黄檗种群间存在较强基因流,当Nm>1时,一定程度上可抵消种群间因遗传漂变而引起的遗传分化[21],这也是黄檗种群间遗传分化小,遗传变异主要存在于种群内原因之一。经Mantel检验发现遗传和地理距离呈显著正相关,说明地理隔离对黄檗种群遗传分化影响显著。根据UPGMA聚类结果表明,地理距离较近种群有聚集趋势,17个黄檗种群可分为两大类群即华北种群与东北种群,可能是由于两大类群地理距离较远,影响两者基因交流,长期自然选择进而导致遗传分化。
3.3保护策略
了解物种遗传多样性及其遗传分化可为制定珍稀濒危物种保护策略及栽培利用措施提供重要信息[19]。本研究发现黄檗物种水平上具有较高遗传多样性,遗传多样性主要存在于种群内。因此,黄檗野生种群仍具有较高适应能力和进化潜力。但若不能有效阻止生境恶化及人为破坏,将加速黄檗种群片断化,种群规模进一步减小,引起种群遗传漂变发生,导致当前丰富遗传多样性大量丧失,影响该物种生存及可利用优质资源丧失。因此,基于黄檗遗传多样性及种群生存现状,建议采取以下保护措施:
①应尽快建立黄檗专门保护区,同时重视黄檗栖息地以及周围环境保护,加强宣传力度,禁止乱砍乱伐,保护尽可能多的遗传变异。
②在加强对现有种群保护基础上,进一步加强黄檗繁殖生物学研究,由于黄檗果实粘度大,果皮腐烂速度慢,种子具有胚生理后熟特点,在自然环境下种子发芽率较低,因此有必要建立人工繁育基地以提高种子繁育率和幼苗成活率,扩大黄檗种群规模。
③华北种群和东北种群作为两个独立单元保护,尤其注意优先保护遗传多样性较高种群(WQ、WC、HD、JH)和遗传多样性较低种群(BJ、QL、HN)。
④考虑黄檗遗传变异主要来自于种群内,因此迁地保护时,需尽可能在所有种群内采样,保存遗传资源。
[1]限颖,王立军.黄檗的种质资源学研究[J].北方园艺,2010(20): 189-192.
[2]傅立国.中国植物红皮书-稀有濒危植物(第1册)[M].北京:科学出版社,1991:572.
[3]宋超,余琦殷,于梦凡,等.北京地区黄璧种群数量结构及空间分布特征[J].生态学杂志,2015,34(11):3040-3049.
[4]王瑾.RP-HPLC法测定黄柏中小檗碱、巴马汀和药根碱的含量[J].药物分析杂志,2007,27(4):610-612.
[5]杨周平,武志军.中药黄柏的药理作用和临床应用研究[J].甘肃医药,2010,29(3):329-331.
[6]鲁长虎,常家传,许青.黄檗的更新特点及食果实鸟类对其种子的传播[J].生态学杂志,2004,23(1):24-29.
[7]闫志峰,张本刚,张昭,等.珍稀濒危药用植物黄檗野生种群遗传多样性的AFLP分析[J].生物多样性,2006,14(6):488-497.
[8]李绍臣,李凤明,张立民,等.吉林省天然黄檗种群遗传多样性ISSR分析[J].生态学报,2016,36(13):1-7.
[9]Gilbert JE,Levis R V,Wilkinson M J,et al.Developing an appro⁃priate strategy to assess genetic variability in plant germplasm col⁃lections[J].Theor Appl Genet,1999,98(6):1125-1131.
[10]翟梅枝,肖志娟,许静,等.ISSR和SSR体系优化及在分析不同核桃品种遗传多样性上的应用[J].东北农业大学学报,2014, 45(1):90-97.
[11]刘海广,田贺,宋杨,等.茶藨属植物遗传变异和亲缘关系ISSR标记与表型分析[J].东北农业大学学报,2012,43(10):85-90.
[12]李晓红,张慧,王德元,等.我国特有植物青檀遗传结构的ISSR分析[J].生态学报,2013,33(16):4892-4901.
[13]王爱兰,王贵琳,李维卫.濒危物种珊瑚菜遗传多样性的ISSR分析[J].西北植物学报,2015,35(8):1541-1546.
[14]Doyle J J,Doyle JL.A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J].Phytochem Bull,1987,19:11-15.
[15]陈灵芝.中国的生物多样性现状及其保护对策[M].北京:科学出版,1993:11-15.
[16]张杰,吴迪,汪春蕾,等.应用ISSR-PCR分析蒙古栎种群的遗传多样性[J].生物多样性,2007,15(3):292-299.
[17]王东娜,牟长城,高卓,等.胡桃楸天然种群遗传多样性ISSR的分析[J].经济林研究,2011,29(2):22-29.
[18]穆立蔷,刘赢男.不同地理分布区紫锻种群的遗传多样性变化[J].植物生态学报,2007,31(6):1190-1198.
[19]Hamrick J L,Godt M J W.Effects of life history traits on genetic diversity in plant species[J].Philos Trans R Soc Lond B,1996, 351(1345):1291-1298.
[20]任青山.天然次生林主要种群生态位结构的研究[J].东北林业大学学报,1998,26(2):5-10.
[21]W right S.Evolution in Mendelian population[J].Genetics,1931, 16(1):97-159.
ISSR analysis on genetic diversity in natural populations of rare and endangered species Phellodendron amurense/YANG Hongsheng1,2,LI Fuheng1,
WANG Changbao2,QI Xiaoli2,LUO Zhiwen2,LI Xiuping2,ZHANG Weidong2
(1.School of Life Sciences,Northeast Agricultural University,Harbin 150030,China;2.School of Life Science, Jiamusi University,Jiamusi Heilongjiang 154007,China)
Genetic diversity of 17 natural populations of Phellodendron amurense was assessed by the Inter-Simple Sequence Repeat(ISSR)technique,using eight specific and stable primers,a total of 81 loci were identified,61 of which were polymorphic.The results showed that at the species level,total percentage of polymorphic loci(PPL)was 75.31%,and Shannon's information index(I)and Nei's gene diversity(H) were 0.3045 and 0.2788,respectively.At the population level,the mean value of P,I and H were 43.87%, 0.2243 and 0.2628,respectively.The genetic differentiation coefficient(Gst)among populations was 0.2065, and the value of gene flow(Nm)inferred from Gst was 1.9213.Analysis of molecular variance(AMOVA)demonstrated that most variance occurred within populations,while the variance among populations accounted for 22.35%(Fst=0.2235).Mantel test results showed a significant positive correlation between genetic distance and geographic distance among all populations(R=0.789,P<0.01).Total 17 populations were clustered into two groups by the UPGMA analysis including the North China group and the Northeast China group.Moreover,results of UPGMA clustering analysis showed that the populations tend to cluster together were nearer in geographical distance.The Changbai Mountain may be modern center of genetic diversity for P.amurense.
Phellodendron amurense;genetic diversity;genetic differentiation;ISSR;protection strategy图1黄檗样品采集分布
S792.31
A
1005-9369(2016)06-0026-07
2016-03-17
国家自然科学基金项目(81441132);黑龙江省教育厅科学技术研究项目(12541808);佳木斯大学青年基金项目(Sq2013-026)
杨洪升(1979-),男,讲师,博士研究生,研究方向为植物资源学与分子生态学。E-mail:yhongsheng@126.com
李富恒,教授,博士生导师,研究方向为植物生理生化。E-mail:lifuheng1963@126.com
