玉米光敏色素A1与A2在各种光处理下的转录表达特性
2016-10-19杨宗举闫蕾宋梅芳苏亮孟凡华李红丹1白建荣郭杨建平
杨宗举闫 蕾宋梅芳苏 亮孟凡华李红丹1,白建荣郭 林,*杨建平,*
1中国农业科学院研究生院, 北京 100081;2中国农业科学院作物科学研究所, 北京 100081;3山西大学生物工程学院, 山西太原030006;4北京市辐射中心, 北京 100875;5山西省农业科学院作物科学研究所, 山西太原 030031
玉米光敏色素A1与A2在各种光处理下的转录表达特性
杨宗举1,2,**闫 蕾2,3,**宋梅芳2,4苏 亮2孟凡华2李红丹1,2白建荣5郭 林2,*杨建平2,*
1中国农业科学院研究生院, 北京 100081;2中国农业科学院作物科学研究所, 北京 100081;3山西大学生物工程学院, 山西太原030006;4北京市辐射中心, 北京 100875;5山西省农业科学院作物科学研究所, 山西太原 030031
光敏色素是一类红光/远红光受体, 它们在植物体内有非活性形式的红光吸收型(Pr)和活性形式远红光吸收型(Pfr) 2种状态, 通常其活性形式负责调控植物的种子萌发、株高、开花时间和避荫性等生长发育过程。在禾本科中, 光敏色素只有 PHYA、PHYB和 PHYC三个基因亚家族, 古四倍体化造成的玉米光敏色素基因有 6个成员, 即PHYA1、PHYA2、PHYB1、PHYB2、PHYC1和PHYC2。光敏色素A参与抑制下胚轴的伸长、促进张开子叶和花青素的积累、阻断持续远红光条件下的变绿。为了评价ZmPHYA1和ZmPHYA2对光的响应能力及其功能差异, 本研究采用实时定量PCR技术分析玉米自交系B73和Mo17中ZmPHYA1和ZmPHYA2对不同光照处理响应的表达模式。结果表明玉米光敏色素A主要在叶片和花丝中表达, 并且ZmPHYA1转录丰度是ZmPHYA2的2~8倍; 玉米自交系B73 和Mo17中胚轴在黑暗、远红光和蓝光条件下较红光和白光下更长。ZmPHYA1和ZmPHYA2的转录水平在持续远红光和蓝光条件下均较高; 并且均较迅速响应黑暗到远红光和蓝光光质转换, 但是前者的丰度显著高于后者,ZmPHYA1在远红光下更重要, 而ZmPHYA2在蓝光下更重要。ZmPHYA1和ZmPHYA2同样响应于黑暗到红光和白光的转换, 并且ZmPHYA1和ZmPHYA2表达模式基本一致。ZmPHYA1和ZmPHYA2的表达均能响应长日照和短日照处理, 但是ZmPHYA1转录丰度高于ZmPHYA2的2~5倍。以上结果表明, ZmPHYA1和ZmPHYA2的转录能有效地响应各种光处理, 可能ZmPHYA1在作物改良上比ZmPHYA2更有效。本研究为进一步了解ZmPHYA1和ZmPHYA2基因功能以及评价二者的光反应能力提供了理论基础。
玉米; 光敏色素; 光信号转导; 表达分析; 光处理
植物生长发育过程会受到来自环境的生物及非生物因素影响[1], 其中光作为重要的非生物环境因子, 对植物整个生命过程起至关重要的作用[2]。光不仅是植物光合作用的能量来源, 而且作为环境信号参与调控种子萌发、幼苗去黄化、叶片展开、下胚轴伸长、向光性、气孔开关、叶绿体移动、避荫性、节律、开花等生长和发育过程[2-6]。植物的光受体系统负责感知光的强度、波长、节律和方向[6-11], 高等植物至少存在 3类光受体系统, 以满足它们对不同波长信号(红光/远红光、蓝光/紫外光A, 以及紫外光B)的吸收[12], 其中光敏色素是红光/远红光(600~750 mm)的受体[13]。模式植物拟南芥具有 5个光敏色素基因(PHYA-PHYE)[14], 它们在植物生长和发育过程中明确分工, 又功能冗余[2]。光敏色素A (phyA)和光敏色素 B (phyB)均参与萌发的调节, 但前者主要是负责极低辐照反应(very-low-fluence response, VLFR,0.1~1.0 µmol m-2s-1), 而后者主要负责低辐照反应(low-fluence response, LFR, 1~1000 µmol m-2s-1)[15-17]。光敏色素A属于光不稳定类型[18], 在黑暗和远红光条件下转录和蛋白丰度较高, 而在红光和白光下蛋白质稳定性极差, 转录水平也很低[19]。拟南芥的光敏色素 A不但参与远红光信号途径, 而且介导红光和蓝光信号转导[20]。研究表明, phyA在植物整个生命过程中起重要作用[21]。黑暗条件下丰度最高的光受体 phyA对萌发种子光形态建成的开启起主要作用; 露出土壤后的幼苗通过 phyA感知周围因遮盖导致的弱辐照, 启动极低辐照反应来完成最初的去黄化反应[22]。phyA在黑暗条件下是稳定的红光吸收型(Pr), 受光激活后变为极易被降解的远红光吸收型(Pfr)[23-25]。phyA突变体在短日照处理条件下开花延迟、叶片增多, 表明phyA参与感受光周期的变化[26]。在长日照条件下, phyA 通过增加CONSTANS (CO)蛋白的稳定性来提高FLOWERING LOCUS T (FT)的表达, 进而加速开花[27]。phyA不仅在促进种子萌发和幼苗去黄化过程发挥重要作用[28],而且通过增强 phyB的作用来抑制植物的避荫性反应[29]。马铃薯的 phyA通过促进体内花青素合成和蔗糖磷酸合成酶活性, 来增强幼苗早期对周围环境感知的能力[30]。水稻的 phyA在长日照条件下延迟开花, 却在短日照条件下促进开花[31]。
通过修饰作物的光敏色素途径可以改良株高、增强光反应能力和提高产量。在水稻中过量表达拟南芥PHYA基因, 尽管没有导致转基因植株节数的变化, 但是节间却明显缩短, 导致成株高度显著降低[32]。在马铃薯中转入拟南芥 PHYB基因, 转基因植株在高密度栽培条件(20株 m-2)下, 节间和叶柄缩短而株高明显降低, 叶绿素含量高而光合作用增强, 单株块茎产量得到显著提高[33-35]。在烟草中过量表达燕麦PHYA基因可以增加叶面积指数[36]。最新的研究表明, 在长日照条件下小麦 PHYC的缺失使生物钟及光周期基因表达发生改变, 因而开花延迟[37]。
迄今, 人们对于双子叶植物模式植物拟南芥的光敏色素研究较为深入, 但对于单子叶植物光敏色素的研究还鲜有报道。在禾本科植物中, 光敏色素基因只存在3个亚家族: PHYA、PHYB和PHYC[40-41]。玉米的古四倍体化过程导致其产生光敏色素A有2个拷贝, ZmPHYA1和ZmPHYA2, 它们之间是否存在功能分化, 以及二者之间的相互关系, 一直引起我们的关注。本研究以玉米自交系B73和Mo17为材料, 测量不同光质条件下玉米中胚轴长度; 通过实时定量PCR技术研究不同光照处理下ZmPHYA1和ZmPHYA2的表达差异, 了解它们的表达模式, 为评价二者的光反应能力及在玉米改良中的价值提供科学依据。
1 材料与方法
1.1 试验材料及样品处理
1.1.1 试验材料 选用玉米自交系B73和Mo17(本实验室保存)。
1.1.2 器官特异性表达样品的准备 将自交系B73的种子在北京地区4月下旬播种于花盆中自然条件下生长60 d, 分别取成株的根、茎、叶、叶枕、叶鞘、花丝、花柄、雄花、苞叶和幼穗。
1.1.3 各种持续光质处理 将玉米种子消毒杀菌后, 平放在湿润滤纸的培养皿中28℃催芽3 d, 挑选萌动一致的种子播于装有培养土塑料小盆(长宽高均为8.5 cm)中, 每小盆播9粒, 所有玉米幼苗均生长在22℃。分别放黑暗(Dk)、远红光(FR, 0.5 µmol m-2s-1)、红光(R, 22.3 µmol m-2s-1)、蓝光(B, 13 µmol m-2s-1)和白光(WL, 17 µmol m-2s-1)培养箱中生长13 d。
1.1.4 黑暗转换各种光质 将幼苗自黑暗中生长13 d, 分别转入以上各种光质条件下, 0.25、0.5、1、2、4、8、12和24 h后, 分别取幼苗地上部为试材。
1.1.5 长日照和短日照处理 玉米幼苗在22℃长日(LD, 16 h光照/8 h黑暗)或短日(SD, 8 h光照/16 h黑暗)条件下中生长13 d, 每隔2 h取一次样。
1.2 RNA提取及cDNA合成
用 TRIzol (Invitrogen, USA)法提取各种处理的玉米幼苗总RNA, 经DNase I (RNase-free, TaKaRa大连公司)处理后作为模板, Oligo-dT18为引物, 利用RevertAid First Strand cDNA Synthesis Kit (Thermo Scientific公司)反转录成cDNA备用。
1.3 玉米实时荧光定量RT-PCR (qRT-PCR)分析
以不同处理的B73玉米的cDNA第1链为模板, 玉米 Tubulin基因为内参, 测定 ZmPHYA1和ZmPHYA2的相对表达量。ZmPHYA1和ZmPHYA2 与Tubulin的序列分别来源于PLAZA网站和NCBI网站, 利用Primer Premier 5.0软件设计荧光定量PCR引物(表1)。荧光定量 PCR仪为 Roche 480 (Roche, 瑞士), PCR程序为95℃预变性30 s, 再进行以下循环: 95℃变性5 s, 60℃退火20 s, 72℃延伸10 s, 共进行50个循环; 然后60~95℃绘制溶解曲线。定量PCR试剂为SYBR Premix Ex Taq II (TaKaRa大连公司), 按照商家使用说明操作。采用 2-ΔΔCT的方法计算结果[42]。经 3次独立的生物学重复, 并依此计算标准差。采用SAS9.2软件对数据进行方差及相关分析, 用 Duncan’s新复极差法进行多重比较, 并采用Microsoft Excel 2010软件绘图。
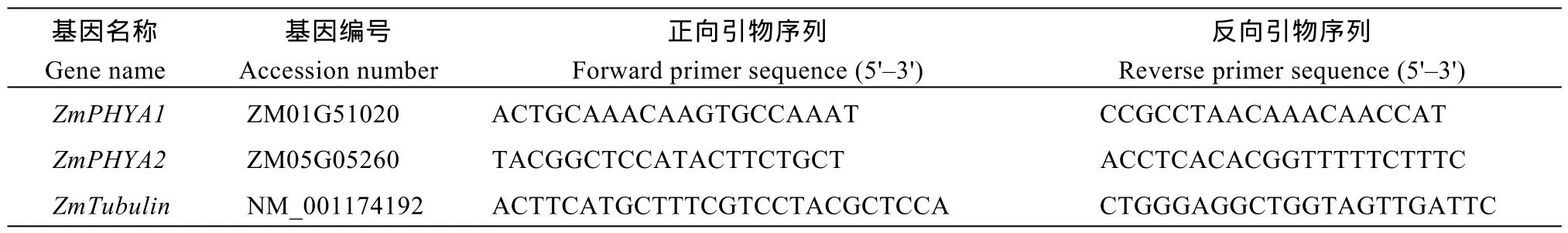
表1 qRT-PCR所用引物Table1 Primers for qRT-PCR
2 结果与分析
2.1 ZmPHYA1和ZmPHYA2器官特异性表达分析
玉米光敏色素A基因在根、茎、叶、叶枕、叶鞘、花丝、花柄、雄花、苞叶和幼穗中均有表达, 尤其在叶片和花丝中的表达量较高, 其中ZmPHYA1在叶片和花丝中分别为在根中的6.5倍和10.9倍, 推测叶片和花丝是玉米光敏色素A主要起作用的部位。从图1可知, 根、茎、叶、叶枕、叶鞘、花丝、花柄和苞叶中ZmPHYA1的转录丰度约为ZmPHYA2的4~10倍,雄花和幼穗中ZmPHYA1的转录丰度约为ZmPHYA2 的1.5倍和3.0倍。ZmPHYA1在各器官中的表达丰度均明显高于ZmPHYA2, 暗示ZmPHYA1可能比ZmPHYA2有更重要的作用。
2.2 玉米中胚轴伸长响应不同光质处理
采用玉米中胚轴作为衡量其光形态建成程度的指标, 为了解它的伸长与光质的关系, 测量了在不同光质持续照射条件下玉米自交系B73和Mo17的中胚轴长度。在黑暗条件下因黄化反应, 中胚轴明显长于其他光质处理(图2)。远红光、红光、蓝光和白光均可以抑制玉米中胚轴伸长, 并且红光和白光条件下对中胚轴的抑制作用较远红光和蓝光更为明显。可见, 玉米中胚轴伸长是响应各种光质处理的。
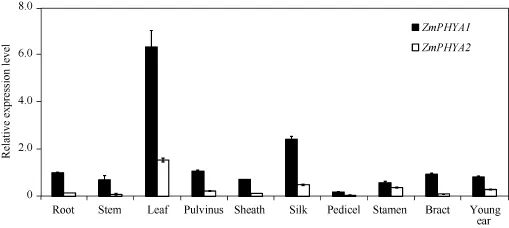
图1 ZmPHYA1和ZmPHYA2器官特异性表达的定量RT-PCR分析Fig.1 qRT-PCR assay of ZmPHYA1和ZmPHYA2 expression in different organs of maize
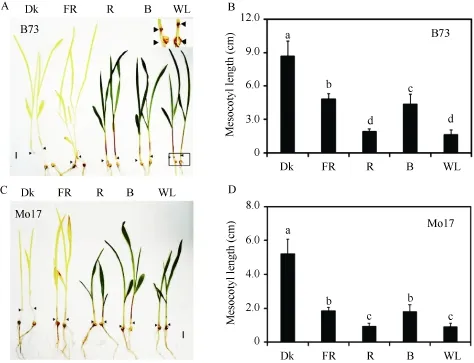
图2 各种持续光质条件下B73和Mo17中胚轴长度Fig.2 Mesocotyl lengths of B73 and Mo17 under different continuous light conditions
2.3 ZmPHYA1和ZmPHYA2对持续不同光质处理的响应
图3-A显示ZmPHYA1在红光和白光下的转录丰度很低, 仅为自身黑暗中的 30%和 40%; 远红光条件下, ZmPHYA1的转录丰度最高, 约为自身黑暗中的1.4倍; 而蓝光下的表达丰度与黑暗条件下相似。ZmPHYA2在红光和白光中表达丰度也非常低(图3-B), 在蓝光下的转录丰度约为自身黑暗中的1.3倍;而远红光下 ZmPHYA2的转录丰度仅为其在黑暗中的 80%。持续光条件下的转录表达分析结果显示,ZmPHYA1和ZmPHYA2均受到红光和白光的强烈抑制, 这与拟南芥的PHYA基因结果类似, 可能与它们编码的蛋白质在光下不稳定有关。另外从二者在远红光和蓝光下的转录丰度推测, 2个基因在远红光和蓝光中均具有作用; ZmPHYA1在远红光下更重要,而ZmPHYA2在蓝光下更重要。
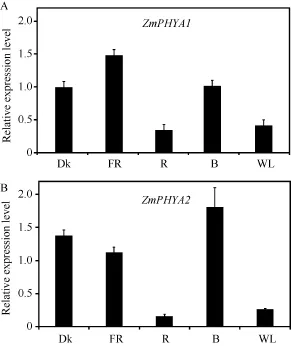
图3 各种持续光质条件下ZmPHYA1和ZmPHYA2转录表达的定量RT-PCR分析Fig.3 qRT-PCR assay of ZmPHYA1 and ZmPHYA2 expression under different continuous light conditions
2.4 ZmPHYA1和ZmPHYA2对黑暗到不同光质转换的响应
我们将黑暗中生长13 d的B73幼苗分别转入到远红光、蓝光、红光和白光下0、0.25、0.5、1、2、4、8、12和24 h, 来进一步探究ZmPHYA1和ZmPHYA2转录丰度对不同光质转换的响应。ZmPHYA1的转录能迅速响应黑暗到远红光的转换, 0.25 h内其转录水平上升到自身黑暗中的1.9倍, 0.5 h内继续上升到自身黑暗中的4.5倍, 之后迅速上升并在1 h达到峰值(黑暗中的18倍); 但在2 h、4 h和8 h分别下降到峰值的70%、60%和6%, 其较低的转录水平一直维持到12 h; 在远红光处理24 h后, 其转录水平又回升到自身黑暗中的3.6倍(图4-A)。ZmPHYA2在黑暗到远红光的转换最初0.5 h之内与ZmPHYA1的转录水平相当, 属于较平缓的上升, 但在1 h上升到仅为黑暗中ZmPHYA1的3.4倍的峰值; 随后缓慢下降, 8 h时达到超低值(黑暗中ZmPHYA1的2%), 并且其转录在24 h内均维持较低水平(黑暗中ZmPHYA1的2%~10%)。由此可见, ZmPHYA1和ZmPHYA2均能迅速响应远红光;ZmPHYA2的转录在24 h内只有一个峰值, 上升和下降均较缓慢; 而ZmPHYA1的转录不但迅速响应黑暗到远红光的转换, 并且峰值达到了自身黑暗中的18倍。这些结果暗示ZmPHYA2在持续远红光中起重要作用, 而ZmPHYA1在对远红光光质转换的响应中更重要。
ZmPHYA1和ZmPHYA2转录丰度能迅速响应黑暗到蓝光的转换(图4-B)。在0.25 h时ZmPHYA1急剧上升至自身黑暗中的15倍, 之后继续飙升, 在0.5 h时达到峰值(约为自身黑暗中的 56倍); 随后其转录水平迅速下降, 在 2 h时已基本恢复至自身黑暗的水平; 在4 h出现第2个峰值(自身黑暗中的5倍,最大峰值的 9%); 随后其转录丰度稳定在相对较低的水平(自身黑暗中的44.6%~71.7%)。同ZmPHYA1类似 ZmPHYA2峰值也出现在 0.5 h (约为黑暗中ZmPHYA1的10倍); 随后迅速下降, 在1 h后稳定在黑暗中ZmPHYA1的6.7%~23.7%的水平。可以看出,二者均能迅速响应黑暗到蓝光的转换, 且光转换早期表达丰度上升快; ZmPHYA2的转录丰度在24 h内只有一个峰值; 而ZmPHYA1出现了两个峰值, 并且峰值极大(自身黑暗中的56倍)。以上结果表明ZmPHYA1 和 ZmPHYA2在蓝光信号途径中起重要作用, 而ZmPHYA1在对蓝光光质转换的响应更为重要。
ZmPHYA1和ZmPHYA2对黑暗到红光转换的转录表达模式基本一致(图4-C)。在转入红光 0.25 h,ZmPHYA1的转录水平未发生变化; 在0.5 h达到第1个峰(黑暗中自身的 3.4倍), 随后迅速下降, 在 2 h恢复至自身黑暗起点时的水平, 而在4 h略有上升;在 8~12 h期间其转录丰度为自身黑暗的 35%左右;之后迅速上升, 在24 h已上升至转入红光24 h内的峰值(自身黑暗的4.5倍)。ZmPHYA2的转录模式整体与 ZmPHYA1一致, 稍有不同的是在刚转入红光0.25 h内, ZmPHYA2的转录丰度先下降至ZmPHYA1黑暗的 31%, 之后在 0.5 h上升至第 1个高于此时ZmPHYA1的峰值; 以后其转录丰度均低于ZmPHYA1, 在24 h, ZmPHYA2仅恢复至自身黑暗时的水平。由此看来, ZmPHYA1和ZmPHYA2在黑暗到红光转换的转录模式基本一致, 但从表达丰度上看,ZmPHYA1在前期作用更重要。
从图4-D可以看出, ZmPHYA1和ZmPHYA2由黑暗到白光转换的转录模式基本一致, 二者均在0.5 h时达到各自唯一的峰值, 此时 ZmPHYA1和ZmPHYA2的转录丰度分别达到黑暗中ZmPHYA1的35和10倍, 之后迅速下降至低于黑暗水平并保持稳定。以上结果表明, ZmPHYA1和ZmPHYA2均迅速响应黑暗到白光的转换。
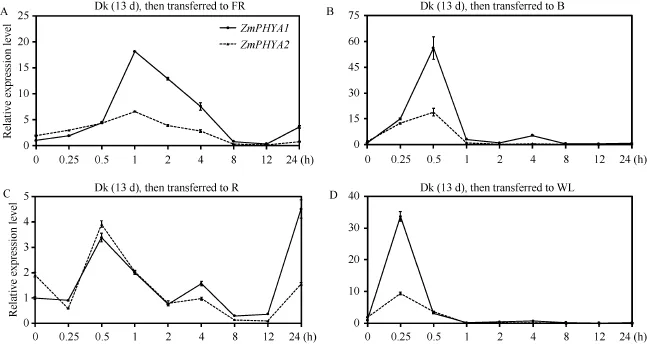
图4 黑暗到不同光质转换条件下ZmPHYA1和ZmPHYA2转录表达的定量RT-PCR分析Fig.4 qRT-PCR assays of ZmPHYA1 and ZmPHYA2 expressions during transition from the dark to different light conditions
2.5 ZmPHYA1和ZmPHYA2在长日照和短日照处理下的响应
长日照处理条件下, ZmPHYA1和ZmPHYA2的转录表达模式一致(图5-A), 但是整体上 ZmPHYA2转录丰度仅为ZmPHYA1的22%~51%。在光照和黑暗阶段各出现一个峰值。在光照阶段峰值出现在8 h,此时ZmPHYA1和ZmPHYA2的转录丰度分别是各自在黑暗阶段结束时的2.4倍和4.6倍。黑暗阶段的峰值出现在进入黑暗后 4 h, 此时 ZmPHYA1和 ZmPHYA2的转录丰度分别是各自在黑暗阶段结束时的 7倍和 5倍。在白光到黑暗转换时 ZmPHYA1 和ZmPHYA2都能迅速上升至较高水平。
ZmPHYA1和ZmPHYA2的转录表达模式对短日照处理的响应亦相似(图5-B), ZmPHYA2转录丰度仅为ZmPHYA1的21%~53%。在光照阶段, 二者转录丰度基本保持稳定状态。在转入黑暗后二者开始波动性上升, 在转入黑暗10 h均出现第1个峰值, 此时ZmPHYA1和ZmPHYA2的转录丰度分别是各自在黑暗阶段结束时的6倍和9倍。随后迅速下降, 2 h后达到最低(恢复至各自在黑暗阶段结束时水平)。在转入黑暗14 h达到第2个峰值(分别是各自在黑暗阶段结束时的 9倍和 5倍)。可见, ZmPHYA1和ZmPHYA2对长日照和短日照处理响应的表达模式极其一致, 前者转录丰度总体上是后者的2~5倍。
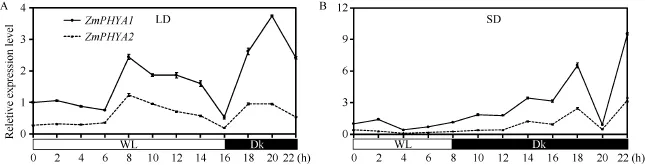
图5 长日照和短日照件下ZmPHYA1和ZmPHYA2转录表达的定量RT-PCR分析Fig.5 qRT-PCR assays of ZmPHYA1 and ZmPHYA2 expression under long day and short day conditions
3 讨论
本研究表明, ZmPHYA1和ZmPHYA2在玉米的叶片以及花丝中表达量较高, ZmPHYA1表达量约是ZmPHYA2的2~8倍, 且ZmPHYA1在各个器官中的表达丰度均显著高于ZmPHYA2 (图1)。玉米中胚轴伸长是响应不同光质处理的, 红光和白光下中胚轴较远红光和蓝光下更短(图2)。ZmPHYA1和ZmPHYA2的转录表达均响应远红光、蓝光、红光和白光处理(图3), 在黑暗到远红光、蓝光、红光或白光转换下 ZmPHYA1的转录丰度均高于 ZmPHYA2(图4)。另外, 二者在长日照和短日照处理下,ZmPHYA1转录丰度总体上是ZmPHYA2的2~5倍(图5), 推测ZmPHYA1和ZmPHYA2在响应光周期上可能是不同的, 也暗示着二者功能可能存在差异。
水稻、玉米、小麦、高粱、谷子等是世界范围的粮食作物, 人类对它们已经进行了 5000~10 000年的人工选择。作物的开花期和株高, 以及产量均与其栽培季节、纬度、海拔相适应, 因此这些性状都受到特定光照(红光/远红光)的调节[43]。光是影响玉米生长发育的重要因素, 减少光照时间会造成玉米早花[44]。玉米在1.4~2.0亿年前发生的异源四倍体化造成玉米中光敏色素基因为双拷贝[45-49]。ZmPHYB1与 ZmPHYB2功能上的差异主要表现在phyB1抑制红光下的中胚轴伸长, 而 phyB2主要负责光周期介导的开花调控[50]。我们的研究表明,ZmPHYA1和ZmPHYA2的转录丰度对黑暗到各种光质的转换, 以及长日照和短日照处理的响应存在差异, 这是否反映二者在功能上也存在差异, 还待进一步研究。
光受体过量表达的转基因植株节间缩短和植株矮化, 暗示修饰光信号转导途径同样能有效地改良株高和株型。在双子叶植物番茄中过量表达单子叶燕麦的 PHYA基因, 导致植株显著降低[51]。在烟草中转化水稻类型 I光敏色素(PHYA)后, 转基因植株表现下胚轴变短, 并且在伸长期生长速率减缓, 造成下胚轴变短的原因是表皮细胞变短而不是细胞数量的减少[52]。水稻phyA参与抑制幼苗中胚轴伸长[53]。玉米elm1突变体表现幼苗中胚轴变长、成株株高增加[54]。单子叶植物玉米中胚轴(mesocotyl)在功能上类似于双子叶植物的下胚轴, 可作为光形态建成的指标[54]。本研究表明玉米中胚轴伸长是响应光质处理的, 这种响应是否与光敏色素活性相关还需要进一步探究。
在拟南芥中, 长日照条件下CRY2与phyA通过与生物钟基因相互作用可以加速开花[55]。短日照植物水稻phyB或phyC突变体在长日照条件下均表现早花, 而phyA突变体却对开花时间没有影响, 但是phyA突变体在phyB或phyC突变体背景下却出现早花现象[56]。研究表明phyA参与光周期调节[26], 在长日照条件下, 水稻的 phyA通过促进开花抑制因子GRAIN NUMBER及降低开花诱导物 EARLY HEADING DATE 1的活性来导致开花推迟[56]。由此可见, 光信号转导途径与植物的开花期诱导紧密相关, 通过修饰光信号转导途径改良植物开花期性状是行之有效的方法[10]。本研究还表明, ZmPHYA1和ZmPHYA2转录丰度均强烈地响应在长日照和短日照处理, 玉米光敏色素是否参与其开花调控, 以及分子机制值得进一步探讨。
4 结论
ZmPHYA1和ZmPHYA2主要在叶片和花丝中表达; 玉米中胚轴伸长是响应不同光质处理的, 远红光和蓝光下中胚轴较红光和白光下更长; ZmPHYA1 和ZmPHYA2转录丰度较强地响应远红光和蓝光, 并呈现不同转录表达模式, ZmPHYA1在远红光下更重要, 而 ZmPHYA2在蓝光下更重要。ZmPHYA1和ZmPHYA2的转录能有效地响应各种光处理, 可能ZmPHYA1在作物改良上比ZmPHYA2更有效。
References
[1] Fankhauser C, Chory J.Light control of plant development.Annu Rev Cell Dev Biol, 1997, 13: 203-229
[2] Deng X W, Quail P H.Signalling in light-controlled development.Semin Cell Dev Biol, 1999, 10: 121-129
[3] Casal J J, Candia A N, Sellaro R.Light perception and signalling by phytochrome A.J Exp Bot, 2014, 65: 2835-2845
[4] Wang H Y, Deng X W.Dissecting the phytochrome A-dependent signaling network in higher plants.Trends Plant Sci, 2003, 8:172-178
[5] Jiao Y, Lau O, Deng X.Light-regulated transcriptional networks in higher plants.Nat Rev Genet, 2007, 8: 217-230
[6] Li J, Li G, Wang H, Deng X W.Phytochrome Signaling Mechanisms.Arabidopsis Book, 2011, 9: e0149
[7] Quail P H.Phytochrome photosensory signalling networks.Nat Rev Mol Cell Biol, 2002, 3: 85-93
[8] Bae G, Choi G.Decoding of light signals by plant phytochromes and their interacting proteins.Annu Rev Plant Biol, 2008, 59:281-311
[9] Quail P H.Phytochrome photosensory signalling networks.Nat Rev Mol Cell Biol, 2002, 3: 85-93
[10] 詹克慧, 李志勇, 侯佩, 习雨琳, 肖阳, 孟凡华, 杨建平.利用修饰光敏色素信号途径进行品种改良的可行性.中国农业科学, 2012, 45: 3249-3255 Zhan K H, Li Z Y, Hou P, Xi Y L, Xiao Y, Meng F H, Yang J P.A new strategy for crop improvement through modification of phytochrome signaling pathways.Sci Agric Sin, 2012, 45:3249-3255 (in Chinese with English abstract)
[11] Boylan M T, Quil P H.Oat phytochrome is biologically active in transgenic tomatoes.Plant Cell, 1989, 1: 765-773
[12] Gyula P, Schäfer E, Nagy F.Light perception and signalling in higher plants.Curr Opin Plant Biol, 2003, 6: 446-452
[13] Quail P H.Photosensory perception and signalling in plant cells:new paradigms? Curr Opin Plant Biol, 2002, 14: 180-188
[14] Sharrock R A, Clack T, Goosey L.Differential activities of the Arabidopsis PHYB/D/E phytochromes in complementing PHYB mutant phenotypes.Plant Mol Biol Rep, 2003, 52: 135-142
[15] Reed J W, Nagatani A, Elich T D, Fagan M, Chory J.Phytochrome A and phytochrome B have overlapping but distinct functions in Arabidopsis development.Plant Physiol, 1994, 104:1139-1149
[16] Botto J F, Sanchez R A, Whitelam G C, Casal J J.Phytochrome A mediates the promotion of seed germination by very low fluences of light and canopy shade light in Arabidopsis.Plant Physiol,1996, 110: 439-444
[17] Shinomura T, Nagatani A, Hanzawa H, Kubota M, Watanabe M,Furuya M.Action spectra for phytochrome A- and B-specific photoinduction of seed germination in Arabidopsis thaliana.Proc Natl Acad Sci USA, 1996, 93: 8129-8133
[18] Furuya M.Molecular properties and biogenesis of phytochrome I and II.Adv Biophys, 1989, 25: 133-167
[19] Quail P H.An emerging molecular map of the phytochromes.Plant Cell Environ, 1997, 20: 657-665
[20] Franklin K A, Whitelam G C.Phytochrome a function in red light sensing.Plant Signal Behav, 2007, 2: 383-385
[21] Whitelam G C, Devlin P F.Roles of different phytochromes in Arabidopsis photomorphogenesis.Plant Cell Environ, 1997, 20:752-758
[22] Casal J J, Luccioni L G, Oliverio K A, Boccalandro H E.Light,phytochrome signalling and photomorphogenesis in Arabidopsis.Photochem Photobiol Sci, 2003, 2: 625-636
[23] Clough R C, Vierstra R D.Phytochrome degradation.Plant Cell Environ, 1997, 20: 713-721
[24] Nagy F, Schäfer E.Phytochromes control photomorphogenesis by differentially regulated, interacting signaling pathways in higher plants.Annu Rev Plant Biol, 53: 329-355
[25] Sharrock R A, Clack T.Patterns of expression and normalized levels of the five Arabidopsis phytochromes.Plant Physiol, 2002,130: 442-456
[26] Johnson E, Bradley M, Harberd N P, Whitelam G C.Photoresponses of light-grown phyA mutants of Arabidopsis (phytochrome A is required for the perception of daylength extensions).Plant Physiol, 1994, 105: 141-149
[27] Yanovsky M J, Kay S A.Molecular basis of seasonal time measurement in Arabidopsis.Nature, 2002, 419: 308-312
[28] Yanovsky M J, Casal J J, Whitelam G C.Phytochrome A, phytochrome B and HY4 are involved in hypocotyl growth responses to natural radiation in Arabidopsis: weak de-etiolation of the phyA mutant under dense canopies.Plant Cell Environ, 1995, 18:788-794
[29] Casal J J.Phytochrome A enhances the promotion of hypocotyl growth caused by reductions in levels of phytochrome B in its far-red-light-absorbing form in light-grown Arabidopsis thaliana.Plant Physiol, 1996, 112: 965-973
[30] Yanovsky M J, Alconada-Magliano T M, Mazzella M A, Gatz C, Thomas B, Casal J J.Phytochrome A affects stem growth,anthocyanin synthesis, sucrose-phosphate-synthase activity and neighbour detection in sunlight-grown potato.Planta,1998, 205: 235-241
[31] Takano M, Inagaki N, Xie X, Yuzurihara N, Hihara F, Ishizuka T,Yano M, Nishimura M, Miyao A, Hirochika H, Shinomura T.Distinct and cooperative functions of phytochromes A, B, and C in the control of deetiolation and flowering in rice.Plant Cell,2005, 17: 3311-3325
[32] Garg A K, Sawers R J H, Wang H, Kim J K, Walker J M, Brutnell T P, Wu R J.Light-regulated overexpression of an Arabidopsis phytochrome A gene in rice alters plant architecture and increases grain yield.Planta, 2006, 223: 627-636
[33] Thiele A, Herold M, Lenk I, Quail P H, Gatz C.Heterologous expression of Arabidopsis phytochrome B in transgenic potato influences photosynthetic performance and tuber development.Plant Physiol, 1999, 120: 73-82
[34] Heyer A G, Mozley D, Landschutze V, Thomas B, Gatz C.Function of phytochrome A in potato plants as revealed through the study of transgenic plants.Plant Physiol, 1995, 109: 53-61
[35] Boccalandro H E, Ploschuk E L, Yanovsky M J, Sánchez R A,Gatz C, Casal J J.Increased phytochrome B alleviates density effects on tuber yield of field potato crops.Plant Physiol, 2003,133: 1539-1546
[36] Robson P R, McCormac A C, Irvine A S, Smith H.Genetic engineering of harvest index in tobacco through overexpression of a phytochrome gene.Nat Biotechnol, 1996, 14: 995-998
[37] Chen A, Li C X, Hu W, Lau M Y, Lin H Q, Rockwell N C, Martin S S, Jernstedt J A, Lagarias J C, Dubcovsky J, Lagarias J C.PHYTOCHROME C plays a major role in the acceleration of wheat flowering under long-day photoperiod.Proc Natl Acad Sci USA, 2014, 111: 10037-10044
[38] Mathews S, Sharrock R A.The phytochrome gene family in grasses (Poaceae): a phylogeny and evidence that grasses have a subset of the loci found in dicot angiosperms.Mol Biol Evol,1996, 13: 1141-1150
[39] Mathews S, Sharrock R A.Phytochrome gene diversity.Plant Cell Environ, 1997, 20: 666-671
[40] Liu X B, Zhang X Y, Wang Y X, Sui Y Y, Zhang S L, Herbert S J,Ding G.Soil degradation: a problem threatening the sustainable development of agriculture in Northeast China.Plant Soil Environ, 2010, 56: 87-97
[41] Gao Y, Jiang W, Dai Y, Xiao N, Zhang C, Li H, Lu Y, Wu M, Tao X, Deng D, Chen J.A maize phytochrome interacting factor 3 improves drought and salt stress tolerance in rice.Plant Mol Biol,2015, 87: 413-428
[42] Rajeevan M S, Ranamukhaarachi D G, Vernon S D, Unger E R.Use of real-time quantitative PCR to validate the results of cDNA array and differential display PCR technologies.Methods, 2001, 25: 443-451
[43] Sheehan M J, Farmer P R, Brutnell T P.Structure and expression of maize phytochrome family homeologs.Genetics, 2004, 167:1395-1405
[44] Markelz N H, Costich D E, Brutnell T P.Photomorphogenic responses in maize seedling development.Plant Physiol, 2004,133: 1578-1591
[45] Basu D, Dehesh K, Schneider-Poetsch H J, Harrington S E,McCouch S R, Quail P H.Rice PHYC gene: structure, expression,map position and evolution.Plant Mol Biol, 2000, 44: 27-42
[46] Childs K L, Miller F R, Cordonnier-Pratt M M, Pratt L H, Morgan P W, Mullet J E.The sorghum photoperiod sensitivity gene,Ma3, encodes a phytochrome B.Plant Physiol, 1997, 113:611-619
[47] Gaut B S.Patterns of chromosomal duplication in maize and their implications for comparative maps of the grasses.Genome Res,2001, 11: 55-66
[48] Gaut B S, Doebley J F.DNA sequence evidence for the segmental allotetraploid origin of maize.Proc Natl Acad Sci USA, 1997,94: 6809-6814
[49] Wilson W A, Harrington S E, Woodman W L, Lee M, Sorrells M E, McCouch S R.Inferences on the genome structure of progenitor maize through comparative analysis of rice, maize and the domesticated panicoids.Genetics, 1999, 153: 453-473
[50] Sheehan M J, Kennedy L M, Costich D E, Lee M, Sorrells M E,McCouch S R.Subfunctionalization of PhyB1 and PhyB2 in the control of seedling and mature plant traits in maize.Plant J, 2007,49: 338-353
[51] Boylan M T, Quail P H.Oat phytochrome is biologically active in transgenic tomatoes.Plant Cell, 1989, 1: 765-773
[52] Nagatani A, Kay S A, Deak M, Chua N H, Furuya M.Rice type I phytochrome regulates hypocotyl elongation in transgenic tobacco seedlings.Proc Natl Acad Sci USA, 1991, 88: 5207-5211
[53] Takano M, Kanegae H, Shinomura T, Miyao A, Hirochika H, Furuya, M.Isolation and characterization of rice phytochrome A mutants.Plant Cell, 2001, 13: 521-534
[54] Sawers R J H, Linley P J, Farmer P R, Hanley N P, Costich D E,Terry M J, Brutnell T P.Elongated mesocotyl1, a phytochromedeficient mutant of maize.Plant Physiol, 2002, 130: 155-163
[55] Halliday K J, Salter M G, Thingnaes E, Whitelam G C.Phytochrome control of flowering is temperature sensitive and correlates with expression of the floral integrator FT.Plant J, 2003, 33:875-885
[56] Asami O, Hironori I, Kyoko I K, Takano M, Izawa T.Molecular dissection of the roles of phytochrome in photoperiodic flowering in rice.Plant Physiol, 2011, 157: 1128-1137
Transcription Characteristics of ZmPHYA1 and ZmPHYA2 under Different Light Treatments in Maize
YANG Zong-Ju1,2,**, YAN Lei2,3,**, SONG Mei-Fang2,4, SU Liang2, MENG Fan-Hua2, LI Hong-Dan1,2, BAI Jian-Rong5, GUO Lin2,*, and YANG Jian-Ping2,*1Graduate School of Chinese Academy of Agricultural Sciences, Beijing 100081, China;2Institute of Crop Science, Chinese Academy of Agricultural Sciences, Beijing 100081, China;3College of Biology Engineering, Shanxi University, Taiyuan 030006, China;4Beijing Radiation Center, Beijing 100875, China;5Institute of Crop Science, Shanxi Academy of Agricultural Sciences, Taiyuan 030031, China
Plant phytochromes are a family of red/far-red light photoreceptors, which have two forms in plant: inactive red light absorbing form (Pr) and active far-red light absorbing form (Pfr).During plant growth and developmental processes, phytochromes playpivotal roles in regulations of seed germination, plant height, flowering time, and shade-avoidance.In the grasses, three subfamilies are present: PHYA, PHYB and PHYC.In maize, an ancient genome duplication has increased the family member to six: PHYA1,PHYA2, PHYB1, PHYB2, PHYC1, and PHYC2.Phytochrome A facilitates the inhibition of hypocotyl elongation, opening of the apical hook, expansion of cotyledons, accumulation of anthocyanin and blocking of greening by continuous FR (FRc) light.In order to evaluate the light response capability and difference of transcription abundance between ZmPHYA1 and ZmPHYA2, we employed quantitative real-time PCR (qRT-PCR) assay to investigate the expression patterns of ZmPHYA1 and ZmPHYA2 in the inbred line B73 and Mo17 with different light treatments.The results indicated that both ZmPHYA1 and ZmPHYA2 had a high expression level in leaf and silk, and the transcription abundance of ZmPHYA1 was 2-8 times higher than that of ZmPHYA2.Inbred lines of both B73 and Mo17 possessed longer mesocotyls in dark, far-red and blue light conditions than in red or white light conditions.Both ZmPHYA1 and ZmPHYA2 had a high expression level in far-red and blue lights and rapidly responded to dark-to-far-red and dark-to-blue transitions.ZmPHYA1 was more important under far-red light, so was ZmPHYA2 in blue light.Both of the genes could rapidly respond to transitions from dark to red or white light with similar expression pattern.The both genes also respond to long-day or short-day treatments,however the transcription abundance of ZmPHYA1 was 2-5 times higher than that of ZmPHYA2 during the treatments.All the results suggested that the transcription of both ZmPHYA1 and ZmPHYA2 could rapidly responded to different light treatments; ZmPHYA1 might be more effective than ZmPHYA2 in crop improvement.Our results provide a theoretical basis for the function study and evaluation of light response ability for both ZmPHYA1 and ZmPHYA2.
Maize; Phytochrome; Light signaling transduction; Expression analysis; Light treatment
10.3724/SP.J.1006.2016.01462
本研究由国家重点研发计划试点专项(SQ2016ZY03002918), 国家转基因生物新品种培育重大专项(2016ZX08010002-003-002), 北京市自然科学基金(重点)项目(6151002)和中国农业科学院科技创新工程项目资助。
This study was supported by the National Research and Development Program (SQ2016ZY03002918), the Genetically Modified Organisms Breeding Major Projects of China (2016ZX08010002-003-002), the Key Project of Beijing Natural Science Foundation (6151002) and the Agricultural Science and Technology Innovation Program of CAAS.
(Corresponding authors): 郭林, E-mail: guolin@caas.cn, Tel: 010-82105851; 杨建平, E-mail: yangjianping02@caas.cn, Tel/Fax:010-82105859
**同等贡献(Contributed equally to this work)
联系方式: 杨宗举, E-mail: zongjuyang@163.com; 闫蕾, E-mail: yanlei2723@126.com, Tel: 010-82105851
Received(): 2016-02-06; Accepted(接受日期): 2016-05-09; Published online(网络出版日期): 2016-06-06.
URL: http://www.cnki.net/kcms/detail/11.1809.S.20160606.0855.004.html
