珍稀植物海南草海桐基于ITS序列分析的系统位置研究*
2016-06-05何思宁李炜曦谈凤笑黄椰林
粟 米,何思宁,李炜曦,王 舒,谈凤笑,黄椰林
(1. 中山大学生命科学学院∥广东省植物资源重点实验室,广东 广州510275;2. 华南农业大学资源环境学院,广东 广州 510642)
珍稀植物海南草海桐基于ITS序列分析的系统位置研究*
粟 米1,何思宁1,李炜曦2,王 舒2,谈凤笑2,黄椰林1
(1. 中山大学生命科学学院∥广东省植物资源重点实验室,广东 广州510275;2. 华南农业大学资源环境学院,广东 广州 510642)
通过测定来自国内5个分布地点的海南草海桐ScaevolahainanensisHance群体核核糖体基因间隔区序列(nuclear ribosomal internal transcribed spacer region,ITS),一方面评估海南草海桐的遗传多样性大小,另一方面,结合GenBank中已有的草海桐属物种ITS序列来共同进行分子系统发育分析,旨在确定海南草海桐在草海桐属中的系统位置。研究结果表明海南草海桐遗传多样性水平极低,作为一种珍稀植物在分子水平上同样面临灭绝的风险。而分子聚类结果发现海南草海桐位于Scaevola组里的北半球谱系,并与夏威夷本地物种的亲缘关系最近,两地草海桐属物种之间可能存在祖先—后代(progenitor-derivative)的扩散模式。研究结果同时否定了Scaevola组的单系性以及海南草海桐由同域广布种草海桐演化而来的假说。
海南草海桐ScaevolahainanensisHance;ITS;系统发育分析
海南草海桐ScaevolahainanensisHance,又名小草海桐,属于草海桐科Goodeniaceae草海桐属Scaevola。该属全世界大约130个种,该属的起源以及现存物种都集中在澳大利亚。分布在澳大利亚以外的大约有40个种,其中草海桐S.taccada和非洲草海桐S.plumieri均为热带广布种,其余物种大多零散分布在热带海岛地区[1],仅海南草海桐是唯一的一种澳大利亚以外沿大陆沿岸狭域分布的物种,分布于中国南部沿海地区以及越南与中国相邻的北部湾地区[1],生于海边盐田或与红树林同生,该地区同域分布的同属植物只有草海桐[2]。从海南草海桐的分类学研究历史来看,海南草海桐学名的命名者Hance于1878年命此学名,也就是以采集地海南岛来命名种小名,但命名当时,以及往后的一段时日,包括命名者在内的分类学者都不大确定其是否可放在草海桐这一属[3]。1915年日本植物学家早田文藏解剖来自台湾的海南草海桐标本的花朵之后,强烈主张其属于草海桐属无误[3],但随后的几十年里,对该种的研究限于停顿,直到Carolin[4]于1990年在研究草海桐属的系统分类时,在没有见到该种标本的情况下,按前人的描述将海南草海桐归到了Scaevola组,但他承认其植株形态和果实等特点并不完全与Scaevola组的特点相符合;2003年Howarth等[1]利用ITS分子数据对草海桐属的系统分类进行了研究,尤其是对分布在澳大利亚以外的所有该属物种的演化进行了深入探讨,这也是唯一的关于草海桐属分子系统学的报道,但唯独就缺少海南草海桐的样品和数据。作为只限于在中国南部和越南沿岸分布的一种本地草海桐属植物,海南草海桐是否是由本地区与之同域分布,但同时又是泛热带广布的唯一一种草海桐属植物——草海桐演化而来?该问题是Howarth在他们研究中所提出的但尚未有数据验证的一个假说[1];此外,由于近年来海岸生态系统的破坏,目前海南草海桐的分布范围不断缩减,种群数量极其稀少,以前有海南草海桐记录分布的区域如今大多数难以寻找踪迹。依据世界自然保育联盟(IUCN)的保育等级分类标准该种已被台湾列为珍稀濒危种(Critically Endangered,CR)[5],在大陆也被列为珍稀易危植物(Vulnerable species,VU)[6]。针对以上问题,本研究将测定来自国内5个分布地点的海南草海桐的群体ITS序列,一方面评估该物种在物种水平和群体水平上的遗传多样性,另一方面,结合GenBank中已有的且唯一可获得的草海桐属各物种ITS序列来共同进行分子系统发育分析,旨在确定海南草海桐在草海桐属中的系统位置。
1 材料和方法
1.1 实验材料
从国内南部沿海地区(包括广东、广西、台湾、和海南)采集了5个海南草海桐的群体,共50个个体(表1)。同时在广东和台湾各采集草海桐个体6个,采样时用GPS定位仪记录经纬度,严格按照群体采样原则,个体间距离不小于5 m,叶片置于密封的硅胶袋中干燥保存,凭证标本存放于中山大学生物适应性进化研究室。
1.2 方法
1.2.1 植物总DNA提取 采用改进的CTAB法[7]用紫外分光光度法测定其核酸含量以及吸光比值。用琼脂糖电泳检测其含量。
1.2.2 ITS 序列的扩增及测序 本研究所用ITS引物(ITS4,ITSLeu)同Howarth(2003)(上海生工合成)[1]。反应体系为30 μL,包括DNA 20~40 ng,引物0.15 μmol/L,MgCl21.5 mol/L,dNTP 0.1 mmol/L,TaqDNA聚合酶1.5 U。扩增程序为:94 ℃,4 min → 94 ℃,45 s → 60 ℃,50 s→72 ℃,80 s →32个循环→72 ℃,6 min。PCR产物进行电泳检测后,送华大基因公司进行双向测序。
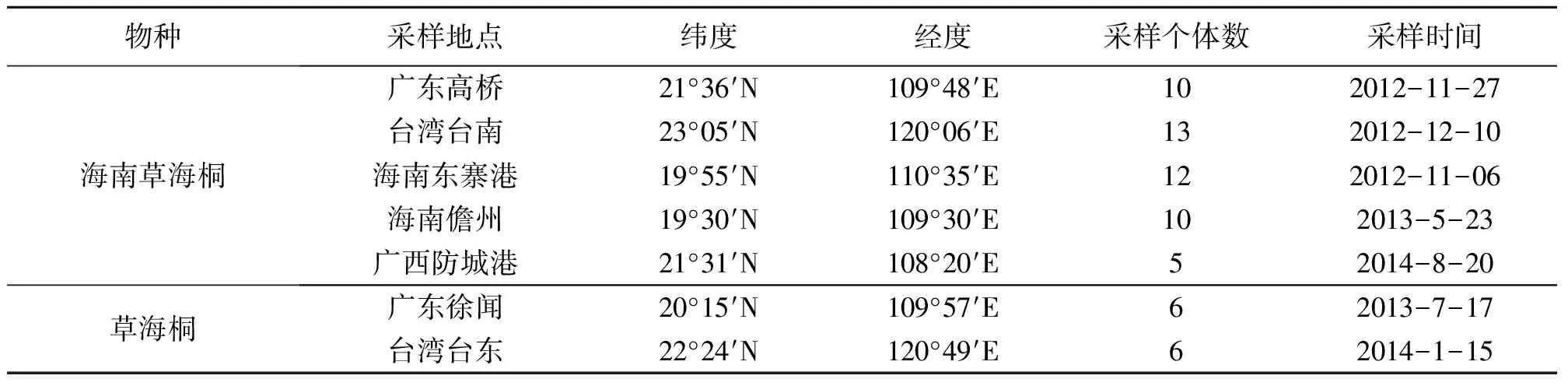
表1 实验材料采集信息
1.2.3 数据分析 首先采用Clustal-X软件[8]和POPGENE[9]对所测得的海南草海桐和草海桐群体ITS序列进行对位排列和多样性检测,在获得相应的单倍型信息后,用Clustal X软件将本研究所测序列与GenBank已有的66个草海桐属及其相关物种的ITS序列(见表2)共69个序列进行共同排序,再用PAUP 4.0 b10 软件[10]进行最大简约分析,外类群选取参照Howarth[1]研究中的设置,简约法构树中空位(gap)处理作缺失(missing)状态。最大简约树(Maximum parsimony:MP)采用Heuristic法寻找。用bootstrap 值检验简约树中各分支的置信度,自展数据集为1 000次。此外,该数据集也采用贝叶斯法推断法(Bayesian inference:BI)进行构树,采用MrBayes3.1.1软件[11],用MODELTEST 3.7[12]选择最适模型。模型选择的标准基于贝叶斯信息准则(Bayes information criterion,BIC)[13]。所有的贝叶斯分析均以随机树作为起始树,4条链(3条热链 1条冷链)同时运行5×106代,每1 000代对系统树抽样一次,其它参数的先验值设定为默认值。为了进一步获得分辨率更高的系统树,排除差异较大的序列的分析干扰,我们在前面大数据集构树结果的基础上选取部分序列(共27个序列)构成小数据集,并采用同样的两种分析方法进行构树,后者将Scaevolataccada和S.repens作为外类群。
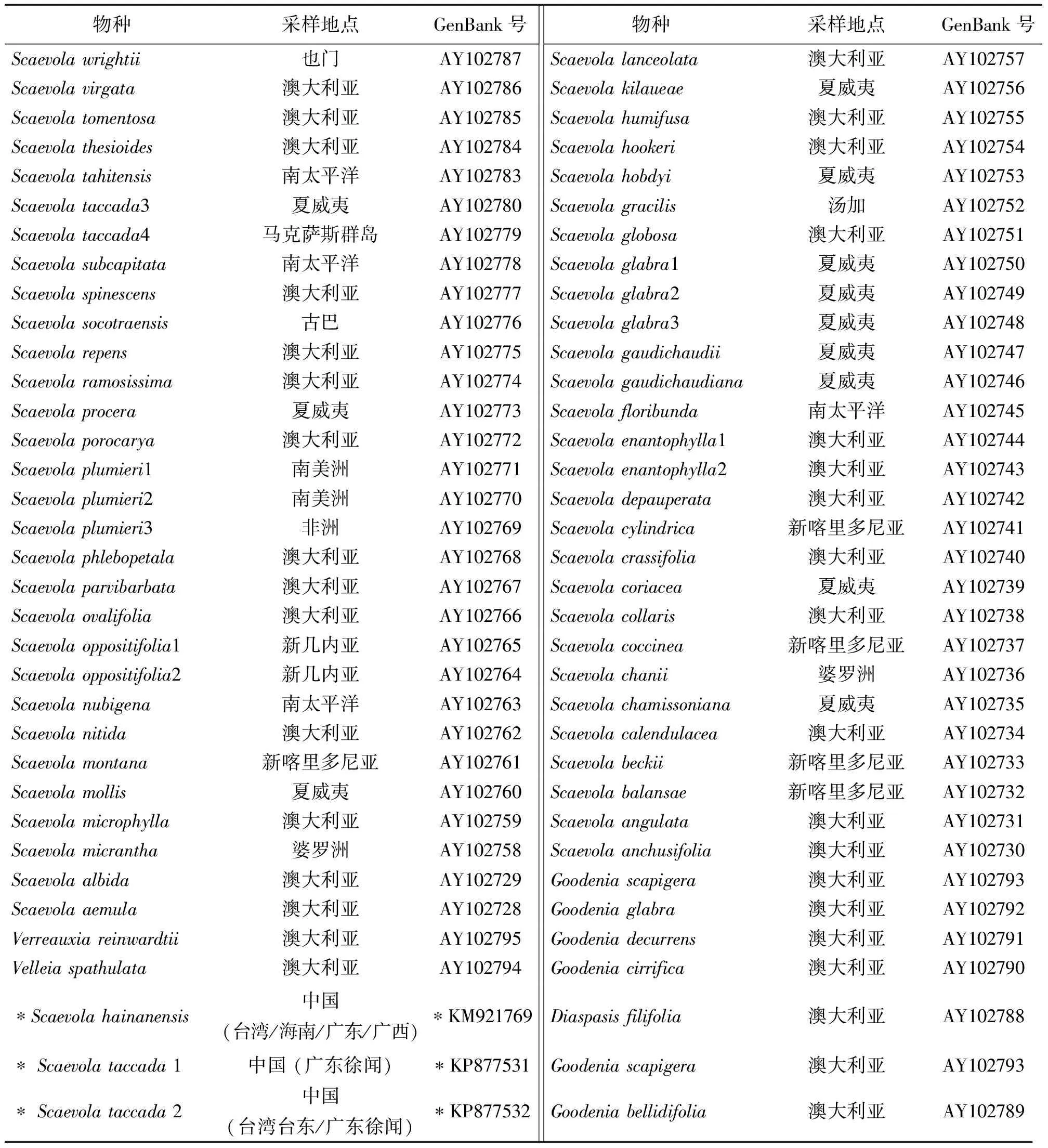
表2 本研究所用序列的物种样品信息及GenBank号(*代表自测序列)
2 结果与分析
2.1 基于ITS序列的海南草海桐群体遗传多样性检测
本研究测定的海南草海桐5个种群共50个个体的ITS序列完全一致,具有唯一的单倍型和基因型,Nei遗传多样性指数为零,因此提交给GenBank唯一的登录号KM921769。而本研究测定的草海桐2个群体共12个个体共含有两种单倍型,登录号分别为KP877531和KP877532,对应的基因型共3种。其中广东徐闻群体同时存在2种单倍型和3种基因型,Nei遗传多样性指数为0.53。遗传多样性检测结果表明:即使所检测的草海桐群体和个体数均显著少于海南草海桐,从遗传多样性水平上也明显高于海南草海桐。这一检测结果同 Ho等[14]针对海南草海桐台湾群体的ISSR研究结果一致,同样揭示了海南草海桐无论在群体水平还是在物种水平上的遗传多样性均极低。
2.2 基于大数据集(69个序列)的ITS序列分析结果
69个草海桐属及其外类群物种序列(加入本研究所获得的海南草海桐和中国分布草海桐的序列在内)的ITS全序列排序后的长度为803 bp,用PAUP 4.0 b10进行运算。有335个变异位点,243个为信息位点,分别占41.7%和30.2%。Heuristic 法找到90个最简约树,用严格一致性树(strict consensus tree)进行系统分析。树长为370,一致性指数(CI)和保持指数(RI)分别为0.629 3和0.830 2。MP树图与使用贝叶斯推断法分析构建的系统树基本一致,但各分支的支持率都较低,因此图1未显示MP树,只显示了具有较高分辨率的贝叶斯树(BI)。实际上,在分析比较中考虑到各种之间较大的序列差异,采用复杂模型的贝叶斯法更为合理,构树所对应的核苷酸模型按贝叶斯信息准则为GTR+I+G。从BI图中可以得知,4个草海桐S.taccada个体序列聚在一起(支持率为100%),1和2来自中国广东和台湾,3来自夏威夷,4来自太平洋中的Marquesas Islands。同域分布的草海桐和海南草海桐并不构成姐妹群关系,否定了海南草海桐是由该地区另一物种草海桐演化而来的设想。与之相对比的是,海南草海桐在系统树上被归为Howarth等建议的北半球谱系(Northern Hemisphere lineage)[1],该分支包括非洲草海桐S.plumieri,S.socotraensis,S.wrightii,S.nitida和分布在夏威夷的8个本地种,但支持率只有0.53,除夏威夷本地物种形成单系类群外,北半球谱系内部各分支间均为平行关系。该分支网结于Howarth等所划出的Subglobulifera类群(支持率为0.98),该类群包括了大多数属于S. sect.Xerocarpaser.Globuliferae的澳大利亚物种以及来自于婆罗洲的物种。
2.3 基于小数据集(27个序列)的ITS序列分析结果
确定了海南草海桐以高支持率属于Subglobulifera类群后,为了进一步确定海南草海桐在Subglobulifera类群中的位置,排除大数据集内差异较大物种在系统分析上的干扰,将Subglobulifera类群内的所有25个物种(包括海南草海桐, 见图1),加上该类群之外的S.taccada和S.repens作为外类群,一共27个序列重新排序以进行系统发育分析。由于去除了序列差异极大的物种所带来的信号干扰,两种方法都获得了分辨率和支持率得到提高的系统发育树。其中图2为采用bootstrap法抽样检验所得的一致性MP树,图中可以看到海南草海桐与夏威夷的8个本地物种构成姐妹群关系,且有较高的自展支持率:70%。使用贝叶斯法构建的BI树见图3,分辨率更高,Howarth等建议的北半球谱系(已标出)支持率为67%,在此谱系中,海南草海桐与夏威夷各物种所形成的分枝支持率为83%。
3 讨 论
海南草海桐是唯一分布在澳大利亚以外沿大陆沿岸狭域分布的草海桐属物种,仅分布于中国南部沿海地区以及越南与中国相邻的北部湾地区,但近几十年种群数量急剧减少,Ho等[14]的研究就指出海南草海桐在台湾历来的栖息地,如云林县、嘉义县及澎湖地区均已不见踪迹,如今只在台南将军乡剩最后一个群体,因此在台湾已被列为珍稀濒危种(CR)[5]。在大陆曾经有海南草海桐记录分布的区域如今大多数也难以寻找踪迹,因此该种也已被列为珍稀易危种(VU)[6]。我们在华南沿海的采样过程同样见证了海南草海桐种群的消失。海南草海桐濒危的原因前人推测是由于体型不高,本身不能被遮光,生态幅度窄隘,因而在海岸大规模开发过程中逐渐消失[3]。本研究通过检测来自国内五个分布地点的海南草海桐的群体ITS序列,揭示了海南草海桐在遗传多样性上同样面临灭绝的风险,尽管这只是基于一种分子标记——ITS序列的检测结果,但ITS序列在草海桐属的分子研究中被普遍认为是适合的分子标记[1]。此外,在对比研究中,即使所检测的同域草海桐群体和个体数均显著少于海南草海桐,但草海桐在ITS序列上也显示出更高的遗传多样性水平。该遗传多样性检测结果也与Ho等[14]利用ISSR对台湾海南草海桐群体进行研究的结果相一致,在他们所获得的233个条带中,只发现6个多态性条带。海南草海桐遗传多样性极低的检测结果将为制定这一珍稀植物的保育策略提供重要信息。
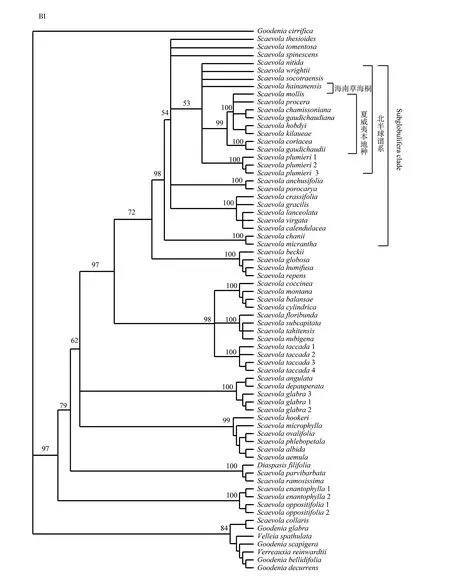
图1 基于69个ITS序列(大数据集)所构建的BI树图(数字代表后验概率,只显示主要分支),图中部分物种的代表个体多于1个,因此在物种名后附加数字以示区分,相对应的物种及其个体具体地理信息见表1Fig.1 The phylogenetic tree resulting from the Bayesian inference (BI) of 69 ITS gene sequences (large dataset). Posterior probability values are given above major branches. The species name with number means more than one individual for this species were used in this study. The information for all taxa was listed in the supplementary Table 1

图2 基于27个ITS序列(小数据集)所构建的MP树(数字代表bootstrap自展支持率)Fig.2 The phylogenetic tree resulting from the Maximum parsimony (MP) analysis of 27 ITS gene sequences (small dataset). Bootstrap values are given at nodes

图3 基于27个ITS序列(小数据集)所构建的BI树(数字代表后验概率)Fig.3 The phylogenetic tree resulting from the Bayesian inference (BI) of 27 ITS gene sequences (small dataset). Posterior probability values are given at nodes
依据Carolin[15]所提出的经典的草海桐属的分类系统,该属被分成了3个组,分别是Scaevola组,Xerocarpa组和Enantiophyllum组。后两组主要由分布在澳大利亚的物种所组成,包括了130个种中的大多数物种(约90种),形态上主要以干外果皮、蔓生草本和灌木为最重要特征。而分布在澳大利亚以外的物种绝大部分被归为Scaevola组,该组的主要特点是果实具肉质外果皮,植株常为高大的灌木甚至是乔木。Carolin[4]于1990年在研究草海桐属的系统分类中,在没有见到海南草海桐标本的情况下,按前人的描述将该种归到了Scaevola组,但他承认其植株形态和果实等特点并不完全与Scaevola组的特点相符合;实际上按朱芳莹[16]以及我们的观察,海南草海桐实为蔓生的小灌木,肉质核果成熟后变干。但Howarth[1]在他们研究中发现Carolin所提出的3个组除Enantiophyllum组外都不为单系类群,因此单凭形态证据就对海南草海桐进行系统位置的确定是不太准确的。本研究基于ITS序列所得出的聚类结果发现海南草海桐位于Scaevola组里的一个分支谱系——北半球谱系(Northern Hemisphere lineage)。在Howarth[1]的研究中该谱系包括S.plumieri,S.socotraensis,S.wrightii,S.nitida和分布在夏威夷的8个本地种,但他的研究中未包括海南草海桐。本研究的结果支持海南草海桐属于这一谱系,但该谱系总体的支持率并不高,在MP树上支持率小于50%,在BI树上支持率为67%, 在Howarth的研究中该谱系支持率同样较低[1]。Howarth[1]认为是由于来自于澳大利亚的物种S.nitida的数据异常造成的,因此随后将S.nitida排除在北半球谱系外。从北半球谱系所包括的几个物种来看,分布地点分别是也门索科特拉岛S.socotraensis,古巴S.wrightii,夏威夷(标记的7个本地种),中国越南沿岸(海南草海桐),相互之间地理距离相隔极远。而该谱系中唯一的广布种S.plumieri尽管广布在热带美洲和非洲,但除了古巴外并未与北半球谱系其它物种在分布范围上存在交集,因此Howarth[1]在研究中认为北半球谱系的祖先仍然位于澳大利亚,而现存的各个狭域物种均来自于鸟类对肉质果实的长距离扩散和随后的本地适应,而狭域物种之间同样存在与扩散相对应的祖先—后代(progenitor-derivative)形成模式,但广布种的扩散更依赖于洋流。此外,分子系统发育树显示依据形态特征被归入Scaevola组里的草海桐,尽管与海南草海桐同域分布,却在系统位置上相隔较远,这一结果否定了Scaevola组的单系性以及海南草海桐最有可能由草海桐演化而来的假说。这与前人针对夏威夷群岛草海桐属的研究结果相一致。他们的研究证实:在夏威夷群岛分布的草海桐,也并未参与演化形成夏威夷的其他本地种[17]。本研究中,4个分布在全球不同地域的草海桐个体形成单系类群,在分子系统树上获得的极高支持(100%)。这一结果进一步反映了该广布种并未在分布的不同地点独立演化而表现出遗传上的分化。
本研究基于ITS序列所得出的聚类结果还进一步揭示了海南草海桐与夏威夷本地种的紧密关系,尤其是与S.coriacea和S.gaudichaudii这两个本地种在系统树上组成的分支最近[1]。在形态上海南草海桐与其中S.coriacea最接近。二者均为匍匐肉质小灌木,茎均光滑,叶肉质,叶基不抱茎,花单生或呈简单的总状花序;苞叶与叶大小相近,小苞片生于花梗顶端,萼片波状缘,花冠筒内部有毛,花柱下部有短毛;果实光滑无毛[2]。但相比海南草海桐的腋生花,S.coriacea的花是顶生的;S.gaudichaudii在形态特点上同前两者大多类似,与S.coriacea和海南草海桐所不同的只是其花冠筒内部无毛。从生境来看,海南草海桐生长在较为湿润低海拔的海边湿地,而S.coriacea生长在海边较干燥的沙丘,S.gaudichaudi则长在更为干燥和高海拔的山脊和灌木丛林中[17],这种从海边到内陆的生境变化反映了新种产生所依赖的生态位的必要性,而亚洲与夏威夷之间鸟类的迁移和对果实的长距离扩散则从另一个方面解释了两地植物存在祖先-后代(progenitor-derivative)扩散模式的可能性,这一推测与Howarth对草海桐属北半球谱系其它物种在起源和适应上的讨论高度一致[1],这在一定程度上支持了海南草海桐在系统上位于北半球谱系,并更靠近夏威夷本地物种的结论。针对海南草海桐的系统发育研究,下一步将尽可能采用多基因数据联合分析法[18],尤其是加入叶绿体基因的数据进行研究[19]。但由于草海桐属物种分布范围极广,采样受限以及前人所测的基因数据只限于ITS序列,故短期内无法完成ITS外的其他基因的分析。今后应进一步加大采集力度,收集其他基因数据,以进一步验证海南草海桐的系统位置,同时也为草海桐属的起源和进化提供重要信息。
[1] HOWARTH D G,GUSTAFSSON M H G,BAUM D A,et al. Phylogenetics of the genusScaevola(Goodeniaceae):Implication for dispersal patterns across the Pacific basin and colonization of the Hawaiian Islands [J]. American Journal of Botany,2003,90(6):915-923.
[2] 中国科学院植物志编委. 中国植物志 [M]. 北京:科学出版社,1983:179.
[3] 陈玉峰. 台湾生态大变迁之海南草海桐 [J/OL]. 生态台湾电子季刊,2007(15). [2015-02-18]. http:∥ecology.org.tw/publication/15/issue15-3.htm.
[4] CAROLIN R C. Nomenclatural notes,new taxa and the systematic arrangement in the genusScaevola(Goodeniaceae) including synonyms [J]. Telopea,1990,3:477-516.
[5] 吕勝由,邱文良. 台湾稀有及濒危植物之分级彩色图鉴(III)[M]. 台北:行政院农业委员会,1998:143.
[6] 邢福武. 中国的珍稀植物 [M]. 长沙:湖南教育出版社,2005:216.
[7] DOYLE J J. DNA protocols for plants—CTAB total DNA isolation [M] ∥ HEWITT G M, JOHNSTON A, et al. Molecular techniques in taxonomy. Berlin:Springer-Verlag,1991:283-293.
[8] THOMPSON J D,GIBSON T J,PLEWNIAK F,et al. The Clustal X windows interface:flexible strategies for multiple sequences alignment aided by quality analysis tools [J]. Nucleic Acids Research,1997,25:4876-4882.
[9] YEH F C,BOYLE T J B. Population genetic analysis of codominant and dominant markers and quantitative traits [J]. Belgian Journal of Botany,1997,129:157.
[10] SWOFFORD D L. PAUP*:Phylogenetic analysis using Parsimony (and other methods) [M]. 4th ed. Sunderland:Sinauer Associates,2002.
[11] RONQUIST F,HUELSENBECK J P. MRBAYES 3:Bayesian phylogenetic inference under mixed models [J]. Bioinformatics,2003,19:1572-1574.
[12] POSADA D,CRANDALL K A. Modeltest:testing the model of DNA substitution [J]. Bioinformatics,1998,14:817-818.
[13] POSADA D,BUCKLEY T R. Model selection and model averaging in phylogenetics:advantages of akaike information criterion and Bayesian approaches over likelihood ratio tests [J]. Systematic Biology,2004,53:793-808.
[14] HO K Y,DENG S L,CHANG Y H,et al. Genetic variation of the endangeredScaevolahainanensis(Goodeniaceae) in the Jiangjun stream mouth,Taiwan [J]. Taiwan Journal of Forest Science,2005,20 (3):193-202.
[15] CAROLIN R C,MORRISION D A,RAJPUT T. Flora of Australia volume 35,Brunoniaceae,Goodeniaceae [M]. Canberra:Australian Government Publishing Service,1992.
[16] 朱芳莹. 海南草海桐之生殖特性与植群组成 [D]. 台南:台南大学材料科学系,2007.
[17] HOWARTH D G, BAUM D A,Genealogical evidence of homoploid hybrid speciation in an adaptive radiation ofScaevola(Goodeniaceae) in the Hawaiian Islands [J]. Evolution,2005,59(5):948-961.
[18] 陈兴汉,郭梁,李明明,等. 中国南方沿海13种石斑鱼类的分子系统进化关系分析 [J]. 中山大学学报(自然科学版),2014,53(4):123-130.
[19] 赖燕玲,王晓明,陈国培,等. 利用trnL-trnF探讨文定果属的系统地位及其与若干近缘科属的系统发育关系 [J]. 中山大学学报(自然科学版),2012,51(3):98-107.
Systematic position of a rare plant speciesScaevolahainanensisbased on ITS sequence analysis
SUMi1,HESining1,LIWeixi2,WANGSu2,TANFengxiao2,HUANGYelin1
(1. Guangdong Provincial Key Laboratory of Plant Resources,School of Life Sciences,Sun Yat-sen University, Guangzhou 510275,China;2. South China Agricultural University, Guangzhou 510642, China)
Genetic diversity ofScaevolahainanensisHance is estimated based on the sequences of nuclear ribosomal internal transcribed spacer region (ITS) from five populations distributed in China. Further, phylogenetic analysis of theS.hainanensisis performed based on combined ITS sequences data with other species ofScaevolaand outgroups available from GenBank. The results shows that the genetic diversity ofS.hainanensisis very low, implying that it is at risk of extinction at the genetic level as a rare plant. In the phylogenetic tree,S.hainanensisis located in the Northern Hemisphere lineage and closest to the clade consisting of local species in Hawaii with high support value, which imply the progenitor-derivative dispersal pattern ofScaevolaspecies between Asia and Hawaii. The results did not support the monophyly ofS. sectScaevolaand the hypothesis thatS.hainanensisis derived from a widespread and sympatric speciesS.taccadais rejected based on the present result.
ScaevolahainanesisHance;ITS;phylogenetic analysis
10.13471/j.cnki.acta.snus.2016.01.003
2015-03-11
国家自然科学基金资助项目(41276107,31200466,J1310025)
粟米(1994年生),女;研究方向:生态与进化基因组;通讯作者:黄椰林;E-mail:lsshyl@mail.sysu.edu.cn
Q949.783
A
0529-6579(2016)01-0016-08
