不同好氧颗粒污泥中微生物群落结构特点
2016-05-27侯爱月王昌稳岳耀冬赵昕燕张彦灼北京工业大学水质科学与水环境恢复工程北京市重点实验室北京004临沂大学资源环境学院山东省水土保持与环境保育重点实验室山东临沂76005
侯爱月,李 军*,王昌稳,岳耀冬,赵昕燕,卞 伟,张彦灼(.北京工业大学水质科学与水环境恢复工程北京市重点实验室,北京 004;.临沂大学资源环境学院,山东省水土保持与环境保育重点实验室,山东 临沂76005)
不同好氧颗粒污泥中微生物群落结构特点
侯爱月1,李 军1*,王昌稳2,岳耀冬1,赵昕燕1,卞 伟1,张彦灼1(1.北京工业大学水质科学与水环境恢复工程北京市重点实验室,北京 100124;2.临沂大学资源环境学院,山东省水土保持与环境保育重点实验室,山东 临沂276005)
摘要:为了探讨活性污泥好氧颗粒化过程对微生物种群的影响、不同底物及不同颗粒化方法培养的好氧颗粒污泥中微生物群落结构的差异,以接种污泥、模拟废水好氧颗粒污泥和分别投加粉末活性炭和硅藻土的实际生活污水好氧颗粒污泥为研究对象,利用PCR-DGGE对比分析了接种污泥和好氧颗粒污泥中的微生物群落结构.结果表明:活性污泥好氧颗粒化过程会减少微生物种群多样性,影响颗粒污泥稳定性的细菌被淘汰,而聚磷菌、反硝化菌、难降解有机物降解菌等污水处理功能微生物都在颗粒化过程中得到保留.活性污泥好氧颗粒化过程中能够实现亚硝化细菌(AOB)一定程度的富集.与接种活性污泥相比,好氧颗粒污泥中AOB的多样性指数与均匀性指数均有提高.好氧颗粒污泥中的优势菌群主要分布于变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和未培养菌(uncultured bacterium).其中AOB均属于β-Proteobacteria的亚硝化单胞菌属(Nitrosomonas).
关键词:好氧颗粒污泥;微生物群落结构;PCR-DGGE
* 责任作者, 教授, jglijun@bjut.edu.cn
好氧颗粒污泥具有沉降性能好、污泥密度大、生物量高、抗冲击负荷和耐有毒物质的优势.目前好氧颗粒污泥的应用研究多集中在高浓度有机废水,富含有毒有害污染物废水以及生活污水的处理中[1-3].好氧颗粒污泥内部不同的微环境能够为各功能微生物提供适宜的生存条件,应用于生活污水处理时,异养菌、硝化细菌和反硝化细菌协同作用,可以较好的实现碳氮的同步去除.以典型低有机物浓度城市生活污水培养好氧颗粒污泥的研究还较少[4],以低有机物浓度城市生活污水快速培养好氧颗粒污泥对于该技术的应用至关重要.
好氧颗粒污泥的形成过程是一个较为复杂的过程,其形成受很多操作条件制约,比如接种污泥,底物组成,有机负荷,反应器类型和运行等.SBR的运行模式一直被广泛用来培养好氧颗粒污泥.城市生活污水的有机物浓度较低,且水质成分波动较大,活性污泥好氧颗粒化时间较长.根据颗粒污泥形成的“晶核假说[5]”并结合工程实践,在接种污泥的同时添加惰性载体,可以缩短活性污泥好氧颗粒化的时间.高阳等[6]证实在接种活性污泥的同时,投加一定量活性炭可以加速好氧颗粒污泥的颗粒化进程.硅藻土作为吸附剂已被广泛用在废水处理中.樊红辉[7]针对焦化废水,采用两级微氧颗粒污泥+硅藻土得到了较好的氨氮去除效果.硅藻土的多孔隙结构和良好吸附性能有助于活性污泥微生物的附着聚集,有助于强化好氧颗粒化过程,但是目前对这方面的研究还鲜有报道.
聚合酶链式反应—变性梯度凝胶电泳技术(PCR-DGGE),可以同时快速地对多个环境样品的微生物群落结构进行比较分析,提供微生物群落调控方面的理论依据,已成为水处理中微生物检测的常用方法之一.
本试验采用SBR反应器,通过控制沉淀时间,分别培养模拟废水好氧颗粒污泥、投加粉末活性炭的实际生活污水好氧颗粒污泥和投加硅藻土的实际生活污水好氧颗粒污泥.为了了解活性污泥好氧颗粒化过程对微生物种群的影响、不同底物及不同颗粒化方法培养出来的好氧颗粒污泥中微生物群落结构差异,以接种污泥和不同方法培养出来的好氧颗粒污泥为对象,利用PCR-DGGE技术对比分析接种污泥和好氧颗粒污泥中的微生物群落结构.
1 材料与方法
1.1 样品采集
好氧颗粒污泥的培养采用SBR反应器,高120cm,直径18cm,总体积30.5L,有效容积为25.4L,由有机玻璃制成.SBR的运行通过定时控制器实现对反应过程的控制.每天运行3个周期,每个周期包含5min进水,7.5h好氧曝气,沉淀时间由各部分研究内容确定,5min排水和5min闲置.
好氧颗粒污泥培养的接种污泥取自北京高碑店污水处理厂回流污泥,该污泥具有良好的脱氮除磷性能,MLSS为3930mg/L,SVI为157.人工配水培养好氧颗粒污泥,进水水质指标如表1所示.通过控制沉淀时间,将试验开始时的15min逐步缩短到3min,系统运行20d后出现颗粒污泥,稳定运行后COD平均去除率为94.7%,NH4+-N去除率接近100%,NO2--N积累率达到83%左右,实现稳定的短程硝化.

表1 人工模拟废水成分组成Table 1 Compositions of synthetic wastewater
实际生活污水取自北京某高校教工生活区化粪池,水质指标如表2所示.由于直接以实际生活污水为底物较难培养好氧颗粒污泥,因此考虑投加粉末活性炭(PAC)和硅藻土强化活性污泥好氧颗粒化过程.投加粉末活性炭实际生活污水好氧颗粒污泥培养过程:反应器接种活性污泥后即投加4000mg/L PAC,控制沉淀时间,将试验开始的30min初步缩短到3min,系统运行20d后形成颗粒,运行60d后,粉末活性炭好氧颗粒污泥对COD、NH4+-N的去除率分别为85.3%和87.2%;投加硅藻土实际生活污水好氧颗粒污泥培养过程为:进水水质、硅藻土投加量及沉淀时间控制条件与投加粉末活性炭过程相同,系统运行60d形成颗粒污泥,运行72d后,硅藻土好氧颗粒污泥对COD、NH4+-N去除率分别为53.2%和34.5%.
取接种污泥及各反应器中形成并稳定运行后的好氧颗粒污泥样品,分别编号为样品R1(接种污泥)、R2(模拟废水好氧颗粒污泥)、R3(投加PAC实际生活污水好氧颗粒污泥)和R4(投加硅藻土实际生活污水好氧颗粒污泥),样品采集后在-20℃下保存.
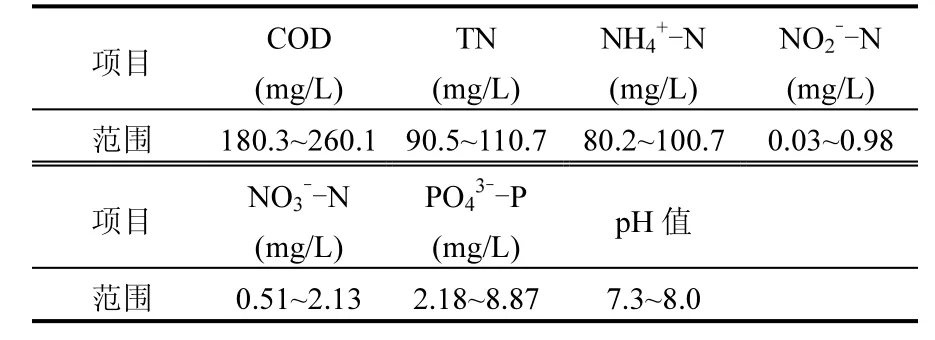
表2 实际生活污水水质Table 2 The quality of real domestic wastewater
1.2 样品总DNA提取
泥样在1.5mL离心管中静沉30min后舍去上清液,15000r/min高速离心5min后,去除上层清液,取0.3g用于总DNA提取,剩余污泥冷冻备用.采用上海生工的“Ezup柱式基因组DNA抽提试剂盒(土壤)”提取后,取5µL用1.2%琼脂糖凝胶检测.其余置于-20℃储存备用.
1.3 聚合酶链式反应(PCR)
针对总细菌的PCR扩增,以提取的细菌总DNA作为模板,采用对大多数细菌16S rRNA基因V3区都具有特异性的引物对F357-GC(5'-CGCCCGCCGCGCCCCGCGCCCGGCCCGCCGCCCCCGCCCCCCTACGGGAGGCAGCAG-3') 和R518(5'-ATTACCGCGGCTGCTGG-3').反应体系(50µL)为:10×PCR buffer 5µL,dNTP(各2.5mmol/L) 4µL,F357-GC(20µmol/L) 0.5µL, R518(20µmol/L) 0.5µL,Taq DNA聚合酶(5U) 0.5µL,DNA模板1µL,加ddH20至50µL.反应程序为:94℃预变性5min;94℃变性1min,65℃退火1min,72℃延伸1min,共进行20个循环,每个循环降低0.5℃;94℃变1min,55℃退火1min,72℃延伸1min,共进行10个循环;72℃延伸8min.
针对亚硝化细菌(AOB)的PCR扩增,采用巢式PCR扩增,第一轮扩增以提取的细菌总DNA作为模板,采用特异性引物对CTO189fA/B/C (CTO189fA:5'-GGAGAAAAGCAGGGGATCG-3'、CTO189fB:5'-GGAGGAAAGCAGGGGATCG-3'、CTO189fC:5'-GGAGGAAAGTAGGGGATCG-3')和CTO654r(5'-CTAGCYTTGTAGTTTCAAACGC-3');第二轮扩增以第一轮PCR扩增的产物为模板,采用引物对F357-GC和R518.第一轮反应体系(50µL)为:10×PCR buffer 5µL,dNTP(各2.5mmol/L)4µL,CTO189fA/B/C(20µmol/L)0.5µL, CTO654r(20µmol/L) 0.5µL,Taq DNA聚合酶(5U) 0.5µL,DNA模板1µL,加ddH20至50µL.反应程序为:94℃预变性5min;94℃变性45s,57℃退火45s,72℃延伸90s,共进行35个循环,72℃延伸10min.第二轮反应体系(50µL)为:10×PCR buffer 5µL,dNTP(各2.5mmol/L) 4µL,F357-GC (20µmol/L) 2µL,R518(20µmol/L) 2µL,Taq DNA聚合酶(5U) 0.5µL,DNA模板1µL,加ddH20至50µL.反应程序为:94℃预变性5min;94℃变性1min,65℃退火1min,72℃延伸1min,共进行20个循环,每个循环降低0.5℃;94℃变性1min,55℃退火1min,72℃延伸1min,共进行10个循环;72℃延伸8min.
1.4 变性梯度凝胶电泳(DGGE)
采用美国Bio-Rad公司生产的DCodeTM基因突变检验系统对PCR扩增产物进行分离.电泳条件:凝胶变性梯度30%~65%(总细菌)和30%~ 55%(AOB),电压105V,电泳缓冲液1×TAE,电泳温度60℃,电泳时间10h.电泳结束后,用Gel Red核酸凝胶染料染色30min,在凝胶成像系统(Gel DocTMXR+, Bio-Rad)中进行拍照.
1.5 克隆与测序
将凝胶中较亮的条带切下置于1.5mL的离心管中,加入50uL灭菌的去离子水,4℃下放置过夜,使DNA片段从胶中慢慢扩散出来,以此为模板,采用不带GC夹结构的F357和R518引物进行PCR扩增,扩增体系和程序同之前带GC夹的引物对F357-GC和R518.PCR产物用1.2%琼脂糖凝胶电泳检测并采用上海生工提供的SanPrep柱式DNA胶回收试剂盒纯化,将纯化后的DNA片段连接至pMD18-T Vector(TaKaRa, Japan),转化到JM109感受态细胞中,在含有Amp/XGal/IPTG的LB平板上进行蓝白斑筛选.挑选白斑做菌落PCR验证,片段大小正确的PCR产物送往上海生工生物公司进行测序.将测序所得基因序列利用NCBI网站上BLAST程序与GenBank中已登录的序列进行同源性比较,下载具有代表性的同源性较高的序列,并利用MEGA5.0软件中的邻接算法构建系统发育树.
1.6 样品的生物多样性分析
应用凝胶分析软件Quantity One对扫描所得的DGGE图谱进行分析.将DGGE图谱中每一个单独的条带看作是一个单独的菌种,并且该条带的密度等于该菌种的丰度.Shannon多样性指数(SDI)由样品中微生物种属的数量和丰度所决定,SDI数值越大,种群的多样性越高.Pielou均匀性指数(EI)表示菌种分布的均匀程度.因此可利用Shannon多样性指数和Pielou均匀性指数讨论活性污泥中微生物多样性特征.计算公式为

式中:Pi为菌种i在菌落中所占的百分比,Pi=ni/N, ni为峰面积,N为所有峰的总面积;S为样品中所有菌种的总数,即Patrick丰富度指数.
2 结果与讨论
2.1 污泥样品总DNA提取和PCR扩增结果
污泥样品提取的DNA经1.2%的琼脂糖凝胶电泳检测,得到的总DNA片段大小约为23kbp,属于较完整的细菌总DNA,适合做下一步的PCR扩增.以提取的样品总DNA为模板,通过特异性引物对总细菌16S rRNA基因进行PCR扩增,得到约250bp的片段,与目的基因片段长度相当,适合后续进行DGGE.以提取的样品总DNA为模板,通过两轮特异性引物对AOB 16S rRNA基因进行PCR扩增,分别得到约500bp(第一轮)和250bp(第二轮)的片段,为扩增的目的条带,适合后续进行DGGE.
2.2 DGGE指纹图谱结果
将样品R1~R4基因组DNA的PCR扩增产物通过变性梯度凝胶电泳分离,获得DGGE图谱.如图1和图2所示.根据DGGE技术原理,图谱中分离出来的条带都是不同种类的微生物的特定区的DNA片段,每个条带原理上可以代表一个微生物种属,条带信号强度越大表示该细菌在污泥中的优势地位越大[8].因此DGGE图谱显示了样品R1~R4所代表的接种污泥和不同底物及不同颗粒化方法培养出的好氧颗粒污泥中微生物群落结构的差异.

图1 好氧颗粒污泥总细菌DGGE图谱Fig.1 Total bacteria DGGE profile of the aerobic granular sludge
总细菌DGGE图谱中,有些菌属是接种污泥和3种好氧颗粒污泥所共有的优势菌属,如条带1、2、3、4、7、18,其所代表的微生物对环境条件具有较强的适应能力,表明接种污泥中的这部分细菌能够适应不同底物及不同颗粒化培养方法,是各种好氧颗粒污泥中的常见优势细菌;样品R1中特有条带9和10,其所代表的微生物只在接种污泥中出现,表明这些细菌不利于颗粒污泥的稳定,在活性污泥好氧颗粒化过程中被淘汰;样品R2中包含有条带1、2、3、4、5、6、7、8、11、15、16、17、18、19、20、21,其中样品2所特有的条带包括8、11、16、17,表明接种污泥采用模拟废水培养成熟的好氧颗粒污泥中的优势菌属发生了变化,并形成该条件下特有的优势菌属;样品R3中包含有条带1、2、3、4、5、7、12、13、14、18、22,这些条带在接种污泥样品中均能够找到,表明接种污泥投加粉末活性炭培养形成的实际生活污水好氧颗粒污泥中部分细菌淘汰,部分细菌得到富集,且没有新的优势细菌出现;样品R4中包含有条带1、2、3、4、7、14、18,该样品中条带数最少,且这些条带均能在样品R1中找到,表明接种污泥投加硅藻土培养形成的实际生活污水好氧颗粒污泥中细菌的种类最少,这种培养方法对微生物具有较高的选择性.

图2 好氧颗粒污泥AOB DGGE图谱Fig.2 AOB DGGE profile of the aerobic granular sludge
AOB DGGE图谱中,有些菌属是接种污泥和3种好氧颗粒污泥所共有的优势菌属,如条带6、7,其所代表的微生物对环境条件具有较强的适应能力,表明接种污泥中的这部分细菌能够适应不同底物及不同颗粒化培养方法,是活性污泥与各种好氧颗粒污泥中常见的优势AOB菌属.有些菌属是接种污泥培养成好氧颗粒污泥后所形成的新的优势菌属,如条带2在样品R3和R4中出现,表明投加粉末活性炭和硅藻土所形成的实际生活污水好氧颗粒污泥环境中更适合这种细菌的生长;条带4、9和10均在样品R2和R4中出现,表明这部分细菌能够适应不同的底物环境;条带11在样品R2、R3、R4中均出现,表明这部分细菌是各种好氧颗粒污泥中常见的优势细菌;条带12只出现在样品R4中,表明这种细菌是硅藻土好氧颗粒污泥中常见的优势细菌.有些菌属是接种污泥和特定几种好氧颗粒污泥中所共有的菌属,如条带1在样品R1、R3和R4中出现,表明接种污泥中这部分细菌在实际生活污水好氧颗粒污泥中得到了富集;条带3和8在样品R1和R3中出现,表明这部分细菌是活性炭好氧颗粒污泥中常见的优势细菌;条带5在样品R1、R2和R3中出现,表明这部分细菌能够适应不同的底物环境.
2.3 细菌种群多样性
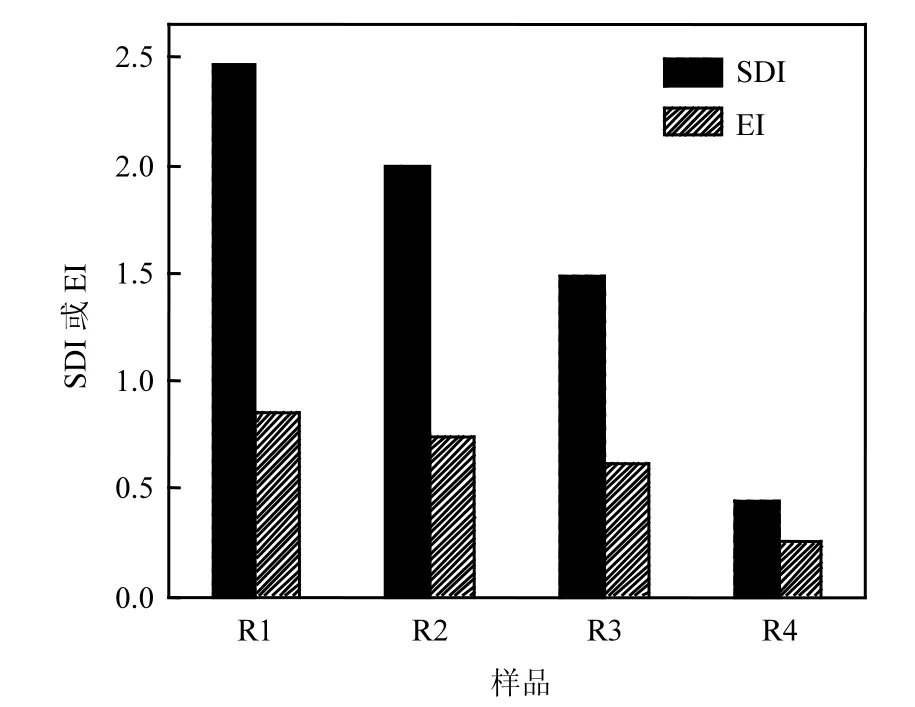
图3 样品R1~R4总细菌多样性Fig.3 The diverisity of total bacteria in sample R1~R4

图4 样品R1~R4AOB多样性特性Fig.4 The diverisity of AOB in samples R1~R4
从图3可以看出:4个样品中接种污泥对应的样品R1的SDI和EI均最高,分别为2.46和0.85,表明接种污泥中微生物种群相对丰富;颗粒污泥对应的样品R2~R4的SDI和EI均有不同程度下降,其中模拟废水好氧颗粒污泥(对应样品R2)的2个指数最高、活性炭好氧颗粒污泥(对应样品R3)次之、硅藻土好氧颗粒污泥(对应样品R4)最低.表明污泥好氧颗粒化的过程会淘汰部分种群从而降低污泥中微生物种群的多样性.模拟废水培养的颗粒污泥微生物种群多样性丰富、分布均匀,而实际生活污水培养的好氧颗粒污泥微生物种群多样性较低、分布集中,表明颗粒化条件越适宜,形成的颗粒污泥的微生物种群结构越多样.
从图4可以看出:4个样品中颗粒污泥多样性指数、均匀性指数均高于接种的活性污泥,模拟废水好氧颗粒污泥(对应样品R2)的多样性指数和均匀性指数最高、硅藻土好氧颗粒污泥(对应样品R4)次之、活性炭好氧颗粒污泥(对应样品R3)最低.表明好氧颗粒污泥能够富集AOB,这为好氧颗粒污泥能够维持稳定的短程硝化提供了微生物学基础.
2.4 特征条带的回收测序和系统发育分析
在对DGGE图谱分析的基础上,割取代表性优势条带,进行克隆测序,将测序所得基因序列输入到NCBI网站,利用BLAST程序与数据库中已有的序列进行比对分析,同时用MEGA5.0软件构建进化树.总细菌和AOB的部分条带16S rDNA序列比对结果分别见表3.总细菌和AOB的系统发育树分别见图5和图6.
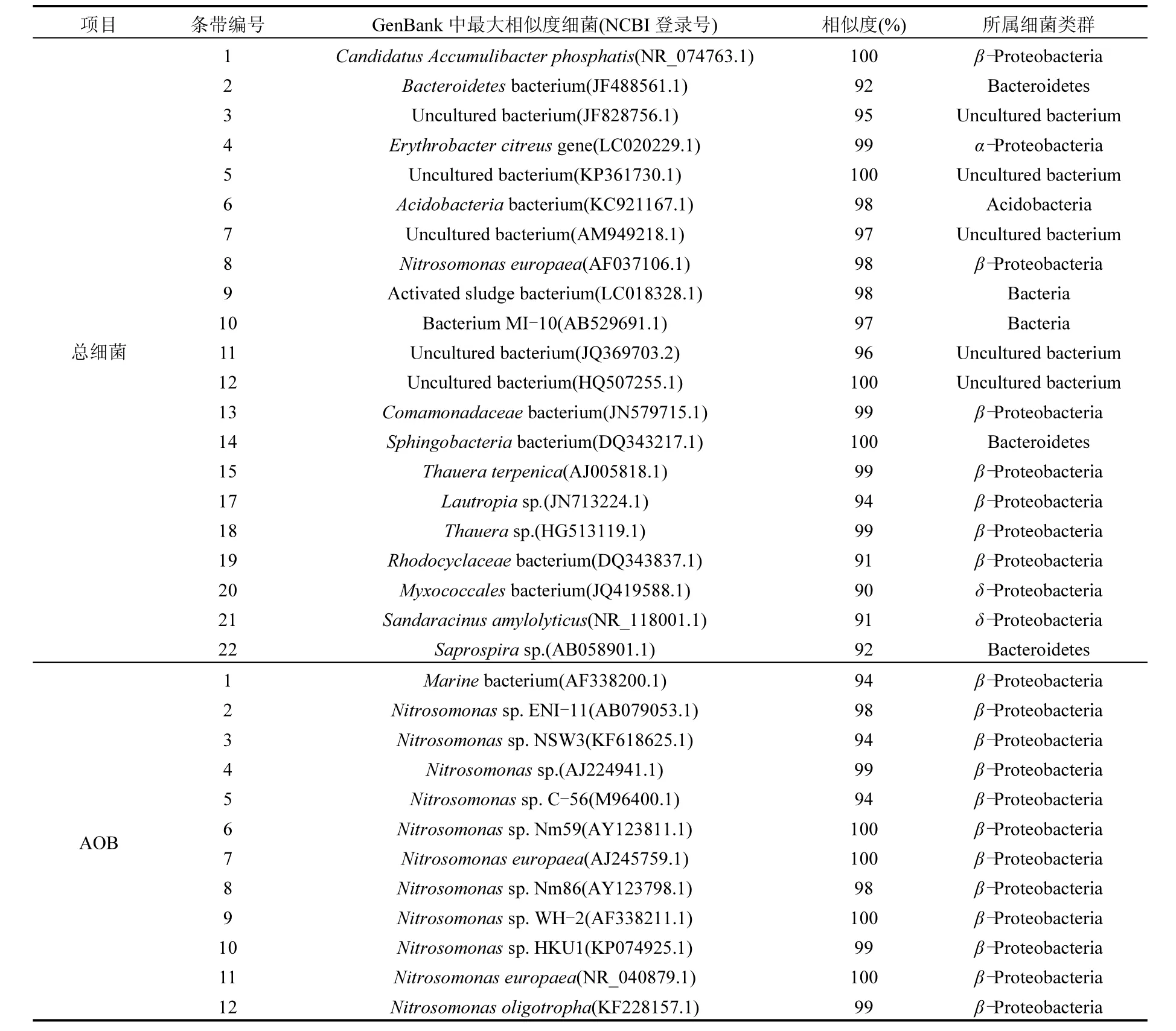
表3 总细菌与AOB部分条带 16S rDNA 序列比对结果Table 3 16S rDNA sequencing results of total bacteria and AOB bacteria

图5 总细菌系统发育树Fig.5 Phylogenetic tree of total bacteria
从表3的总细菌片段测序比对结果和图5的总细菌系统发育树可以看出,获得的各条序列的同源性信息,这些优势细菌基本都能在Genbank数据库中找到同源性较好的细菌,序列相似性范围在90%~100%.由表3可知,4个样品中的微生物群落主要分布于变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和未培养菌(uncultured bacterium).这一结果与Wagner[9]对多篇关于废水生物处理反应器中细菌群落结构的研究结果一致,即变形菌门和拟杆菌门一直是废水处理系统中的优势菌群.4个样品共有的条带中,条带1与Candidatus Accumulibacter phosphatis (NR_074763.1)同源性达到了100%,Candidatus Accumulibacter phosphatis是一种广泛存在于脱氮除磷工艺中的聚磷菌[10];条带4与Erythrobacter citreus(LC020229)同源性达到99%,Erythrobacter citreus据报道是一种石油污染生物修复细菌[11];条带18与Thauera sp. (HG513119)同源性达到99%,Thauera属细菌是一类广泛存在于各种类型的废水处理装置中的反硝化菌,并具有相当广泛的污染物降解能力的重要功能类群[12];条带3和条带7分别与Uncultured bacterium(JF828756.1)和Uncultured bacterium(AM949218.1)的同源性达到95%和97%,表明好氧颗粒污泥中潜藏着许多未被人们认识的微生物新资源.这几种细菌存在于接种污泥、模拟废水好氧颗粒污泥、活性炭实际生活污水好氧颗粒污泥和硅藻土实际生活污水好氧颗粒污泥中,表明其具有较强的环境适应能力,而他们具有的聚磷功能、难降解有机物生物降解功能和脱氮功能都是水处理中功能菌的生化特性.样品R2中特有的条带8与Nitrosomonas europaea (AF037106.1)同源性高达98%,β-Proteobacteria的亚硝化单胞菌属(Nitrosomonas)在反应器内将氨氧化为亚硝酸盐,作为短程硝化系统中的优势菌群已经见诸多报道[13-14].

图6 AOB系统发育树Fig.6 Phylogenetic tree of AOB
从表4的AOB片段测序比对结果和图6的AOB系统发育树可以得到,本研究中分离到的亚硝化细菌(AOB)均与β-Proteobacteria的亚硝化单胞菌属(Nitrosomonas)有着较高的相似性(94%~100%),而没有与亚硝化球菌属(Nitrosococcus)和亚硝化螺菌属(Nitrosospira)有一定相似性的序列.其中条带2、3、4、5、6、8、9、10均与Nitrosomonas sp.有着较高的相似性,条带11与Nitrosomonas europaea(NR_040879.1)同源性达到了100%,条带12与Nitrosomonas oligotropha(KF228157.1)同源性达到99%. Nitrosomonas europaea对 NH4+-N有较高的半饱和常数(Ks)(0.42~0.85mg/L),它们对NH3的底物亲和力较低,适合生存在富营养环境中[15-16]; Nitrosomonas oligotropha对NH4+-N的Ks较低, 在0.027~0.059mg/L,常被发现于贫营养的淡水环境和中性土壤中[17-18].本研究的优势AOB均属于亚硝化单胞菌属(Nitrosomonas),且出现对基质要求不同的菌种,表明好氧颗粒污泥内部形成的基质浓度梯度能够为多种AOB提供生存环境.
3 结论
3.1 活性污泥好氧颗粒化过程会减少微生物种群多样性,影响颗粒污泥稳定性的细菌被淘汰,而难降解有机物降解菌、聚磷菌、反硝化菌等污水处理功能微生物都在颗粒化过程中得到保留.模拟废水培养的颗粒污泥微生物种群多样性丰富、分布均匀,而实际生活污水培养的好氧颗粒污泥微生物种群多样性较低、分布集中.好氧颗粒污泥中的优势菌群主要分布于变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和未培养菌(uncultured bacterium).
3.2 活性污泥好氧颗粒化过程实现了对AOB一定程度的富集,与接种活性污泥相比,好氧颗粒污泥中AOB的多样性指数、均匀性指数均有提高,模拟废水好氧颗粒污泥的多样性指数和均匀性指数最高、硅藻土好氧颗粒污泥次之、活性炭好氧颗粒污泥最低.AOB种群分析表明好氧颗粒污泥中AOB均属于β-Proteobacteria的亚硝化单胞菌属(Nitrosomonas),且出现对基质要求不同的菌种,表明好氧颗粒污泥内部形成的基质浓度梯度能够为多种AOB提供生存环境.
参考文献:
[1] Moy B Y P, Tay J H, Toh S K, et al. High organic loading influences the physical characteristics of aerobic sludge granules [J]. Letters Applied Microbiology, 2002,34(6):407-412.
[2] 王 杰,彭永臻,杨 雄,等.不同碳源种类对好氧颗粒污泥合成PHA的影响 [J]. 中国环境科学, 2015,35(8):2360-2366.
[3] Yang S F, Tay J H, Liu Y. A novel granular sludge sequencing batch reactor for removal of organic and nitrogen from wastewater [J]. Journal of Biotechnology, 2004,106(1):77-86.
[4] Ni B J, Xie W M, Liu S G, et al. Granulation of activated sludge in a pilot-scale sequencing batch reactor for the treatment of low-strength municipal wastewater [J]. Water Research, 2008, 43(3):751-761.
[5] Heijnen J J, Loosdrecht M C M V, Mulder R, et al. Development and scale-up of an aerobic biofilm air-lift suspension reactor [J]. Water Science and Technology, 1993,27(5/6):253-261.
[6] 高 阳,徐晓晨,徐光景.活性炭为载体的好氧颗粒污泥培养及性能研究 [J]. 水资源与水工程学报, 2012,23(5):25-28.
[7] 樊红辉.微氧颗粒污泥+硅藻土去除焦化废水中氨氮的研究[D]. 太原:太原理工大学, 2012.
[8] 陈 谊.利用 PCR-DGGE 研究膜生物反应器中微生物的群落结构 [D]. 天津:天津大学, 2009.
[9] Wagner M, Loy A. Bacterial community composition and function in sewage treatment systems [J]. Current Opinion in Biotechnology, 2002,13(3):218−227.
[10] Nguyen H T, Nielsen J L, Nielsen P H. 'Candidatus Halomonas phosphatis', a novel polyphosphate-accumulating organism in full-scale enhanced biological phosphorus removal plants [J]. Environmental Microbiology, 2012,14(10):2826-2837.
[11] Ebm D, Vybiral D, Koblízek M P, et al. Erythrobacter citreus sp nov., a yellow-pigmented bacterium that lacks bacteriochlorophyll a, isolated from the western Mediterranean Sea [J]. International Journal of Systematic and Evolutionary Microbiology, 2002,52(5): 1655-1661.
[12] 毛跃建.废水处理系统中重要功能类群Thauera属种群结构与功能的研究 [D]. 上海:上海交通大学, 2009.
[13] Liu S, Yang F, Xue Y, et al. Evaluation of oxygen adaptation and identification of functional bacteria composition for anammox consortium in non-woven biological rotating contactor [J]. Bioresource Technology, 2008,99(17):8273-8279.
[14] Limpiyakorn T, Kurisu F, Sakamoto Y, et al. Effects of ammonium and nitrite oncommunities and populations of ammonia-oxidizing bacteria in laboratory-scale continuous-flow reactors [J]. Fems Microbiology Ecology, 2007,60(3):501-512.
[15] 王晓慧,文湘华,杨宁宁.污水处理工艺对氨氧化菌及细菌群落的影响 [J]. 中国环境科学, 2009,29(6):622-628.
[16] Persson F, Wik T, Sorensson F, et al. Distribution and activity of ammonia oxidizing bacteria in a large full-scale trickling filter [J]. Water Research, 2002,36(6):1439-1448.
[17] Lydmark P, Almstrand R, Samuelsson K, et al. Effects of environmental conditions on the nitrifying population dynamics in a pilot wastewater treatment plant [J]. Environmental Microbiology, 2007,9(9):2220-2233.
[18] Rittmann B E, Laspidou C S, Flax J, et al. Molecular and modeling analyses of the structure and function of nitrifying activated sludge [J]. Water Science & Technology, 1999,39(1): 51-59.
Characteristics of microbial community structure in different aerobic granular sludge.
HOU Ai-yue1, LI Jun1*, WANG Chang-wen2, YUE Yao-dong1, ZHAO Xin-yan1, BIAN Wei1, ZHANG Yan-zhuo1(1.Key Laboratory of Beijing for Water Quality Science and Water Environment Recovery Engineering, Beijing University of Technology, Beijing 100124, China;2.Key Laboratory of Shandong Provincial for Water and Soil Conservation & Environmental Protection, School of Resources & Environment, Linyi University, Linyi 276005, China). China Environmental Science, 2016,36(4):1136~1144
Abstract:In order to investigate the influences of activated sludge aerobic granulation process on microbial diversity and the differences of microbial community structure in aerobic granular sludge (AGS) which was cultivated by different carbon sources or different granulation methods, the microbial community structure of seed sludge and AGS (AGS with artificial wastewater, AGS with real domestic wastewater by adding diatomite and powered activated carbon respectively) were analyzed by PCR-DGGE. The activated sludge aerobic granulation process led to a low microbial diversity. The bacteria which might impact the stability of granular sludge were eliminated. However, the bacteria which were capable of phosphorus accumulation, denitrification or bio-degradation of refractory organic substances were retained. In the activated sludge aerobic granulation process the ammonia oxidation bacteria (AOB) were enriched to a certain extent. Compared with the seed activated sludge, the diversity index and evenness index of AOB were increased. The dominant bacterial communities in AGS were Proteobacteria, Bacteroidetes and uncultured bacterium. The dominant AOB in AGS were all belonged to Nitrosomonas.
Key words:aerobic granular sludge;microbial community structure;PCR-DGGE
作者简介:侯爱月(1990-),女,河北张家口人,硕士研究生,主要从事污水生物处理研究.
基金项目:国家水体污染控制与治理科技重大专项资助项目(2014ZX07201-011)
收稿日期:2015-09-06
中图分类号:X172
文献标识码:A
文章编号:1000-6923(2016)04-1136-09
