链状亚历山大藻不同生长状态差异表达microRNA的筛选和验证*
2016-04-06耿慧利隋正红任源源杜青伟
耿慧利, 隋正红, 任源源, 杜青伟, 刘 源
(中国海洋大学海洋生物遗传学与育种教育部重点实验室, 山东 青岛 266003)
链状亚历山大藻不同生长状态差异表达microRNA的筛选和验证*
耿慧利, 隋正红**, 任源源, 杜青伟, 刘源
(中国海洋大学海洋生物遗传学与育种教育部重点实验室, 山东 青岛 266003)
摘要:从链状亚历山大藻(Alexandrium catenella)延迟期、对数期2个小RNA文库中选择了2个差异表达的microRNA(osa-miR2876和rgl-miR5139),设计f/2培养下延迟期、对数期和高氮、高磷、高锰对数期5个条件,用qPCR的方法对其表达进行了检测。研究建立了以5.8srRNA为内参基因,以 SYBR Green I 为荧光染料,用加尾法荧光定量PCR检测microRNA在不同生长状态相对表达的方法。microRNA的表达量在对数期和诱导下对数期均低于延迟期,高锰条件下对数期均为最低,且延迟期的表达量分别是高锰条件表达量的18.63和14.12倍。microRNA负调控靶基因的表达,而osa-miR2876和rgl-miR5139在对数期均为下调表达,说明其靶基因参与的细胞生理过程:DNA复制、修复、嘌呤、嘧啶碱基的代谢、RNA聚合酶的代谢等过程呈现激活状态,而上述生理过程的激活均有利于藻细胞增殖。这些结果表明:osa-miR2876和rgl-miR5139可能在链状亚历山大藻的快速分裂和增殖中发挥着非常重要的调控作用。本文研究结果为甲藻兼性营养型正确性提供了进一步的证据,同时rgl-miR5139在细胞快速生长阶段的下调表达提示在该阶段下异养营养型可能占优势。本研究说明了microRNA在链状亚历山大藻快速生长过程中可能发挥着重要的调节作用。也为其他物种microRNA的研究和检测提供了快速简便、特异性好的研究方法。
关键词:链状亚历山大藻; microRNA; qPCR; 差异表达
甲藻是一类具有2个鞭毛能够自由游动的单细胞原生生物,在海洋生态系统的初级生产中发挥着重要的作用[1]。最初,甲藻被认为只能进行自养作用,但在近二十年来,研究发现甲藻是兼性营养型的,既能够进行光合作用又能通过鞭毛孔分泌细胞质到胞外形成捕食笼将猎物包裹并进行消化[2]。甲藻是数量最多的能够产生毒素的物种类型,占有害赤潮藻类的75%[3]。由产毒甲藻引起的赤潮给渔业生产、海洋环境和人类的健康带来了严重的影响。甲藻属中的许多物种都能够产生一种神经性麻痹贝毒PSTs(Paralytic shellfish toxins),能够引发贝类麻痹性中毒,而误食这种贝类将给人的神经系统带来极大的损害[4]。链状亚历山大藻(Alexandriumcatenella)是一种典型的能够产生PSTs的有毒甲藻,属于甲藻门,甲藻纲,多甲藻目,膝沟藻科,亚历山大藻属,研究者做了很多努力以期找到链状亚历山大藻爆发性增殖的分子机制,包括:转录组测序及分析[5],蛋白质组分析[6],与藻细胞增殖有关基因[7]的表达分析等。但是,至今在赤潮爆发过程中藻细胞大量增殖的机制仍然不清楚。
microRNA(Micro ribonucleic acids)是真核生物中普遍存在的一类长约18~25个核苷酸的内源性非编码RNA,它能在转录水平或转录后水平通过mRNA剪切和抑制蛋白质翻译的方式负调控靶基因的表达[8]。在植物中,microRNA基因首先被转录成microRNA前体(pri-microRNA)的初级转录物,随后pri-microRNA经过Dicer酶的剪切产生成熟的microRNA,成熟的microRNA会选择性整合入RISC(RNA induced silencing complex)中特异性识别靶基因。绝大多数植物microRNA都与靶基因mRNA近乎完全匹配进而引发靶mRNA的降解[9]。研究表明,microRNA在植物的多种生长代谢过程中发挥着至关重要的调节作用,如植物的生长、发育、细胞的分化、信号转导及响应各种生物的或非生物的胁迫[10]。
目前,microRNA已成功地在多种高等植物中得到鉴定,如:拟南芥、花生、水稻,小麦等[11-14]。但是关于藻类microRNA的发现仅仅在近几年才有报道,单细胞藻类的更少,仅有莱茵衣藻,三角褐指藻等[15-16]。为了研究microRNA在链状亚历山大藻不同生长时期的调控作用,我们构建了延迟期、对数期2个小RNA文库,并用Illumina测序平台进行了高通量测序,从中发现了在2个不同生长时期差异表达的microRNA: osa-miR2876和rgl-miR5139,经靶基因预测和KEGG注释推测它们可能在该藻快速生长过程中具有重要调控作用[17]。
在本研究中,为验证高通量测序结果及解析osa-miR2876和rgl-miR5139的调控作用,用qPCR的方法对其在链状亚历山大藻中的不同生长时期的表达进行了检测,并对其调控功能进行了分析。这是首次在甲藻类群中用实验的方法对microRNA 的表达进行的检测。我们的研究结果不仅能够证实microRNA在甲藻中的存在,及在链状亚历山大藻不同生长时期差异表达,而且为阐明链状亚历山大藻爆发性增殖的分子机理提供了依据。
1材料与方法
1.1 藻细胞的培养和收集
链状亚历山大藻种来自于中国海洋大学海洋生命学院教育部遗传育种重点实验室。用f/2培养基(不含硅)对藻进行培养,温度为(20±1)°C,光暗周期控制在12 h/12 h,光量子密度为30~35μmol·m-2·s-l[18]。用于实验的藻首先经过48 h的黑暗处理,即同步化处理[19],随后处理过的藻细胞以2×106细胞/升的密度接入新的f/2培养基。在中午分别收集生长至延迟期(培养2d)和对数期(分别培养至第7天和第12天)的藻细胞,每个样品约含5×106个藻细胞。有研究表明,高浓度的氮、磷、锰能促进藻细胞的生长[20-21],因此除延迟期、对数期2个样品外,又选择了3个诱导对数期进行qPCR试验,分别是:高氮诱导对数期(NaNO3终浓度为2.304mmol/L),高磷诱导对数期(NaH2PO4·2H2O终浓度为0.144mmol/L)高锰诱导对数期(MnCl2·4H2O终浓度为2.730μmol/L)。5个不同生长时期和条件的样品(约含5×106个藻细胞)用离心机以4 000g的速度离心6 min,然后将形成藻球的1.5mL EP管及时放入液氮中并于-80 °C冰箱保存。
1.2 引物设计
本实验采用的是加尾法qPCR[22],反向通用引物由试剂盒采用SYBR○RPrimeScriptxTMmiRNA RT-PCR Kit (日本的TaKaRa公司)。特异的正向引物根据miRNA的序列进行设计,基本与miRNA的序列一致。当退火温度Tm值偏低时,在序列5′端增加几个G或者C碱基,保证PCR的退火温度在60 °C左右。设计好的引物由华大公司进行合成。引物序列见表1。
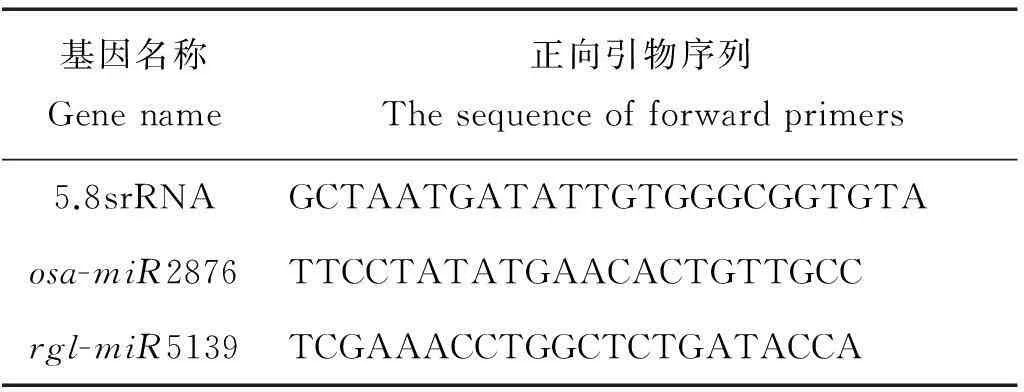
表1 用于荧光定量PCR的引物序列
1.3 样品RNA的提取及反转录
采用日本的TaKaRa公司提供的RNAiso for Small RNA试剂对20~200个核苷酸的小RNA进行提取。提取的RNA样品用1.5%的琼脂糖凝胶电泳对其完整性进行检测,并用NanoDrop 2000检测其纯度和浓度。仅高质量的RNA(OD 260/280在1.9 ~2.1之间))才能被用来进行实验。依据试剂盒SYBR○RPrimeScriptxTMmiRNA RT-PCR Kit,反转录体系如下所示:10 μL 的 2× miRNA Reaction Buffer Mix, 2μL 的 0.1%BSA, 2μL 的 miRNA PrimeScript○RRT Enzyme Mix, 大约 1μg 的 总 RNA以及 5 μL 的RNase-free ddH2O。合成的cDNA保存于-20 °C冰箱。用内参5.8srRNA检测cDNA的合成效率。3%琼脂糖凝胶电泳检测PCR结果。
1.4 目的序列的回收和克隆测序
将内参5.8srRNA及osa-miR2876和rgl-miR5139的PCR产物按照普通琼脂糖凝胶DNA回收试剂盒(天根公司提供)所提供的说明进行胶回收,对回收得到的DNA进行浓度和纯度检测。并将连接好的产物导入大肠杆菌DH5α进行克隆并送给华大公司进行测序。测序完全正确的引物才可被用来进行qPCR实验。
1.5 qPCR溶解曲线和标准曲线的制作
依据TaKaRa公司所提供的miRNA定量试剂盒制qPCR体系:SYBR○RPremix Ex TaqTMII 10L,10 mmol/L 特异的正向引物0.4L, 10mmol/L 通用反向引物0.4L, RNase-free H2O 7.2L,cDNA约2L。制备好的体系在ABI 7500 荧光定量PCR仪上进行反应。反应程序为:95 °C 30 s, 95 °C 5 s, 65 °C 34 s,40个循环。反应结束后对扩增曲线、溶解曲线进行分析,每个反应包括3个重复。若溶解曲线峰单一尖锐,则将目的片段克隆后根据天根的质粒小提试剂盒(提供公司提供)说明步骤进行质粒的提取。将5.8srRNA及osa-miR2876和rgl-miR5139的质粒逐级稀释10倍,作为qPCR的模板进行PCR反应(体系及程序同上),通过各级反应的Ct值进行标准曲线的绘制。
1.6 qPCR检测基因的差异表达
以延迟期、对数期、以及高氮、高磷、高锰诱导对数期为5个样品进行RNA的提取及cDNA的合成(同1.3,1.4),以5.8srRNA为内参基因,用1.2中设计的引物进行荧光定量PCR,每个样品设置3个生物学重复,3个技术重复(反应体系及测序见1.6)对实验结果用2-△△CT相对定量法进行计算[23]。
2结果分析
2.1 osa-miR2876和rgl-miR5139高通量测序差异结果
通过对延迟期、对数期2个小RNA文库高通量测序结果分析发现,osa-miR2876和rgl-miR5139 2个microRNA在延迟期均呈上调表达状态,且延迟期的表达量分别是对数期的正无穷倍和1.12倍。具体结果如表2所示。

表2 osa-miR2876和rgl-miR5139测序结果
2.2 一链合成效率的检测
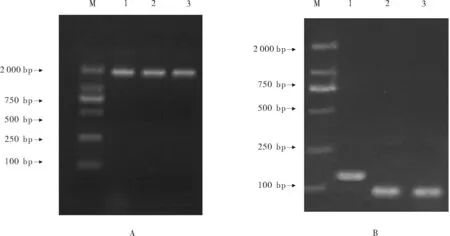
使用内参基因5.8srRNA的引物对所合成的一链cDNA进行检测(见图1)。不同时期的一链cDNA进行检测结果说明RNA的完整性较好,且cDNA的合成效率较高。

(1:延迟期cDNA; 2:对数期cDNA; 3:高磷诱导对数期; 4:高氮诱导对数期; 5:高锰诱导对数期;M:DNA分子标记DL2000.)
图1用内参5.8srRNA的引物对cDNA进行检测电泳图
Fig.1Gel analysis of the resulting cDNA by
the reference gene 5.8srRNA
2.3 质粒的检测
将5.8srRNA及osa-miR2876和rgl-miR5139目的片段克隆后,分别提取的质粒电泳图见图2A。提取带有目的片段的质粒,然后利用PCR通过目的片段的特异引物对相应的质粒进行检测,电泳结果显示,得到的5.8srRNA、osa-miR2876和rgl-miR5139长度分别为140 bp和约100 bp(见图2B),与预设的长度相同,说明质粒可用。
2.4 目的基因溶解曲线和标准曲线的制作
经分析,5.8srRNA,osa-miR2876和rgl-miR5139溶解曲线的峰单一尖锐,说明扩增产物单一,无非特异性扩增。用无菌的超纯水对获得的质粒按101、102、103、104和105梯度逐级稀释后,作为荧光定量PCR的模板进行PCR反应。每个梯度都会有一个Ct值,以此为纵坐标,以相应的稀释倍数为横坐标,绘制标准曲线。得到的5.8srRNA的R2值为0.998 5,扩增效率为101%;osa-miR2876的R2值为0.993,扩增效率为101%;rgl-miR5139的R2值为0.994,扩增效率为105%。目的基因和内参基因的扩增效率接近100%,且差值在5%以内,满足荧光定量体系的要求[24](见图3)。
2.5 2个差异microRNA表达量的变化
应用荧光定量PCR对osa-miR2876和rgl-miR5139在链状亚历山大藻延迟期、f/2对数期和3个诱导下对数期(高磷、高氮和高锰)的RNA表达量进行相对定量分析。结果显示,microRNA的表达量在对数期和诱导下对数期均低于延迟期,osa-miR2876的表达量在高锰条件下对数期最低,在延迟期时表达量最高,是高锰条件表达量的18.63倍。rgl-miR5139的表达量同样在高锰条件下最低,在延迟期表达量最高,是高锰条件表达量的14.12倍。经过SPSS软件分析,osa-miR2876和rgl-miR5139在延迟期的表达量均显著高于诱导条件下对数期的表达量(P<0.05)。
(M:DNA分子标记DL2000,DNA marleer DL 2000,1:5.8s;2:osa-miR2876;3:rgl-miR5139)

(A:5.8s溶解曲线;B:osa-miR2876溶解曲线;C:rgl-miR5139溶解曲线;D:5.8s标准曲线;E:osa-miR2876标准曲线;F:rgl-miR5139标准曲线。A:The dissociation curve of 5.8 s,B:The dissociation curve of osa-miR2876,C:The dissociation curve of rgl-miR5139,D:The standard curve of 5.8 s,E:The standard curve of osa-miR2876,F:The standard curve of rgl-miR5139.)
图3内参基因和目的基因的溶解曲线和标准曲线
Fig.3The melting curves and standard curve
3讨论
3.1 加尾法qPCR验证microRNA的优越性
高通量测序提供一种快速高效的鉴定microRNA的方法,但所得到的microRNA具有一定的假阳性,因此通过实验的方法对测序鉴定出来的microRNA进行验证是非常必要而且重要的。目前,microRNA本身的验证方法有多种:如real-time PCR(qPCR)、Northern blotting、原位杂交和基因芯片等。Northern blotting验证方法可靠性高,但要使用放射性同位素,而且难以检测低丰度的microRNA,大大降低了其使用范围。原位杂交方法因其操作复杂、耗时长而应用较少,同样的,基因芯片方法需要以大量已知的miRNA序列为前提设计探针而且成本较高使用较少[25]。qPCR因具有特异性强、通量高、灵敏度高、操作简便、成本较低等优点而成为大量使用的microRNA检测方法。但microRNA不仅表达量低且长度只有16~28个核苷酸,没有Poly(A)尾,因此无法用普通的反转录和荧光定量PCR检测miRNA。目前,microRNA的反转录主要有茎环反转录法和加尾反转录法。前者因需要合成特异的茎环结构引物而使成本相对较高。后者利用Poly(A)聚合酶为成熟的microRNA加上多聚Poly(A)尾,同时引入通用引物的结合为位点,具有成本较低,操作简便,结果可靠等优点,也是检测microRNA的最常用的方法[26]。

(*标识的为经SPSS软件分析得出的与延迟期表达量具有显著差异(p<0.05)的生长条。The significant difference resulted from SPSS was marked by *.)
图4osa-miR2876和rgl-miR5139在不同
生长时期和生长条件中的相对表达情况
Fig.4Relative expression level of different growth stages and
different growth conditions of osa-miR2876 and rgl-miR5139
SYBR Green I因成本较低、简单易行、荧光信号强,不需要特别优化条件和不需要另外设计荧光探针等优点而被本实验采用。但该染料专一性不如探针法,对引物的特异性要求较高。因为荧光染料可嵌入任何双链DNA中,如翻译组有非特异性扩增或引物二聚体也会产生假阳性信号[27]。因此本实验通过引物设计、调整退火温度和引物浓度来确保无非特异性扩增。首先对PCR产物进行琼脂糖电泳检测,条带单一正确后再进行溶解曲线分析。经电泳和溶解曲线分析,本实验设计的引物无非特异性扩增,可用于osa-miR2876和rgl-miR5139在不同生长时期表达量的定量分析。
3.2 osa-miR2876的qPCR实验结果与高通量测序结果比较分析
通过高通量测序得到的osa-miR2876在延迟期、对数期2个不同生长时期差异表达倍数是正无穷,而qPCR得到的结果是2.28倍,二者差异较大的原因主要有以下2点:第一,2种方法对差异表达倍数的计算方法不同:高通量序技术单次反应可以对数以百万计的序列进行分析,其表达量可从1个拷贝至数10万个不等,因而为了比较2个样品间microRNA表达量是否存在显著性差异,首先将2个样品归一化到同一数量级,方法是该microRNA的实际表达量/该样品总的表达量;然后对2个样本进行Fisher检验和T检验(得出p值)以及计算差异倍数(log2fold change):延迟期表达量/对数期表达量[28],同时满足|log2fold change|>1和P< 0.05的microRNA被认为是差异表达的[29]。由于osa-miR2876在对数期的实际表达量为3个拷贝,对数期microRNA总的表达量为7 226,因而均一化后其表达量约为0,所以差异倍数(log2fold change)为无穷大。该结果反映了osa-miR2876在延迟期和对数期的表达趋势,即对数期的表达量远低于延迟期。而在本次qPCR实验中,作者对实验数据的分析采用的是相对定量2-△△CT,以内参5.8s的表达量校准osa-miR2876在延迟期和对数期的表达量,其结果更接近实际。第二是高通量测序技术的原理与qPCR实验原理的不同。二代高通量测序的基本思想是边合成边测序( Sequencing by synthesis),即生成新DNA互补链时直接加入被荧光标记的dNTP或半简并引物,在合成或连接生成互补链时释放出荧光信号,通过捕获光信号并转化为一个测序峰值,获得互补链序列信息[30]。在信号读取过程中,会遇到N碱基等无信号采集的情况,且测序质量从5’到3’逐渐降低,因而在获得大批量数据的背后也存在着一定的假阳性。高通量测序的数据为我们研究基因的差异表达提供了方向和表达趋势,但必须结合生物学实验的验证才能更深层次地挖掘其功能信息。关于高通量测序所得的差异倍数与实验所得相差较大的结果在microRNA高通量测序的研究中相对是常见的,如在Eriocheirsinensis[31]中高通量测序得到的差异倍数接近300,而qPCR结果也在10以下。
本实验中,对高通量测序的结果进行了分析,并通过qPCR实验验证了osa-miR2876的相对表达量,二者差异趋势是一致的,也为研究其功能提供了一定的证据。
3.3 osa-miR2876和rgl-miR5139对藻细胞快速生长的调节作用
靶基因KEGG注释结果显示osa-miR2876的靶基因为DNA复制过程中的DNA复制执照因子MCM5和限制性核酸内切酶。DNA复制执照因子作为DNA复制过程中一种重要的起始因子,在细胞周期的M期,核膜破裂后可与染色质结合从而确保细胞周期进入S期[32]。研究表明:DNA复制执照因子可通过阻止染色体DNA的重复制从而精确控制每个细胞周期只能进行1次DNA复制[33];并且osa-miR2876的靶基因参与碱基错配修复过程的调节。
rgl-miR5139的靶基因涉及嘌呤、嘧啶碱基的代谢、RNA聚合酶的代谢、多种氨基酸合成、细胞骨架的调节、及氧化磷酸化、光合作用、三羧酸循环等多种与藻细胞生长密切相关的代谢过程。嘌呤、嘧啶等碱基的合成、氨基酸的合成与氧化磷酸化、光合作用等为细胞的快速分裂和增殖提供必备的原料和能量;而细胞骨架不仅可以维持细胞形态,保持细胞内部结构的有序性而且在细胞分裂中染色体的分离中发挥着重要的牵引作用[34]。
microRNA负调控靶基因的表达,osa-miR2876和rgl-miR5139在对数期均为下调表达,说明其靶基因参与的细胞生理过程:DNA复制、修复、嘌呤、嘧啶碱基的代谢、RNA聚合酶的代谢、多种氨基酸合成、细胞骨架的调节、及氧化磷酸化、光合作用、三羧酸循环过程呈现激活状态,而上述生理过程的激活均有利于藻细胞增殖。这些结果表明:osa-miR2876和rgl-miR5139可能在链状亚历山大藻的快速分裂和增殖中发挥着非常重要的调控作用。
3.4 甲藻的兼性营养型
近二十年来,研究证明甲藻是兼性营养型的,即:甲藻既能通过光合作用进行自养又能通过吞噬摄食其他浮游生物进行异养,打破了之前甲藻只能进行自养的结论[2]。KEGG注释结果显示rgl-miR5139参与吞噬作用的调节,我们的研究结果为甲藻兼性营养型正确性提供了进一步的证据,同时rgl-miR5139在细胞快速生长阶段的下调表达提示在该阶段下异养营养型可能占优势。
本研究建立了 microRNA 加尾法实时定量 PCR 的检测方法,具有成本较低、特异性好、快速准确、灵敏度高等特点,不仅证明了链状亚历山大藻中osa-miR2876和rgl-miR5139在不同生长时期和生长条件下的差异表达水平,而且为其他物种microRNA的研究和检测提供了切实可靠的方法。同时,osa-miR2876和rgl-miR5139在链状亚历山大藻的快速增殖生长中发挥的重要调控作用将有助于我们研究有赤潮爆发的分子机理,从而为可能的赤潮预报提供科学依据。
4结语
本文选择了2个在延迟期、对数期文库中差异表达的microRNA(osa-miR2876和rgl-miR5139),研究了其在5个不同生长条件下的相对表达情况,并对其功能进行了分析。2个microRNA在对数期和诱导下对数期的表达量均低于延迟期,且高锰条件下对数期的表达量均为最低。由于microRNA负调控靶基因的表达,而osa-miR2876和rgl-miR5139在对数期均为下调表达,这说明其靶基因参与的有利于藻细胞增殖的生理过程如DNA复制、修复、嘌呤、嘧啶碱基的代谢等过程均呈现激活状态,因此这些结果表明:osa-miR2876和rgl-miR5139在链状亚历山大藻的快速分裂和增殖中可能发挥着非常重要的调控作用。且本文的研究结果也为甲藻兼性营养型的正确性提供了进一步的证据。同时,本研究也为其他物种microRNA的研究和检测提供了快速简便、特异性好的研究方法。
参考文献:
[1]Sournia A. Red tide and toxic marine phytoplankton of the world ocean: an inquiry into biodiversity [C]. Harmful Marine Algal Blooms. Paris: Technique et Documentation -Lavoisier, Intercept Ltd, 1995: 103-112.
[2]Stoecker D K. Mixotrophy among Dinoflagellates [J]. Journal of Eukaryotic Microbiology, 1999, 46: 397-401.
[3]Toulza E, Shin M S, Blanc G, et al. Gene expression in proliferating cells of the dinoflagellateAlexandriumcatenella(Dinophyceae) [J]. Applied and Environmental Microbiology, 2010, 76: 4521-4529.
[4]Burkholder J M. Implications of harmful microalgae and heterotrophic dinoflagellates in management of sustainable marine fisheries [J]. Ecological Applications, 1998, 8: S37-S62.
[5]Zhang S, Sui Z H, Chang L P, et al. Transcriptome de novo assembly sequencing and analysis of the toxic dinoflagellateAlexandriumcatenellausing the Illumina platform [J]. Gene, 2014, 537: 285-293.
[6]Wang D Z, Lin L, Wang M H, et al. Proteomic analysis of a toxic dinoflagellateAlexandriumcatenellaunder different growth phases and conditions[J]. Chinese Science Bulletin, 2012, 57: 3328-3341.
[7]Huang J, Liang S, Sui Z H, et al. Cloning and characterization of proliferating cell nuclear antigen gene ofAlexandriumcatenella(Dinoflagellate) with respect to cell growth[J]. Acta Oceanologica Sinica, 2010, 29: 90-96.
[8]Bartel D P. MicroRNAs: Genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116: 281-297.
[9]Jones-Rhoades M W, Bartel D P, Bartel B. MicroRNAs and their regulatory roles in plants[J]. Annu Rev Plant Biol, 2006, 57: 19-53.
[10]Kidner C A and Martienssen R A. The developmental role of microRNA in plants [J]. Current Opinion in Plant Biology, 2005, 8: 38-44.
[11]Fahlgren N,Howell M D, Kasschau K D, et al. High-Throughput Sequencing ofArabidopsismicroRNAs: Evidence for for frequent birth and death of MIRNA Genes evidence for frequent birth and death of MIRNA Genes [J]. PloS One, 2007, 2: e219.
[12]Chi X Y, Yang Q L, Chen X P, et al. Identification and characterization of microRNAs from peanut (ArachishypogaeaL. ) by high-throughput sequencing [J]. Plos One, 2011, 6: e27530.
[13]Sunkar R, Zhou X F, Zheng Y, et al. Identification of novel and candidate miRNAs in rice by high throughput sequencing [J]. BMC Plant Biology, 2008, 8: 25.
[14]Su C, Yang X Z, Gao S Q, et al. Identification and characterization of a subset of microRNAs in wheat (TriticumaestivumL. )[J]. Genomics, 2014, 103: 298-307.
[15]Zhao T, Li G L, Mi S J, et al. A complex system of small RNAs in the unicellular green algaChlamydomonasreinhardtii[J]. Genes & Development, 2007, 21: 1190-1203.
[16]Huang A Y, He L W and Wang G C. Identification and characterization of microRNAs fromPhaeodactylumtricornutumby high-throughput sequencing and bioinformatics analysis[J]. BMC Genomics, 2011, 12: 337.
[17]耿慧利. 链状亚历山大藻microRNA高通量测序及生物信息学分析[D]. 青岛: 中国海洋大学, 2015.
Geng H L. Identification of MicroRNAs in the Toxigenic DinoflagellateAlexandriumcatenellaby High-Throughput Illumina Sequencing and Bioinformatic Analysis[D]. Qingdao: Ocean University of China, 2015
[18]Guillard R R L. Cultureof phytoplankton for feeding marine invertebrates[C]. In Smith W L. Chanley M H. Culture Ofmarine Invertebrate Animals. Plenum: New York Press, 1975: 29-60.
[19]Galleron C. Synchronization of the marine dinoflagellateamphidiniumcarteriin dense clutures[J]. Journal of Phycology, 1976, 12(1): 69-73.
[20]Matsuda A, Nishijima T, Fukami K. Effects of nitrogenous and phosphorus nutrients on the growth of toxic dinoflagellateAlexandriumcatenella[J]. Nippon Suisan Gakkaishi, 1999, 65: 847-855.
[21]Brand L E, Sunda W G, Guillard R R. Limitation of marine phytoplankton reproductive rates by zinc, manganese, and iron[J]. Limnology and Oceanography, 1983, 28: 1182-1198.
[22]Shi R, Chiang V L. Acile means for quantifying microRNA expression by real-time PCR[J]. BioTechniques, 2005, 39: 519-525
[23]Yao C L, Kong P, Wang Z Y, et al. Molecular cloning and expression of MyD88 in large yellow croaker,Pseudosciaenacrocea[J]. Fish & Shellfish Immunology, 2009, 26(2): 249-255.
[24]张吉凤. Rho激酶对大鼠海马神经元CRMPs mRNA表达的调节[D]. 暨南大学, 2009.
Zhang J F. The expression regulation of Rho kinase on rat hippocampal neurons CRMPs mRNA[D]. Jinan University, 2009.
[25]王磊, 范云六. 植物微小RNA(microRNA)研究进展[J]. 中国农业科技导报, 2007, 9(3): 18-23.
Wang L, Fan Y L. The research advances in plant mocroRNA [J]. Journal of Agricultural Science and Techology , 2007, 9(3): 18-23.
[26]成永强, 李正平, 王愈聪 . MicroRNA分析方法进展[J]. 化学进展, 2010, 8, 22 (8): 1509-1517. Cheng Y Q, Li Z P, Wang Y C. The advance of microRNA analysis method[J]. Progress in Chemistry, 2010, 22(8): 1509-1517.
[27]赵丽媛, 于志刚, 甄毓. 东海原甲藻线粒体细胞色素 b( Cy t b) 基因的定量检测[J]. 中国海洋大学学报(自然科学版), 39(3) : 448-452.
Zhao L Y, Yu Z G, Zhen L. The quantitative detection of mitochondrial cytochrome b (Cytb) [J]. Periodical of Ocean University of China, 39(3) : 448-452.
[28]D’haene B, Mestdagh P, Hellemans J, et al. MiRNA expression profiling: From reference genes to global mean normalization[J]. Methods Mol Biol, 2012, 822: 261-272.
[29]Audic S and Claverie JM. The significance of digital gene expression profiles[J]. Genome Research, 1997, 7: 986-995.
[30]王兴春, 杨致荣, 王敏, 等. 高通量测序技术及其应用 [J]. 中国生物工程杂志, 2012, 32(1): 109-114.
Wang X C, Yang Z R, Wang M, et al. High-throughput sequencing technology and its application [J]. China Biotechnology, 2012, 32(1): 109-114.
[31]Ou J, Meng Q, Li Y, et al. Identification and comparative analysis oftheEriocheirsinensismicroRNA transcriptome response toSpiroplasmaeriocheirisinfection using a deep sequencing approach[J]. Fish & Shellfish Immunology, 2012, 32(2): 345-352.
[32]Alberts B, Bray D, Lewis J, et al. Molecular Biology of the Cell [M]. 3rd Ed. NewYork: Garland Publishing , 1994: 139-194.
[33]Thömmes P, Kubota Y, Takisawa H, et al. The RLF-M component of the replication licensing system forms complexes containing all six MCM/P1 polypeptides [J]. The EMBO Journal, 1997, 16: 3312-3319.
[34]李兰. 两个棉花 (Gossypiumhirsutum) LIM 蛋白调控肌动蛋白细胞骨架及其在花药发育中的功能研究[D]. 武汉: 华中师范大学, 2014.
Li L. The Regulation of TwoGossypiumhirsutumLIM Protein on Actin Cytoskeleton and Iits Function Research on Anther Development[D]. Wuhan: Central China Normal University, 2014.
责任编辑高蓓
The Screen and Verification of Differentially Expressed MicroRNAs Under Different Growth Conditions inAlexandriumcatenella
GENG Hui-Li, SUI Zheng-Hong,REN Yuan-Yuan, DU Qing-Wei, LIU Yuan
(The Key Laboratory of Marine Genetics and Breeding, Ministry of Education, Ocean University of China, Qingdao 266003, China)
Abstract:As a class of relatively conservative regulator, accumulated reports demonstrated that miRNAs play an important role in a large variety of biological and metabolic processes, including cell proliferation, organ maturation, signal transduction and response to stress. Dinoflagellates is a kind of important organisms that can cause harmful algal blooms (HABs). Alexandrium catenella is a representative of dinoflagellates that is widely spread all over the world and has the ability of producing PSTs. In this paper, two differentially expressed microRNAs (osa-miR2876 and rgl-miR5139) were selected from constructed small RNA libraries of A. catenella under the lag and the logarithmic phases. Five conditions including the lag phase and the logarithmic phase under f/2 medium, high N, high P and high Mn induced logarithmic phase were designed for expression detection via qPCR technique. The study established a poly(A)-tailing qPCR method with SYBR Green I to detect the differential expression of microRNA under different growth conditions in A. catenella. 5.8srRNA gene was used to normalize the expression of microRNAs. The expression level of microRNA under the logarithmic and induced logarithmic (eg. high N, high P and high Mn induced logarithmic phase) phases were all lower than those under the lag phase. The lowest expression level was shown in the loagrithmic phase under high Mn. The expression level in the lag phase was 18.63 (for osa-miR2876) and 14.12 (for rgl-miR5139), respectively of that under high Mn condition. microRNA regulated gene expression via a negative pattern. The result of real-time PCR was consist with high-throughput sequencing. osa-miR2876 and rgl-miR5139 in the logarithmic phase were both down-regulated, which suggested that the target genes of two microRNAs involved, eg. DNA replication, repairing, the metabolism of purine and pyrimidine, the metabolism of RNA polymerase, may be activated. The activation of the above physiological processes were all benefit for the proliferation of the cells. Our results suggested that osa-miR2876 and rgl-miR5139 might play important roles during the process of rapid division and proliferation of A. catenella. Moreover our study provided further evidence of mixotrophic modes in dinoflagellates. The heterotrophic mode may be dominate in the logarithmic phase as revealed by the down regulation of rgl-miR5139 under fast growth phases of the cells. The study demonstrated that microRNAs played an important role in the blooming growth of A. catenella and provided an rapid, simple and specific method to detect and study microRNA in other organisms.
Key words:Alexandrium catenella; microRNAs; qPCR; differential expressed
中图法分类号:S917.3
文献标志码:A
文章编号:1672-5174(2016)01-064-08
作者简介:耿慧利(1989-),女,硕士生。E-mail:genghuili2009@163.com
收稿日期:2015-03-11;
修订日期:2015-04-18
*基金项目:国家自然科学基金项目(41176098);山东省自然科学基金重点项目(ZR2011DZ002)资助
DOI:10.16441/j.cnki.hdxb.20150069
引用格式:耿慧利, 隋正红, 任源源, 等. 链状亚历山大藻不同生长状态差异表达microRNA的筛选和验证[J]. 中国海洋大学(自然科学版), 2016, 46(1):64-71.
GENG Hui-Li, SUI Zheng-Hong,REN Yuan-Yuan,et al.The screen and verification of differentially expressed microRNAs under different growth conditins inalexandriumcatenella[J]. Periodical of Ocean University of China, 2016, 46(1): 64-71.
Supported by the Natural Science Foundation of China(41176098);The key research project supported by Shan Dong Natural Science Foundation(ZR2011DZ002)
**通讯作者:E-mail:suizhengh@ouc.edu.cn
