米贝链霉菌DSM41911的米尔贝霉素生物合成的初步研究
2016-03-08吕振斌曾小东田孝东赵昌明段建利邓子新
吕振斌, 曾小东, 田孝东, 赵昌明, 虞 沂, 段建利, 邓子新
(武汉大学药学院 组合生物合成与新药发现教育部重点实验室,湖北 武汉 430071)
米贝链霉菌DSM41911的米尔贝霉素生物合成的初步研究
吕振斌, 曾小东, 田孝东, 赵昌明, 虞 沂, 段建利*, 邓子新
(武汉大学药学院 组合生物合成与新药发现教育部重点实验室,湖北 武汉 430071)
对米贝链霉菌DSM41911进行初步研究,通过对16S rRNA及相关功能基因序列进行生物信息学分析,表明米贝链霉菌DSM41911与冰城链霉菌具有较高同源性。设计16种固体和液体培养基对菌株的发酵条件进行筛选,在MB4、MB6培养基中成功检测到milbemycin B5、milbemycin E、milbemycin VM44864和milbemycin B1。参考近似菌株冰城链霉菌的合成基因并结合文献推测出米尔贝霉素在米贝链霉菌DSM41911中的合成途径,为后续高产菌株的筛选提供参考。
米尔贝霉素;发酵;生物合成;16S rRNA
1967年,日本Sankyo公司的青木(Aoki)等人从链霉菌Streptomyceshygroscopicussubsp.aureolacrimosus的发酵液中发现米尔贝霉素(milbemycin)[1]。米尔贝霉素属于十六元大环内脂化合物,目前已报导60多种组分[2-4]。米尔贝霉素作为γ-氨基丁酸(GABA)激动剂,引发突触前体释放GABA[5],通过引起谷氨酸门控Cl-通道的开放,使Cl-内流量增加,从而使神经元休止电位超级化,不能释放正常的动作电位,最终导致昆虫死亡[6-7]。由于哺乳动物体内GABA仅存在于中枢神经系统,而米尔贝霉素不能通过哺乳动物的血脑屏障,所以米尔贝霉素类抗虫药对哺乳动物具有很高的安全性[8]。米尔贝霉素作为一种广谱、高效、安全、易降解的生物杀虫剂,对各种螨虫、农业害虫及家畜寄生虫均有很好的治疗效果,被广泛应用于抗寄生虫药和作物保护农药,具有广阔的市场前景和巨大的经济价值[9-10]。国外公司如日本三共株式会社、美国默克公司和瑞士诺华制药厂均生产米尔贝霉素及相关制剂,但由于菌株保护,知识产权等问题,国内市场基本被跨国公司垄断,国内相关研究仍处在起步阶段。向文胜等[11-12]从冰城链霉菌(Streptomycesbingchenggensis)发酵产物中成功分离出多种米尔贝霉素组分,并公布了冰城链霉菌全基因组序列,但米尔贝霉素产量仍有待提高。要想实现米尔贝霉素生产的工业化,就必须对米尔贝霉素的生物合成途径进行研究,进而筛选出米尔贝霉素的高产菌株。本研究将对米贝链霉菌DSM41911进行初步发酵研究,并对米尔贝霉素生物合成途径进行初步探索,为后续高产菌株的筛选提供参考。
1 材料与方法
1.1 材料
1.1.1 菌株来源 米贝链霉菌DSM41911,来自德国DSMZ保藏中心。
1.1.2 培养基(g/L) ①产孢培养基:黄豆饼粉20,甘露醇20,琼脂20;②TSBY:Oxoid胰蛋白胨大豆肉汤30,蔗糖103,酵母提取物 5;③种子液培养基:蔗糖5,聚蛋白胨1.75,K2HPO40.25,pH 7.2;④液体发酵培养基:MB1:蔗糖120,棉子粉11,黄豆饼粉11,脱脂奶粉11,K2HPO41,FeSO4·7H2O 0.1,CaCO32.5,pH 7.2;MB2:玉米粉100,黄豆饼粉10,棉子粉10,α-淀粉酶0.2,NaCl 1,K2HPO42,FeSO4·7H2O 1,CaCO37,pH 7.0;MB3:蔗糖80,黄豆饼粉10,酵母提取物2,肉提取物1,CaCO33,K2HPO40.3,FeSO4·7H2O 1,FeSO4·7H2O 0.05,pH 7.2;MB4:可溶性淀粉70,酵母提取物16,K2HPO40.5,MgSO4·7H2O 0.5,KCl 4,CoCl2·6H2O 0.01,CaCO32,pH 7.2;MB5:蔗糖160,黄豆饼粉20,酵母提取物5,肉提取物5,K2HPO40.5,MgSO4·7H2O 0.5,FeSO4·7H2O 0.05,CaCO33,pH 7.2;MB6:蔗糖70,黄豆饼粉14,酵母提取物2.5,NaCl 0.1,CaCO31,K2HPO40.5,MOPS(丙磺酸) 10,pH 7.2;MB7:玉米粉65 ,黄豆饼粉10,酵母粉1,K2HPO40.5,α-淀粉酶0.5,pH 7.2;MB8:葡萄糖60,酵母提取物2,NaCl 2,K2HPO40.5,MgSO4·7H2O 0.1,(NH4)2SO42,FeSO4·7H2O 0.05,MnSO4·4H2O 0.05,ZnSO4·7H2O 0.05,CaCO35,pH 7.2;⑤固体发酵培养基:NO.1:麦芽糖20,葡萄糖10,酵母粉4,棉子粉7.5,青岛琼脂20, pH 7.0;NO.2:蔗糖40,燕麦粉7.5,番茄酱5,CaCO33,青岛琼脂20,pH 7.0;NO.3:蔗糖20,黄豆饼粉10,玉米浆10,KCl 8,青岛琼脂20, pH 6.5;NO.4:葡萄糖4,麦芽抽提物10,CaCO32,青岛琼脂20,pH 7.0;NO.5:蔗糖20,黄豆饼粉10,玉米浆10,KCl 8,青岛琼脂20,pH 6.5;NO.6:酵母提取物4,麦芽提取物10,葡萄糖4,青岛琼脂20,pH 8.0;NO.7:葡萄糖10,黄豆饼粉10,米浆10,甘油5,酵母粉5,NaCl 5,CaCO32,青岛琼脂20,pH 7.0;NO.8:可溶性淀粉20,子粉5,酵母抽提物2.5,NaCl 1,K2HPO40.75,MgSO4·7H2O 1,CaCO33,青岛琼脂20,pH 7.5。
1.1.3 仪器设备 PCR扩增仪(德国Eppendorf公司);Genenap凝胶成像系统(美国Syngene公司);台式冷冻离心机Sorvall Legend MICRO 17(美国Thermo公司);恒温培养振荡器ZHWY-2102C(武汉大风生物科技有限公司);旋转蒸发仪BUCHI Rotavator R-200 (瑞士BUCHI公司);液质联用仪Thermo Scientific LTQ Orbitrap (美国Thermo 公司);高压灭菌器HVE-50(日本HIRAYAMA公司)。
1.2 方法
1.2.1 引物设计 根据已公布的冰城链霉菌全基因组,分别设计扩增16S rDNA、I型PKS 加载域以及C-5氧甲基转移酶基因的引物(表1)。
1.2.2 米贝链霉菌DSM41911的初步鉴定 采用Chelex 100法提取链霉菌DSM41911的基因组DNA,采用1.2.1中设计的引物分别对16S rRNA、I型PKS加载域以及C-5氧甲基转移酶的基因进行PCR扩增,将PCR产物连接到pMD19-T载体上,转至DH10B感受态细胞中,挑取阳性单克隆,提取质粒后由南京金斯瑞公司测序,测序结果在NCBI进行BLAST 比对。

表1 基于冰城链霉菌全基因组设计的引物
1.2.3 米贝链霉菌DSM41911的发酵培养 液体发酵根据培养基的不同进行的发酵天数不同,首先接种100 μL的孢子液于装有50 mL的种子液培养基中,待菌体生长一定时间后以1∶10的比例转接发酵培养。对于种子液培养基和产孢培养基进行7 d培养,MB1、MB2、MB3、MB5、MB7培养基进行5、6、7、8 d培养,MB4、MB6、MB8培养基进行5、6、7 d培养。对于固体发酵,所有培养基均进行5、7、10 d发酵。
1.2.4 发酵产物的处理 对液体培养基:离心使上清和菌丝体分开,分别收集上清和菌丝体,上清冷冻干燥,用色谱级甲醇溶解制样;菌丝体用V(甲醇)∶V(丙酮)=1∶1振荡,超声30 min进行细胞破碎,过滤,滤液旋干,用甲醇溶解制样。对固体培养基:将培养基切碎,甲醇浸泡5 h,过滤,滤液旋干后用甲醇溶解制样。
1.2.5 发酵产物LC-MS检测 HPLC分离条件:Dikma 公司的Diamonsil C18反相柱(250 mm×4.6 mm,5 μm),柱温:25 ℃,洗脱条件:A为0.1%的乙酸水溶液,B为乙腈。0~3 min:5%B~20%B,3~15 min:20%B~60%B,15~20 min:60%B~80%B,20~35 min:80%B~20%B;流动相的流速为800 μL/min,样品进样量为10 μL。发酵产物MS-ESI检测参数:离子源:电喷雾ESI;雾化气压力:0.8×105Pa;毛细管电压:4.5 kV;干燥气:N2;干燥气流速:8 L/min;干燥气温度:275 ℃;扫描分子量范围:200~1 000。
2 结果与分析
2.1 米贝链霉菌DSM41911基因水平的验证
在NCBI上的比对结果显示米贝链霉菌DSM41911的16S rRNA基因序列与冰城链霉菌的相似性为99%,同源性较高(见表2);且功能基因I型PKS加载域序列与冰城链霉菌完全一致,MilD同源性高达99%(见表3),表明米贝链霉菌DSM41911具有生产米尔贝霉素的潜力。而对于合成途径中其他功能基因由于在冰城链霉菌全基因组的报道中并没有明确的指出,所以暂时也无法设计引物对其他基因进行特异性扩增。
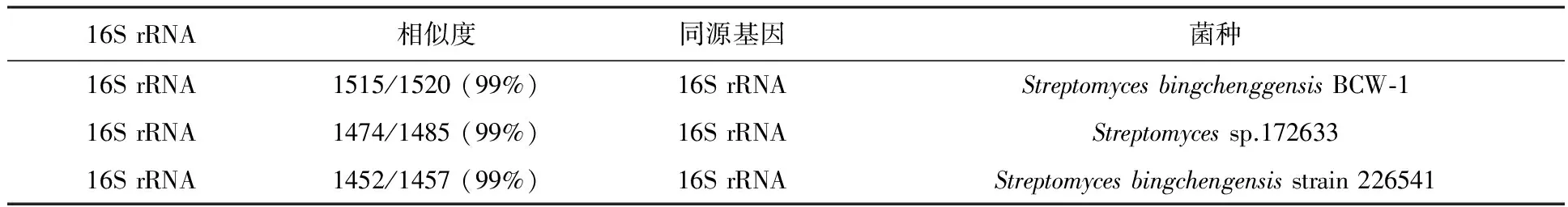
表2 16S rRNA同源比对结果
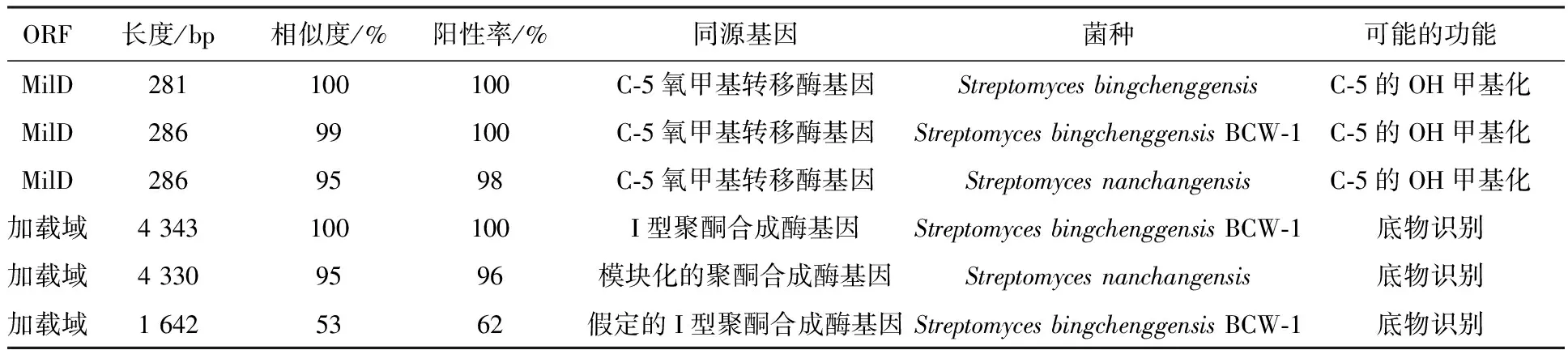
表3 MilD和加载域的同源比对结果
2.2 米贝链霉菌DSM41911发酵结果分析
对米贝链霉菌DSM41911进行了大量固体和液体发酵验证,在固体发酵培养基中均未检测出米尔贝霉素产物;对液体发酵培养基产物进行LC-MS数据分析,对于培养5、6、7 d的液体发酵培养基MB4、MB6、MB8,在发酵7 d的MB4菌丝体中检测到milbemycin B5[M+Na]+(如图1,Δppm值为-6.0),在发酵5 d的MB6菌丝体中检测milbemycin E[M+Na]+(如图2,Δppm值为-7.7)、milbemycinVM44864[M+H]+(如图3,Δppm值为-3.6)和milbemycin B1 [M+H]+(如图4,Δppm值为-3.2)。但是在培养6、7、8 d的液体发酵培养基MB1、MB2、MB3、MB5、MB7中均未检测到米尔贝霉素产物,对此批培养基进行5 d的液体发酵对比,仍未检测出米尔贝霉素产物。未检测到milbemycin组分的培养基,可能是米尔贝霉素产量过低,也可能是受培养基成分影响未产生米尔贝霉素。
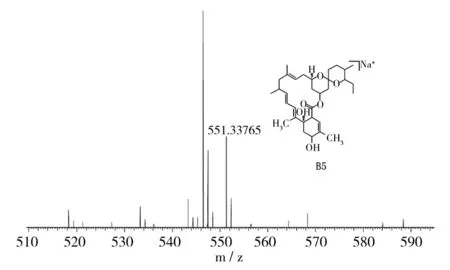
图1 MB4发酵7 d LC-MS检测结果Fig.1 LC-MS testing result of MB4 fermentation for seven days
2.3 米贝链霉菌生物合成基因簇和生物合成途径的推测
米尔贝霉素属于I型PKS的阿维菌素的结构类似物,其生物合成基因簇中包含一条特异性强的PKS途径。根据东北农业大学向文胜教授课题组公布的冰城链霉菌的生物合成基因簇(如图5)和米尔贝霉素类似物阿维菌素(Avermectin)和伊维菌素(Ivermectin)的合成途径[13-14],我们以milbemycin D为对象,对其生物合成途径做出如下推测:米尔贝霉素通过Ⅰ型聚酮合酶(PKS typeⅠ)途径合成,该聚酮合酶由1个起始模块和12个延伸模块组成,分别由milA1、milA2、milA3和milA4四个基因编码。其生物合成过程:首先起始模块加载1分子异丁酸形成异丁酰-ACP,在延伸过程中先后加载7分子乙酸和5分子丙酸,经最后一个延伸单元上的硫酯酶(TE)催化,脂肪长链从ACP上解离并由milC催化闭合成环,再经过MilF(酮基还原酶)、MilE(细胞色素P450羟化酶)、MilD(C-5氧甲基转移酶)催化最终形成milbemycin D(如图6)。
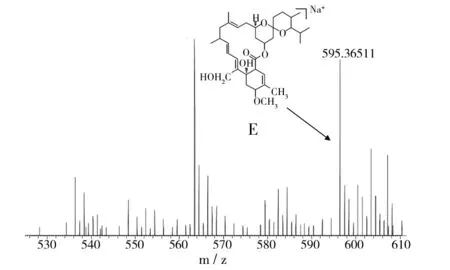
图2 MB6发酵5 d LC-MS检测结果(I)Fig.2 LC-MS testing result of MB6 fermentation for five days(I)
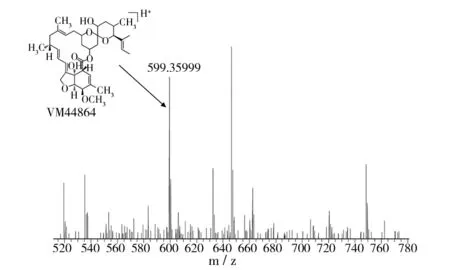
图3 MB6发酵5 d LC-MS检测结果(II)Fig.3 LC-MS testing result of MB6 fermentation for five days(II)
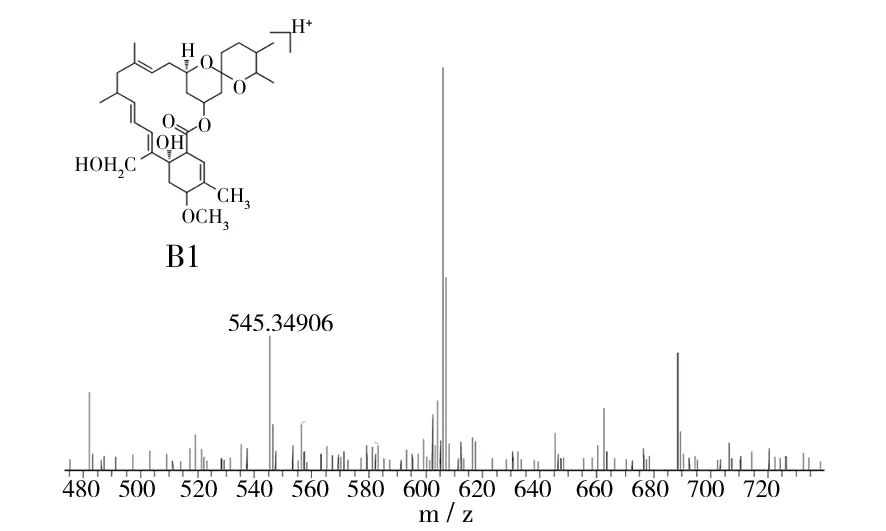
图4 MB6发酵5 d LC-MS检测结果(III)Fig.4 LC-MS testing result of MB6 fermentation for five days(III)
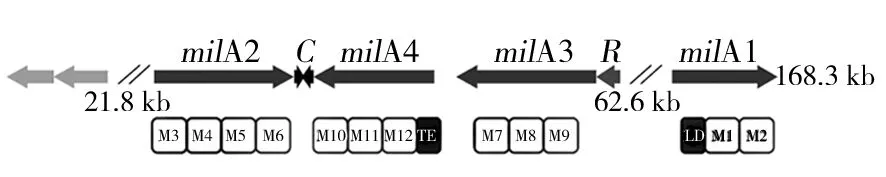
图5 米尔贝霉素生物合成基因簇的推测Fig.5 The conjecture on Biosynthetic gene cluster of milbemycin
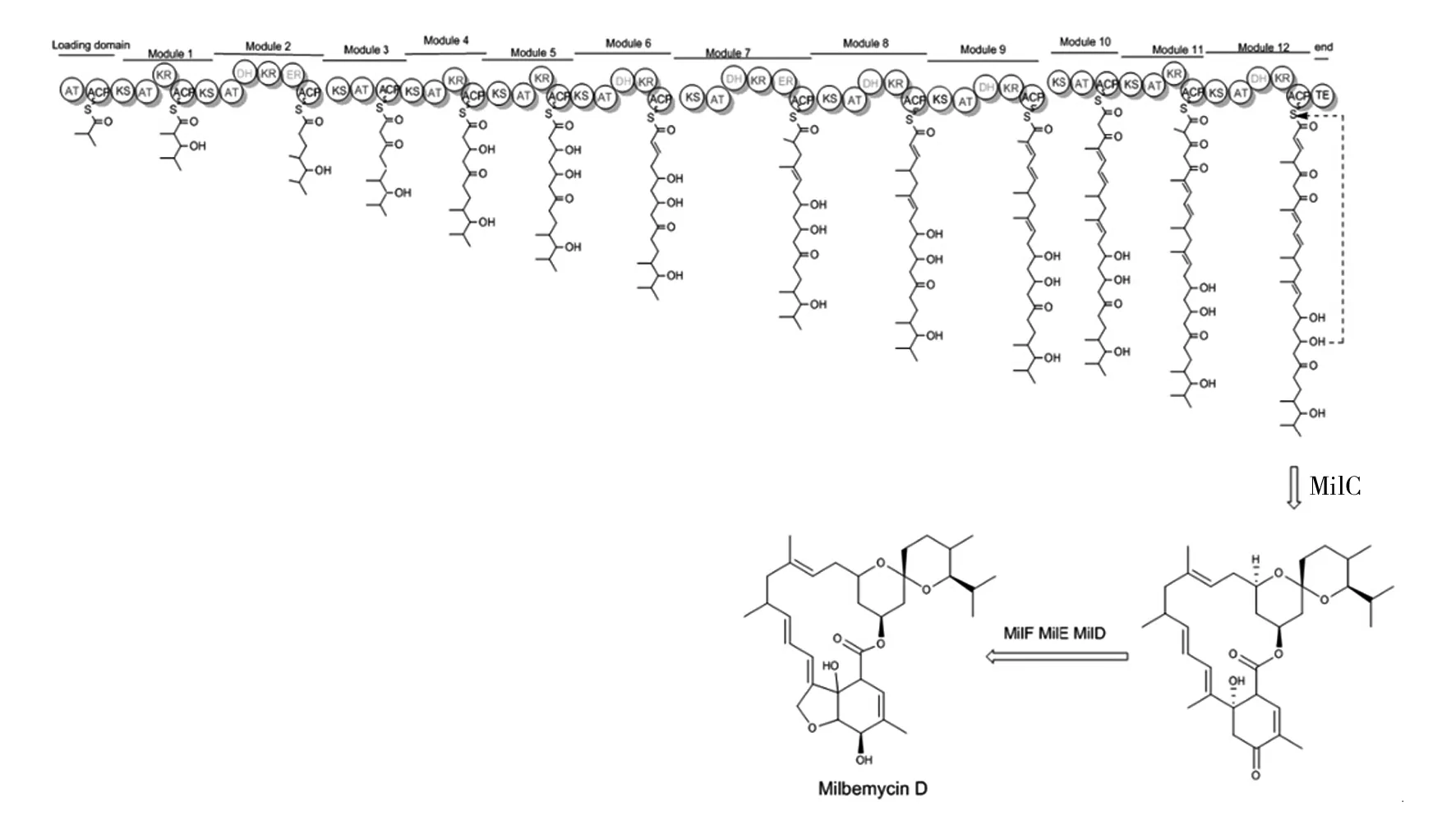
图6 米尔贝霉素生物合成途径的推测Fig.6 The conjecture on biosynthetic pathway of milbemycin
3 讨 论
目前米尔贝霉素类药品已经在世界范围内实现了商业化,米尔贝霉素发酵也成为国内研究的热点领域。本研究通过对特异性较高的16S rRNA、I型PKS加载域和MilD基因测序并进行生物信息学比对,发现米贝链霉菌DSM41911与冰城链霉菌同源性较高。同时对其进行一系列固、液发酵,得到milbemycin B5、milbemycin B1、milbemycin VM4486 和 milbemycin E等4种米尔贝霉素,初步确认菌株的发酵条件。结合冰城链霉菌的合成基因簇和米尔贝霉素类似物阿维菌素(Avermectin)和伊维菌素(Ivermectin)的合成途径推测出米尔贝霉素的生物合成途径,为后续高产菌株的筛选提供理论依据,为米尔贝霉素在国内的工业生产打下基础。
[1] 陈小龙,王远山,郑裕国. 生物农药米尔贝霉素的研究进展[J]. 农药,2003,42(4):5-9.
[2] Nonaka K,Tsukiyama T,Okamoto Y,et al. New milbemycins fromStreptomyceshygroscopicussubsp.aureolacrimosus: fermentation,isolation and structure elucidation[J]. J Antibiot(Tokyo),2000,53(7):694-704.
[3] 林秀萍,刘永宏,李季伦. 米尔贝霉素的产生菌、理化性质、生物学活性及应用研究进展[J].中国抗生素杂志,2013,38(4):314-320.
[4] Li L, Zhou D,Chen A, et al. Two new α-class milbemycin metabolites from mutantStreptomycesavermitilisNEAU1069-3[J].J Antibiot(Tokyo),2015,68(5):354-356.
[5]McKellar QA,Benchaoui HA. Avermectins and milbemycins[J].J Vet Pharmacol Ther,1996,19(5):331-351.
[6]Shoop WL,Mrozik H,Fisher MH. Structure and activity of avermectins and milbemycins in animal health[J].Vet Parasitol, 1995, 59(2):139-156.
[7] Deng Y,Casida JE. House fly head GABA-gated chloride channel: toxicologically relevant binding site for avermectins coupled to site for ethynylbicycloorthobenzoate[J].Pestic Biochem Physiol,1992,43(2):116-122.
[8] 张宝新,王相晶,向文胜. 米尔贝霉素毒理学研究进展[J]. 世界农药,2009,31(4):11-12.
[9] 夏广欣,刘重喜,王相晶,等. 微生物源农药米尔贝霉素的应用[J].世界农药,2012,33(3):35-39.
[10]陈园,张晓琳,黄颖,等. 杀虫抗生素的研究进展[J].农业生物技术学报,2014,22(11):1455-1462.
[11]Xiang WS,Wang JD,Wang XJ,et al.Two new beta-class milbemycins fromstreptomycesbingchenggensis: fermentation, isolation, structure elucidation and biological properties[J].J Antibiot(Tokyo),2007,60(6):351-356.
[12]Wang XJ,Yan YJ,Zhang B,et al. Genome sequence of the milbemycin-producing bacteriumStreptomycesbingchenggensis[J].J Bacteriol,2010,192(17):4526-4527.
[13]Ikeda H,Nonomiya T,Usami M,et al. Organization of the biosynthetic gene cluster for the polyketide anthelmintic macrolide avermectin inStreptomycesavermitilis[J].Proc Natl Acad Sci U S A,1999,96(17):9509-9514.
[14]Ji Zhang,Yi Jun Yan,Jing An,et al. Designed biosynthesis of 25-methyl and 25-ethyl ivermectin with enhanced insecticidal activity by domain swap of avermectin polyketide synthase[J].Microb Cell Fact,2015,24(1):152-163.
Preliminary Study on Milbemycin Biosynthesis ofStreptomycesmilbemycinicusDSM41911
LYU Zhen-bin, ZENG Xiao-dong, TIAN Xiao-dong, ZHAO Chang-ming, YU Yi, DUAN Jian-li, DENG Zi-xin
(KeyLab.ofCombinat’lBiosynth.andDrugDiscov.,Minist.ofEducat’n,Schl.ofPharm.Sci.,WuhanUni.,Wuhan430071)
StreptomycesmilbemycinicusDSM41911 was carried out initial study, through bioinformatics analysis of 16S rDNA sequence and sequence of other functional genes indicated that there were a high homology possessed betweenS.milbemycinicusDSM41911 andS.bingchenggensis. 16 different kinds of solid and liquid media were designed for screening suitable for fermentation condition of the strain, milbemycin B5, milbemycin B1, milbemycin VM4486 and milbemycin E were successfully detected in MB4 and MB6 medium. A possible biosynthesis pathway of milbemycin inS.milbemycinicusDSM41911 was speculated referring to synthetic gene ofS.bingchenggensiscombined with literatures, providing theoretic foundations for the screening of milbemycin high-production strains.
milbemycin; fermentation; biosynthesis; 16S rRNA
教育部高等学校博士学科点专项科研基金项目(20110141120017);武汉市科技局应用基础研究计划项目(2013020501010176)
吕振斌 男,硕士研究生。研究方向为生物制药。E-mail:zhenbinlv@163.com
* 通讯作者。男,博士,讲师。研究方向为生物制药。E-mail:kitty_fen@126.com
2016-01-25;
2016-03-11
Q936
A
1005-7021(2016)06-0024-05
10.3969/j.issn.1005-7021.2016.06.004
