糙柱花草遗传多样性SSR分析
2016-01-15丁西朋罗小燕张龙王文强白昌军
丁西朋+罗小燕+张龙+王文强+白昌军


摘 要 为鉴定糙柱花草种质的遗传背景和亲缘关系,提高其利用效率,利用来自不同柱花草种中的16个SSR标记对14份糙柱花草种质进行遗传多样性和聚类分析。结果表明:在16个SSR标记中,10个SSR标记在14份糙柱花草种质间具有多态性。10个多态性SSR标记共检测到32个等位基因,每个标记可检测到2~8个等位基因,平均为3.20个;每个SSR标记的Shannon信息指数(I)和多态性信息含量(PIC)分别为0.191~0.796和0.173~0.769,平均为0.474和0.411。14份糙柱花草种质间的遗传相似系数为0.438~0.938,平均为0.733。聚类分析结果显示,在遗传相似系数为0.74处,14份供试糙柱花草种质可明显被分为3类,其中III类中的CPI 93116柱花草与其他种质遗传关系较远。
关键词 糙柱花草 ;种质资源 ;SSR标记 ;遗传多样性
分类号 S54
Stylosanthes scabra Genetic Diversity Analysis by SSR Markers
DING Xipeng LUO Xiaoyan ZHANG Long WANG Wenqiang BAI Changjun
(Institute of Tropical Crops Genetic Resources, CATAS, Danzhou, Hainan 571737)
Abstract To identify the genetic background and relationship of Stylosanthes scabra varieties and promote the further utilization of them, the genetic diversity of 14 S. scabra varieties was analyzed using 16 SSR markers developed from other different Stylosanthes species. Result: Among 16 SSR markers, 10 markers were polymorphic among 14 S. scabra materials. A total of 32 alleles were generated by the 10 polymorphic markers, and the number of alleles of SSR markers ranged from 2 to 8 with an average of 3.20 alleles. The Shannon's information index and polymorphic information content each polymorphic SSR marker varied from 0.191 to 0.796 and 0.173 to 0.769, with an average of 0.474 and 0.411, respectively. The genetic similarity coefficients of 14 S. scabra materials were from 0.438 to 0.938 with an average of 0.733. The cluster analysis showed that the 14 S. scabra materials could be obviously divided into 3 clusters at genetic similarity coefficient of 0.74. The lower genetic relation was presented between S. scabra CPI 93116 in cluster III and other S. scabra materials.
Keywords Stylosanthes scabra ;germplasm ; SSR marker ; genetic diversity
糙柱花草具有很强的抗旱性,是热带地区放牧草地应用最广泛的柱花草种[1]。西卡柱花草(Styosanthes scabra‘Seca)是由中国热带农业科学院热带作物品种资源研究所引进选育而成的优良糙柱花草品种,是华南干旱、半干旱地区人工草地建设和天然草地改良的首选草种,在海南、云南、广东、广西、四川、贵州、福建等省区累计推广面积超过1万hm2[2-3]。糙柱花草为异源四倍体,其二倍体祖先为粘质柱花草(Styosanthes viscosa)和灌木柱花草(S. seabrana)[4]。糙柱花草为典型的自花授粉植物,异交率仅为1%~2%[5]。目前为止,对糙柱花草种质资源遗传多样性研究的报道非常少。Maass[6]通过对糙柱花草的形态指标及农艺性状经常考察及聚类分析,最终将糙柱花草资源分为4类;Liu[1]利用21对RAPD标记将93份糙柱花草资源分为5类。SSR标记作为一种重要的分子标记技术,具有数量丰富、多态性高、共显性、操作简单等优点,已经被广泛于遗传学研究的诸多领域[7]。在柱花草中已报道的可用SSR标记共139个,其中44个来自圭亚那柱花草、26个来自大头柱花草(S. macrocephala)、23个来自灌木柱花草、23个来自头状柱花草(S. capitata),其他的来自其他14个不同的柱花草种[8-14],这些SSR标记主要应用于柱花草种质资源遗传多样性分析和核心种质的构建。Chandra等[8]利用开发的41个SSR标记对灌木柱花草的20份种质进行了遗传多样性分析。Santos-Garcia等[15]利用20个SSR标记对圭亚那柱花草资源进行了遗传多样性及聚类分析,将150圭亚那柱花草分为9 类。Santos-Garcia等[16]利用SSR标记对134份大头柱花草种质和192份头状柱花草种质进行了遗传多样性分析,构建了分别含有23份大头柱花草和13份头状柱花草的核心种质。但SSR标记在糙柱花草种质资源中的开发及应用尚未见报道。本课题组对123个在其他柱花草种中开发的SSR标记进行筛选,结果获得了79个在糙柱花草中能有效扩增的SSR标记[17]。在本研究中,利用其中16个在糙柱花草中能有效扩增、条带清晰且多态性较好的SSR标记对14份糙柱花草种质资源的遗传背景及亲缘关系进行鉴定,为糙柱花草资源的引进及利用提供理论依据。
1 材料与方法
1.1 材料
本研究所用14份糙柱花草种质包含3个栽培品种(西卡柱花草、费特罗雷柱花草和斯伦柱花草)和其他11份资源(表1),其中3份糙柱花草种质为中国热带农业科学院(CATAS)热带作物品种资源研究所收集,6份引自澳大利亚国际热带农业研究中心(ACIAR),5份引自哥伦比亚国际热带农业中心(CIAT)。14份糙柱花草种质资源均种植在中国热带农业科学院热带作物品种资源研究所热带牧草研究中心基地,按照日常大田管理办法进行管理。
1.2 SSR扩增与分析
采集糙柱花草苗期的新鲜叶片置于液氮中,按本实验室改良CTAB 法提取柱花草的基因组DNA[17],然后用1%琼脂糖凝胶电泳和NanoVue超微量分光光度计(GE Healthcare)检测DNA的质量和浓度,将DNA稀释至50 ng/μL后置于-20℃备用。从张龙等[17]筛选获得的在8个不同柱花草种中都能有效扩增且条带清晰的SSR标记中,挑选16个SSR标记对14份糙柱花草种质资源进行遗传多样性分析(表2),引物由上海生工生物工程公司合成。PCR扩增条件及程序根据丁西朋等[18]描述的方法进行,采用20 μL的PCR反应体系:50 ng/μL模板DNA 1 μL,5 μmol/μL上下游混合引物0.8 μL,2.5 μmol/μL dNTPs 1.2 μL,10×PCR Buffer 2 μL,5 U/μL Taq聚合酶0.2 μL,最后用ddH2O补足。PCR反应程序为:94℃预变性4 min;94℃变性40 s,55℃退火40 s,72℃延伸45 s,35个循环;最后72℃延伸10 min。扩增产物用8% PAGE(Polyacrylamide Gel Electrophoresis,聚丙烯酰胺凝胶电泳)电泳后,采用硝酸银染色法进行显影,最后拍照记录。
1.3 数据分析
以DL2000 DNA marker作为扩增产物片段大小的参考,对大小合适、清晰且易于辨认的谱带采用“0,1”系统记录,无条带记为“0”,有条带记记为“1”。将记录的原始数据汇总到Excel表格中生成数据矩阵,用于扩增条带的多态性比较和聚类分析。利用NTSYS-pc 2.10e软件分析糙柱花草种质间的遗传相似系数(Genetic similarity coefficient,GS),然后根据GS值采用非加权组平均法(Unweighted Pair-group Method with Arithmetic Means,UPGMA)对供试糙柱花草材料进行聚类分析[19]。利用POPGENE 1.32软件计算每个位点的遗传多样性参数,包括等位基因数、等位基因频率和Shannon信息指数(I)[20]。每个位点的多态性信息含量(Polymorphic Information Content,PIC)按如下公式进行计算:
PIC=1-(Pi2)-2Pi2Pj2
其中,n表示每个位点检测到的等位基因数目,Pi、Pj表示第i、j个等位基因在供试材料中出现的频率[21]。
2 结果与分析
2.1 SSR扩增结果
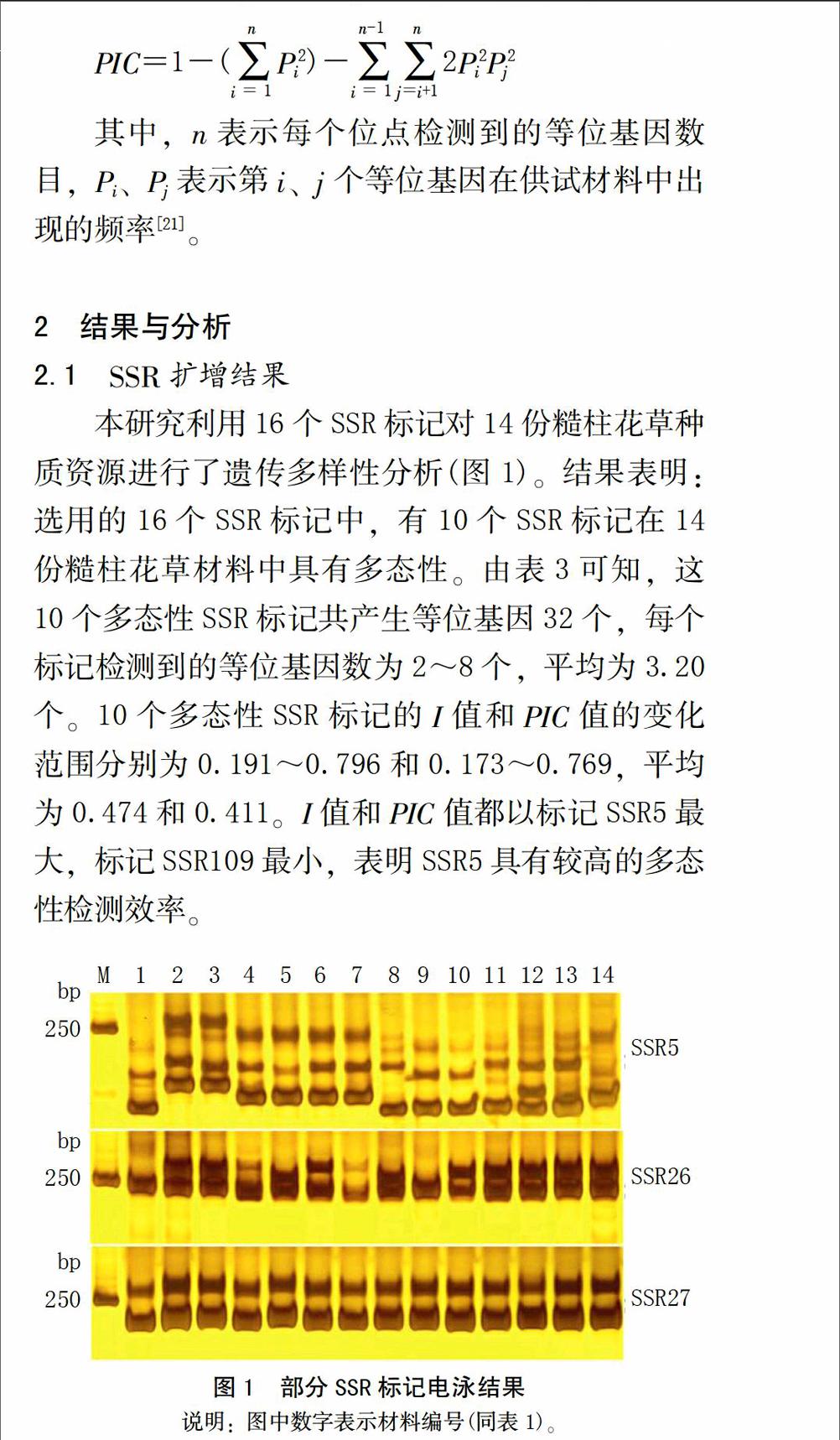
本研究利用16个SSR标记对14份糙柱花草种质资源进行了遗传多样性分析(图1)。结果表明:选用的16个SSR标记中,有10个SSR标记在14份糙柱花草材料中具有多态性。由表3可知,这10个多态性SSR标记共产生等位基因32个,每个标记检测到的等位基因数为2~8个,平均为3.20个。10个多态性SSR标记的I值和PIC值的变化范围分别为0.191~0.796和0.173~0.769,平均为0.474和0.411。I值和PIC值都以标记SSR5最大,标记SSR109最小,表明SSR5具有较高的多态性检测效率。
2.2 遗传多样性分析结果
基于10个多态性SSR标记的带谱,利用NTSYS-pc 2.10e对14份糙柱花草种质资源进行了遗传多样性分析。结果表明,14份糙柱花草种质资源间的遗传相似系数GS为0.375~0.938,平均为0.733(表4)。其中CPI 33260与费特罗雷柱花草(0.938)、CIAT 1077与CIAT 1080(0.938)的遗传相似性最大。而费特罗雷柱花草(0.375)、TPRC250(0.438)、CPI 33260(0.438)、Q10042(0.469)、CIAT 1077(0.469)和CIAT 1080(0.469)与CPI 93116的遗传相似系数最低,均在0.500以下。从各糙柱花草种质间平均相似系数来看,CPI 93116与其他材料的平均相似系数最低,为0.488,表明CPI 93116与其他种质的亲缘关系最远;其次为品153柱花草,为0.685。其余材料的平均相似系数均大于0.700,其中Q10042、CIAT 1077和CIAT 1080最高,达0.796。
2.3 聚类分析结果
由图2可知,14份糙柱花草种质资源在相似系数为0.74处可明显分为3大类群。第I类群包括10份材料,在相似系数为0.80处可分为2个亚类:I-A亚类包含6份材料,其中西卡柱花草和CIAT 66聚在一起,CPI 40292、CIAT 1077、CIAT 1077和CIAT 1080聚在一起;I-B亚类包含4份材料,其中Q10042与斯伦柱花草的亲缘关系较近,而CPI 33260与费特罗雷柱花草的亲缘关系更近。第II类群包括3份材料,在相似系数为0.80处,品153和TPRC 250可归为II-A亚类,而CIAT 1355单独归为II-B亚类。第III类群仅1份材料CPI 93116,这份材料同其他供试材料的遗传关系最远。
3 讨论与结论
分子标记是作物遗传学研究和分子育种的重要工具,相对于其他基于PCR的分子标记,SSR标记具有数量丰富、多态性高、共显性、操作简单、重复性好等优点,这使SSR技术在种质资源遗传多样性分析、指纹图谱构建、基因定位及遗传图谱的构建等多个领域得以应用[7]。糙柱花草是热带地区重要的放牧型豆科牧草,当前糙柱花草SSR标记的缺失极大地限制了人们对糙柱花草分子水平的认识。SSR标记的开发一般需要大量的碱基序列作为基础,然而SSR引物在近缘种间具有一定的通用性,因此,对遗传学基础较弱的物种,近缘物种转移法已成为一种成本低廉、简便快速获得SSR标记的有效途径[22]。钟敏等[23]通过对1 205个绿豆SSR标记进行筛选,获得了603、883和983个分别在豇豆、小豆和饭中可用的SSR标记。Sharma等[24]研究结果表明,98个水稻SSR标记和20个甘蔗SSR标记在竹子中的通用性比率分别为44.9%和75%。本课题组对123个在其他柱花草种中开发的SSR标记进行了筛选,获得了79个在糙柱花草中能有效扩增的SSR标记[17]。利用其中16个SSR标记对14份糙柱花草种质资源的遗传背景及亲缘关系进行鉴定,结果表明,在16个SSR标记中,有10个SSR标记在14份糙柱花草材料中具有多态性,每个多态性标记可检测到2~8个等位基因,PIC值的变化范围为0.173~0.769,平均为0.411。利用这10个多态性SSR标记能够有效地对14份糙柱花草种质资源进行区别鉴定,表明利用近缘物种转移法获得的糙柱花草SSR标记不仅具有较高的多态性,且能够成功用于糙柱花草种质资源的遗传多样性分析。
由于柱花草属植物分布广泛、种类繁多、植物学特征复杂多变,所以基于传统的形态学特征分类方法很难对柱花草属内种和亚种进行区分鉴定,所以许多学者通过分子标记方法对柱花草种间遗传关系进行了研究[25-26]。如灌木柱花草和糙柱花草有着非常相似的生态学特征,包括生长习性、荚果性状、均为多年生等[27],所以根据形态特征很难对它们进行区分,直到2002年,灌木柱花草才被确定为一个新种[28]。Chandra等[27]利用STS标记和流式细胞仪从48份糙柱花草种质资源中鉴定出10份灌木柱花草。Liu[1]利用RAPD标记对100份糙柱花草资源进行分析,从中分离鉴定出7个错误种质,将剩下的93份种质分为5个类群,说明相对于形态学指标,分子标记技术在种质资源鉴定中更有效。本研究利用16个SSR标记对14份糙柱花草种质进行了遗传多样性分析,将它们分为3个大类,5个小群,该结果与Liu的研究结果类似。14份糙柱花草种质的来源和聚类结果并不一致,这可能是不同地区种质资源互相交流的原因。
综上所述,对于遗传学基础较弱的糙柱花草,利用同属柱花草的SSR标记来分析糙柱花草的遗传信息,是一种成本低廉、简便快速的有效途径。本课题组利用近缘物种转移法从其他柱花草中获得了糙柱花草SSR标记,本研究将其中16个条带清晰且多态性较好的SSR标记成功应用于糙柱花草种质资源的遗传多样性分析,能有效地对糙柱花草资源进行区分鉴定。本研究结果将有助于糙柱花草种质资源遗传多样性、亲缘关系鉴定、基因发掘与定位及分子辅助植物育种等研究的开展。
参考文献
[1] Liu C J. Geographical distribution of genetic variation in Stylosanthes scabra revealed by RAPD analysis[J]. Euphytica, 1997, 98(1): 21-27.
[2] 白昌军,刘国道.西卡柱花草[J].农家顾问,2004,1(1):28-29.
[3] 白昌军,刘国道,王东劲,等.西卡柱花草选育及其利用评价[J].草地学报,2004,12(3):170-175.
[4] Liu C J, Musial J M. Stylosanthes sp. aff. S. scabra:a putative diploid progenitor of Stylosanthes scabra (Fabaceae)[J]. Plant Systematics and Evolution, 1997, 208(1): 99-105.
[5] Stace H M. Breeding systems in Stylosanthes. I. Observations of outcrossing in S. scabra at an alcohol dehydrogenase locus[J]. Australian Journal of Agricultural Research, 1982, 33(1): 87-96.
[6] Maass B. The tropical forage legume Stylosanthes scabra Vog.-variability, performance and possibilities for improvement by plant breeding[D].Gottingen: Universitat Gottingen, 1989.
[7] Kalia R K, Rai M K, Kalia S, et al. Microsatellite markers:an overview of the recent progress in plants[J]. Euphytica,2011,177(3):309-334.
[8] Chandra A,Tiwari K K,Nagaich D,et al. Development and characterization of microsatellite markers from tropical forage Stylosanthes species and analysis of genetic variability and cross-species transferability[J]. Genome, 2011, 54(12): 1 016-1 028.
[9] Santos M O, Karia C T, Resende R M, et al. Isolation and characterization of microsatellite loci in the tropical forage legume Stylosanthes guianensis(Aubl.)Sw[J]. Conservation Genetics Resources,2009, 1(1): 43-46.
[10] Vander S J, Weltjens I, Volckaert G. Microsatellite markers in Stylosanthes guianensis[J]. Molecular Ecology, 1999, 8(3): 514-517.
[11] Santos M O, Sassaki R P, Ferreira T H S, et al. Polymorphic microsatellite loci for Stylosanthes macrocephala Ferr. et Costa, a tropical forage legume[J]. Conservation Genetics Resources, 2009, 1(1): 481-485
[12] Santos M O, Sassaki R P, Chiari L, et al. Isolation and characterization of microsatellite loci in tropical forage Stylosanthes capitata Vogel [J]. Molecular Ecology Resources, 2009, 9(1): 192-194.
[13] Billotte N, Lagoda P J R, Risterucci A M, et al. Microsatellite-enriched libraries:applied methodology for the development of SSR markers in tropical crops[J]. Fruits, 1999, 54(2): 277-288.
[14] Mace E S, Varshney R K, Mahalakshmi V, et al. In silico development of simple sequence repeat markers within the aeschynomenoid/dalbergoid and genistoid clades of the Leguminosae family and their transferability to Arachis hypogaea, groundnut[J]. Plant Science, 2008, 174(1): 51-60.
[15] Santos-Garcia M O, Karia C T, Resende R M, et al. Identification of Stylosanthes guianensis varieties using molecular genetic analysis[J]. AoB Plants, 2012: pls001.
[16] Santos-Garcia M O, Toledo-Silva G, Sassaki R P, et al. Using genetic diversity information to establish core collections of Stylosanthes capitata and Stylosanthes macrocephala[J]. Genetic and Molecular Biology, 2012, 35(4): 847-861.
[17] 张 龙,丁西朋,严琳玲,等. 8种柱花草属牧草SSR-PCR反应体系优化及引物筛选[J]. 草业科学,2014,31(2):233-242.
[18] 丁西朋,罗小燕,邵辰光,等. Genomic-SSR和EST-SSR在柱花草种间的遗传差异[J]. 广东农业科学,2015,42(14):106-113.
[19] Rohlf F J. NTSYS-pc Numerical taxonomy and multivariate analysis system, version 2.10[M]. New York:Exeter Software, 2000.
[20] Yeh F C, Yang R C, Boyle T. POPGENE, version 1.32: the user friendly software for population genetic analysis[M]. Canada:Molecular Biology and Biotechnology Centre, University of Alberta, Edmonton, AB, 1999.
[21] Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. The American Journal of Human Genetics, 1980, 32(3): 314-331.
[22] 邓绍勇,温 强,李康琴,等. 大青属植物通用性SSR引物筛选及应用[J]. 中草药,2014,45(22):3 317-3 322.
[23] 钟 敏,程须珍,王丽侠,等. 绿豆基因组SSR引物在豇豆属作物中的通用性[J]. 作物学报,2012,38(2):223-230.
[24] Sharma R K, Gupta P, Sharma V, et al. Evaluation of rice and sugarcane SSR markers for phylogenetic and genetic diversity analyses in bamboo[J]. Genome, 2008, 51(2): 91-103.
[25] Liu C J, Musial J M, Thomas B D. Genetic relationships among Stylosanthes species revealed by RFLP and STS analyses[J]. Theoretical and Applied Genetics, 1999, 99(7-8): 1 179-1 186.
[26] Vander S J, Marant S, Volckaert G. Molecular characterization and phylogenetic utility of the rDNA external transcribed spacer region in Stylosanthes(Fabaceae)[J]. Theoretical and Applied Genetics, 2003, 107(2): 291-298.
[27] Chandra A, Kaushal P. Identification of diploid Stylosanthes seabrana accessions from existing germplasm of S. scabra utilizing genome-specific STS markers and flow cytometry, and their molecular characterization[J]. Molecular Biotechnology, 2009, 42(3): 282-291.
[28] Maass B L, Mannetje L. Stylosanthes seabrana(leguminosae:papilionoideae), a new species from Bahia, Brazil[J]. Missouri Botanical Garden, 2002, 12(4): 497-500.
