基因芯片检测鱼类病毒的方法建立与优化
2015-12-16王胜强耿伟光粟子丹史成银
王胜强,耿伟光,李 晋,粟子丹,史成银
(1.农业部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所,山东青岛 266071;2.上海海洋大学水产与生命学院,上海 201306)
基因芯片检测鱼类病毒的方法建立与优化
王胜强1,2,耿伟光1,2,李 晋1,2,粟子丹1,2,史成银1
(1.农业部海洋渔业可持续发展重点实验室 中国水产科学研究院黄海水产研究所,山东青岛 266071;2.上海海洋大学水产与生命学院,上海 201306)
本研究建立并优化了一种基因芯片检测方法,可以同步检测7种重要的海水养殖鱼类病毒(淋巴囊肿病毒、细胞肿大病毒属虹彩病毒、赤点石斑鱼神经坏死病毒、传染性造血器官坏死病毒、传染性胰脏坏死病毒、病毒性出血败血症病毒、传染性鲑贫血病毒)。该基因芯片包含10条病毒特异性的寡核苷酸探针(25~30 mer)分别与病毒的CP、N、VP5、G、NS、MA等基因位点互补。将各病毒基因探针以50% DMSO稀释至20 μmol/L,使用PersonalArrayer 16个人点样仪在醛基修饰玻片上点样,然后与Cy3标记的扩增产物在47℃条件下杂交1.5 h,最后在LuxScan 10K扫描仪上采集荧光信号、判断检测结果。初步应用表明,该基因芯片检测方法具有良好的特异性和可靠性,在鱼类病毒高通量检测技术领域有广泛的应用前景。
病毒;基因芯片;高通量检测;鱼类
建立对多种病毒的快速、高通量、低成本的分子检测方法,对保障养殖鱼类健康、防止疾病发生具有重要的意义。淋巴囊肿病毒(lymphocystis disease virus,LCDV)、大菱鲆红体病虹彩病毒(turbot reddish body iridovirus,TRBIV)、细 胞肿大病毒属虹彩病毒(Megalocytivirus,Mega)、赤点石斑鱼神经坏死病毒(red-spotted grouper nervous necrosis virus,RGNNV)、传染性造血器官坏死病毒(infectious haematopoietic necrosis virus,IHNV)、传染性胰脏坏死病毒(infectious pancre-
atic necrosis virus,IPNV)、病毒性出血败血症病毒(viral hemorrhagic septicemia virus,VHSV)、传染性鲑鱼贫血病毒(infectious salmon anaemia virus,ISAV)是养殖鱼类主要的病毒性病原,均能引起传染性、暴发性疾病。国内外关于这些病毒的毒力基因分析以及分子检测已有较多的文献报道。其中LCDV、TRBIV、Mega和RGNNV都含有衣壳蛋白(CP),在病毒颗粒中负责包被病毒核酸和核酸-蛋白质复合体,由于不同病毒的CP基因序列相似性较低,因此该基因可用于这些病毒的特异性鉴定[1-3]。核蛋白基因(N)是IHNV的主要毒力基因之一,通常被用于IHNV的分子鉴定以及其毒性强弱的判断[4]。VP5是IPNV的主要毒力基因之一,因此VP5基因可作为检测IPNV的生物学标记[5]。糖蛋白基因(G)是VHSV的主要毒力基因之一,常被用于VHSV的分子鉴定以及其毒性强弱的判断[6]。核蛋白NS、基质蛋白MA是ISAV的主要毒力基因,因此NS、MA基因可作为检测ISAV的生物学标记[7]。
基因芯片作为生物芯片的一种,最初是在20世纪80年代初由Bains W.等人提出的概念。目前是在生命科学领域应用的最为广泛也最为成熟的一种对大量的遗传信息进行高效、快速的检测和分析的方法。在表达谱的构建[8]、筛选相关基因[9]、临床诊断上都有广泛的研究和应用[10]。基因芯片在临床医学检测上的应用已有相关的报道,但在鱼类病毒的检测方面研究很少。本研究针对上述鱼类病毒,建立了基因芯片检测方法,对其反应参数进行优化并进行了初步应用,现将结果报道如下。
1 材料与方法
1.1 材料
1.1.1 含病毒基因的克隆质粒。含有LCDV、TRBIV、Mega、RGNNV、IHNV、IPNV、VHSV、ISAV-NS、ISAV-MA相关基因的T-A克隆质粒和菌株均由本实验室构建,用作基因芯片检测的阳性模板(各基因的GenBank检索号见表1)。
1.1.2 主要试剂、耗材。实验中所用到的Trans-StartTMTop Taq DNA Polymerase、High Pure dNTPs购自北京全式金生物技术有限公司,海洋动物组织基因组DNA提取试剂盒购自天根生化科技(北京)有限公司,质粒小量快速提取试剂盒购自博迈德生物,氨基、醛基修饰片基购自博奥生物。
1.2 方法
1.2.1 病毒基因阳性模板的制备。分别将含有LCDV、TRBIV、Mega、RGNNV、IHNV、IPNV、VHSV、ISAV-NS、ISAV-MA等病毒相关基因的克隆菌株过夜培养。对扩大培养的菌液提取质粒,用超微量核酸蛋白测定仪(美国Nano-Drop2000)测定核酸的浓度和纯度,保存于-20 °C备用。
1.2.2 基因芯片探针的设计。各病毒探针的设计方法是:依据病毒特异性的基因序列,使用AlleleID 7.0软件设计寡核苷酸探针(25 ~ 30 mer),使其分别与病毒的CP、N、VP5、G、NS、MA等基因位点互补。此外,在各探针5′端连上一段长度为15 mer的poly(dT)作为间隔臂,以减少探针与片基之间的空间位阻,提高其杂交效率(检测各鱼类病毒的探针序列见表1)。

表1 用于基因芯片的鱼类病毒探针序列
本研究还设计了基因芯片的3套质控探针,即表面化学质控探针、阳性对照探针、阴性对照探针。表面化学质控探针(QC-1):随机'写出一段寡核苷酸序列(5'-ACAAGGGATATCGCTGGGT-3'),该序列不产生稳定的二级结构,与病原基因序列也没有同源性。阳性对照探针(QC-2):根据牛的β-珠蛋白基因(HBB)设计扩增引物和阳性对照探针,扩增得到的标记产物与阳性对照探针杂交产生阳性信号。阴性对照探针(QC-3):根据牛的β-珠蛋白基因(HBB)进行阴性探针的设计。另外设置一套空白对照(QC-4),即以ddH2O代替探针点样。
1.2.3 基因芯片和Cy3标记产物的制备。芯片制备:将各探针以50% DMSO稀释至20 μmol/L,操作PersonalArrayer 16 个人点样仪点样于醛基修饰玻片,每种探针并排点3份,即设置3个平行。将玻片置于湿盒中室温放置4 h,0.5% SDS清洗10 min,超纯水洗涤2 min,离心后4℃保存。采用多重PCR技术对鱼类病毒的相关基因片段进行扩增,并对扩增产物进行同步Cy3标记。
1.2.4 杂交体系组成。杂交体系总体积15 μL:含100%甲 酰 胺3.75 μL,20×SSC 2.25 μL,10% SDS 0.3 μL,50×Denhardt’s 1.5 μL,Cy3标记产物7.2 μL。
1.2.5 氨基片基和醛基片基对比。氨基片基探针的固定:将未修饰的LCDV、RGNNV探针以50% DMSO稀释至20 μmol/L,点样后将玻片置于80°C烘箱80 min,浸入超纯水中1 min,再迅速浸入-20 °C无水乙醇中1 min,超纯水清洗2 min,离心后4 °C保存。醛基片基探针的固定:将5′端带有氨基修饰的LCDV、RGNNV探针按1.2.3制备流程进行探针的固定。最后取LCDV、RGNNV的Cy3标记产物按1.2.4配制成杂交液与基因芯片进行杂交、清洗和扫描,分析结果。
1.2.6 不同探针点样液的对比。取LCDV、RGNNV、IHNV、IPNV氨基修饰的探针,分别用50% DMSO、3×SSC和ddH2O稀释至20 μmol/L,进行醛基片基点样。取等量LCDV、RGNNV、 IHNV、IPNV的Cy3标记产物,按1.2.4配制杂交液,进行杂交、清洗,最后用LuxScan 10K扫描仪扫描,分析结果。
1.2.7 芯片杂交探针浓度的优化。取LCDV、RGNNV、IHNV、IPNV的氨基修饰探针以50% DMSO分别稀释成20、15、10、5 μmol/L,进行醛基片基点样。取等量LCDV、RGNNV、IHNV、IPNV的 Cy3标记产物,按1.2.4配制杂交液,进行杂交、清洗和扫描,分析结果。
1.2.8 芯片杂交温度的优化。取LCDV、RGNNV、IHNV、IPNV的氨基修饰探针点样,取等量LCDV、RGNNV、IHNV、IPNV的Cy3标记产物,按1.2.4配制交体系,每次取一张基因芯片进行杂交,每次的杂交温度分别设定为37 °C、42 °C、47 °C、52 °C、57 °C,最终清洗、扫描,分析结果。
1.2.9 芯片杂交时间的优化。取LCDV、RGNNV、IHNV、IPNV的氨基修饰探针点样,取等量LCDV、RGNNV、IHNV、IPNV的Cy3标记产物,按1.2.4配制杂交体系,取四张芯片进行杂交,杂交过程中每隔0.5 h取出一张芯片进行清洗,最终进行芯片的扫描,分析结果。
1.2.10 优化后芯片的整体杂交效果。根据上述优化得到的最佳反应参数,对LCDV、TRBIV、Mega、RGNNV、IHNV、IPNV、VHSV、ISAVNS、ISAV-MA进行Cy3标记产物的制备、与芯片进行杂交、清洗和扫描,评价杂交效果。
1.2.11 基因芯片对病鱼样品的检测。取患病鱼的脑、脾、肾等组织,分别提取DNA和RNA。对DNA和反转录生成的cDNA进行多重PCR扩增,制备Cy3标记产物。将扩增产物按照1.2.10所述进行基因芯片检测,分析检测结果。同时使用常规PCR方法进行病毒检测,作为对比。
2 结果与分析
2.1 氨基片基与醛基片基的杂交效果对比
醛基片基的点样结果与氨基片基相比,点阵更加均一,杂交信号强度明显高于氨基片基(图1)。表明醛基片基的探针结合能力更强,因此本研究选用醛基片基进行基因芯片的制备。

图1 醛基片基(左)和氨基片基(右)对杂交结果的影响
2.2 不同点样液的对比
通过对不同点样液制备的芯片进行杂交、清洗及扫描,结果显示:50% DMSO效果最好,该点样液的点圆润均一,杂交信号最强;而3 × SSC作为点样液杂交信号强度较弱,且部分点出现扩散、大小不均一的情况;ddH2O作为点样液的杂交结果几乎看不到信号(图2)。因此选用50 % DMSO作为基因芯片制备的点样液。
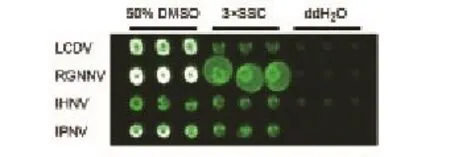
图2 点样液对杂交结果的影响
2.3 芯片杂交探针浓度的优化
通过对不同浓度探针制备的芯片进行杂交、清洗及扫描,结果显示:随着探针浓度的逐渐降低,杂交信号强度与杂交信号均值大致呈下降趋势。由于各探针的点样均一程度等问题,个别杂交点出现了低浓度探针杂交信号高于高浓度探针杂交信号的情况(图3)。综合比较,探针终浓度为20 μmol/L时杂交效果最好,因此选用该浓度的探针用于基因芯片的检测。
2.4 杂交温度的优化
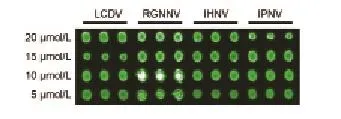
图3 探针浓度对杂交结果的影响
当杂交温度为37 °C、42 °C、47 °C时,样品的荧光信号均清晰可见。随着温度的升高,信号强度也相应增强,在47 °C时达到最强。当杂交温度升高至52 °C时,信号强度急剧下降。其中LCDV和RGNNV的信号强度很弱;当杂交温度升高至57 °C时,IHNV和IPNV的信号强度也变得很弱(图4)。因此最终选择47 °C作为基因芯片的最佳杂交温度。

图4 不同杂交温度的结果
2.5 杂交时间的优化
当杂交时间为0.5 h时,几乎看不到样品的荧光信号;杂交1.0 h后,荧光信号有所增强。随着杂交时间延长至1.5 h时,荧光信号强度也达到了峰值。继续延长杂交时间至2.0 h时,荧光信号强度并无明显增加(图5)。综合考虑,选用1.5 h作为基因芯片的最佳杂交时间。
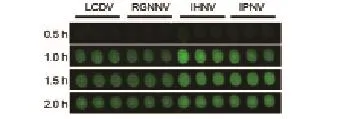
图5 不同杂交时间的结果
2.6 参数优化后的基因芯片杂交效果
制备鱼类病毒相关基因Cy3标记产物之后,应用上述优化后的反应参数,进行基因芯片的杂交。结果显示:病毒探针均能与对应的Cy3标记产物杂交,并产生均匀的荧光信号,整体杂交效果非常理想(图6)。

图6 整体杂交结果
2.7 对病鱼样品的检测结果
在对病鱼样品的实际检测中,该基因芯片对本实验室收集到的19份样品的检测结果为:养成期牙鲆中检出LCDV的感染,养成期大菱鲆中检出TRBIV的感染,在半滑舌鳎鱼苗中检出RGNNV的感染,在石斑鱼中检出细胞肿大属(Mega)病毒(图7)。该检测结果与使用常规PCR的检测结果完全一致。
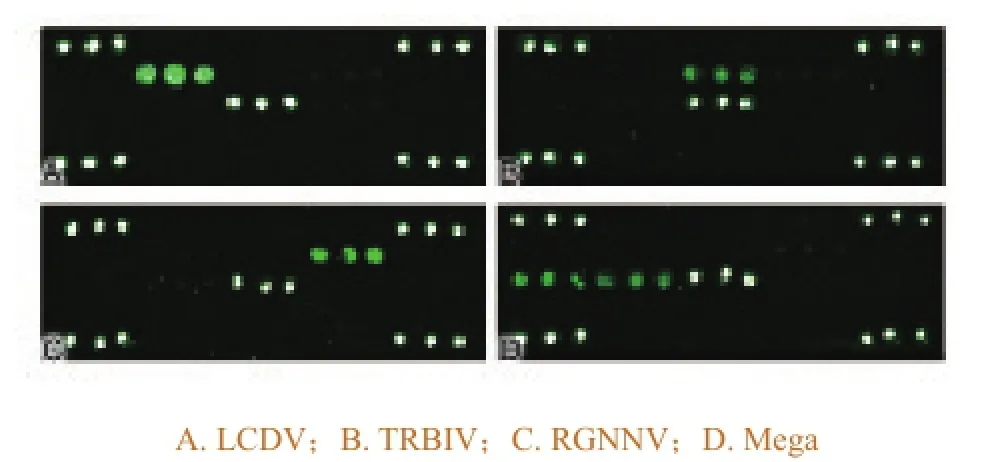
图7 应用基因芯片在19份鱼类样品中检测出的4份病毒阳性结果
3 讨论
在鱼类的养殖过程中,多种病毒病的发生往往会带来巨大的经济损失,严重影响了养殖业的健康、可持续发展,因此研发快速、可靠、高通量的病毒检测方法具有重要价值。生物芯片技术已被广泛应用在基因工程和临床医学等领域[9,11],在水产方面的研究和应用也日益增多,比如在水母分型及进化地位[12]、贝类致病性弧菌[13]、对虾致病菌[14]、鱼类病原菌[15-16]等方面,但在鱼类病毒方面的研究和应用却很少。本研究建立了基因芯片技术检测鱼类病毒的方法,优化了反应参数,得到了稳定、清晰的杂交结果,并在鱼类病毒检测中的进行了初步应用。
本研究建立的基因芯片检测技术能够在一个微阵列中与7种病毒10个基因的标记扩增产物同时进行杂交,并得到理想的杂交效果,这在很大程度上提高了重要海水养殖鱼类病原筛查工作的效率,与已报道的仅能同时检测2种鱼类病毒的基因芯片相比具有明显的优势[17]。在探针设计方面,本研究在每条探针的5'端连上一段长度为15 mer的poly(dT)作为间隔臂,从而有效地降低了探针与片基间的空间位阻,提高了标记产物与其杂交的效率。总之,本研究建立的基因芯片检测鱼类病毒的方法,具有通量高、特异性强的优势,为鱼类的多病原检测及流行病调查提供了快速、有效的工具,对保障养殖鱼类健康、防治疾病发生具有重要的意义。
本研究的不足之处是对建立并优化的基因芯片方法应用不足,今后需要使用更多的样品对该基因芯片进行验证和测试,以便持续改进。
[1] 闫秀英,吴灶和,简纪常,等. 淋巴囊肿病毒中国分离株基因组序列和结构分析[J]. 广东海洋大学学报,2011,31(1):1-6.
[2] 吴成龙,史成银,黄倢,等. 大菱鲆红体病虹彩病毒主要衣壳蛋白基因在毕赤酵母中的重组分泌表达[J]. 渔业科学进展,2009(3):55-61.
[3] Choi Y R,Kim H J,Lee J Y,et al. Chromatographicallypurifi ed capsid proteins of red-spotted grouper nervous necrosis virus expressed in Saccharomyces cerevisiae form virus-like particles[J]. Protein Expression and Purifi cation,2013,89(2):162-168.
[4] 韩硕,赵前程,吴斌,等. 传染性造血器官坏死病毒毒株核蛋白基因片段的克隆及序列分析[J]. 大连海洋大学学报,2011,26(3):232-237.
[5] Hong J,Gong H,Wu J. IPNV VP5,a novel anti-apoptosis gene of the Bcl-2 family,regulates Mcl-1 and viral protein expression[J]. Virology,2002,295(2):217-229.
[6] Martinez-Lopez A,Garcia-Valtanen P,Ortega-Villaizan M,et al. VHSV G glycoprotein major determinants implicated in triggering the host type I IFN antiviral response as DNA vaccine molecular adjuvants[J]. Vaccine,2014,32(45):6012-6019.
[7] Aspehaug V,Falk K,Krossøy B,et al. Infectious salmon anemia virus(ISAV)genomic segment 3 encodes the viral nucleoprotein(NP),an RNA-binding protein with two monopartite nuclear localization signals(NLS)[J]. Virus Research,2004,106(1):51-60.
[8] Duggan D J,Bittner M,Chen Y,et al. Expression profi ling using cDNA microarrays[J]. Nature genetics,1999,21:10-14.
[9] 赵国红,王晟,贾银华,等. cDNA芯片筛选亚洲棉短绒分化发育相关基因[J]. 中国农业科学,2010,43(2):430-437.
[10] Chen W,Xiang J,Chen D,et al. Screening for differentially methylated genes among human colorectal cancer tissues and normal mucosa by microarray chip[J]. Molecular Biology Reports,2013,40(5):3457-3464.[11] Cao X,Wang Y,Zhang C,et al. Visual DNA microarrays for simultaneous detection of Ureaplasma urealyticum and Chlamydia trachomatis coupled with multiplex asymmetrical PCR[J]. Biosensors and Bioelectronics,2006,22(3):393-398.
[12] Lee G,Park S Y,Hwang J,et al. Development of DNA chip for jellyfish verification from South Korea[J]. BioChip Journal,2011,5(4):375-382.
[13] Panicker G,Call D R,Krug M J,et al. Detection of pathogenic Vibrio spp.in shellfi sh by using multiplex PCR and DNA microarrays[J]. Applied and Environmental Microbiology,2004(12):7436-7444.
[14] 李晨,黄倢,谢国驷,等. 3种水产病原菌简型基因芯片检测技术的建立[J]. 中国海洋大学学报(自然科学版),2011,41(3):37-42.
[15] González S F,Krug M J,Nielsen M E,et al. Simultaneous detection of marine fi sh pathogens by using multiplex PCR and a DNA microarray[J]. Journal of Clinical Microbiology,2004(4):1414-1419.
[16] Warsen A E,Krug M J,LaFrentz S,et al. Simultaneous discrimination between 15 fi sh pathogens by using 16S ribosomal DNA PCR and DNA microarrays[J]. Applied and Environmental Microbiology,2004(7):4216-4221.
[17] 尹伟力,耿金培,刘宁,等. 液相芯片技术同时检测真鲷虹彩病毒和锦鲤疱疹病毒方法的建立[J]. 水产科学,2014(3):157-162.
(责任编辑:白雅娟)
Establishment and Optimization of Gene Microarray for Detection of Fish Viruses
Wang Shengqiang1,2,Geng Weiguang1,2,Li Jin1,2,Su Zidan1,2,Shi Chengyin1
(1. Key Laboratory of Sustainable Development of Marine Fisheries,Ministry of Agriculture,Yellow Sea Fisheries Research Institute,Chinese Academy of Fishery Sciences,Qingdao,Shangdong 266071;2. College of Fisheries and Life Science,Shanghai Ocean University,Shanghai 201306)
A gene microarray was established and optimized in the study for spontaneous detection of seven marine fi sh viruses in(lymphocystis disease virus,Megalocytivirus,red-spotted grouper nervous necrosis virus,infectious haematopoietic necrosis virus,infectious pancreatic necrosis virus,viral hemorrhagic septicemia virus and infectious salmon anemia virus). The microarray was composed of ten short oligonucleotide probes(25 ~ 30 mer)complementary to specifi c genes of above viruses. For gene microarrays,oligonucleotide probes(20 μmol/L in 50% DMSO)for these targeted genes were spotted onto aldehyde-masked slides by using a PersonalArrayer 16. Amplifi ed PCR products were hybridized to arrays at 47℃ for 1.5 h and detected by using signal amplifi cation with Cy3 fl uorescent dye. Slides were imaged by using a LuxScan 10K. The established gene microarray showed good specifi city and reliability by preliminary application,and could be widely used in high throughput detection of fi sh viruses.
virus;gene microarray;high throughput detection;Fish
S941.41;Q78
A
1005-944X(2015)07-0077-06
国家科技支撑计划课题(2012BAD17B01)
史成银
