枫香树DNA提取及SRAP-PCR反应体系的建立与优化
2015-06-27李芳芳杨少宗柳新红李海波王丽玲李永华
李芳芳, 杨少宗, 柳新红, 李海波, 王丽玲, 李永华
(1.河南农业大学,河南 郑州 450002;2.浙江省林业科学研究院,浙江 杭州 310023)
枫香树DNA提取及SRAP-PCR反应体系的建立与优化
李芳芳1,2, 杨少宗2, 柳新红2, 李海波2, 王丽玲2, 李永华1
(1.河南农业大学,河南 郑州 450002;2.浙江省林业科学研究院,浙江 杭州 310023)
通过不同处理的枫香树叶片及基因组DNA提取方法的对比,并采用L16(45)正交试验设计,对影响SRAP-PCR反应体系的Mg2+,dNTPs,引物浓度及Taq DNA聚合酶和模板DNA用量等5个因素进行优化,确立枫香树SRAP-PCR最佳反应体系。结果表明,改良CTAB法提取枫香树基因组DNA的产率和纯度均可满足下游试验需要。SRAP-PCR最优反应体系为:总体积20 μL,含 1×PCR Buffer,1.5 mmol·L-1Mg2+,0.16 mmol·L-1dNTPs,0.5 μmol·L-1引物,0.9 U Taq DNA聚合酶和40 ng模板DNA。各因素对反应结果的影响大小顺序为Taq DNA聚合酶量>dNTPs浓度>Mg2+浓度>模板DNA用量>引物浓度。运用优化的SRAP-PCR反应体系对10个不同居群的枫香树基因组DNA扩增检测,结果均能获得稳定性高、多态性丰富和重复性好的条带图谱。
枫香树;DNA提取;SRAP-PCR;正交试验设计;反应体系优化
枫香树(LiquidambarformosanaHance.)系金缕梅科(Hamamelidaceae)枫香树属落叶乔木,分布于秦岭淮河以南地区,跨北热带和南、中、北亚热带等4个气候带,是中国重要的乡土阔叶速生树种,集观赏、药用和材用等多种价值于一身。多年来,对枫香树的研究主要集中在无性繁殖技术[1-3]、人工林和混交林技术[4-6]、叶片的叶色分析[7]、种子生物学特性[8, 9]、枫香树林的生理生化研究及园林应用[10]等方面,而对其分子水平的遗传多样性研究较少,目前,仅见枫香树群体细胞学水平的同工酶[11]和特定群体的ISSR分析[12, 13]等。相关序列扩增多态性(Sequence Related Amplified Polymorphism,SRAP) 标记是2001年发展的一种新型分子标记技术[14],具有简便、快捷、共显性高、重复性好、易于分离条带及测序等优点,已成功用于多种植物,如棉花[15, 16]、桃和油桃[17]、西葫芦[18]、草坪草[19-21]、菊花[22]、一串红[23]、黄瓜[24]及槭属[25]和安息香属[26, 27]等,均显示其可作为遗传多样性评价、品种鉴定、遗传图谱构建和研究系统发生的有效工具[28]。该标记在金缕梅科的研究则未见报道。SRAP标记作为一种有效的分子标记技术,其重要前提条件是研究物种的基因组DNA高质量提取方法及其反应体系的优化和确立。本研究在传统小量法提取植物基因组DNA的CTAB方法基础上加以改良和优化,建立适合枫香树基因组DNA的提取方法:运用正交试验设计,对影响枫香树SRAP-PCR体系的Taq DNA聚合酶,Mg2+,dNTPs,引物和模板DNA用量等5种因素进行优化试验,获得适合枫香树SRAP-PCR反应的最优体系,并对此优化体系进行检测验证,以期为枫香树不同居群的遗传多样性分析、种质鉴定、遗传连锁图谱构建和分子育种等领域研究提供参考。
1 材料与方法
1.1 材料
1.1.1 试验材料 分别于2013年9月和2014年3月采集不同居群枫香树生长健壮、无病虫害的幼叶和老叶,置于保鲜袋中,于超低温冰箱中保存备用。
1.1.2 试剂设备 为Taq DNA聚合酶, Mg2+, dNTPs和10×Buffer等,均购自TaKaRa公司,其他试剂均为分析纯;引物合成由上海生物工程有限公司完成;主要仪器有Nano Drop 2000C微量核酸蛋白测定仪、ABI Veriti 96孔多重控温梯度PCR仪、BIO-RAD水平电泳系统及Syngene G: BOX凝胶成像系统等。
1.2 方法
1.2.1 枫香树基因组DNA的提取与检测 采用十六烷基三甲基溴化胺(CTAB)和试剂盒等方法[29-31],并改进试剂质量浓度和处理方法,用ddH2O溶解稀释至20 ng·μL-1,25 ℃保存备用。纯化后分别用1.5%的琼脂糖凝胶电泳和微量核酸蛋白测定仪对DNA进行定性和定量检测(表1)。
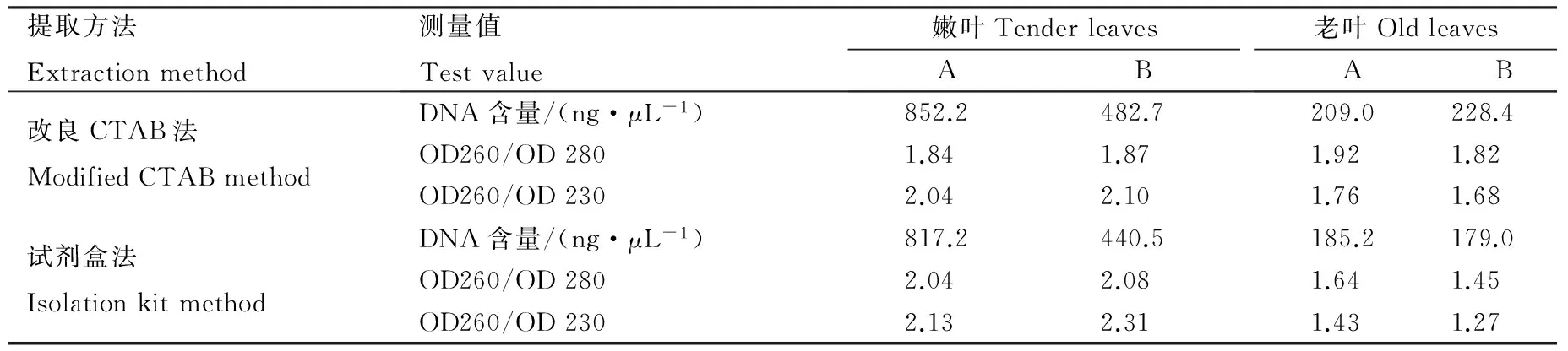
表1 不同提取方法的枫香树基因组DNA纯度和含量的测定结果Table 1 Determination of purity and content of genomic DNA of Liquidambar formosana by different methods
注:A.新鲜叶片;B.硅胶干燥处理叶片。
Note: A. Fresh leaves; B. Dried leaves saved in silica.
1.2.2 SRAP-PCR 反应体系的正交试验优化与检测根据盖钧镒等[32]的方法对Taq DNA聚合酶,Mg2+浓度,dNTPs浓度,SRAP引物质量浓度,模板DNA用量等5因素在4个水平上设计L16(45) 正交试验方案(表2)。
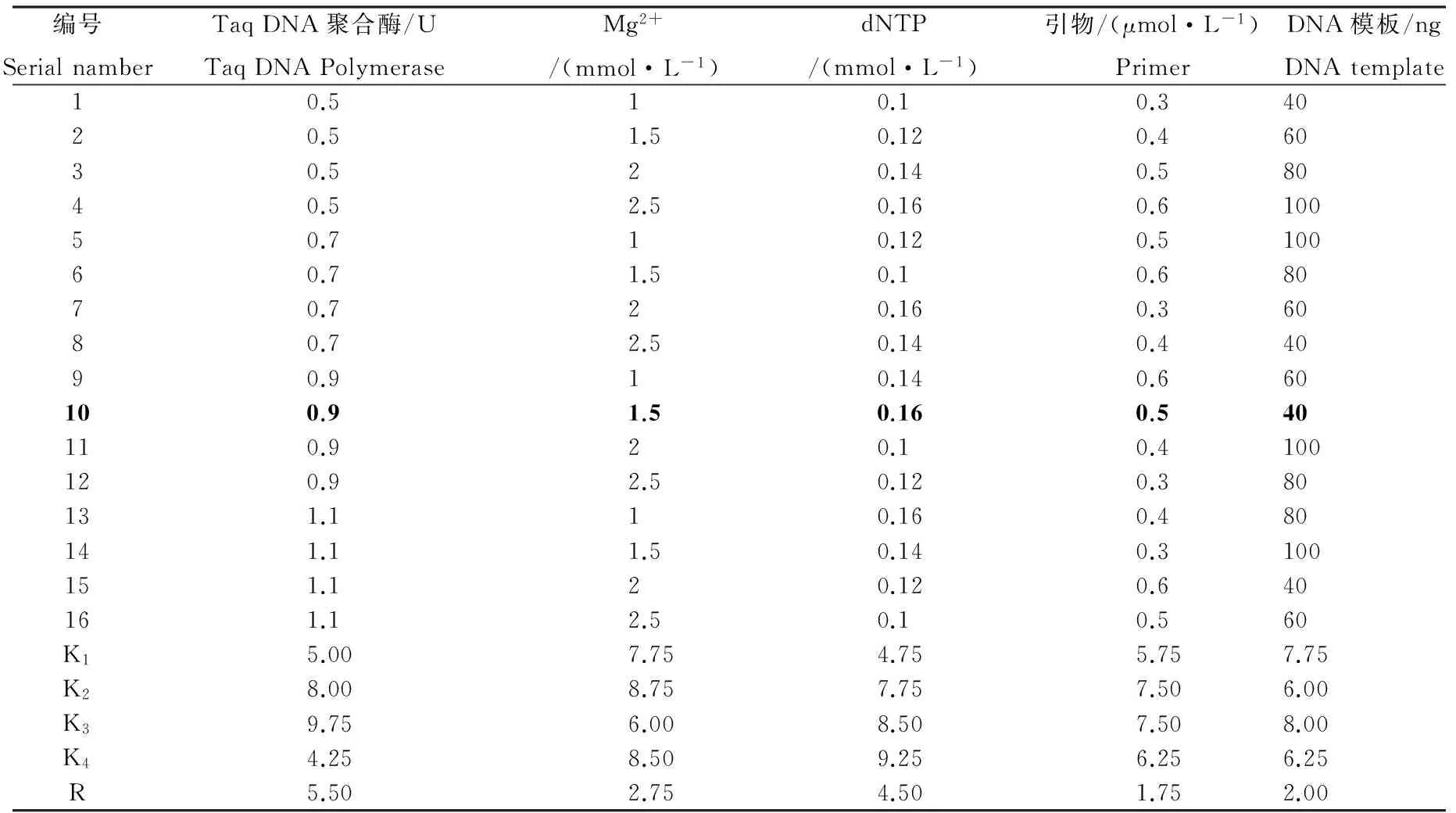
表2 枫香树SRAP-PCR反应因素水平L16(45)正交设计Table 2 Orthogonal design for SRAP-PCR optimization on Liquidambar formosana
其中,Taq DNA聚合酶用量分别为0.5、0.7、0.9、1.1 U;dNTPs浓度分别为0.1、0.12、0.14、0.16 mmol·L-1;Mg2+浓度分别为1、1.5、2、2.5 mmol·L-1;SRAP引物浓度分别为0.3、0.4、0.5、0.6 μmol·L-1;模板DNA含量分别为40、60、80、100 ng,反应总体积为20 μL。筛选SRAP引物Me3-Em11组合进行试验验证,3次重复。SRAP-PCR扩增程序为:95 ℃预变性5 min,94 ℃变性30 sec,35 ℃退火1 min,72 ℃延伸2 min,5次循环;94 ℃变性30 s,50 ℃ 变性30 s,72 ℃ 延伸1 min,30次循环;72 ℃延伸 10 min,4 ℃保存。扩增产物于1.5%的琼脂糖凝胶(含0.05%溴化乙锭)电泳检测,凝胶成像分析结果并拍照。
1.2.3 SRAP-PCR反应体系的稳定性检测 随机选择不同居群枫香树10个单株的基因组DNA为模板,筛选SRAP引物组合(Me3-Em11, Me8-Em8),按照上述最优SRAP-PCR反应体系扩增检测,对优化确立的枫香树SRAP-PCR反应体系的稳定性进行检验。
1.2.4 数据统计处理 采用正交直观分析法和新复极差法对所得数据进行处理分析[32, 33]。
2 结果与分析
2.1 2种方法提取枫香树基因组DNA的纯度和产率
不同提取方法、叶片发育时期和处理保存方式所提取的DNA质量和产率不同(表2)。由表2可知,对不同处理的嫩叶,试剂盒法所得DNA的OD260/OD280为2.04和2.08,改良CTAB法为1.84和1.87,2者OD260/OD230都接近于2,电泳条带明亮清晰,无明显弥散拖尾现象(图1)。
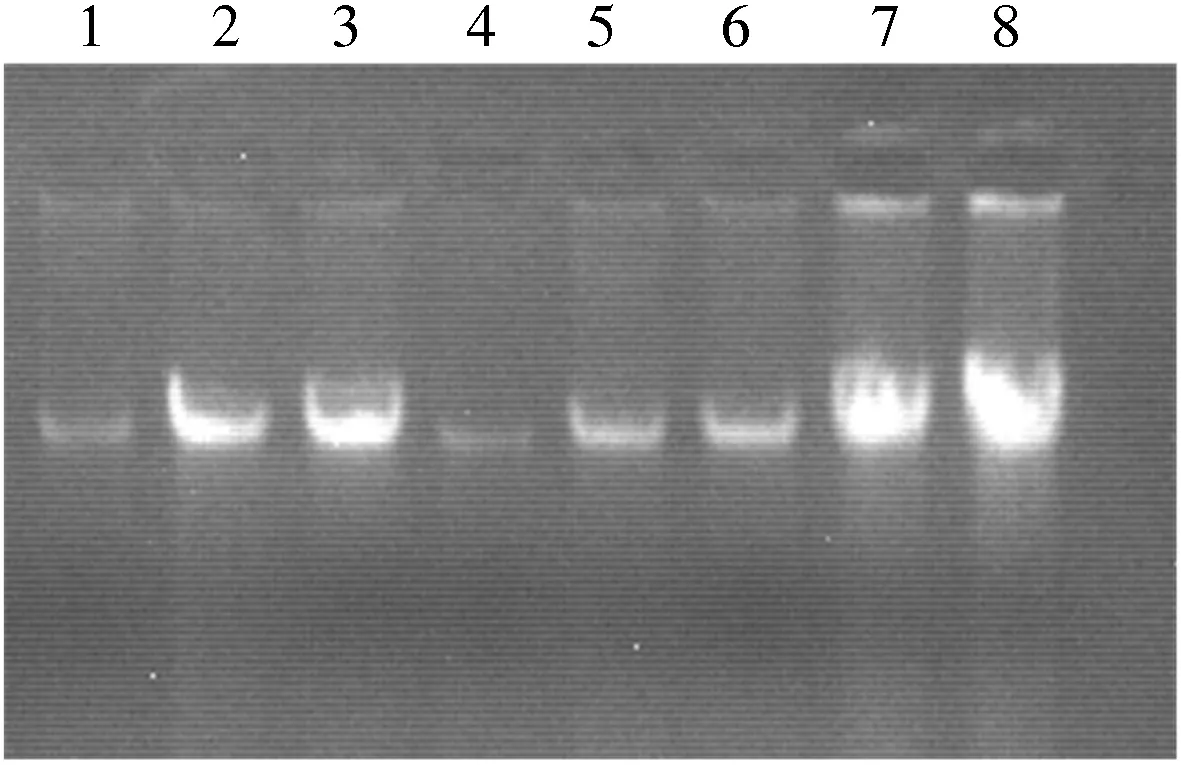
1,4,5,6:硅胶干燥处理和新鲜老叶(试剂盒法和改良CTAB法);2,3,7,8:硅胶干燥处理和新鲜嫩叶(试剂盒法和改良CTAB法)。
1,4,5,6: Dried old leaves saved in silica and fresh old leaves (Isolation kit method and modified CTAB method); 2,3,7,8: Dried tender leaves saved in silica and fresh tender leaves (Isolation kit method and modified CTAB method).
图1 不同提取方法的枫香树基因组DNA电泳检测图
Fig.1 Electrophoresis map ofLiquidambarformosanagenomic DNA extracted by different methods
2种方法提取的枫香树嫩叶DNA在含量和纯度上相差不大,均能满足后续PCR扩增要求。而对于老叶,改良CTAB法所得DNA的OD260/OD280为1.92和1.82,试剂盒法为1.64和1.45,结合OD260/OD230值分析表明,改良CTAB法提取老叶基因组DNA的产率和纯度均较试剂盒法高。后者不仅含量较低,且含有其他杂质,DNA降解较严重,将对下游PCR反应和其他DNA分子操作产生不利影响。
2.2 SRAP-PCR扩增体系的确立
2.2.1 正交试验结果分析 参照刘萌芽等[33]的方法,对枫香树基因组DNA的SRAP-PCR反应体系中Mg2+浓度, dNTPs浓度,引物浓度,Taq DNA聚合酶和模板DNA用量等设计5个因素4个水平组合的正交试验结果进行统计分析(表2)。R值结果表明,对枫香树基因组DNA的SRAP-PCR反应体系影响程度由大到小依次为:Taq DNA聚合酶,dNTPs浓度,Mg2+浓度,模板DNA用量和引物浓度;从K值来看,Taq DNA聚合酶用量为0.9 U,Mg2+浓度为1.5 mmol·L-1,dNTPs浓度为0.16 mmol·L-1,引物浓度在0.4~0.5 μmol·L-1范围都较好,模板DNA用量在80 ng最好。根据3次正交试验重复结果,初步确立枫香树基因组DNA的SRAP-PCR扩增的适宜反应体系应为:0.9 U Taq DNA聚合酶、1.5 mmol·L-1Mg2+、0.14 mmol·L-1dNTPs、80 ng模板DNA及0.4~0.5 μmol·L-1引物浓度。
按照表2正交试验设计的16个反应体系,随机选用Me3-Em11引物进行SRAP-PCR反应(图2),结果表明,采用不同浓度的Mg2+,dNTPs,引物及不同用量的Taq DNA聚合酶和模板DNA的16个反应体系,其扩增结果存在明显差异。第1、14、15和16号反应体系的扩增效果较差,条带弱且多态性也较低;第2、3、4、6、7、8、9、13号反应体系多态性好,但条带均较弱;第5、10、11、12号4个反应体系的扩增结果不仅条带清晰且多态性丰富。根据遗传多样性分析要求初步选定10号反应体系:0.9 U Taq DNA聚合酶、1.5 mmol·L-1Mg2+、0.16 mmol·L-1dNTPs、 0.5 μmol·L-1引物及40 ng模板DNA。
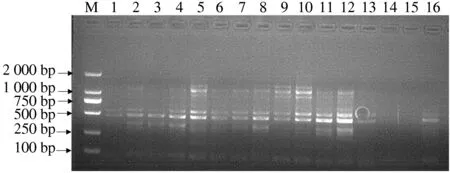
1-16:不同的SRAP-PCR反应体系; M:DL2000 DNA marker.下同。
1-16:SRAP-PCR amplification patterns in different reaction systems);
M:DL2000 DNA marker. The same as below.
图2 不同反应体系中枫香树SRAP-PCR扩增图谱(引物组合Me3-Em11)
Fig.2 SRAP-PCR amplification patterns in different reaction systems (primer combination Me3-Em11)
2.2.2 SRAP-PCR 扩增体系的确立及引物筛选
由表2,图2知,考虑扩增效果及试验成本,将正交试验的模板用量由80 ng调为40 ng,而将10号反应体系的引物浓度由0.5 μmol·L-1调为0.4 μmol·L-1。最终确定枫香树基因组DNA的SRAP-PCR最优反应体系为总体积20 μL中含有1×PCR Buffer, 1.5 mmol·L-1Mg2+, 0.16 mmol·L-1dNTP, 0.5 μmol·L-1引物, 0.9 UTaq DNA聚合酶和40 ng模板DNA。在该体系下随机选用枫香树单株基因组DNA进行SRAP引物筛选,按照多态性丰富、条带清晰和重复性好的原则,在96对SRAP引物组合中筛选出18对条带清晰、稳定且多态性丰富的引物,作为枫香树基因组的扩增引物组合(图3,表3)。
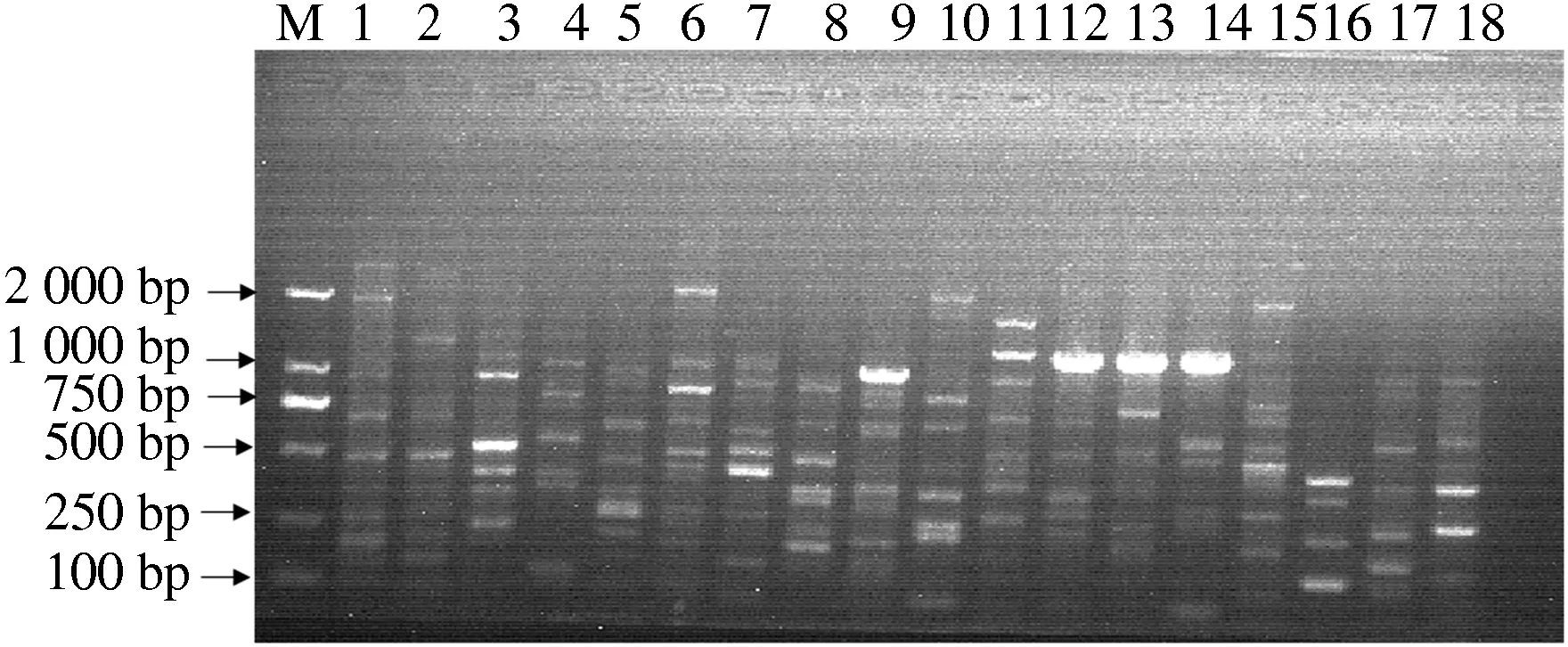
图3 筛选出的18对SRAP引物Fig.3 18 pairs of screened SRAP primers
表3 SRAP引物及序列Table 3 Primer sequence(5′-3′)applied for SRAP-PCR
2.3 SRAP-PCR反应体系稳定性分析
运用上述最优反应体系,随机选择SRAP引物组合Me3-Em11和Me8-Em8,在不同居群枫香树随机选择10个单株基因组DNA进行SRAP-PCR扩增(图4)。结果显示,随机选择的引物对随机选择的基因组DNA均可扩增出多态性丰富且条带清晰的DNA片段。说明该SRAP-PCR反应体系稳定可靠,适用于枫香树基因组DNA的SRAP-PCR扩增反应。

图4 枫香树基因组DNA 的SRAP-PCR扩增图谱Fig.4 SRAP-PCR amplificaion pattern of genomic DNA from L. formosana
3 结论和讨论
改良的CTAB法可获得含量、纯度和完整性较高的枫香树基因组DNA,且实用性和性价比较高。枫香树叶片中含有大量的萜类、黄酮类、苯丙素类、多糖类、酚酸类化合物及其苷类等次生代谢产物,并且这些物质的含量随叶片成熟逐渐增多[7, 34-37]。而这些物质易与DNA结合形成极难溶解的黏稠状物质,并抑制PCR反应过程中Taq DNA酶的活性,使扩增条带模糊甚至消失,严重影响PCR的反应效果。KIM等[38]研究表明,使用抗氧化剂PVP可以提高DNA的提取产率,陈大明等[30]利用细胞区室化多酚类物质来排除干扰也获得了较好的基因组DNA。改良CTAB法可以采用针对性的手段和步骤去除枫香树基因组DNA杂质,更适合枫香树叶片基因组DNA的提取,尤其是对于硅胶快速干燥处理的枫香树老叶来说,无论DNA的产率还是电泳结果都明显优于试剂盒法,这对于特定时间内大规模调查和采样来研究DNA分子水平上的枫香树群体遗传多样性来说,具有节省时间和经费的优势。枫香树基因组DNA的SRAP-PCR最优反应体系扩增结果显示其具有多态性丰富、条带清晰、重复性和稳定性好等特点。该标记克服了RAPD (Random Amplified Polymorphic DNA) 重复性差、SSR (Simple Sequence Repeat) 位点较少和AFLP (Amplified Fragment Length Polymorphism) 成本高等缺点。SRAP-PCR反应体系易受反应条件和扩增程序变化及物种的影响。对影响枫香树SRAP-PCR反应的5因素4水平的正交试验结果表明,Taq DNA聚合酶的用量直接影响SRAP-PCR扩增结果的重复性和稳定性:用量过低将降低反应的灵敏度,减少扩增量;用量过多则会降低反应的特异性,易出现非特异性扩增,且试验成本增高。dNTPs质量浓度是影响PCR反应的另一关键因素,该结果与樊洪泓等[39]对石斛属SRAP-PCR扩增体系的影响因素实验结果相一致,但与楚爱香[40]和任绪瑞等[41]研究结果不同,这可能是不同物种对SRAP-PCR反应体系中的影响因素敏感性差异所致。此外,多数研究结果表明引物浓度对扩增结果的影响很小[42],也与本研究结果一致。由此可知,不同物种的基因组结构和组成有其自身的特点,从而对PCR扩增体系的敏感性有所差异,既有相似的影响因素,也有各自特殊的反应条件。因此,应对不同的物种设计不同的PCR反应条件。
以不同居群枫香树的10个随机选择个体基因组DNA为材料,采用正交试验设计建立适宜于枫香树SRAP-PCR扩增的最优反应体系,并分别利用2对SRAP引物组合进行稳定性检测。结果表明,本试验建立的枫香树SRAP-PCR反应体系稳定,可靠,可进一步应用于枫香树群体遗传多样性分析、种质鉴定、遗传连锁图谱构建和分子育种等方面研究。
[1] 陈登雄,李玉蕾,池丽月. 枫香播种苗培育技术研究[J]. 福建林学院学报,1998,18(1):1923.
[2] 林昌礼,温丽娜. 枫香不同基质扦插实验研究[J]. 林业科技开发,2012 (5):11-12.
[3] 龚 峥,王洪峰. 枫香组织培养快繁育苗技术研究[J]. 广州林业科技,2012 (5):45-50.
[4] 黄勇来. 枫香与不同树种混交林的生长及生物生产力研究[J]. 西南林学院学报,2006 (2):15-18.
[5] 易利萍,文仕知,王珍珍,等. 枫香人工林的生物产量及生产力[J]. 中南林业科技大学学报,2008(2):50-53.
[6] 徐高福,肖建宏,毛显锋. 枫香人工造林技术与效果初报[J]. 浙江林业科技,2000 (2):39-42.
[7] 李效文,陈秋夏,郑 坚,等. 枫香秋叶变化与环境因子关系研究[J]. 浙江亚热带作物通讯,2011(1):29-33.
[8] 高捍东,陈凤毛,施季森,等. 枫香种子成熟期的研究[J].南京林业大学学报,2005 (3):26-28.
[9] 刘 就,刘和平,陈考科,等. 枫香种子形状研究进展[J]. 福建林业科技,2007(2):190-192.
[10]江 聂,姜卫兵,翁忙玲,等. 枫香的园林特性及其开发利用[J]. 江西农业学报,2008,20(12):46-49.
[11]柴国峰,郑勇奇,王良桂,等. 枫香同工酶遗传多样性研究[J]. 林业科学研究,2013,26(1):15-20.
[12]毕泉鑫,金则新,李钧敏,等. 枫香自然种群遗传多样性的ISSR分析[J]. 植物研究,2010,30(1):120-125.
[13]林昌礼,张大伟,吴丽萍,等. 优良观赏枫香种质的遗传多样性研究[J]. 湖北林业科技,2013 (2):7-10.
[14]LI G, QUIROS C F. Sequence-related amplified polymorpgism (SRAP) -a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica [J]. Theoretical and Applied Genetics. 2001, 103: 455-461.
[15]林忠旭,张献龙,聂以春.棉花SRAP遗传连锁图构建[J].科学通讯,2003,48(15):1676-1679.
[16]林忠旭,张献龙,聂以春.新型标记SRAP在棉花F2分离群体及遗传多样性评价中和适用性分析[J].遗传学报, 2004 ,31(6):622-626.
[17]RIAZ A, DAN P, STEPHEN M S. Genotyping of peach and nectarine cultivars with SSR and SRAP molecular marker [J]. The American Society for Horticultural Science. 2004, 129 (2): 204-210.
[18]FERRIOL M, PICO B, NUEZ F. Genetic diversity of a germ plasm collection of cucurbita pepo using SRAP and AFLP markers [J]. Theoretical and Applied Genetics. 2003,107:271-282.
[19]BUDAK H, SHEARLNAN R C, PARMAKSIZ I. Moleeular characterization of Buffalo grass germplasm using sequence related amplified polymorphism markers [J]. Theoretical and Applied Genetics. 2004,108: 328-334.
[20]BUDAK H, SHEARNLALL R C, GAUSSOIN R E. Application of sequence-related amplified polymorphism markers for characterization of turf grass species [J]. HortScience. 2004,39(5): 955-958.
[21]BUDAK H, SHEARNLALL R C, PARMAKSIZ I. Comparative analysis of seeded and vegetative biotype buffalo grasses based on phylogenetic relationship using ISSRs, SSRs, RAPDs and SRAPs [J]. Theoretical and Applied Genetics,2004,109: 280-288.
[22]张 飞,陈发棣, 房伟民,等.菊花SRAP-PCR反应体系的优化与确立[J]. 植物资源与环境学报,2009,18(3):44-49.
[23]董爱香,王 涛,徐 进,等. 用SRAP分子标记分析一串红品种资源的亲缘关系[J].北京林业大学学报,2012, 34(5):134-138.
[24]潘俊松,王 刚,李效尊,等.黄瓜SRAP遗传连锁图的构建及始花节位的基因定位[J]. 自然科学进展,2005,15(2):167-172.
[25]胡 颖. 槭属植物SRAP技术体系的建立及其遗传多样性研究[D]. 郑州:河南农业大学,2009.
[26]柳新红,李 楠,李因刚,等. 白花树SRAP-PCR反应体系的建立与优化[J]. 浙江林业科技,2011,31(6):30-34.
[27]李 楠,柳新红,李因刚,等. 白花树天然群体的遗传多样性[J]. 林业科学,2012 (11):49-55.
[28]邹喻苹,葛 颂,王晓东,等.系统与进化植物学中的分子标记[M].北京:科学出版社,2001.
[29]马学军,舒跃龙,颜子颖,等.精编分子生物学实验指南[M].北京:科学出版社,2005.
[30]陈大明,张上隆,金勇丰,等.一种木本果树基因组DNA提取方法研究[J]. 浙江农业大学学报,1997, 23(6):621-624.
[31]KHANUJA S P S, SHASANY A K, DAROKAR M P. Rapid isolation of DNA from dry and fresh samples of plants producing large amounts of secondary metabolites and essential oils [J]. Plant Molecular Biology Reporter, 1999, 17: 1-7.
[32]盖钧镒.实验统计方法[M]. 北京:中国农业出版社,2000.
[33]刘萌芽,黄如葵,黄玉辉,等. 正交直观分析法和新复极差法优化苦瓜SRAP反应体系研究[J]. 北方园艺,2014 (5):85-88.
[34]刘玉民,刘亚敏,李鹏霞,等. 枫香叶精油抑菌活性及抗氧化活性研究[J]. 食品科学,2009,30(11):134-137.
[35]刘亚敏,刘玉民,马 明,等. 枫香叶总黄酮提取工艺优化及含量动态变化[J]. 食品科学, 2010,31(4):35-38.
[36]胡敬志,田 旗,鲁心安,等. 枫香叶片色素含量变化及其与叶色变化的关系[J]. 西北农林科大学学报:自然科学版,2007,35(10):219-223.
[37]郑 毅,张 青. 江西野生枫香活性成分提取及鞣质含量研究[J]. 江西化工,2004(6):99-102.
[38]KIM C S,LEE C H, SHIN J S, et al. A simple and rapid method for isolation of high quality genomic DNA from fruit trees and conifers using PVP [J]. Nucleic Acids Research, 1997, 25: 1085-1086.
[39]樊洪泓,李延春,邱 婧,等. 石斛属植物SRAP反应体系的建立与优化[J]. 分子植物育种,2006,52 (4):14-18.
[40]楚爱香,汤庚国. 观赏海棠SRAP-PCR反应体系优化及引物筛选[J]. 湖北农业科学,2008,48 (12):1394-1397.
[41]任绪瑞,刘艳玲,杨 美,等. 菰SRAP-PCR反应体系的优化与建立[J]. 热带作物学报,2014,35 (2):299-306.
[42]赵 杨,李玉璞,代毅,等.华山松SRAP-PCR反应体系的优化[J]. 西北林学院学报,2012,27(5):87-90.
(责任编辑:梁保松)
Genomic DNA extraction and establishment of the optimal SRAP-PCR reaction system forLiquidambarformosanaHance.
LI Fangfang1,2, YANG Shaozong2, LIU Xinhong2, LI haibo2, WANG liling2, LI Yonghua1
(1.Henan Agricultural University, Zhengzhou 450002, China; 2.Zhejiang Forestry Academy, Hangzhou 310023, China)
In order to investigate theL.formosanagenomic DNA extraction methods and the optimal SRAP-PCR reaction system, two kinds of genomic DNA extraction methods for different materials were contrasted. By the orthogonal experiment design Ll6(45), five factors including Mg2+concentration, dNTPs concentration, primer concentration, Taq DNA polymerase and template DNA amount in SRAP-PCR reaction system were optimized, and the optimal SRAP-PCR reaction system suitable for genomic DNA extracted fromL.formosanawas also established. The results showed that the modified CTAB method was better than the isolation kit one, and the optimized SRAP-PCR reaction system was as follows: total volume 20 μL, containing 1×PCR Buffer, 1.5 mmol·L-1Mg2+, 0.16 mmol·L-1dNTPs, 0.9 U Taq DNA polymerase, 0.5 μmol·L-1primer, 40 ng template DNA. The effects of the five factors on SRAP-PCR amplification are different, in which, Taq DNA polymerase was the greatest, but that of template DNA amount was the least. The optimal SRAP-PCR reaction system is tested by means of ten random selected genomic DNA ofL.formosana., and the amplification pattern with rich polymorphism and clear band was obtained.
LiquidambarformosanaHance; genomic DNA extraction; SRAP-PCR; orthogonal experiment design; optimization and test of reaction system
1000-2340(2015)01-0046-06
2014-06-10
林业公益性行业科研项目(201404312);浙江省重大科技项目(2012C12908-14);浙江省重大科技项目(2012C12909-21)
李芳芳(1987-),女,河南尉氏县人,硕士研究生,从事观赏园艺植物研究。
李永华(1976-),男,河南周口人,副教授,博士。
S 722.3
A
