岗梅实时荧光定量PCR内参基因的筛选
2015-05-30郑夏生等
郑夏生等
摘 要 为筛选适用于岗梅实时荧光定量PCR的内参基因。应用实时荧光定量PCR技术,分析18S rRNA(18S)、Actin(ACT)、α-tubulin(TUA)、β-tubulin(TUB)、Cyclophili(CYP)、Elongation factor 1-α(EF-1α)和Polyubiquitin(UBQ)共7个看家基因在岗梅的8个不同组织部位中的表达情况,并利用GeNorm、NormFinder和BestKeeper共3个软件对每个看家基因的表达稳定性进行评价。3个软件的分析结果均表明:18S、ACT和TUA看家基因的稳定性和相关性均较好,可在这3个看家基因中选择1个或以18S-ACT、18S-TUA组合作为岗梅基因研究的内参基因。
关键词 岗梅;qRT-PCR;内参基因
中图分类号 R28 文献标识码 A
Abstract This study aimed to select reference gene(s) for real time quantitative PCR(qRT-PCR)research in I. asprella. The expression levels of seven housekeeping genes (HKGs), including 18S rRNA(18S), Actin(ACT), α-Tubulin (TUA), β-Tubulin (TUB), Cyclophili (CYP), Elongation factor 1-α (EF 1α) and Polyubiquitin(UBQ), in eight different tissues of I. asprella, were analyzed by qRT-PCR. Furthermore, their expression stabilities were evaluated by three software, including GeNorm, NormFinder and BestKeeper. Analysis data showed that 18S, ACT and TUA expressed stably and were related to each other in different tissues of I. asprella. 18S, ACT and TUA, combination of 18S-ACT or 18S-TUA, could be considered as candidate reference gene(s) for qRT-PCR study of I. asprella.
Key words Ilex asprella;qRT-PCR;Reference gene
实时荧光定量PCR(Real-time fluorescence quantitative PCR,qRT-PCR)是目前公认的用于分析基因表达水平的重要手段,具有灵敏度高、快速准确和重复性好等优点[1-2]。高质量的实验材料和可信可靠的定量方法是对目的基因进行准确、客观定量的前提[3]。定量PCR实验数据发表所必需实验信息最低限度标准(Minimum Information for Publication of Quantitative Real-Time PCR Experiments, MIQE)对于qRT-PCR实验设计中必须考虑的因素和水平做出了具体的规定[4]。为了达到准确的基因定量数据,必须充分考虑以下几种因素的可靠性:起始模板的质量和数量,存在于不同样品间的抑制因子,另外,为尽量减少实验样本间的误差,采取适当的数据校正也很有必要。通常利用一个或几个表达较为稳定的看家基因(Housekeeping Genes,HKGs)作为内参基因来对目的基因进行校正和标准化[5]。
理想的内参基因应在生物体各类型的组织、细胞和各种实验因素条件下均恒定表达[6]。然而,大量的实验结果表明,看家基因的表达稳定性并没有很好的普遍性,即同一看家基因在不同物种或相同物种的不同组织中的表达水平可能存在较大的差异[7]。因此,通常需要从多个看家基因中筛选出表达相对稳定的一个或一个以上的看家基因作为内参基因。常用于筛选内参基因的看家基因包括18S核糖体RNA(18S rRNA)、肌动蛋白基因(Actin)、亲环素蛋白(Cyclophili)、微管蛋白基因(Tubulin)和多素泛聚酶基因(UBQ)等。
冬青科植物岗梅Ilex asprella(Hook.et Arn.)Champ.ex Benth. 是岭南地区民间常用的一种草药。岗梅的根入药即岗梅,具有清热解毒、消肿祛瘀的功能[8]。根据文献报导,岗梅的主要化学成分为乌索烷型三萜皂苷。本课题组在前期的研究中利用转录组测序和生物信息分析,揭示了一系列可能参与岗梅根中三萜皂苷生物合成的候选基因片段[9];对岗梅不同组织部位中三萜皂苷积累量的研究结果表明,岗梅的根和叶子很可能是三萜皂苷主要的合成和贮存部位,而茎的三萜皂苷贮存量却很低[10]。因此,进一步对岗梅不同组织部位中三萜皂苷合成酶相关基因进行鉴定和功能验证有助于阐明岗梅中三萜皂苷的生物合成途径,进而为乌索烷型三萜皂苷的生物合成研究奠定基础。基因的表达水平研究可有效帮助鉴定出实际参与三萜皂苷生物合成的基因。本研究的目的是利用qRT-PCR评价18S、ACT、TUA、TUB、CYP、EF-1α和UBQ共7个看家基因的表达稳定性,以筛选在岗梅不同组织器官中稳定表达的内参基因。
1 材料与方法
1.1 材料
实验所用的岗梅I. asprella为种植两年生的盆栽植株,来源于广东省梅州市平远县南岭药业种植基地,由广州中医药大学中药资源科学与工程研究中心詹若挺研究员鉴定。植株分为须根(Fibrous roots,FR)、根皮(Root bark,RB)、根木质部(Root xylem,RX)、茎皮(Stem bark,SB)、茎木质部(Stem xylem,SX)、嫩枝(Twig,TW)、幼叶(Tender leave,TL)和成熟叶(Mature leave,ML)等8个不同的取材部位。除叶子外,其他部位均用干净的解剖刀切成厚约1 mm的薄片,液氮处理后于-80 ℃贮存备用。
RNA提取试剂RNAiso Plus、Fruit-mate for RNA Purification及反转录试剂盒PrimeScriptTM RT Reagent with gDNA Eraser、RNase-free H2O、DNA Marker等均购自大连宝生物工程有限公司(TaKaRa),2×power Taq Master Mix PCR试剂购自北京百泰克生物公司,qRT-PCR试剂Sso Fast Eva Green购自美国伯乐生物科技有限公司(BioRad),其余试剂均为分析纯。
1.2 方法
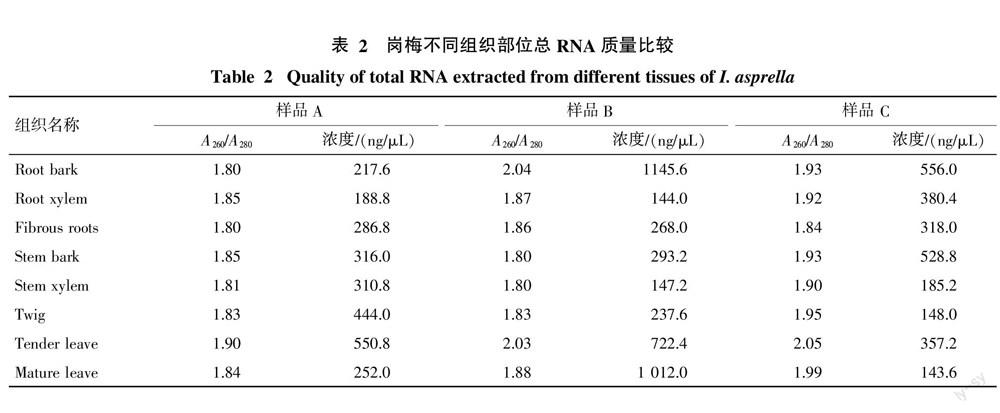
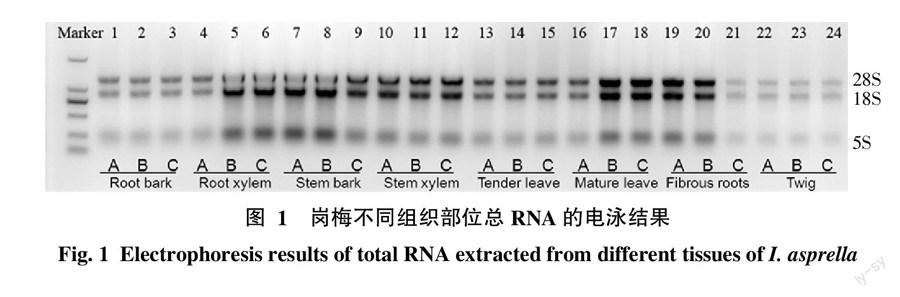
1.2.1 总RNA的提取 每个组织部位取3个不同的生物重复,分别为样品A、样品B和样品C。总RNA的提取参照上述试剂盒说明书进行(改善之处:在加入异丙醇沉淀核酸之前,先加入上清液1/4体积的高盐溶液),制得的总RNA直接用于反转录或保存于-80 ℃备用。取适量总RNA用RNase-free H2O稀释10倍,以RNase-free H2O为空白,用紫外分光光度仪测定其OD260、OD280、OD230和OD320的值;并取适量总RNA加 6×Loading buffer于1%琼脂糖凝胶中电泳。
1.2.2 反转录合成cDNA 按照PrimeScript RT Reagent with gDNA Eraser试剂盒说明书,各取样品的总RNA 1 μg反转录成cDNA第一链。所得cDNA产物直接用于PCR或保存于-20 ℃备用。
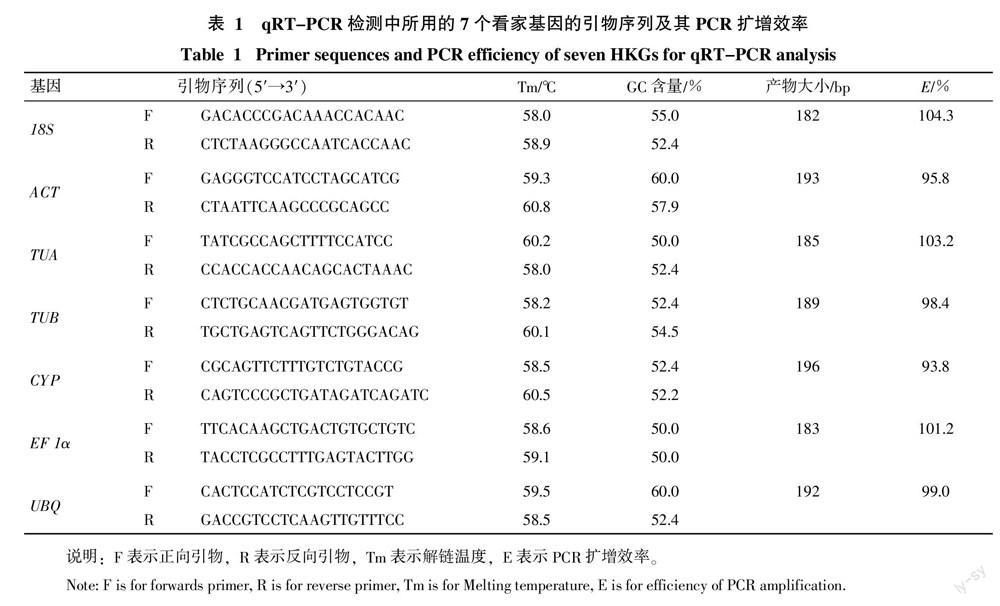
1.2.3 引物设计 根据Technote qPCR MIQE Guide(BioRad)中引物设计的原则,采用Primer Premier 5.0软件设计7个看家基因的qRT-PCR引物,引物序列见表1。引物由华大基因公司合成,采用PAGE纯化。
1.2.4 看家基因的普通PCR 利用表1中的各引物对各内参基因进行普通PCR以验证其可用性。PCR体系为2×power Taq Master Mix 10 μL,混合cDNA 1 μL,Forward 和Reverse引物(浓度10 μmol/L)各0.4 μL,用ddH2O补足体积至20 μL。反应温度程序为:95 ℃预变性2 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸30 s,32个循环;72 ℃延伸5 min。反应完成后用3%琼脂糖凝胶电泳检测各PCR产物。
1.2.5 看家基因的qPCR分析 在荧光定量PCR管(BIO-RAD)中加入体积为20 μL的反应体系:Sso Fast Eva Green 10 μL,各样品的cDNA 1 μL(阴性对照NTC为ddH2O 1 μL),正向和反向引物(浓度10 μmol/L)各0.4 μL,用ddH2O补足体积至20 μL。反应温度程序为:95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火30 s,40个循环;65~95 ℃溶解曲线分析。实验设3个技术重复。加样完成后迅速放入CFX 96 real-time quantity PCR仪上进行反应。数据读取由仪器自动完成。
1.2.6 看家基因的扩增效率和线性考察 随机选取岗梅的第一链cDNA样品,用ddH2O依次稀释5个梯度,每个梯度稀释5倍,即起始模板的浓度分别为1、1/5、1/25、1/125和1/625。每个反应设3个技术重复,按1.2.5的反应体系和程序进行分析。
1.3 数据处理及分析
根据公式Q=E△Ct,利用Excel 2010对每个扩增样品的Ct值计算各内参基因的相对表达量Q。其中E为基因扩增效率(一般情况下将基因扩增设为理想扩增,扩增效率E默认为2),△Ct=Ctmin-Ct样品(Ctmin为所有样品中最低的Ct值,Ct样品为每个样品的Ct值)[11]。
根据7个常用的看家基因在岗梅不同组织部位中的相对表达量,利用GeNorm、NormFinder和BestKeeper共3个软件对每个看家基因的表达稳定性进行统计学分析。
2 结果与分析
2.1 总RNA样品的质量分析
各总RNA样品的OD值见表2,A260/A280和A260/A230均>1.8,且浓度适宜,表明其纯度较高,均可满足下游反转录实验的要求。由图1可知,各总RNA样品均呈现28S、18S和5S共3条清晰的条带,28S条带亮度约为18S条带的2倍,表明各样品的完整性较好。
2.2 看家基因的普通PCR分析
各看家基因的普通PCR结果均可见预期大小的PCR产物(图2),且条带单一,无非特异性扩增,表明引物设计成功,各引物可用于后续荧光定量PCR实验。
2.3 看家基因的扩增效率和线性分析
以5倍梯度稀释的cDNA为模板,对7个看家基因作标准曲线分析,结果显示,各看家基因标准曲线的相关系数R2≥0.990,说明它们的线性关系很好。另外,各看家基因的扩增效率均在90%~105%,且熔解曲线均只产生单一的熔解峰,不存在引物二聚体等非特异扩增,各技术重复的样品间的重复性良好。上述结果表明各看家基因的扩增效率和线性均符合要求。
2.4 看家基因的Ct值分析
qRT-PCR分析结果表明,7个看家基因在岗梅8个不同部位不同组织部位中的Ct值在21~35(图3)。基因的表达量与其Ct值呈反比关系。表达量最高的是18S,其Ct值在21~25;表达量最低的是EF-1α,其Ct值在29~35。说明各看家基因的表达量存在较大的差别。
2.5 内参基因的筛选
GeNorm软件的计算结果是以M值来反映基因的稳定性,M值越小则表明基因的稳定性越高[12]。利用GeNorm软件对各内参基因的相对表达量Q进行统计学分析,结果表明7个看家基因的M值均小于1.5(图4)。各看家基因的表达稳定性各异,表达稳定程度由高到低的顺序为TUA=18S>ACT>EF-1α>CYP>UBQ>TUB。
NormFinder基于方差分析评价内参基因在不同样品中的表达稳定性[13]。利用NormFinder分析7个看家基因在岗梅不同部位中的表达稳定性,结果表明各看家基因的表达稳定性均低于1.2(图5),稳定性大小顺序为:ACT>TUA>18S>EF-1α>CYP>UBQ>TUB。
BestKeeper软件可直接根据看家基因的Cp值(或称Ct值),分析标准变异系数和变异相关系数来判断其表达稳定性[14]。上述2个参数的值越小则表示基因越稳定;而标准偏差>1则被认为该基因的表达不稳定。另外,BestKeeper将候选基因组合成一个指数,并通过重复配对相关性分析评价每个候选基因与该指数的相关性。从表3可看出,TUB、EF-1α和UBQ的Cp值标准偏差均>1,较为不稳定;CYP虽然Cp值变异小,但相关性分析结果表明它与其他的基因相关性较差(表4);移除上述4个不合格的基因后,剩下的18S、TUA和ACT基因的相关系数均>0.900(表5)。上述分析结果表明,7个看家基因的稳定性大小顺序为:ACT>TUA>18S>CYP>TUB>EF-1α>UBQ。
综上所述,3个软件的分析结果均表明,18S、ACT和TUA看家基因的稳定性和相关性均较好,可作为岗梅基因研究的内参基因;其他4个看家基因的稳定性相对较差,不适合作岗梅基因研究的内参基因。
3 讨论与结论
GeNorm、NormFinder和BestKeeper共3个软件是最常用的基因稳定性的分析软件。在本研究中,3个软件的分析结果并不完全一致,可能是由于这3个软件的算法和评价指标不同所致,而且这种现象也存在于其他物种的研究中[15]。尽管如此,3个软件的分析结果均表明,表达相对稳定的前3个基因均为18S、ACT和TUA。18S是细胞胞质核糖体小亚基,在多数物种中均有较高的表达水平,已成为水稻等物种的内参基因[16];而ACT和TUA均编码细胞骨架结构蛋白,在功能上具有相似性,分别是葡萄和番茄的内参基因[17-18]。如果考虑用2个编码不同生物功能蛋白的基因配对作为岗梅的内参基因,可选择18S-ACT或18S-TUA组合。
理想的内参基因,不仅在生物体的不同类型细胞和组织中稳定表达,同时应不受环境、生物和非生物胁迫等的影响[12]。但实际上,内参基因的表达稳定性是相对的。研究者应根据各自的实验样品类型和实验条件来筛选合适的内参基因[7]。本研究筛选得到的3个表达相对稳定的看家基因,是适用于岗梅不同组织部位中基因表达水平研究的内参基因,为岗梅不同组织部位的基因表达水平研究提供了实验依据。另外,利用茉莉酸甲酯刺激有利于诱导生物合成类基因的表达水平调整。因此,对岗梅进行不同组织或不同浓度茉莉酸甲酯刺激,考察上述3个稳定性较好的内参基因的表达水平,有助于进一步筛选最稳定的内参基因。
参考文献
[1] Radonic A, Thulke S, Mackay I M, et al. Guideline to reference gene selection for quantitative real-time PCR[J]. Biochemical and Biophysical Research Communications, 2004, 313(5): 856-862.
[2] Grant P R, Garson J A, Ayliffe U, et al. Real-time PCR quantitation of hepatitis B virus DNA using automated sample preparation and murine cytomegalovirus internal control[J]. J Virol Methods, 2005, 126(1-2): 207-213.
[3] Ginzinger D G. Gene quantification using real-time quantitative PCR: An emerging technology hits the mainstream[J]. Exp Hematol, 2002, 30(6): 503-512.
[4] Bustin S A. The MIQE guidelines: Minimum information for publication of quantitative real-time PCR experiments[J]. Clin Chem, 2009, 55(4): 611-622.
[5] 万红建, 袁 伟,杨悦俭. 植物实时荧光定量PCR内参基因的特点及选择[J]. 植物学报, 2012, 47(4): 427-436.
[6] Bustin S A. Quantification of mRNA using real-time reverse trascription PCR RT-PCR: trends and problems[J]. J Mol Endocrinol, 2002, 29(1): 23-29.
[7] 范成明, 胡瑞波,傅永福. 植物实时荧光定量PCR内参基因的选择[J]. 中国农业科技学报, 2009, 11(6): 30-36.
[9] Zheng X S, Xu H, Ma X Y, et al. Triterpenoid saponin biosynthetic pathway profiling candidate gene mining of the Ilex asprella root using RNA-Seq[J]. Int J Mol Sci, 2014, 15(4): 5 970-5 987.
[10] 郑夏生. 岗梅转录组及三萜皂苷生物合成相关酶基因的挖掘[D]. 广州: 广州中医药大学, 2014.
[11] 刘成前, 吴文凯,周志刚. 用于莱茵衣藻荧光定量PCR分析的内参基因选择[J]. 植物生理学通讯, 2009, 45(4): 667-672.
[12] De Preter K, Vandesompele J, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biol, 2002, 3(7):1-11.
[13] Jensen J L, Andersen C L, Qrntoft T F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Res, 2004, 64(12): 5 245-5 250.
[14] Tichopad A, Michael W, Neuvians P, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-Excel-based tool using pair-wise correlations[J]. Biotechnology Letters, 2004, 26(3): 509-515.
[15] 孙 鹏, 侯维海, 陈全家,等. 地黄实时定量PCR内参基因的筛选[J]. 中国农学通报, 2011, 27(17): 76-82.
[16] Nam H Y, Kim B R, Kim S U, et al. Normalization of reverse transcription quantitative-PCR with housekeeping genes in rice[J]. Biotechnol Lett, 2003, 25(10): 1 869-1 872.
[17] Olsson N, Reid K E, Schlosser J, et al. An optimized grapevine RNA isolation procedure and statistical determination of reference genes for real-time RT-PCR during berry development[J]. BMC Plant Biol, 2006, 6(1): 27.
[18] Davis E, Coker J S. Selection of candidate housekeeping controls in tomato plants using EST data[J]. Biotechniques,2003,35(4):740-742,744,746.
