大鼠全基因组微卫星分布特征研究
2015-05-15涂飞云刘晓华杜联明严超超黄晓凤
涂飞云,刘晓华,杜联明,严超超,黄晓凤*
(1.江西省林业科学院,江西 南昌 330013;2.四川大学 生命科学学院/四川省濒危野生动物保护生物学重点实验室,四川 成都 610064)
微卫星又称简单序列重复(simple sequence repeats,SSRs),是指1~6 bp的核苷酸为基本重复单位的串联重复序列,其长度大多在100 bp以内[1],广泛存在于真核生物和一些原核生物的基因组中[2]。微卫星因突变速率快、多态性高等特性,广泛应用于动物个体识别[3-5]、群体遗传研究[6-7]、基因定位[8]、系统发育[9]及遗传疾病研究[10]。
随着基因测序技术发展,更多物种的全基因组被测定和公布,有关动物基因组微卫星报道也越来越多。到目前为止,有关哺乳动物全基因的序列公布有43种(http://asia.ensembl.org/info/data/ftp/index.html),但哺乳动物全基因微卫星分布规律研究报道文献仅见4篇[11-14]。大鼠是继人类、小鼠之后第三个被测定全基因组的哺乳动物,是极好的模式生物,作为实验材料广泛地应用于医学、药学研究中。Tóth等[15]利用啮齿类81个物种的基因序列(其中大鼠基因序列贡献率是18.25%)进行微卫星序列分析。迄今为止,大鼠全基因组微卫星分布状况未见报道。本研究通过生物信息学方法搜索大鼠全基因组微卫星,并对微卫星数量、分布、频率进行分析,为进一步开发大鼠微卫星的标记提供数据支持。
1 材料与方法
1.1 大鼠基因组序列
大鼠全基因组序列是从NCBI数据库下载的版本名为Rnor_6.0(2014年8月发布)的基因组序列(ftp://ftp.ncbi.nlm.nih.gov/genomes/Rattus_norvegicus)。该基因组包括 20 个常染色体、2 个性染色体(X,Y)和未定位到染色体上的序列(其中已经定位到染色体上的序列占全基因组的99%),基因组总长度约2.8 Gb,所有序列都以FASTA格式保存。
1.2 数据处理
1.2.1 微卫星序列统计术语的定义 微卫星序列统计术语的定义参照戚文华等[12-13]的研究。
1.2.2 微卫星搜索及统计分析 MSDB是用Perl语言编开发的界面友好的分析软件,能快速识别和搜索全基因组微卫星。与其他微卫星搜索软件如TRF、SSRTT、MSATCOMMANDER等软件相比,MSDB具有运算速度快,操作简便,易于分析等特点[16]。本研究利用微卫星搜索软件MSDBv2.4.3[16]对不同染色体基因序列进行微卫星搜索及统计。本研究的搜索标准采用软件默认设置,模式设置为Perfect Search,最小重复(Minimum repeats)设置为单碱基12次重复以上、二碱基7次重复以上、三碱基5次以上、四碱基、五碱基和六碱基4次以上[16]。利用SPSS软件对大鼠染色体序列长度与微卫星数量分析。参照碱基互补配对原则和统计拷贝数起始碱基顺序的排列差异,利用Perl语言脚本将同类重复兼并为一种重复拷贝类型代表,如三碱基AAC,可以与之兼并的有ACA、CAA、TTG、TGT和GTT。
2 结果与分析
2.1 大鼠微卫星在全基因组及染色体分布特征
大鼠基因组全长2 860 318 831 bp,微卫星总数1 483 525个,其总长度是40 198 048 bp,占全基因组的1.41%(表1)。大鼠各染色体中,1号染色体的微卫星数量最多,有144 384个,占微卫星总数的9.7%,出现的频率是511个/Mb,其长度合计3 941 664 bp,其次是2号、4号、3号染色体,分别占微卫星总数的8.5%、6.3%、6.2%(表1),20号染色体分布的微卫星数量最低,仅占总数的2.3%。从各染色体微卫星分布频率看,12号染色体最高,其次是10号,最低X性染色体。通过线性回归分析表明,大鼠染色体DNA序列越长,所含微卫星数量越多(r=0.978,P<0.000 1)。大鼠性染色体微卫星数量存在显著差异,X染色体微卫星数量有62 578,占总数的4.2%,而Y染色体微卫星数量仅有35 067,仅占2.4%(表1)。
2.2 不同重复类型微卫星分布特点
从表1看,整个大鼠全基因组中不同重复类型微卫星占全基因组微卫星比例表现为二碱基(46.9%)>单碱基(22.6%)>四碱基(17.4%)>三碱基(8.4%)>五碱基(2.4%)>六碱基(2.0%)。
不同重复类型微卫星在染色体分布数量上,6种重复类型(单碱基、二碱基、三碱基、四碱基、五碱基、六碱基)微卫星总数在1号染色体分布最多,其次是2号染色体。不同重复类型微卫星分布数量最少的染色体并不一致(表1)。
从不同重复类型微卫星重复拷贝类别数量比例看,单碱基以A微卫星数量占优势,分别占单碱基微卫星总数的46.6%。二碱基微卫星类型搜索到4种,有AC、AG、AT、CG,整个大鼠全基因组序列以AC类型最多,占二碱基微卫星总数的64.6%,其次是AG,占24.9%,CG最少,仅占0.6%。共兼并及统计三碱基微卫星重复类型 10 种,包括 AAC、AAG、AAT、ACC、ACG、ACT、AGC、AGG、AGT、CCG,其中AGG类型数量最多,占三碱基微卫星总数的 21.2%,依次是 AAC、AAT,占20.8%、13.5%;CCG 类型最少,其次是AGC,分别占0.9%和3.4%。四碱基微卫星类型以AAAC类型最多,占四碱基微卫星总数的14.9%,其次是AGAT和AAAG,占11.9%和11.1%。五碱基微卫星以AAACA占优势,占39.8%。六碱基以ACAGAG占优势,占18.9%。
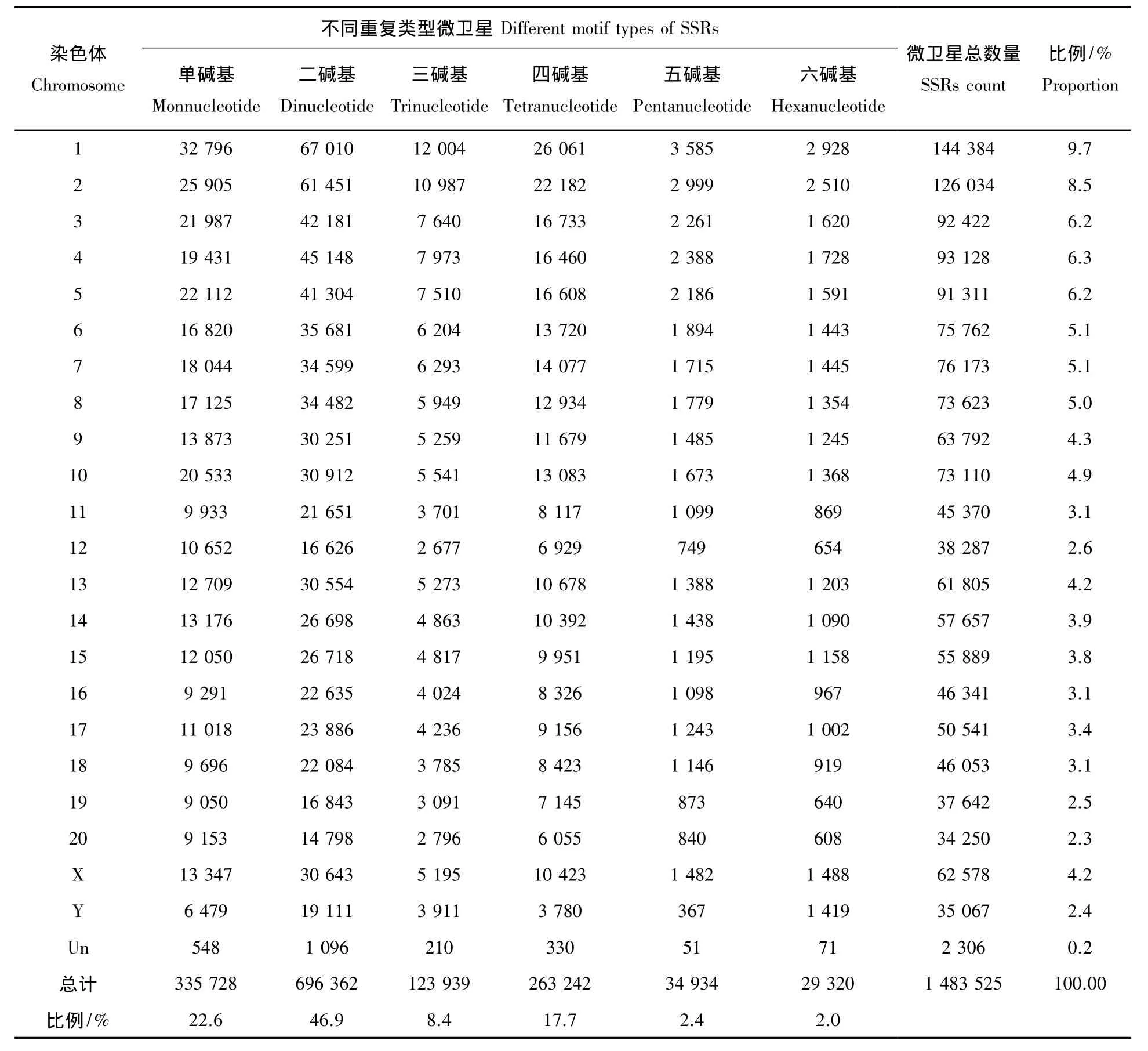
表1 大鼠不同染色体上不同类型微卫星数量的分布情况Tab.1 Distribution of SSRs in each chromosome of rat
3 讨论
本研究首次利用生物信息学方法搜索、统计、分析大鼠全基因组微卫星数量、分布及频率。大鼠全基因组共搜索到微卫星总数1 483 525,其序列长度占全基因组的1.41%。本研究表明微卫星数量与其所在染色体DNA序列长度具有相关性(r=0.978,P<0.000 1),反映了SSRs在染色体上的分布具有随机性,与先前其他研究结果一致[13、17],进一步支持Hancock[18]提出的微卫星数量与染色体大小相关的假说。
不同物种基因组优势类型微卫星有所不同。秀丽隐杆线虫Caenorhabditis elegans[18]、肩突硬蜱Ixodes scapularis[19]、红原鸡 Gallus gallus[17]、四川山鹧鸪 Arborophila rufipectus[20]、牛 Bos taurus[12]、绵羊Ovis aries[12]、猪 Sus scrofa[13]等全基因组均以单碱基占优势;二斑叶螨 Tetranychus urticae[19]全基因组以三碱基占优势;蚊子Anopheles gambiae[21]以六碱基占优势。本研究大鼠基因组微卫星以二碱基微卫星占绝对优势,与果蝇 Drosophila melanogaster[22]、家蚕 Bombyx mori[23]、蜜蜂 Apis mellifera[24]、河豚 Fugu rubripes[25]、全基因组及中国对虾 Fenneropenaeus chinensis[26]、啮齿类[19]部分基因组中二碱基微卫星占主导结果一致。
本研究中大鼠全基因组单碱基微卫星以A占优势,与人类、红原鸡、猪等物种研究结果一致[15],其余不同重复类型微卫星在不同类群中具有差异。大鼠二碱基微卫星以AC最为丰富,与人类、果蝇、蚊子、猪、牛和绵羊的结果一致,不同于中国对虾、蜜蜂、红原鸡二碱基以AT最丰富。三碱基、四碱基、五碱基、六碱基分别以 AGG、AAAC、AAACA、ACAGAG占优势,而其他物种如猪、牛、羊并非以此四种(AGG、AAAC、AAACA、ACAGAG)类型微卫星占优势,可能是不同物种优势类型微卫星有所不同。
致谢:特别感谢四川大学生命科学学院李午佼博士对研究的帮助。
[1]Li Y C,Korol A B,Fahima T,et al.Microsatellites:genomic distribution,putative functions and mutational mechanisms:a review[J].Molecular Ecology,2002,11(12):2453-2465.
[2]Tautz D.Hypervariability of simple sequences as a general source for polymorphic DNA markers[J].Nucleic Acids Research,1989,17(16):6463-6471.
[3]Amavet P,Rosso E,Markarian R,et al.Microsatellite DNA markers applied to detection of multiple paternity in Caiman latirostris in Santa Fe,Argentina[J].Journal of Experimental Zoology,2008,309A:637-642.
[4]王静,刘丑生,张利平,等.微卫星在种公牛个体识别与亲缘鉴定方面的应用[J].遗传,2009,31(3):285-289.
[5]管昊,林清贤,周晓平,等.白鹭脱落羽毛的微卫星个体识别研究[J].厦门大学学报:自然科学版,2013,52(5):710-717.
[6]Hu Y B,Guo Y,Qi D W,et al.Genetic structuring and recent demographic history of red pandas(Ailurus fulgens)inferred from microsatellite and mitochondrial DNA[J].Molecular Ecology,2011,20:2662-2675.
[7]Wu S J,Luo J,Li Q Q,et al.Ecological genetics of Chinese rhesus macaque in response to mountain building:all things are not equal[J].Plos One,2013,8(2):55315.
[8]徐宁迎,Thomsen H,Reinsch N,等.利用微卫星进行奶牛数量形状基因定位的研究[J].遗传学报,2000,27(9):772-776.
[9]Estoup A,Lionel G.Microsatellite variation in honey bee(Apis mellifera L.)populations:hierarchical genetic structure and test of the infinite allege and stepwise mutation models[J].Genetics,1995,140(2):679-695.
[10]陈翠敏,府伟灵,张晓莉.应用微卫星多态位点对X连锁型视网膜色素变性疾病进行遗传连锁分析的研究[J].重庆医学,2006,35(6):501-502.
[11]童晓玲,代方银,李斌,等.小鼠基因组中的微卫星重复序列的数量、分布和密度[J].动物学报,2006,52(1):138-152.
[12]戚文华,蒋雪梅,肖国生,等.牛和绵羊全基因组微卫星序列的搜索及其生物信息学分析[J].畜牧兽医学报,2013,44(11):1724-1733.
[13]戚文华,蒋雪梅,肖国生,等.猪全基因组中微卫星分布规律[J].畜牧与兽医,2014,46(8):9-12.
[14]李午佼,李玉芝,杜联明,等.大熊猫和北极熊基因组微卫星分布特征比较分析[J].四川动物,2014,33(6):874-878.
[15]Tóth G,Gáspári Z,Jurka J.Microsatellites in different eukaryotic genomes:survey and analysis[J].Genome Research,2000,10:967-981.
[16]Du L M,Li Y Z,Zhang X Y,et al.MSDB:a user-friendly program for reporting distribution and building databases of microsatellites from genome sequences[J].Journal of Heredity,2013,104(1):154-157.
[17]黄杰,杜联明,李玉芝,等.红原鸡全基因组中微卫星分布规律研究[J].四川动物,2012,31(3):358-363.
[18]Hancock J M.Simple sequences and the expanding genome[J].Bioessays,1996,18(5):421-425.
[19]汪自立,黄杰,杜联明,等.二斑叶螨和肩突硬蜱基因组微卫星分布规律研究[J].四川动物,2013,32(4):481-486.
[20]黄杰,周瑜,刘与之,等.基于454 GS FLX高通量测序的四川山鹧鸪基因组微卫星特征分析[J].四川动物,2015,34(1):8-14.
[21]余泉友,李斌,李关荣,等.蚊子全基因组中微卫星的丰度及其分布[J].生物化学与生物物理进展,2005,32(5):435-441.
[22]Schug M D,Wetterstrand K A,Gaudette M S,et al.The distribution and frequency of microsatellite loci in Drosophila melanogaster[J].Molecular Ecology,1998,7(1):57-70.
[23]Li B,Xia Q Y,Lu C,et al.Analysis on frequency and density of microsatellites in coding sequences of several eukaryotic genomes[J].Genomics,Proteomics and Bioinformatics,2004,2(1):24-31.
[24]魏朝明,孔光耀,廉振民,等.蜜蜂全基因组中微卫星的丰度及其分布[J].昆虫知识,2007,44(4):501-504.
[25]崔建洲,申雪艳,杨官品,等.红鳍东方鲀基因组微卫星特征分析[J].中国海洋大学学报,2006,36(2):249-254.
[26]高焕,刘萍,孟宪红,等.中国对虾(Fenneropenaeus chinensis)基因组微卫星特征分析[J].海洋与湖沼,2004,35(5):424-431.
