杉木纤维素合成酶基因CesA的克隆及表达分析
2015-03-23童再康黄华宏林二培刘琼瑶
庞 景,童再康,黄华宏,林二培,刘琼瑶
杉木纤维素合成酶基因CesA的克隆及表达分析
庞 景,童再康,黄华宏,林二培,刘琼瑶
(浙江农林大学 亚热带森林培育国家重点实验室培育基地,浙江 临安 311300)
通过逆转录聚合酶链式反应(RT-PCR)结合cDNA末端快速扩增(RACE)技术,从杉木Cunninghamia lanceolata中克隆到2个全长为3 235 bp和3 876 bp的纤维素合成酶基因cDNA序列,被分别命名为ClCesA1和ClCesA2,GenBank登录号分别为JQ844574和JQ844575。相应编码蛋白分别包含992和1 092个氨基酸残基,推测分子量为111 845.3 D和123 105.7 D,等电点为6.04和6.65。氨基酸序列分析发现,它们具有植物纤维素合成酶基因相应的结构特征——锌指结构,2个易变区CSRI和CSRII,2个保守区CRI和CRII,纤维素合成酶底物结合结构域 “D, D,D,QVLRW”,以及8个跨膜区。荧光定量PCR分析表明: ClCesA1和ClCesA2基因在茎中表达丰度均为最高,且成熟木质部中表达量均高于皮层中的相应数值;2个基因在根和针叶中的表达量相对较低。以具特殊材质性状的矮生杉木和正常杉木木质部为材料的进一步表达分析发现:ClCesA1和ClCesA2在正常杉木木质部中的表达量大约是矮生杉木表达量的2~12倍。2个杉木CesA基因可能在杉木木材形成过程中具有重要作用。图5参19
林木育种学;杉木;CesA基因;基因克隆;序列分析;表达分析
纤维素是木材的主要成分之一,约占木材干质量的40%以上,是木材品质的主要决定因子。纤维素是由β-1,4-葡萄糖残基组成的不分支多糖,它的合成涉及一个复杂的酶系,其中由纤维素合成酶催化亚基(CESA)组成的复合酶是关键酶之一。因此,CesA基因的克隆及其功能解析一直是植物分子生物学领域中的一个研究热点。1996年,Pear等[1]分离鉴定了高等植物中的第1个CesA基因,即来自棉花Gossypium hirsutum的GhCesA1。美国的密歇根大学2000年从欧洲颤杨Populus tremuloides中克隆到了林木中的第1个纤维素合成酶基因PtrCesA1基因,它是一个在木质部特异表达的纤维素合成酶基因,与次生壁的形成有关[2]。此后来自拟南芥Arabdopsis thaliana的10个AtCesA,毛果杨Populus trichocarpa 的18个PtriCesA,以及欧洲颤杨、赤桉Eucalyptus camaldulensis等植物的部分CesA基因相继被鉴定[3-5],尤其是通过突变体分析,来自拟南芥的9个AtCesA功能已基本被解析[6],其中AtCesA1,AtCesA3,AtCesA6与细胞初生壁的形成相关,而AtCesA4,AtCesA7,AtCesA8[7]都只在有次生壁的细胞中表达,可能与细胞次生壁的形成有关。欧洲颤杨中的PtrCesA2[8]和PtrCesA5[9]基因主要在正在发育的木质部表达,参与次生壁的形成。来自赤桉的EcCesA4,EcCesA5与初生壁合成有关,EcCesA1,EcCesA2和EcCesA3[10]则参与次生壁合成。这些基因的研究为其他植物纤维素合成机制的解析奠定了坚实的基础。杉木Cunninghamia lanceolata是中国南方重要的针叶速生用材树种,用途广泛。近年来有关杉木材性性状形成分子机制的研究取得了明显进展[11-13],然而涉及其纤维素生物合成分子机制的研究报道甚少,仅彭沙沙等[14]克隆了杉木纤维素合成酶类似蛋白ClCslD1,并对它进行了生物信息学分析。本研究利用逆转录聚合酶链式反应(RT-PCR)结合cDNA末端快速扩增(RACE)技术,分离克隆了2个杉木CesA基因,对它们进行了生物信息学分析,以及在不同器官(或组织)表达差异分析,以期为进一步的功能解析提供实验依据,同时也为杉木分子辅助育种提供重要基因资源。
1 材料与方法
1.1 植物材料
试验材料为杉木速生无性系B057和矮生无性系,均栽植于浙江省临安市横畈林场。在2012年6月下旬,采集正常速生无性系的当年生成熟针叶、茎、根,以及正常速生杉木和矮生杉木1年生茎段的皮层、正常发育的木质部。所有材料液氮速冻之后存放在-80℃冰箱中备用。
1.2 实验试剂
克隆用的大肠埃希菌Escherichia coli DH5α为本实验室保存;SMARTTMRACE cDNA-amplification-kit (Clontech);PureLinkTMplant RNA reagent提取试剂盒(上海英骏生物技术有限公司);Peasy-T1 simple cloning kit(TransGen Biotech);AxyPrepDNA凝胶回收试剂盒(AxyGEN);PCR反应试剂10×buffer(缓冲液),脱氧核苷三磷酸(dNTPs),Taq DNA聚合酶均购自上海生物工程技术有限公司;引物由南京金斯瑞生物科技公司合成。
1.3 总RNA的提取和cDNA合成
总RNA的提取采用PureLinkTMplant RNA reagent试剂盒进行;cDNA第1链的合成则采用购自大连TaKaRa公司的PrimeScriptTMRT reagent kit反转录试剂盒进行。
1.4 ClCesA基因的扩增
根据已报道的其他植物CesA基因序列,在纤维素合成酶保守区域设计RT-PCR上游引物RR:5′>TGYTAYGTNCARTTYCCWC<3′,下游引物RF:5′>GANCCRTADATCCANCC<3′(Y:C or T;N:A or T or C or G;R:A or G;W:A or T;D:A or G or T)。ClCesA基因中间片段的获得参照RrimscriptTMRTPCR kit试剂盒(TaKaRa)步骤来进行。PCR产物回收后连接到Peasy-T1 simple cloning vector(TransGen Biotech)上,转化大肠埃希菌DH5α,筛选阳性克隆后送华大基因公司测序。根据RT-PCR片段的序列测定结果以及局部序列的比对结果,分别合成ClCesA5′RACE和3′RACE特异引物。
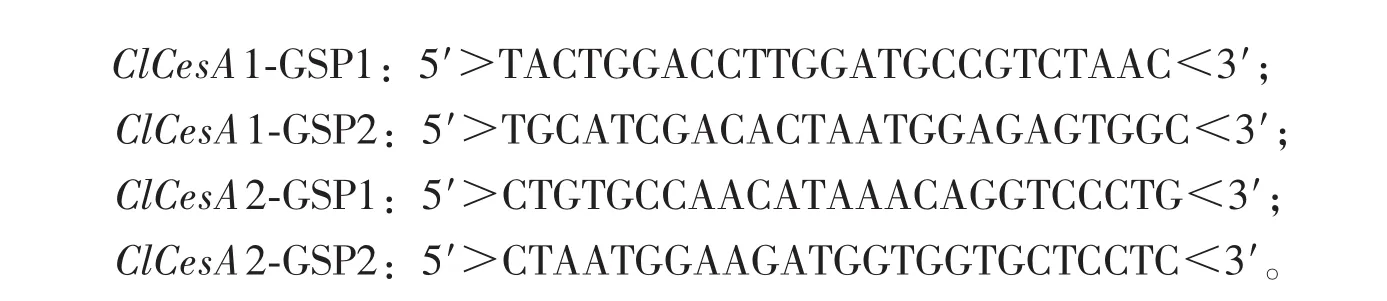
5′RACE和3′RACE具体步骤均参照SMATRTMRACE cDNA amplification kit(ClonTech)的说明书进行,PCR产物经回收测序后,与已克隆的ClCesA基因片段拼接,从而获得全长cDNA序列。根据拼接得到的ClCesA全长cDNA序列设计特异引物,经PCR扩增、测序,验证其准确性。相应引物序列如下。
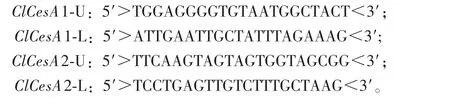
1.5 生物信息学分析
利用Nucleic Tools(http://srs.ebi.ac.uk/tooLs)分析目的基因编码蛋白质的氨基酸组成;用ProtParam (http://www.expasy.org/tooLs/protparam.html)工具推测其分子量和等电点(PI);用ProtScale(http://ca.expasy. org/tools/protscale.html)工具推测其疏水性;用TMHMM(http://www.cds.dtu.dk/services/TMHMM-2.0/)对其跨膜区进行预测;并用SignalP(http://www.cds.dtu.dk/services/SignaLP/)软件对其N端进行分析;在(http:// www.bioinfo.tsinghua.edu.cn/Subloc/)进行蛋白质亚细胞定位;同时用ExPASy的HNN(http://www.expasy. org/tooLs/HNN.html)预测其二级结构进行;利用蛋白质保守结构域推测工具InterProScan(http://ebi.ac.uk/ Tools./InterProScan/)分析结构域;利用MEGA 4.0的邻接(NJ)算法构建CesA蛋白的系统进化树。
1.6 基因表达的定量PCR分析
以正常杉木根、茎、叶、木质部和皮层,矮生杉木木质部的cDNA为模板,利用实时荧光定量PCR检测ClCesA1和ClCesA2在不同器官和组织中的表达变化,采用TaKaRa公司的定量PCR专用的PrimeScriptRreagent kit和SYBRRPremix ex TaqTM(Tli RNaseH Plus)试剂盒,以ClActin作为内参。
ClCesA1基因引物序列5′端引物序列:5′>GAGCAAATCTCAGACTCCTACTC<3′;3′端引物序列:5′>TATATCCCTACGACTCTGTCCAC<3′。ClCesA2基因引物序列 5′端引物序列:5′>GCATCCATTTTCTCCTTGCTTTG<3′;3′端引物序列:5′>ATTGCTGTTTGCCCAACATCAGT<3′。ClActin基因引物序列 5′端引物序列:5′>CAGCAACTGGGATGATATGG<3′;3′端引物序列:5′>ATTTCGCTTTCAGCAGTGGT<3′。
2 结果与分析
2.1 杉木ClCesA1,ClCesA2基因的克隆与生物信息学分析
本研究通过RACE技术,成功克隆到杉木的2个ClCesA基因,cDNA长度分别为3 235 bp和3 876 bp,分别编码992,1 092个氨基酸残基,其在美国国家生物技术信息中心(NCBI)上的登录号分别为JQ844574,JQ844575。经与 NCBI数据库比对分析,ClCesA1氨基酸序列与火炬松 Pinus taeda (AAX18647.1),光皮桦Betula luminifera(ACJ38664.1),巨桉Eucalyptus grandis(AAY60843.1)等CesA家族中部分成员序列同源性高达 82%,73%,73%。ClCesA2氨基酸序列与苎麻 Boehmeria nivea (AAY78952.3),马占相思Acacia mangium(AAT66940.1),巨桉(AAY60847.1)等CesA家族中部分成员序列同源性高达84%,81%,81%。
2个推测ClCESA蛋白相对分子质量分别为111 845.3 D,123 105.7 D,等电点分别为6.04和6.65;半衰期都大于30 h,不稳定系数分别为35.15和39.02,属于稳定蛋白。蛋白保守功能结构域的分析结果显示:这2个蛋白都具有1个纤维素的合成功能域,该结构域为纤维素合成酶蛋白家族的1个特征性结构域,具有催化尿苷二磷酸形成的功能。通过与来自白桦Betula platyphylla,欧洲颤杨,马占相思和巨桉等植物中已鉴定的CESA进行氨基酸序列比对和结构分析,发现2个CESA蛋白与来自其他植物的CESA蛋白一样都含有锌指结合域;2个易变区域CSRI和CSRII,CSRI紧跟锌指结构域后面,由88~166个氨基酸残基组成,CSRII位于CRI和CRII之间,相比CSRI略短;2个高度保守区域CRI和CRII,CRI位于CSRI之后,由394个氨基酸残基组成,而CRII则邻近碳端,由316或317个氨基酸残基组成。这2个保守区内含有纤维素合成酶底物结合结构域 “D,D,D,QVLRW”(图1,D:天冬氨酸,Q:谷氨酸,V:缬氨酸,R:精氨酸,W:色氨酸),其第1个和第2个D残基位于CRI内,第3个D残基和QVLRW序列位于CRII内。跨膜结构预测表明:ClCESA1蛋白拥有8个跨膜区,分别是182~204,211~230,767~789,801~823,838~860,885~907,917~939和952~971,跨膜螺旋长度最小19个氨基酸残基,最大22个氨基酸残基,平均长度为21.25个氨基酸残基。所有这些结果基本符合目前已知高等植物CESA蛋白的特征,进一步说明了分离的2个ClCesA基因序列确实为杉木CesA基因。ClCESA1蛋白的二级结构中,а-螺旋(helix)占34.51%,β-折叠(sheet)占14.03%,无规则卷曲(coil)占51.46%;疏水性/亲水性分析结果表明:疏水性最大值为3.311,最小值为-3.222,总体看属于亲水性蛋白。ClCESA2中а-螺旋、β-折叠、无规则卷曲分别占33.09%,15.95%,50.96%;疏水性最大值和最小值分别为3.278和-3.544,也属亲水性蛋白。此外,蛋白质亚细胞定位预测认定这2个蛋白都属于细胞质蛋白质(可靠性指标为2;预期准确性为74%),而且SignaLP软件分析2个蛋白都为非分泌蛋白,不具有信号肽结构。
2.2 系统进化树的分析
将2个预测的CLCESA蛋白与来自4种双子叶植物、2种单子叶植物和3种裸子植物全长CESA氨基酸序列进行比对和构建进化树[15](图2)。可以看出:ClCesA1和ClCesA2与对应的火炬松、拟南芥、欧洲颤杨、巨桉、水稻Oryza sativa等直系同源蛋白聚在一起,分属于2个与细胞壁物质形成相关的类群。在每个类群中,来自单、双子叶植物和裸子植物的CesA基因都分别聚类在一起,暗示在裸子植物与被子植物,或单子叶植物与双子叶植物分化之前,多数CesA进化产生了不同的类型。ClCesA1与PtCesA1同源性最高,并与欧洲颤杨PtrCesA1,水稻OsCesA4,巨桉EgCesA1,拟南芥AtCesA8等聚类在一个大的分支内;ClCesA2与玉米Zea mays ZmCesA2,欧洲颤杨PtrCesA4,水稻OsCesA1,巨桉EgCesA5,拟南芥AtCesA1等聚类在一个大的分支内。
2.3 不同器官中ClCesA的表达差异
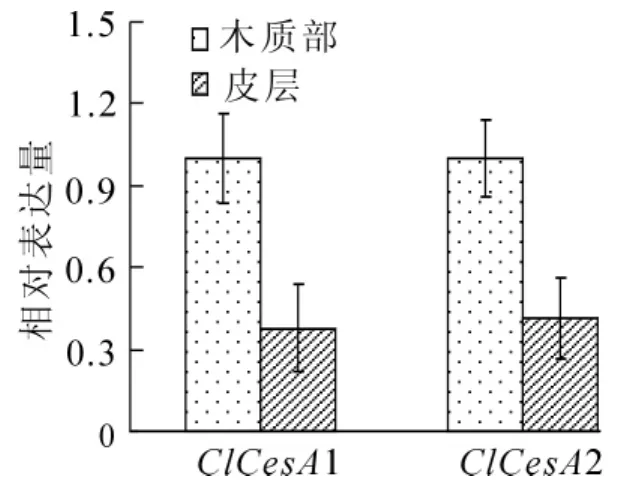
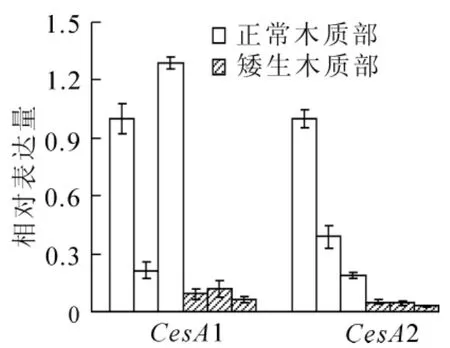
首先,利用RT-PCR和定量PCR产物的融解曲线分析和克隆测序等方法,对设计的2对ClCesA定量PCR引物的特异性和正确性进行了验证,结果发现:扩增特异且产物大小、序列正确,可用于后续定量PCR分析。通过对不同器官中ClCesA基因表达情况分析得出(图3),目的基因在杉木的根、茎、叶中都有表达,但表达模式却不同:ClCesA1,ClCesA2基因在茎中表达丰度最高,ClCesA1在根中的表达量最低,而ClCesA2基因在根和叶中的表达丰度无显著差异。为了初步探讨目的基因在木材形成中的作用,进一步以茎的木质部和皮层为材料进行表达分析,结果发现:2个ClCesA基因在木质部中表达丰度均显著高于在皮层中的相应数值(图4)。同时,本研究以具特殊材质表型的矮生杉木木质部为材料进行目的基因表达量测定,发现ClCesA1和ClCesA2在正常杉木木质部中的表达量明显高于矮生杉木,相应数值是矮生杉木表达量的2~12倍(图5)。
3 讨论
本实验通过应用RACE技术,2个杉木ClCesA基因的cDNA序列被成功分离,它们均具有完整的开放阅读框(ORF),且编码的CESA均包含高等植物CESA蛋白应有序列特征。从基于氨基酸序列相似性的分析结果看,分别来自拟南芥、杨树和巨桉的AtCesA8,PtrCesA1,EgCesA1等是ClCesA1的直系同源基因。拟南芥AtCesA8[17]是次生壁形成所必需的基因,主要在花序茎中表达,在缺少次生木质部的幼叶和花等器官中微弱表达。从欧洲颤杨中克隆的PtrCesA1[8]基因在已发育木质部的次生壁合成中特异表达,但在韧皮部纤维中却不表达。EgCesA1也主要在木质部中表达,而在无次生壁发生的组织中微弱表达[17]。荧光定量PCR分析发现:ClCesA1在茎中的表达量最高,且在茎的木质部部位优势表达。矮生杉木在解剖与生理方面不同于正常杉木,矮生杉木木质部细胞分化缓慢,木材中纤维素含量相对低[18]。进一步的表达分析也发现:ClCesA1在矮生杉木木质部的转录丰度显著低于正常杉木的相应数值。因此,可以推测ClCesA1是参与杉木木材次生壁形成的重要基因。进化树分析显示ClCesA2与AtCesA1,PtrCesA4,EgCesA5等聚在一起。AtCesA1[16]几乎在拟南芥的各个部位都表达,其表达量远远高于AtCesA9[19],被认为主要参与细胞初生壁的形成。PtrCesA4[8]和EgCesA5[17]也被认为控制初生细胞壁中纤维素的合成。然而,定量PCR分析结果显示,ClCesA2呈现与ClCesA1相似的表达模式,也主要在茎的木质部表达。可见,ClCesA2是否直接参与细胞次生壁的形成有待验证。

图1 不同植物CESA氨基酸序列比对及其基序分析Figure 1 Amino acid residue alignment and motif analysis of different plant CESA sequences
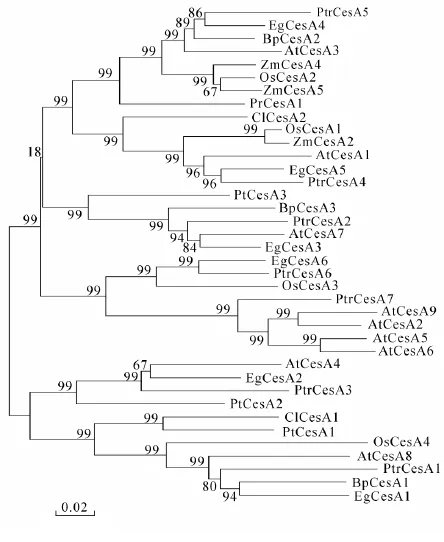
图2 植物CESA蛋白的系统进化分析Figure 2 Phylogenetic tree analysis of CESA from different plant species

图3 不同器官中ClCesA1和ClCesA2基因的相对表达量Figure 3 Relative expression levels of ClCesA1,ClCesA2 in different organs
植物CesA基因是以超基因家族存在的,含有许多的基因成员,不同的成员参与不同组织,不同器官或不同细胞壁层次的纤维素合成。本研究虽从杉木中克隆到了2个CesA基因,但其他CesA基因成员还未克隆到,同时这2个基因的功能还需进行更深入的研究。
[1] PEAR J R,KAWAGOE Y,SCHRECKENGOST W E,et al.Higher plants contain homologs of the bacterial celA genes encoding the catalytic subunit of cellulose synthase[J].Proc Nat Acad Sci USA,1996,93(22):12637-12642.
[2] WU Luguang,JOSHI C P,CHIANG V L.A xylem-specific cellulose synthase gene from aspen(Populus tremuloides)is responsive to mechanical stress[J].Plant J,2000,22(6):495-502.
[3] SUZUKI S,LI Laigeng,SUN Y H,et al.The cellulose synthase gene superfamily and biochemical functions of xylemspecific cellulose synthase-like genes in Populus trichocarpa[J].Plant Physiol,2006,142(3):1233-1245.
[4] ENDLER A,PERSSON S.Cellulose synthases and synthesis in Arabidopsis[J].Mol Plant,2011,4(2):199-211.
[5] RICHMOND T A,SOMERVILLE C R.The cellulose synthase superfamily[J].Plant Physiol,2000,124(2):495-498.
[6] BURN J E,HOCART C H,BIRCH R J,et al.Functional analysis of the cellulose synthase genes CesA1,CesA2 and CesA3 in Arabidopsis[J].Plant Physiol,2002,129(2):797-807.
[7] 陈鹏飞.白桦纤维素合成酶基因克隆与表达特征分析[D].哈尔滨:东北林业大学,2008. CHEN Pengfei.Cloning of Cellulose Synthase Gene from Betula platyphylla and the Analysis of Their Expression Characteristic[D].Harbin:Northeast Forestry University,2008.
[8] SAMUGA A,CHANDRASHEKHAR P.A new cellulose synthase gene(PtrCesA2)from aspen xylem is orthologous to Arabidopsis AtCesA7(irx3)gene associated with secondary cell wall synthesis[J].Gene,2002,296(1/2):37-44.
[9] KALLURI U C,JOSHI C P.Isolation and characterization of a new,full-length cellulose synthase cDNA,PtrCesA5 from developing xylem of Aspen trees[J].J Exp Bot,2003,54(390):2187-2188.
[10] LIN Yen,KAO Yuying,CHEN Zenzong,et al.cDNA cloning and molecular characterization of five cellulose synthase a genes from Eucalyptus camaldulensis[J].J Plant Biochem Biotechnol,2014,23(2):199-210.
[11] 蒋向辉,佘朝文,许栋,等.杉木CCoAOMT基因部分cDNA克隆与序列分析[J].中南林业科技大学学报,2009,29(6):24-29. JIANG Xianghui,SHE Chaowen,XU Dong,et al.Partial cDNA cloning and sequence analysis of CCoAOMT gene of Cunninghamia lanceolata(lamb.)[J].J Central South Univ For&Technol,2009,29(6):24-29.
[12] HUANG Huahong,XU Lili,TONG Zaikang,et al.De novo characterization of the Chinese fir(Cunninghamia lanceolata)transcriptome and analysis of candidate genes involved in cellulose and lignin biosynthesis[J].BMC Genomics,2012,13(1):648.
[13] 吕运舟,郑佳,陈金慧,等.参与杉木次生壁合成调控的转录因子ClMYB4的克隆及在大肠杆菌中表达[J].分子植物育种,2012,10(5):512-519. LÜ Yunzhou,ZHENG Jia,CHEN Jinhui,et al.Cloning transcription factor ClMYB4 involving in secondary cell wall biosynthesis from Cunninghamia lanceolata (Lamb.)Hook and expressing in E.coli[J].Mol Plant Breed,2012,10 (5):512-519.
[14] 彭沙沙,童再康,黄华宏,等.杉木纤维素合成酶类似蛋白基因CLCslD1的克隆及其生物信息学分析 [J].浙江农林大学学报,2012,29(1):1-6. PENG Shasha,TONG Zaikang,HUANG Huahong,et al.Clone and analysis of cellulose synthase-like protein D gene (CSLD)in Cunninghcunia lanceolata[J].J Zhejiang A&F Univ,2012,29(1):1-6.
[15] TAMURA K,DUDLEY J,NEI M,et al.MEGA4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Mol Biol Evol,2007,24(8):1596-1599.
[16] TAYLOR N G,SCHEIBLE W R,CUTLER S,et al.The irregular xylem 3 locus of Arabidopsis encodes a cellulose synthase are required for secondary cell wall synthesis[J].Plant Cell,1999,11(5):769-779.
[17] RANIK M,MYBURG A A.Six new cellulose synthase genes from Eucalyptus are associated with primary and secondary cell wall biosynthesis[J].Tree Physiol,2006,26(5):545-556.
[18] 陈奋学,黄华宏,童再康,等.矮生杉木的解剖特性[J].浙江林学院学报,2008,25(5):619-623. CHEN Fenxue,HUANG Huahong,TONG Zaikang,et al.Anatomical characteristics of dwarf Cunninghamia lanceolata[J].J Zhejiang A For Coll,2008,25(5):619-623.
[19] BEECKMAN T,PRZEMECK G K H,STAMATIOU G,et al.Genetic complexity of cellulose synthase a gene function in Arabidopsis embryogenesis[J].Plant Physiol,2002,130(4):1883-1893.
Isolation and expression analysis of cellulose synthase genes in Chinese fir(Cunninghamia lanceolata)
PANG Jing,TONG Zaikang,HUANG Huahong,LIN Erpei,LIU Qiongyao
(The Nurturing Station for the State Key Laboratory of Subtropical Silviculture,Zhejiang A&F University,Lin’an 311300,Zhejiang,China)
In this study,two full-length cDNAs encoding cellulose synthase(CESA)were isolated from Chinese fir(Cunninghamia lanceolata)using RT-PCR and RACE technology,and their sequence lengths were 3 235 bp and 3 876 bp,respectively.They were named ClCesA1 and ClCesA2,and the corresponding GenBank accession numbers were JQ844574 and JQ844575 respectively.The encoded proteins are composed of 992 and 1 092 amino acids,with theoretical molecular weight of 111 845.3 and 123 105.7 D,whose isoelectric points are 6.04 and 6.65,respectively.Amino acid sequences contain typical motifs of plant CESA proteins:a ring finger domain,two class specific regions CSRI and CSRII,two conserved regions CRI and CRII,cellulose synthase substrate binding domain “D,D,D,QVLRW”,and eight transmembrane regions.The expression analysis showed that ClCesA1 and ClCesA2 genes exhibited the high transcript abundance in stem,the corresponding expression levels in mature xylem were higher than that in cortex.The two genes had the lower expression levels in root and leaf.The further expression analysis was conducted using the dwarf Chinese fir with the specific wood properties.The results showed that the expression levels of ClCesA1 and ClCesA2 in the xylem of normal fir were about 2-12 times than that of the dwarf.Two ClCesA genes may be play importantroles in the wood formation of Chinese fir.[Ch,5 fig.19 ref.]
forest tree breeding;Cunninghamia lanceolata;CesA gene;cloning;sequence analysis;expression analysis
S722.3;Q943
A
2095-0756(2015)01-0040-07
浙 江 农 林 大 学 学 报,2015,32(1):40-46
Journal of Zhejiang A&F University
10.11833/j.issn.2095-0756.2015.01.006
2013-12-30;
2013-05-16
国家高技术研究发展计划(“863”计划)项目(2011AA100203);浙江省农业科技重点项目(2012C12908-11,2011C12014);浙江农林大学亚热带森林资源培育研究中心预研项目(CCSFR2013002)
庞景,从事林木遗传育种研究,E-mail:294144351@qq.com。通信作者:童再康,教授,博士,博士生导师,从事林木遗传育种研究。 E-mail:zktong@zafu.edu.cn
