基于微卫星多重PCR技术的黄喉拟水龟亲子鉴定
2015-03-01萍赵建李伟洪孝友朱新平
文 萍赵 建李 伟洪孝友朱新平
(1. 中国水产科学研究院珠江水产研究所, 农业部热带亚热带水产资源利用与养殖重点实验室, 广州 510380; 2. 上海海洋大学水产与生命学院, 上海 201306)
基于微卫星多重PCR技术的黄喉拟水龟亲子鉴定
文 萍1, 2赵 建1李 伟1洪孝友1朱新平1, 2
(1. 中国水产科学研究院珠江水产研究所, 农业部热带亚热带水产资源利用与养殖重点实验室, 广州 510380; 2. 上海海洋大学水产与生命学院, 上海 201306)
利用黄喉拟水龟(Mauremys mutica)微卫星标记, 筛选出16对微卫星引物, 通过优化各引物比例、荧光接头浓度、退火温度和循环次数, 建立了基于2组各含8个微卫星位点多重PCR体系的黄喉拟水龟亲子鉴定技术。应用2组微卫星多重PCR体系, 通过ABI3130遗传分析仪以及cervus3.0软件对428只黄喉拟水龟进行了个体基因型检测和遗传多样性分析, 结果显示, 群体的平均等位基因数为14.190, 平均多态信息含量为0.748, 平均观测杂合度和期望杂合度分别为0.687、0.771。对89只候选母本及296只子代进行亲子鉴定分析, 结果显示: 在父本信息未知时, 母本鉴定率为 87%; 母本获得的子代个数范围为 1—12, 个体间表现出巨大的差异, 这为选择育种提供了物质基础。黄喉拟水龟多重 PCR亲子鉴定技术的建立为群体遗传多样性分析、家系鉴定管理和选择育种提供了有效的技术手段。
黄喉拟水龟; 微卫星多重PCR; 亲子鉴定
微卫星作为一种分子标记在生物遗传学研究中得到广泛应用, 特别是 Chamberlian等[1]在20世纪80年代发展了微卫星多重PCR (Multiplex PCR) 反应模式后, 更是因其高效已广泛应用于生命科学的各个领域, 其中包括一些水生生物, 如鲢(Hypophthalmichthys molitrix)[2]、斑节对虾(Penaeus monodon)[3]的遗传多样性分析; 三疣梭子蟹(Portunus trituberculatus)[4]、大菱鲆(Scophthalmus maximus)[5]、美洲牡蛎(Crassostrea virginica)[6]、中国明对虾(Fenneropenaeus chinensis)[7]的亲子鉴定; 皱纹盘鲍(Haliotis discus hannai)[8]的遗传分离模式分析等。微卫星的开发, 为水生生物群体遗传多样性、亲子鉴定、家系管理等研究提供了有力的工具。
黄喉拟水龟(Mauremys mutica)隶属龟鳖目(Testudines)、潮龟科(Geoemydidae)、拟水龟属(Mauremys), 具有较高的食用、药用和观赏价值, 在我国主要分布在华东与华南地区, 国外主要分布在越南和日本[9], 已被列入濒危野生动植物种国际贸易公约附录 II[10], 属易危物种。为保护该物种及满足人们的需求, 我国在20世纪90年代开展了黄喉拟水龟人工养殖, 目前已形成了产业。但黄喉拟水龟性成熟时间长, 繁殖力低, 导致苗种供应不足,严重限制了产业规模的发展[11, 12]。本实验室近几年致力于提高黄喉拟水龟繁殖力的研究, 高繁殖力黄喉拟水龟品系选育是其中的一个研究方向。选育需要一个较大群体, 选育过程中需要由后代的数量性状来确定母本的优良性, 而由大群体自由交配产生的后代其系谱难以确定, 导致后续工作难以开展。因此, 有必要开发黄喉拟水龟亲子鉴定技术, 而基于微卫星的亲子鉴定技术则成为首选。
目前关于黄喉拟水龟微卫星标记的报道较少,仅见Zhang等[13]关于黄喉拟水龟微卫星的分离与特性描述。本实验室开发了一些微卫星标记, 结合文献中的微卫星标记, 筛选、优化构建了黄喉拟水龟微卫星多重PCR体系, 用于亲子鉴定及其相关研究,拟以此获得完整、准确的系谱信息, 为高繁殖力黄喉拟水龟品系选育工作打下基础。
1 材料与方法
1.1 试验材料
试验时间在2013年, 试验用父母本材料为人工养殖的9龄黄喉拟水龟, 雌龟89只, 雄龟43只, 共有132只, 采用在背甲上刻字的方法从1—132依次做好标记。养殖场地在中国水产科学研究院珠江水产研究所龟类繁育场。
试验用子代为上述132只父母本2013年在同一个繁育池中产卵, 人工孵化的子代幼龟, 共296只。同一窝龟卵在采集和孵化过程中作好记录, 幼龟孵化出来后, 采用刻字法进行标记。
亲代和子代每个个体抽取10—20 μL血液, 置于装有200 μL buffer TL (MicroElute Genomic DNA Kit试剂盒, OMEGA, USA)的离心管中, 标上与龟个体对应的编号, 冰盒保存, 当天提取DNA。
1.2 基因组DNA的提取
用MicroElute Genomic DNA Kit试剂盒提取样本基因组DNA, 1%琼脂糖凝胶电泳检测DNA的完整性, DNA经NanoQTM微型分光光度计(博奥)检测浓度, 并用去离子水稀释至终浓度 20 ng/μL, 置于–20℃保存备用。
1.3 微卫星引物筛选
从本实验室黄喉拟水龟简化基因组测序数据库及文献中的微卫星标记筛选微卫星引物。一共设计38对微卫星引物, 引物由英潍捷基(上海)贸易有限公司合成。合成时, 每对引物的正向引物 5′端添加相同的接头序列(PET)。通过PCR扩增筛选, 从中选出能稳定扩增、产物条带清晰、产物多态性丰富的16对引物来构建多重PCR体系。16对微卫星引物中, 10对来自实验室开发, 6对来自于发表文献[13]。16对微卫星引物序列信息见表1。
1.4 多重PCR体系构建
根据上述16对引物的退火温度及其PCR产物长度范围进行引物组合, 确定构建2个多重PCR体系, 每个体系包含8个微卫星位点。16对引物由英潍捷基公司合成, 根据添加同种接头的同一体系中的引物其扩增产物不重叠的原则, 合成时在正向引物5′端添加不同的接头序列(PET、VIC、NED、FAM),另外单独的PET、VIC、NED、FAM分别以红色、绿色、黄色、蓝色荧光进行标记, 合成荧光标记接头, 4种接头序列见表2。PCR扩增前将引物稀释至10 μmol/L, 每对引物上下游按1∶40混合; 4种荧光接头均稀释至 20 μmol/L, 并按 1∶1∶1∶1混合,在涡旋混合器上混匀。通过优化体系内各引物混合比例、退火温度、循环次数、荧光接头混合物浓度等条件确定最佳PCR扩增条件。

表1 多重PCR 16对微卫星引物序列Tab. 1 The sixteen microsatellite primers for multiplex PCR

表2 四种荧光标记接头Tab. 2 The four fluorescent joints
PCR 反应体系 10 μL: 包括 1 μL模板 DNA, 5 μL ABI Multiplex PCR Master Mix, 2 μL 10 μmol/L上下游引物混合物, X μL 20 μmol/L荧光标记接头,添加去离子水至10 μL。PCR扩增程序为: 94℃预变性5min, 94℃变性30s, 退火40s, 72℃延伸40s, 若干循环; 94℃变性30s, 53℃退火40s, 72℃延伸30s, 8个循环, 72℃再延伸10min, 4℃保存。
PCR扩增产物加样到 ABI3130遗传分析仪中进行毛细管电泳检测, 同时每个样孔中加入分子量内标 GeneScanTM-500LIZTM和去离子甲酰胺, 微卫星标记基因型采用 Peak Scanner Software V1.0软件分析。
1.5 遗传统计分析及亲子鉴定
采用上述建立的微卫星多重 PCR体系对428只黄喉拟水龟进行微卫星标记基因型检测, 经 Peak Scanner Software V1.0软件分析, 获取其基因型, 并辅以人工校对, 为了保证等位基因分型结果的准确性, 对于不能确定基因型的样品, 可利用完全相同的条件重复 2—3 次扩增和分型。建立亲本(132个体)和子代(296个体)的基因型数据库, 运行Cervus 3.0[14]软件获得各微卫星座位的遗传多样性参数, 包括等位基因数(Number of alleles, Na)、多态信息含量(polymorphism information content, PIC)、观测杂合度(Observed heterozygosity, Ho)、期望杂合度(Expected heterozygosity, He)、哈迪温伯格平衡(Hardy-Weinberg equilibrium, HWE)检测、无效等位基因频率[Null allele frequency, F (Null)]等。采用Cervus 3.0软件进行亲子鉴定, 分析89个母本个体在2013年的繁殖信息。
2 结果
2.1 多重PCR体系构建
通过对多重PCR反应参数的优化, 建立了2组微卫星多重 PCR体系, 每组包含 8个微卫星位点,每组多重 PCR 的具体参数见表 3。其中多重 PCR体系2在黄喉拟水龟中的扩增效果见图1。2.2 群体遗传多样性分析

表3 二组黄喉拟水龟微卫星多重PCR特征Tab. 3 Characteristics of the two multiplex PCRs
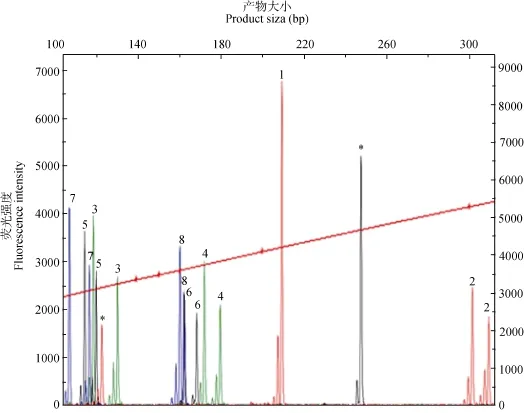
图1 微卫星多重PCR体系2在子代9号中的毛细管电泳图Fig. 1 Electrophorograms of the microsatellite multiple PCR (set 2) of offspring 9
用 2组微卫星多重 PCR体系对 132只亲龟, 296只仔龟进行扩增, 将获得的子代和亲本基因型整理保存为txt格式的文件。采用Cervus 3.0软件分析得到如下结果: 16个微卫星位点共检测到227个等位基因, 单个位点等位基因数(Na)介于 5—26, 平均等位基因数 14.190; 多态信息含量(PIC)介于0.383—0.925, 平均值为0.748, 其中15个微卫星位点PIC>0.5表现为高度多态性, Mmu3位点PIC>0.25表现为中度多态性[15]; 观测杂合度(Ho)介于0.364—0.921, 平均值为 0.687; 期望杂合度(He)介于 0.429—0.930, 平均值为0.771。上述数值表明, 黄喉拟水龟群体遗传多样性较丰富。HWE检验显示: 其中8个位点符合 HWE, 3个位点显著偏离 HWE平衡(P<0.05), 5个位点极显著偏离HWE (P<0.01)。16个位点在428个个体中的具体遗传参数见表4。
2.3 亲子鉴定分析
运行Cervus3.0中Allele frequency analysis程序,获得 16个微卫星座位在群体中各等位基因的频率,以及在下列 3种情况中的非排除概率: 当双亲基因型均未知时, 单个微卫星位点的非排除概率(Nonexclusion probability, NE-1P)介于0.250—0.905, 平均值为0.544, 累积非排除概率(Combined non-exclusion probability, CNE-1P)为0.182×10–4; 已知单亲基因型时单个微卫星位点的非排除概率(NE-2P)介于 0.143—0.778, 平均值为 0.389, 累积非排除概率 (CNE-2P) 为0.004×10–5, 具体参数见表4。
运行Simulation程序中的maternity子程序, 输入等位基因频率文件, 模拟 10000 次亲子鉴定。参数设置为: 母本数 89, 抽样比例为 1, 置信度为95%, 在父本信息未知的情况下进行鉴定, 2个多重PCR 16个微卫星位点的母本模拟鉴定率为100%。
运行Parentage Analysis程序中的maternity子程序, 输入子代及候选母本的基因型文件、等位基因频率分析和模拟分析输出文件, 在父本信息未知的情况下进行鉴定, 结果表明, 在置信水平为 95%时, 258个子代个体找到了母本, 鉴定率为 87%。余下38个子代的母本LOD值小于0, 不宜判定。
将 258个子代的分配情况与原始记录相比较,发现有13个子代鉴定结果与原始记录不一致, 即标记来源于同窝的个体, 其鉴定母本不同。原因可能为: 在产卵期, 不同母龟将卵产在同一地点, 实验人员误认为属于同一窝而放在一起孵化, 或是在孵化出苗过程不慎搞混所导致。258个子代在不同母本中的分配情况如表5所示。
2.4 母本繁殖力分析
本实验89只雌龟成功得到296只子龟, 平均每只母龟有3.3只后代。亲子鉴定可明确分配258只子龟, 这 258个子代只分配到 60个母本中, 有 29个母本没有被分配子代。这结果表明, 有可能一些母龟没有获得后代, 获得后代的不同母本个体也表现出不同的繁殖力, 子代个数介于 1—12, 这些个体在繁殖力上的差异为选择育种提供了物质基础。其中母本 m114的子代数多达 12, 表现出优异的繁殖能力, 这些母本及子代都是黄喉拟水龟高繁殖力品系的选育对象。如果以子代数 7为选择指标, 将有14个母本个体被选择留种, 同时其子代也一同被选择留种以接受下一轮的选择。14个母本占实验群体的 15.7%, 这预示高繁殖力一代的选择率为15.7%。有不同子代数量的母本个数如图2所示。

表4 基于2组多重PCR体系分析的黄喉拟水龟群体遗传多样性参数Tab. 4 The genetic diversity of Asian Yellow Pond Turtles, based on the analysis with two sets of multiplex PCR

表5 子代在不同母本中的分配Tab. 5 The allocation of the offspring in different female parents

图2 有不同子代数量的母本个数Fig. 2 The number of female parents corresponding to different numbers of offspring
3 讨论
3.1 微卫星多重PCR的建立与优化
多重PCR要求多对引物能同时在同一反应体系中进行特异性扩增, 因而技术难度较大, 理想的多重PCR反应体系, 并非单一PCR的简单组合, 需要针对目标产物, 进行全面的分析和反复试验。因此与单个位点PCR相比, 建立多重PCR的前期准备工作比较繁琐, 但技术一旦开发成熟, 可大幅度提高效率。
在影响多重PCR反应的主要因素中, 张毅等[16]认为引物间的兼容性和引物浓度比例是最重要的影响因素; 而黄银花等[17]认为缓冲液的成分对多重PCR的扩增效果产生较大的影响, 而反应体积、循环次数的影响较小。本实验主要对引物组合及其混合比例、退火温度、循环次数进行优化, 结果显示前两个因素影响较大。不同引物组合时, 需注意引物间不能相互作用, 如形成二聚体、发卡结构等, 另外, 激发同种颜色荧光的引物其扩增片段不能重叠,以免造成基因型统计困难; 引物混合比例需根据产物量加以调整, 直至扩增效率相近; 用于构建同一多重PCR体系的引物其退火温度相差不宜超过5℃,在确保每对引物均能扩增出PCR产物前提下, 需尽量将多重PCR退火温度调高, 以免产生杂带。本实验多重 PCR产物采用ABI3130遗传分析仪进行检测、运行Peak Scanner Software V1.0软件分析片段大小, 因此 PCR产物的量应该控制在检测范围内(能够有效检测峰值范围 50—8000), 实验中通过调整模板浓度、循环次数、荧光接头浓度可达到目的。
3.2 试验群体遗传多样性
生物群体的遗传多样性是生物多样性的核心和本质, 也是评估生物资源状况的一个重要指标。一个种群遗传多样性越丰富, 其环境适应能力就越强,生存和进化的潜力也就越大[18]。多态信息含量是基因丰富度的一个指标, 多态信息含量的高低表明了群体遗传基础的丰富多样性, 等位基因数越多, 多态性越丰富。本实验采用的16个微卫星座位的PIC平均值为 0.748, 表明这些基因座位能够提供丰富的信息, 可有效用于黄喉拟水龟遗传多样性分析。杂合度是衡量群体遗传多样性高低的指标。在本研究中 16个微卫星标记的平均观测杂合度(Ho)为0.687, 平均期望杂合度(He)为0.771。以上数据说明该群体遗传多样性水平较高, 与朱新平等[19]运用RAPD分析黄喉拟水龟南北两个群体遗传多样性的结果相类似。
哈迪温伯格平衡检验显示: 16个微卫星位点中有8个位点有不同程度的偏离, 其中5个位点极显著偏离(P<0.01)。杂合子缺失一般是造成群体偏离HWE的主要原因, 5个极显著偏离HWE的位点期望杂合度都远大于它们的观测杂合度, 表明纯合子的个体较多, 说明该试验群体存在一定程度的近交或者存在无效等位基因。在本试验中16个微卫星位点无效等位基因频率介于–0.85%—22.04%, 5个极显著偏离 HWE的位点的无效等位基因频率均大于10%, 说明无效等位基因是造成其偏离HWE一大原因。从本研究的样本来源中可以看出, 非随机交配群体以及育苗过程中的人工选择可能是造成8个位点偏离HWE的另一个主要原因。
3.3 亲子鉴定
本研究使用的亲龟是在2013年3月挑选分池养殖, 由于龟类具有上一年交配的精子可以保留在母体体内至下一年受精的特点[20], 因此有可能部分父本个体不出现在试验群体中, 故本试验只进行了母本群体和子代群体的亲缘关系鉴定。试验中使用 2组微卫星多重PCR体系进行子代与候选母本的关系鉴定, 模拟鉴定率达到100%, 实际鉴定率为87%。
实际鉴定率低于模拟鉴定率, 原因可能是: (1)无效等位基因的存在[21, 22]。本文采用cervus3.0软件分析微卫星位点基因型数据后, 发现16个微卫星位点无效等位基因频率介于–0.85%—22.04%, 其中位点sg8、Mmu7、sg10、Mmu4、sg16的无效等位基因频率大于10%。无效等位基因主要是因为引物无法与微卫星侧翼发生突变的位点相结合, 导致微卫星序列不被扩增而产生的, 且微卫星序列点突变频率很高(10–2—10–5)[23]。(2)基因型统计误差, 本实验多重PCR产物采用ABI3130毛细管电泳检测, 同一样品在不同批次的检测中, 结果有一定的差异, 这可能导致统计结果有一定的误差。另外, 用软件cervus3.0分析时, 需将PCR片段大小化为整数, 也可能会降低鉴定率。另外基因突变, 以及酶在扩增以单、二核苷酸为核心序列的微卫星座位时易发生滑动而引起PCR产物出现同源异型的现象均有可能导致实际鉴定率下降。
亲子鉴定结果显示, 试验群体中不同母本的子代数目有所差异, 这对选择育种有非常大的参考价值。在实际育种过程中, 可以选择子代数目较多的母本及其子代进行留种, 连续几年对其繁殖情况进行观察, 同时观察其子代性成熟之后的繁殖情况,以考察母本高繁殖力能否在子代中遗传。同时根据亲子鉴定, 可将母龟分为高繁殖力与低繁殖力两个群体, 为将来进行分子辅助育种研究提供了重要的研究材料。这些工作将为黄喉拟水龟高繁殖力品系培育打下良好基础。
[1] Chamberlain J S, Gibbs R A, Ranier P N, et al. Deletion screening of the Duchenne muscular dystrophy locus via multiplex DNA amplification [J]. Nucleic Acids Research, 1988, 16(23): 11141—11156
[2] Li X F, Chen X F, Wang S, et al. Optimization and preliminary application of fluorescence multiplex microsatellite PCR for silver carp (Hypophthalmichthys molitrix) [J]. Journal of Agricultural Biotechnology, 2012, 20(7): 822—830 [李小芳, 程晓凤, 王生, 等. 鲢微卫星标记的荧光多重PCR体系建立及其应用.农业生物技术学报, 2012, 20(7): 822—830]
[3] Li Y T, Wong K P, Shekhar M, et al. Development of two microsatellite multiplex systems for black tiger shrimp Penaeus monodon and its application in genetic diversity study for two populations [J]. Aquaculture, 2010, 266(1—4): 279—288
[4] Ren X Y, Liu P, Gao B Q, et al. Microsatellite multiplex PCR gene scanning technology applied for parentage identification of Portunus trituberculatus [J]. Acta Hydrobiologica Sinica, 2012, 36(4): 770—778 [任宪云, 刘萍, 高保全, 等. 微卫星多重PCR基因扫描在三疣梭子蟹个体识别中的应用. 水生生物学报, 2012, 36(4): 770—778]
[5] Miao G D, Du M, Yang J F, et al. Development of two microsatellite multiplex PCR systems for turbot (Scophthalmu smaximus) and its application for family management [J]. Periodical of Ocean University of China, 2011, 41(1—2): 97—106 [苗贵东, 杜民, 杨景峰, 等. 大菱鲆亲子鉴定的微卫星多重 PCR技术建立及应用. 中国海洋大学学报, 2011, 41(1—2): 97—106]
[6] Wang Y, Wang X X, Wang A, et al. A 16-microsatellite multiplex assay for parentage assignment in the easter noyster (Crassostrea virginica Gmelin) [J]. Aquaculture, 2010, 308(S1): 28—33
[7] Gao H, Kong J, Liu P, et al. Establishment of microsatellite-based triplex PCR for parentage analysis of Chinese shrimp Fenneropenaeus chinensis [J]. Acta Oceanologica Sinica, 2007, 26(2): 65—74
[8] Xie H T, L Q, Kong L F. Development of four multiplex PCR panels of microsatellites and application to kinship analysis in the Pacific abalone [J]. Journal of Fisheries of China, 2013, 37(2): 207—215 [聂鸿涛, 李琪, 孔令锋. 皱纹盘鲍微卫星多重 PCR 体系构建及其在家系鉴定中的应用. 水产学报, 2013, 37(2): 207—215]
[9] Zhu X P, Chen Y L, Liu Y H, et al. Comparative study of the growth of Mauremys mutica, Cuora trifasciata and Chlydre serpentine [J]. Journal of Fisheries of China, 2001, 25(6): 507—511 [朱新平, 陈永乐, 刘毅辉, 等.黄喉拟水龟、三线闭壳龟、鳄龟的生长比较.水产学报, 2001, 25(6): 507—511]
[10] Zhao E M. The Red Book about Severe Danger Animals in China Amphibians and Reptiles [M]. Beijing: Science Press. 1998, 120—121 [赵尔宓. 中国濒危动物红皮书.两栖类和爬行类.北京: 科学出版社. 1998, 120—121]
[11] Zhu X P, Chen Y L, Wei C Q, et al. Reproduction of yellow pond turtle, Mauremys mutica [J]. Acta Hydrobiologica Sinica, 2001, 25(5): 449—454 [朱新平, 陈永乐, 魏成清, 等. 黄喉拟水龟的繁殖生物学研究. 水生生物学报, 2001, 25(5): 449—454]
[12] Zhu X P, Chen Y L, Wei C Q, et al. Effects of artificial rearing on fecundity of Mauremys mutica Cantor [J]. Journal of Fishery Sciences of China, 2001, 8(2): 52—54 [朱新平,陈永乐, 魏成清, 等. 人工饲养对黄喉拟水龟繁殖力的影响. 中国水产科学, 2001, 8(2): 52—54]
[13] Zhang Y, Zhang J Y, Zheng R Q. Isolation and characterization of 14 polymorphic microsatellite loci in theAsian yellow pond turtle, Mauremys mutica (Cantor, 1842) [J]. Aquaculture Research, 2010, 41(9), e353—e356
[14] Kalinowski S T, Taper M L, Marshall T C. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment [J]. Molecular Ecology, 2007, 16(5): 1099—1106
[15] Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J]. American Journal of Human Genetics, 1980, 32(3): 314—331
[16] Zhang Y, Sun D X, Zhang Y, et al. Optimized multiplex PCR sets and genetic polymorphism of 30 microsatellite loci in domestic buffalo [J]. Hereditas, 2008, 30(1): 59—64 [张毅, 孙东晓, 张沅, 等.家养水牛 30 个微卫星标记的多重 PCR体系建立及其多态性检测.遗传, 2008, 30(1): 59—64]
[17] Huang Y H, Hu X X, Xu W Z, et al. The factors affecting the efficiency of multiplex PCR [J]. Hereditas, 2003, 25(1): 65—68 [黄银花, 胡晓湘, 徐慰倬, 等.影响多重 PCR 扩增效果的因素. 遗传, 2003, 25(1): 65—68]
[18] Ji W Z, Su B. Principles and Methodologies of Genetic Diversity Studies [M]. Zhejiang: Zhejiang Science and Technology Press. 1999 [季维智, 宿兵. 遗传多样性的研究原理与方法. 浙江: 浙江科学技术出版社. 1999]
[19] Zhu X P, Zhou L, Chen Y L, et al. Phenotypic and genetic variation between two populations of the Chinese yellow pond turtle, Mauremys mutica (Cantor, 1842) [J]. High Technology Letters, 2008, 14(1): 104—111
[20] Pearse D E, Janzen F J, Avise J C. Genetic markers substantiate long-term storage and utilization of sperm by female painted turtles [J]. Heredity, 2001, 86(3): 378—384
[21] Dakin E E, Avise J C. Microsatellite null alleles in parentage analysis [J]. Heredity, 2004, 93(5): 504—509
[22] Wen Y F, K U, Han W J, et al. Null alleles in microsatellite markers [J]. Biodiversity Science, 2013, 21(1): 117—126 [文亚峰, Kentaro Uchiyama, 韩文军, 等. 微卫星标记中的无效等位基因.生物多样性, 2013, 21(1): 117—126]
[23] Vigouroux Y, Jaqueth J S, Matsuoka Y, et al. Rate and pattern of mutation at microsatellite loci in maize [J]. Moleculla Biology and Evolution, 2002, 19(8): 1251—1260
THE PARENTAGE ASSIGNMENT OF MAUREMYS MUTICA USING MULTIPLEX PCR OF MICROSATELLITES
WEN Ping1, 2, ZHAO Jian1, LI Wei1, HONG Xiao-You1and ZHU Xin-Ping1, 2
(1. Key Laboratory of Tropical & Subtropical Fishery Resource Application & Cultivation of Ministry of Agriculture, Pearl River Fisheries Research Institute, Chinese Academy of Fishery Sciences, Guangzhou 510380, China; 2. College of Life Science and Technology, Shanghai Ocean University, Shanghai 201306, China)
In this study, we established a technique for the parentage assignment in Asian Yellow Pond Turtles (Mauremys mutica) based on two multiplex PCR panels. The primer ratio, the concentration of the fluorescent joint, the annealing temperature and the number of cycles were optimized in this technique. Each multiplex PCR panel had eight loci, 10 of which were developed in our lab and the other 6 were chosen from published literatures. With the multiplex PCR tool we performed the genotyping and the genetic diversity analysis on 428 individuals using ABl3I30 genetic analyzer and Cervus 3.0 software. The results showed that the average number of alleles was 14.190; the average polymorphism information content (PIC) was 0.748; the average observed heterozygosity (Ho) and expected heterozygosity (He) were 0.687 and 0.771 respectively. The parentage assignment was performed on 89 candidate female parents and 296 offspring. Without information on the male parent, the identification rate was 87%. The numbers of offspring of different female parents were remarkably various, ranging from 1 to 12. This provided the material foundation for the selective breeding. This technique could be a highly efficient tool for the genetic diversity analysis, the marker-assisted family identification and management, and the selective breeding of Asian Yellow Pond Turtles.
Mauremys mutica; Microsatellite multiplex PCR; Parentage assignment
Q347
A
1000-3207(2015)06-1134-08
10.7541/2015.149
2014-11-18;
2015-01-15
国家自然科学基金(31302176)资助
文萍(1989—), 女, 湖南株洲人; 硕士; 研究方向为种质资源与遗传育种。E-mail: 461211601@qq.com
朱新平(1964—), 男, 博士, 研究员; E-mail: zhuxinping_1964@163.com
