西南印度洋真光层海水中固氮细菌多样性
2015-01-05谢尚微杨俊毅张东声王春生
谢尚微,杨俊毅*,张东声,王春生
(1. 国家海洋局 第二海洋研究所, 浙江 杭州 310012; 2. 国家海洋局 海洋生态系统与生物地球化学重点实验室,浙江 杭州 310012)
西南印度洋真光层海水中固氮细菌多样性
谢尚微1,2,杨俊毅*1,2,张东声1,2,王春生1,2
(1. 国家海洋局 第二海洋研究所, 浙江 杭州 310012; 2. 国家海洋局 海洋生态系统与生物地球化学重点实验室,浙江 杭州 310012)
本文通过构建克隆文库和基因测序的方法研究了西南印度洋真光层海水中固氮细菌nifH基因的多样性。从构建的2个nifH基因克隆文库中共获得76条有效序列,其中46条来自CTD13-30 m文库,分属10个OTUs;30条来自CTD13-125 m文库,分属8个OTUs。系统发育分析结果显示,研究站位nifH基因序列主要分布于Cluster I和Cluster III两个分支,其中Cluster I中包含蓝细菌和变形菌两个分支,蓝细菌以Group B为优势类群,并未获得束毛藻和Group A的nifH基因序列。此外还有少数nifH基因序列分布于Cluster II。总体来看,西南印度洋固氮生物基因与大西洋的亲缘关系更近;固氮生物的多样性较为丰富,受环境条件的影响,群落结构与其它热带、亚热带寡营养海域具有明显的不同。
固氮生物;nifH基因;多样性;西南印度洋
0 引言
海洋生物固氮是海洋新氮的重要来源,据估计全球海洋生物固氮量可达200 Tg N·a-1,这几乎与陆源生物固氮的总和持平[1]。在开放性大洋环境中,生物固氮对真光层新氮输入的贡献可达50%左右[2-3],是海洋氮循环中必不可少的组成部分[4]。
长期以来关于海洋固氮生物的研究主要集中在束毛藻Trichodesmium这一丝状、无异型细胞的蓝藻上,它们是最早发现的海洋固氮生物,广泛分布在全球热带和亚热带海洋,被认为是海洋生物固氮的主要贡献者[5]。然而,基于束毛藻的生物固氮量并不能满足海洋氮预算的平衡[6],本世纪以来,在开放性寡营养大洋环境中发现了大量的单细胞固氮细菌,研究证明这些单细胞细菌在海洋氮循环中发挥着重要作用,其对生物固氮的贡献甚至可能超过束毛藻[7-8]。它们主要包括单细胞蓝细菌和变形菌[9-10]。其中单细胞固氮蓝细菌可分为Group A和Group B两个分支[7-8,11-13]:Group A细胞内缺乏产氧的光合系统II和具有固碳功能的加氧酶,有利于其在白天进行固氮作用,在北大西洋和北太平洋都是重要的固氮生物;Group B以Crocosphaerawatsonii(WH8501)为代表种,具有蓝细菌典型的光合作用机制,但其种群内基因组的多样性极低[14]。与蓝细菌相比,变形菌也是一类重要的固氮生物,且分布更加广泛,具有比蓝细菌更高的多样性[9]。除此之外,海洋环境中的固氮生物还包括厌氧菌(如梭状芽孢杆菌ClostridiumPrazmowski和硫酸盐还原菌Sulfate-Reducing Bacteria等)和古菌等,它们的多样性及其对海洋生物固氮的贡献也不容忽视[9,15-16]。
由于多数固氮生物还不能通过分离培养获得纯种菌株,目前更多的是利用分子生物学技术,以nifH基因为标记物,从分子水平研究固氮生物的多样性。CHIEN和ZINDER[17]在固氮酶基因系统发育分析的基础上将固氮生物分成4个大的分支:Cluster I、II、III、IV。海洋环境中的固氮生物多样性丰富,来自所有4个分支的固氮生物在海洋中均有发现,其中Cluster I是最主要的海洋固氮生物分支,包含了蓝细菌、α-变形菌、β-变形菌和γ-变形菌,在数量和多样性上占有绝对优势。Cluster II包含少数变形菌和古菌,Cluster III中的固氮生物以严格的厌氧微生物为主,Cluster IV以产甲烷古菌为主[9]。
随着分子生物学技术的进步,我们对海洋固氮生物的多样性、系统发育和分布范围有了全新的认识。例如,高通量测序的结果表明,大洋表层海水固氮生物的优势类群可能是变形菌而不是蓝细菌[18];在传统观点中不适合固氮生物生存的极地环境中也发现了nifH基因[19]。这些研究成果进一步证明了固氮生物在海洋氮循环中的重要地位。
尽管已经得到各国学者的重视,但受采样和研究方法等条件限制,针对固氮细菌多样性的研究有限,主要集中在大西洋和太平洋[10,14,20],印度洋的相关报道则相当匮乏,仅有个别报道[21-22]。西南印度洋的营养盐水平较低、水文环境条件复杂[23-24],受西风带影响,海水温度较低、风力较大,并不是传统观点上海洋固氮生物的理想生境[5]。然而,据估计印度洋生物固氮可达25 Tg/a[2],其单位面积的固氮速率甚至高于大西洋[25],关于固氮生物的研究与其在该海域的重要性有明显差距。本文对西南印度洋真光层海水固氮生物进行研究,揭示了该海域固氮生物的多样性和分布特征,为进一步评估海洋固氮生物的分布及其对新氮的贡献提供科学资料。
1 材料与方法
1.1 研究海区和采样分析方法
样品由“大洋一号”科学考察船2010年1月采集自西南印度洋(21V-CTD13站位:37.789 4°S、49.636 7°E,图1)。使用Seabird 911Plus采水器分别采集30 m层和125 m层海水水样。每层取4 L水样,用200 μm筛绢预过滤后,过滤于孔径为0.2 μm的核孔滤膜上,滤膜保存于-20 ℃,带回实验室分析。
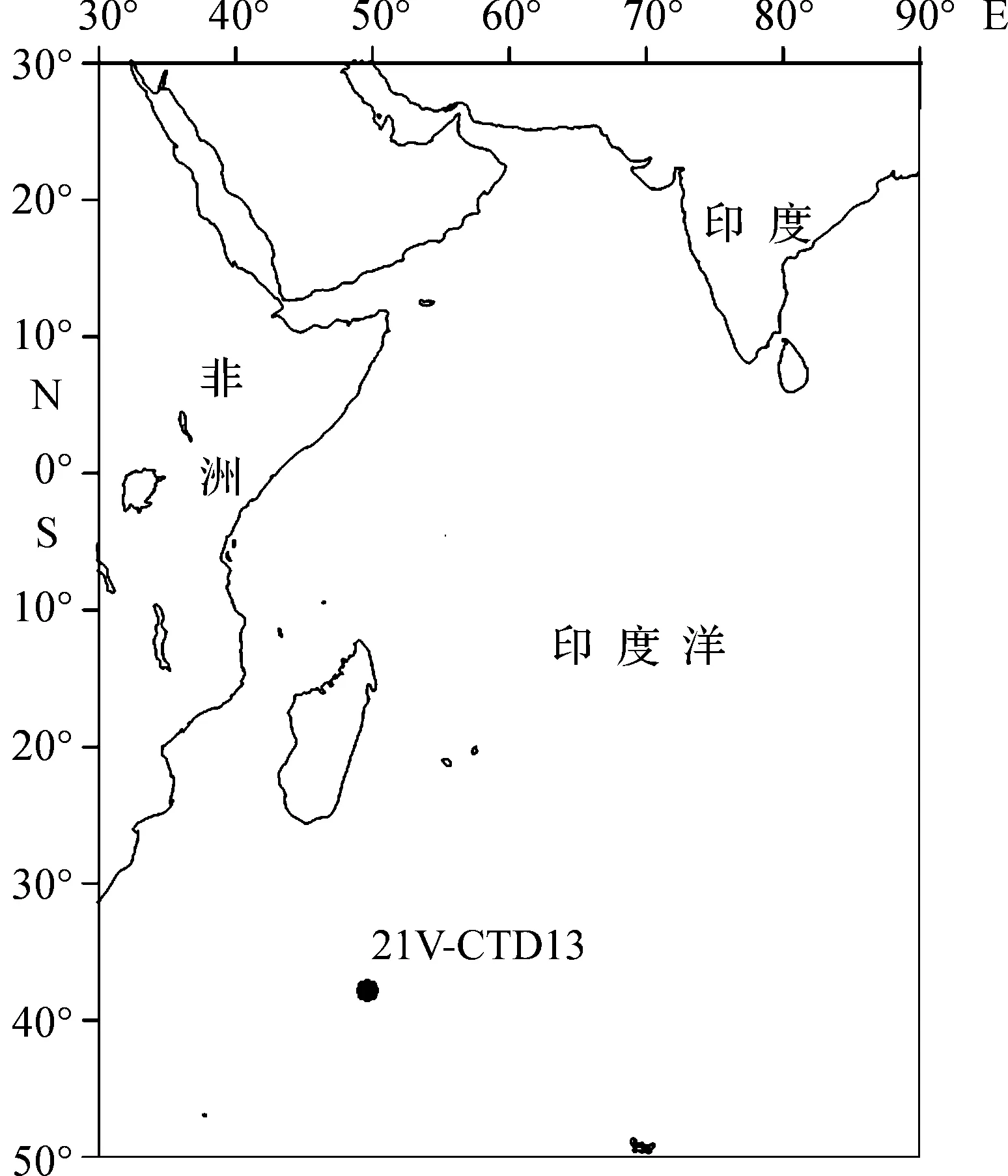
图1 西南印度洋固氮生物采样站位Fig.1 Sampling station in the southwest Indian Ocean
1.2 DNA提取
参照LANGLOIS et al[12]的方法,使用QIAGEN DNA提取试剂盒(DNeasy Plant mini kit)提取滤膜上的环境总DNA。
1.3 nifH基因扩增与克隆
以从滤膜上提取的环境总DNA为模板,对nifH基因进行巢式PCR,两轮引物分别为:第一轮引物[26]:nifH1: 5’-TTYTAYGGNAARGGNGG-3’,nifH2: 5’-ATRTTRTTNGCNGCRTA-3’;第二轮引物[27]:nifH3: 5’-TGYGAYCCNAARGCNG A-3’,nifH4: 5’-ADNGCCATCATYTCNCC-3’。50 μL PCR反应体系:Mg2+(80 nmol/L)、dNTPs(400 μmol/L)、上下游引物(1 μmol/L)、Taq酶(2.5 U)、10×PCR缓冲液 5 μL、DNA模板2 μL,无菌水补充至50 μL。PCR反应条件如下:94 ℃变性4 min;30个循环:94 ℃变性60 s,57 ℃退火30 s,72 ℃延伸40 s;72 ℃延伸10 min。第一轮PCR产物作为DNA模板进行第二轮PCR反应,两轮PCR反应条件相同。PCR产物使用1%琼脂糖凝胶电泳检验。
nifH基因的PCR产物经凝胶回收试剂盒纯化后,连接到pMD20-T Vector(TaKaRa)上,转入大肠杆菌感受态细胞(E.coli JM109 Competent cells, TaKaRa),涂布到含有氨苄青霉素(Ampicillin)/IPTG/X-Gal的LB固体培养基上,37 ℃培养过夜,挑取白色克隆子,4 ℃保存。利用T7和SP6进行菌落PCR,筛选插入目的片段的阳性克隆子并直接利用菌液进行DNA序列测定,构建克隆文库。测序反应由上海生工生物工程公司完成。
1.4 序列分析
将测序所得序列在NCBI中进行同源性比对,挑选与序列亲缘关系最相似序列,利用CHECK-CHIMERA软件分析检查去除嵌合体[28]。使用Clustal X 1.83和Mega 4.0分析多重对比结果,构建系统发育树。将nifH基因序列提交GenBank数据库,登记号为KF740730—KF740805。
1.5 环境参数分析
温度和盐度数据由Seabird 911Plus CTD直接测得。用于叶绿素a分析的样品采集自0、10、20、30、50、75、100、125、150、175和200 m层,每层水样分别取500 mL,依次用孔径为20、2和0.2 μm的滤膜(Millipore NY2004700、TTTP04700、GTTP04700)分级过滤,分析测定细胞粒径大于20、2~20和0.2~2 μm三个粒级叶绿素a质量浓度,各粒级叶绿素a质量浓度之和为总叶绿素a质量浓度。叶绿素a测定采用荧光法[29]。
2 结果
2.1 温度、盐度及叶绿素a质量浓度的垂直分布
调查站位200 m以浅水层温、盐垂直分布较均匀,水温在16~19 ℃之间,随水深增大而逐渐降低;盐度在35.5~35.6之间,垂直分布呈单峰形态,高值在水深40 m左右(图2)。
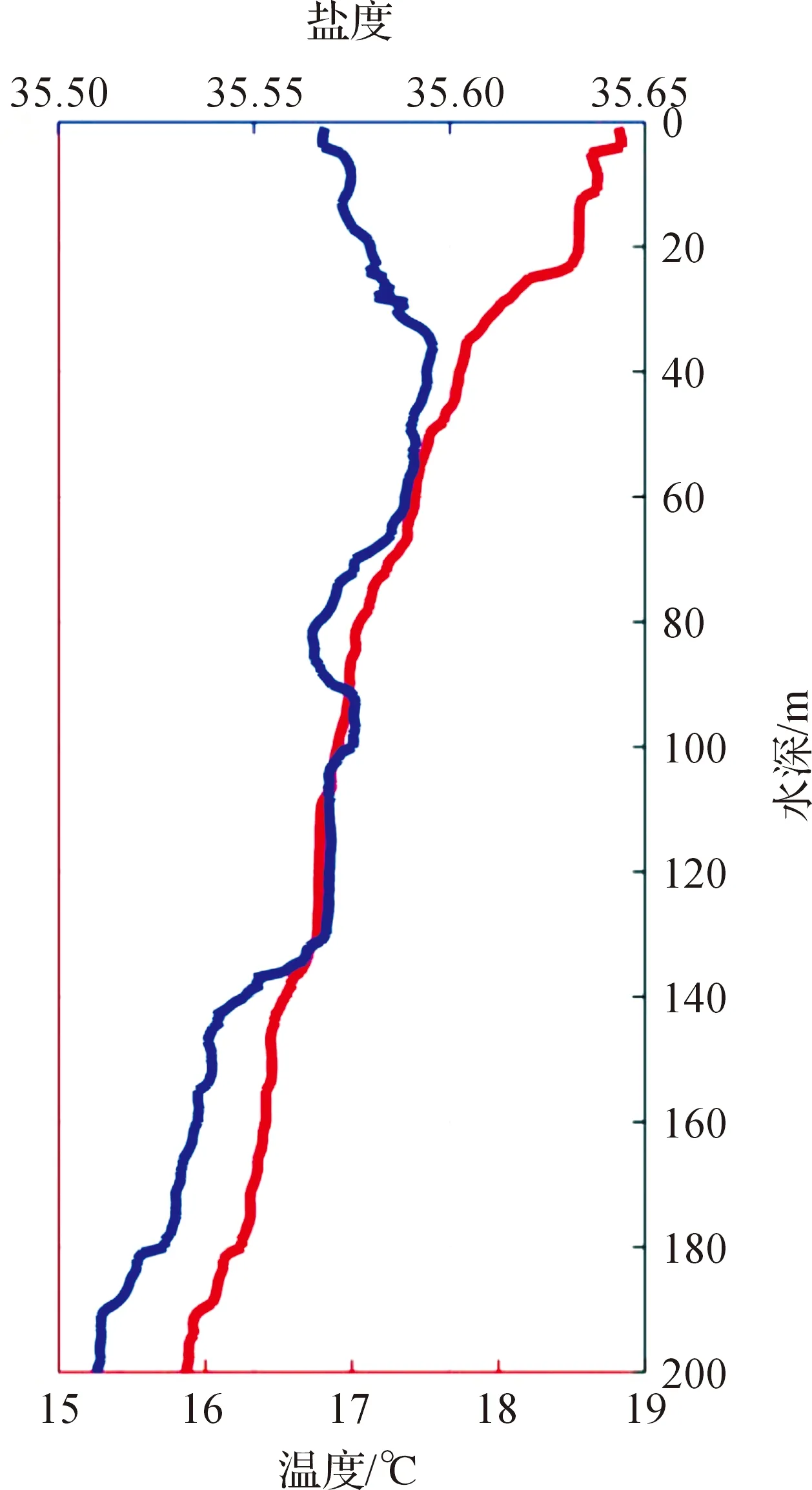
图2 CTD13站位温、盐垂直分布Fig.2 Vertical distribution of temperature and salinity on CTD13 station
叶绿素a质量浓度的垂直分布呈典型的单峰形态(图3),总叶绿素a质量浓度最大值(0.336 μg/L)位于100 m层,30 m层和125 m层的总叶绿素a质量浓度相近,分别为0.106 μg/L和0.134 μg/L。微微型浮游植物(Pico,0.2~2 μm)对总叶绿素a质量浓度的贡献占绝对优势,平均达到70.2%,垂直分布特征也与总叶绿素a质量浓度一致;其次是微型浮游植物(Nano,2~20 μm),占总叶绿素a质量浓度的27.4%;小型浮游植物(Micro,>20 μm)占总叶绿素a质量浓度比例最低,仅2.4%。

图3 CTD13站位各粒级叶绿素a质量浓度垂直分布Fig.3 Vertical distribution of Chlorophyll a concentration of different size on CTD13 station
2.2 nifH基因克隆文库构建和多样性分析
构建CTD13-30 m和CTD13-125 m两个nifH基因文库,对获得的阳性克隆进行测序,共获得76条有效的nifH基因,其中CTD13-30 m文库46条,CTD13-125 m文库30条。以基因相似性≥97%为一个OTU统计,CTD13-30 m文库获得10个OTUs,CTD13-125 m文库获得8个OTUs,稀释度曲线(图4)显示这2个文库均已趋于饱和。
系统发育分析结果显示,CTD13-30 m和CTD13-125 m两个文库中的nifH基因分属于Cluster I、Cluster II和Cluster III这3个已知的分支。CTD13-30 m文库中的nifH基因在3个分支中均有分布,CTD13-125 m文库中的nifH基因仅分布于Cluster I和Cluster III两个分支(图5和图6)。
属于Cluster I的nifH基因大多数与固氮蓝细菌亲缘关系较近,其中25条nifH基因属于Group B分支,该分支以Crocosphaerawatsonii为代表,是寡营养海域单细胞固氮蓝细菌的一个重要分支[7]。另有3条nifH基因不属于蓝细菌nifH基因已知的分支,CTD13-30 m-56与色球藻属Chroococcus和从现代海洋叠层石中获取的未培养nifH基因亲缘关系较近,CTD13-125 m-21与来自中国南海海水中的未培养nifH基因亲缘关系较近,CTD13-125 m-13则与来自土壤和活性污泥等环境中的未培养nifH基因亲缘关系较近(图5)。4条nifH基因与γ变形菌的亲缘关系较近,来自CTD13-125 m文库的1条nifH基因与Vibriodiazotrophicus亲缘关系较近,来自CTD13-30 m文库的3条nifH基因与太平洋和大西洋寡营养海水中的未培养nifH基因亲缘关系较近(图5)。
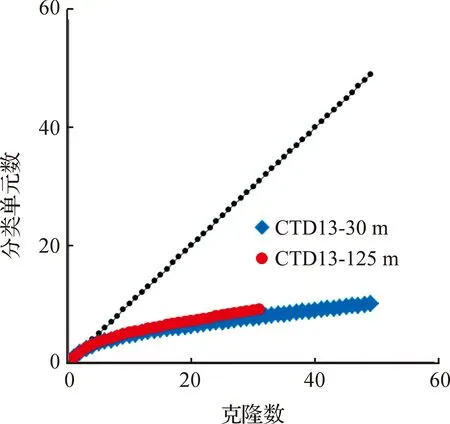
图4 CTD13-30 m和CTD13-125 m nifH基因文库稀释度曲线Fig.4 Rarefaction curves of OTUs of uncultured nifH clones
41条nifH基因序列属于Cluster III,该分支包含厌氧的硫还原菌(如δ变形菌)、梭状芽胞杆菌和紫硫菌(Purple Sulfur Bacteria)等。Cluster III又可以分成2个聚类分支,1支包含CTD13-125 m文库的8条nifH基因,它们与来自开放性大洋和深海沉积物的未培养nifH基因亲缘关系较近,另一支包含CTD13-30 m文库的23条nifH基因和CTD 13-125 m文库的10条nifH基因,它们与来自活性污泥环境中的未培养nifH基因亲缘关系较近(图6)。
Cluster II仅包含3条CTD13-30 m文库的nifH基因,也可分成2个分支,CTD13-30 m-2和CTD13-30 m-44与来自波罗的海的未培养nifH基因亲缘关系较近,CTD13-30 m-13与密螺旋体的nifH基因亲缘关系较近(图6)。
3 讨论与结论
本文结果显示西南印度洋海域固氮细菌nifH基因具有较为独特的多样性和群落结构特征。与热带、亚热带的太平洋[7]和大西洋[30]海域相比,本文构建的克隆文库中未获得来自束毛藻的nifH基因,这可能是受温度限制的结果。温度是限制固氮细菌生长和分布的一个重要因素,例如束毛藻,主要分布在水温大于20 ℃的海域[31],相比较而言,单细胞固氮生物对温度的耐受性更强,是低温环境下固氮生物的优势类群[12]。调查站位200 m以浅水层温度在16~19 ℃之间(图2),30 m和125 m层的温度分别为17.9 ℃和16.6 ℃,均低于束毛藻的适宜温度下限,不利于束毛藻的生长繁殖。
Group B是本文研究站位固氮生物的主要优势类群,温度较高的CTD13-30 m文库中共获得16条属于Group B的nifH基因,占其文库百分比的34.8%,温度较低的CTD13-125 m文库获得9条属于Group B的nifH基因,占文库总序列数的30.0%,前者明显高于后者。有研究表明,单细胞固氮蓝细菌Group B的数量与所在环境水温之间存在显著的正相关[13],温度越高数量越多。这与本文的结果一致,说明本文调查站位中Group B的数量与温度可能也存在正相关关系。
固氮生物是一类全球性分布的生物,LANGLOIS et al[30]认为热带、亚热带大西洋和太平洋的单细胞蓝细菌和Cluster III的nifH基因具有很高的同源性。BIRD et al[32]发现北印度洋阿拉伯海的变形菌nifH基因与大西洋和太平洋的变形菌nifH基因相似性很高。本文获得的Group BnifH基因与大西洋Group B的非培养nifH基因相似性较高(98%),与太平洋Group B的非培养nifH基因相似性较低(93%),这意味着,西南印度洋Group B固氮蓝细菌与大西洋固氮蓝细菌具有更近的亲缘关系。来自变形菌分支的nifH基因与大西洋和太平洋的变形菌nifH基因都具有很高的相似性,而Cluster III的nifH基因与大西洋和太平洋同一分支中的nifH基因相似性都很低。本文还获得了属于Cluster II分支的3条nifH基因,这一分支很少出现在热带、亚热带的太平洋和大西洋,在深海沉积物[33]、热液喷口[34]等海洋环境中更为常见,受这一环境特征影响,可能与产甲烷细菌具有较近的亲缘关系[34]。
尽管固氮生物广泛分布在全球海洋当中,但不同海域固氮生物的群落结构和多样性存在显著差异[18]。在热带、亚热带北大西洋,以束毛藻为主的固氮蓝细菌在固氮生物中占绝对优势,是生物固氮的主要来源[12,35];在亚热带北太平洋单细胞蓝细菌(Group A)则成为固氮生物的优势类群,同时也是生物固氮的主要来源[8,11]。本文的研究结果表明西南印度洋固氮细菌群落结构与西南太平洋[18]相似,以蓝细菌和Cluster III的单细胞固氮细菌为主,这可能是由两者之间相似的环境特征造成的。尽管还缺乏更为直接的证据,本文结果仍表明Cluster III可能在西南印度洋的生物固氮和单循环中扮演重要角色。实际的固氮酶表达活性和细菌丰度仍需要进一步研究,以证明Cluster III固氮细菌的固氮能力。
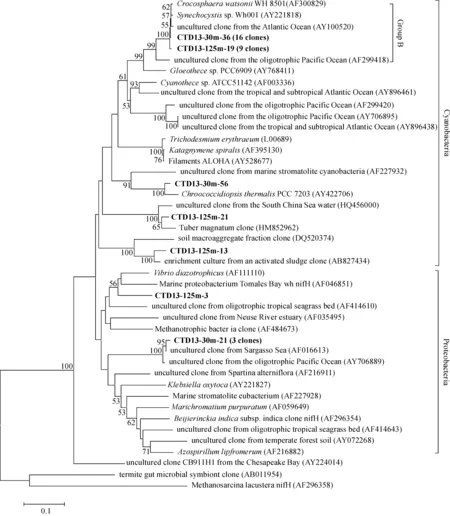
图5 nifH基因Cluster I分支的系统发育树Fig.5 Neighbor-joining phylogenetic tree of Cluster I nifH gene sequences

图6 nifH基因Cluster II和Cluster III分支的系统发育树Fig.6 Neighbor-joining phylogenetic tree of Cluster II and Cluster III nifH gene sequences
综上所述,西南印度洋固氮细菌nifH基因与其他海区相比具有较为独特的群落结构,Group B和Cluster III是固氮细菌的主要优势类群。从系统发育上来看,该海域的nifH基因与大西洋更为接近。
致谢 感谢“大洋一号”全体船员在采样过程中提供的帮助,感谢国家海洋局第二海洋研究所王渊助理研究员提供温、盐数据。
[1] GALLOWAY J, DENTENER F J, CAPONE D G, et al. Nitrogen cycles: Past, present and future[J]. Biogeochemistry,2004,70(2):153-226.
[2] DEUTSCH C A, GRUBER N P, KEY R M, et al. Denitrification and N2fixation in the Pacific Ocean[J]. Global Biogeochem Cycles,2001,15(2):483-506.
[3] MICHAELS A F, BATES N R, BUESSELER K O, et al. Carbon-cycle imbalances in the Sargasso Sea[J]. Nature,1994,372(6506):537-540.
[4] KARL D, MICHAELS A, BERGMAN B, et al. Dinitrogen fixation in the world’s oceans[J]. Biogeochemistry,2002,57(1):47-98.
[5] CAPONE D G, ZEHR J P, PAERL H W, et al.Trichodesmium: a globally significant marine cyanobacterium[J]. Science,1997,276(5316):1 221-1 229.
[6] ZEHR J P, CARPENTER E J, VILLAREAL T A. New perspectives on nitrogen-fixing microorganisms in tropical and subtropical oceans[J]. Trends Microbiol,2000,8(2):68-73.
[7] ZEHR J P, WATERBURY J B, TURNER P J, et al. Unicellular cyanobacteria fix N2in the subtropical North Pacific Ocean[J]. Nature,2001,412(6847):635-638.
[8] MONTOYA J P, HOLL C M, ZEHR J P, et al. High rates of N2fixation by unicellular diazotrophs in the oligotrophic Pacific Ocean[J]. Nature,2004,430:1 027-1 032.
[9] ZEHR J P, JENKINS B D, SHORT S M, et al. Nitrogenase gene diversity and microbial community structure: a cross-system comparison[J]. Environmental Microbiology,2003,5(7):539-554.
[10] FALCON L I, CARPENTER E J, CIPRIANO F, et al. N2fixation by unicellular bacterioplankton from the Atlantic and Pacific Oceans: Phylogeny and in situ rates[J]. Appl Environ Microbiol,2004,70(2):765-770.
[11] CHURCH M J, BJORKMAN K M, KARL D M. Regional distribution of nitrogen-fixing bacteria in the Pacific Ocean[J]. Limnol Oceanogr,2008,53(1):63-77.
[12] LANGLOIS R J, HUMMER D, LAROCHE J. Abundances and distribution of dominantnifHphylotypes in the northern Atlantic Ocean[J]. Appl Environ Microbiol,2008,74(6):1 922-1 931.
[13] MOISANDER P H, BEINART R A, HEWSON I, et al. Unicellular cyanobacterial distributions broaden the oceanic N2fixation domain[J]. Science,2010,327(5972):1 512-1 514.
[14] ZEHR J P, MONTOYA J P, JENKINS B D, et al. Experiments linking nitrogenase gene expression to nitrogen fixation in the North Pacific subtropical gyre[J]. Limnol Oceanogr,2007,52(1):169-183.
[15] BURNS J A, ZEHR J P, CAPONE D G. Nitrogen fixing phylotypes of Chesapeake Bay and Neuse River Estuary sediments[J]. Microb Ecol,2002,44(4):336-343.
[16] DANG Hong-yue, LUAN Xi-wu, ZHAO Jing-yi, et al. Diverse and novelnifHandnifH-like genes sequences in the deep-sea methane seep sediments of the Okhotsk Sea[J]. Appl Environ Microbiol,2009,75(7):2 238-2 245.
[17] CHIEN Y T, ZINDER S H. Cloning, functional organization, transcript studies, and phylogenetic analysis of the complete nitrogenase structural genes (nifHDK2) and associated genes in the archaeonMethanosarcinabarkeri227[J]. J Bacteriol,1996,178(1):143-148.
[18] FARNELID H, ANDERSSON A F, BERTILSSON S, et al. Nitrogenase gene amplicons from global marine surface waters are dominated by genes of non-cyanobacteria[J]. Plos One,2011,6(4):e19223.
[19] POMMIER T, PINHASSI J, HAGSTROM A. Biogeographic analysis of ribosomal RNA clusters from marine bacterioplankton[J]. Aquat Microb Ecol,2005,41(1):79-89.
[20] KITAJIMA S, FURUYA K, HASHIHAMA F, et al. Latitudinal distribution of diazotrophs and their nitrogen fixation in the tropical and subtropical western North Pacific[J]. Limnol Oceanogr,2009,54(2):537-547.
[21] BIRD C, MARTINEZ J M, O’DONNEL A G, et al. Spatial distribution and transcriptional activity of an uncultured clade of planktonic diazotrophic γ-Proteobacteria in the Arabian Sea[J]. Appl Environ Microbiol,2005,71(4):2 079-2 085.
[22] POULTON A J, STINCHCOMBE M C, QUARTLY G D. High numbers ofTrichodesmiumand diazotrophic diatoms in the southwest Indian Ocean[J]. Geophysical Research Letters,2009,36(15):139-156.
[23] RAJAKUMAR A, ALAGARSAMY R, KHARE N, et al. Studies on the nutrient distribution in the Southern Ocean waters along the 45°E transect[J]. Indian Journal of Marine Sciences,2008,37(4):424-429.
[24] READ J F, LUCAS M I, HOLLEY S E, et al. Phytoplankton, nutrients and hydrography in the frontal zone between the Southwest Indian Subtropical gyre and the Southern Ocean[J]. Deep-Sea Research I,2000,47(12):2 341-2 367.
[25] WAJIH S, NAQVI A. Nitrogen in the marine environment[M]. Second Edition. Elsevier,2008:631-671.
[26] ZEHR J P, MCREYNOLDS L A. Use of degenerate oligonucleotides for amplification of thenifHgene from the marine cyanobacteriumTrichodesmiumthiebautii[J]. Appl Environ Microbiol,1989,55(10):2 522-2 526.
[27] ZANI S, MELLON M T, COLLIER J L, et al. Expression ofnifHgenes in natural microbial assemblages in Lake George, New York, detected by reverse transcriptase PCR[J]. Appl Environ Microbiol,2000,66(7):3 119-3 124.
[28] COLE J R, CHAI B, MARSH T L, et al. The Ribosomal Database Project (RDP-II): Previewing a new autoaligner that allows regular updates and the new prokaryotic taxonomy[J]. Nucleic Acids Research,2003,31(1):442-443.
[29] GB/T 12763.6-2007 Specifications for oceanographic survey[S]. Beijing: China Standard Press,2007. GB/T 12763.6—2007 海洋调查规范[S].北京:中国标准出版社,2007.
[30] LANGLOIS R J, LAROCHE J, RAAB P A. Diazotrophic diversity and distribution in the tropical and subtropical Atlantic Ocean[J]. Appl Environ Microbiol,2005,71(12):7 910-7 919.
[31] BREITBARTH E, OSCHLIES A, LAROCHE J. Physiological constraints on the global distribution ofTrichodesmium-effect of temperature on diazotrophy[J]. Biogeosciences Discussions,2006,3(3):779-801.
[32] BIRD C, MARTINEZ J M, O’DONNELL A G, et al. Spatial distribution and transcriptional activity of an uncultured clade of plankton diazotrophic γ-proteobacteria in the Arabian Sea[J]. Appl Environ Microbiol,2005,71(4):2 079-2 085.
[33] DANG Hong-yue, YANG Jin-ying, LI Jing, et al. Environment-dependent distribution of the sedimentnifH-harboring microbiota in the Northern South China Sea[J]. Appl Environ Microbiol,2013,79(1):121-132.
[34] MEHTA M P, BUTTERFIELD D A, BAROSS J A. Phylogenetic diversity of nitrogenase (nifH) genes in deep-sea and hydrothermal vent environments of the Juan de Fuca Ridge[J]. Appl Environ Microbiol,2003,69(2):960-970.
[35] FOSTER R A, SUBRAMANIAM A, ZEHR J P. Distribution and activity of diazotrophs in the Eastern Equatorial Atlantic[J]. Environ Microb,2009,11(4):741-750.
Phylogenetic diversity ofnifHgenes in the euphotic zone of the southwest Indian Ocean
XIE Shang-wei1,2, YANG Jun-yi*1,2, ZHANG Dong-sheng1,2, WANG Chun-sheng1,2
(1.TheSecondInstituteofOceanography,SOA,Hangzhou310012,China; 2.LaboratoryofMarineEcosystemandBiogeochemistry,SOA,Hangzhou310012,China)
Our knowledge on diazotrophs is limited in the Indian Ocean, which is considered an important source of biological N2fixation of the global ocean. Phylogenetic diversity ofnifHgenes in the southwest Indian Ocean was studied through cloning and sequencing methods. After sequences were checked, twonifHgene libraries were established. Overall, 76nifHgene sequences are recovered from Station CTD13, including 46nifHgene sequences of 10 OTUs from libraries CTD13-30 m, and 30nifHgene sequences of 8 OTUs from libraries CTD13-125 m, respectively. Results of phylogenetic analysis show that mostnifHgene sequences fall into Cluster I and III, only 3 recoverednifHgene sequences are grouped with Cluster II. In Cluster I, most ofnifHgene sequences cluster with unicellular cyanobacteria Group B, while in Cluster III, mostnifHgene sequences are closely related with uncultured clone from active sludge. NonifHgene sequences clustered withTrichodesmiumare recovered, which may be attributed to low water temperature. In general, diazotroph community in the southwest Indian Ocean is different from those in tropical and subtropical oligotrophic ocean.
diazotroph;nifHgene; diversity; the southwest Indian Ocean
10.3969/j.issn.1001-909X.2015.03.008.
2015-03-24
2015-05-19
国家自然科学基金项目资助(41206104); 国际海域资源调查与开发“十二五”课题资助(DY125-11-E-03)
谢尚微(1990-),女,浙江温州市人,主要从事海洋生物学方面的研究。E-mail:410074144@qq.com
*通讯作者:杨俊毅(1970-),男,研究员,主要从事海洋生物与生态环境监测技术方面的研究。E-mail:junyiyang@sio.org.cn
Q938.1
A
1001-909X(2015)03-0054-08
10.3969/j.issn.1001-909X.2015.03.008
谢尚微,杨俊毅,张东声,等. 西南印度洋真光层海水中固氮细菌多样性[J]. 海洋学研究,2015,33(3):54-61,
XIE Shang-wei, YANG Jun-yi, ZHANG Dong-sheng, et al. Phylogenetic diversity ofnifHgenes in the euphotic zone of the southwest Indian Ocean[J]. Journal of Marine Sciences, 2015,33(3):54-61, doi:10.3969/j.issn.1001-909X.2015.03.008.
