大口黑鲈北方亚种和佛罗里达亚种mtDNA COⅠ序列的分析
2014-02-08张大莉,董仕,白俊杰等
大口黑鲈北方亚种和佛罗里达亚种mtDNA COⅠ序列的分析
采用PCR技术对大口黑鲈Micropterus salmoides北方亚种 (21尾)和佛罗里达亚种 (19尾)的mtDNA COⅠ区段进行扩增,得到约1400 bp的扩增片段,经测序获得了碱基排列顺序清晰的1337 bp片段。北方亚种个体间没有变异,只检测到1种单倍型,与GenBank中大口黑鲈全序列 (DQ536425)中的COⅠ区段相应部位比对,有5个碱基出现置换;佛罗里达亚种检测到5种单倍型,19个个体与本研究中的北方亚种比对,有41个碱基出现完全置换。将6种单倍型的序列提交GenBank后获得序列号为KF176376~KF176381,运用Mega 4.0软件计算两个亚种的碱基组成和碱基差异,北方亚种的单倍型多样性指数 (H)、核苷酸多样性指数 (Pi)和平均核苷酸差异性 (k)均为0,佛罗里达亚种的H、Pi、k分别为0.784、0.003 42和4.573,两个亚种平均Kimura 2-parameter遗传距离为0.0351。研究表明,大口黑鲈佛罗里达亚种mtDNA COⅠ区段的遗传多样性高于北方亚种,该序列可作为鉴别两个亚种的DNA条形码。
大口黑鲈;COⅠ序列;遗传多样性;遗传距离;NJ系统树
大口黑鲈Micropterus salmoides隶属于鲈形目Perciformes、鲈亚目 Percoidei、太阳鱼科 Centrarchidae、黑鲈属 Micropterus,原产于北美地区,又称加州鲈[1]。根据大口黑鲈的形态和地理分布可将其分为两个亚种:北方亚种M.s.salmoides和佛罗里达亚种M.s.floridanus,北方亚种分布于美国中东部、墨西哥东北部、加拿大东南部,佛罗里达亚种分布于佛罗里达州南部[2-3]。大口黑鲈是肉食性鱼类,肉质鲜美,生长速度快,耐低温,是重要的淡水养殖鱼类之一。中国大陆于1983年引入大口黑鲈,目前国内大部分地区均有养殖[4]。
鱼类线粒体 DNA(Mitochondrial DNA,mtDNA)是一种环状共价闭合的双链DNA,具有自身转录RNA与翻译蛋白质的体系[5]。线粒体DNA呈母系遗传、结构简单、分子量小、进化速度快且不发生重组,被广泛应用于物种起源、遗传分化、种内种间系统发生关系和种类鉴定等方面[6-13]。与其他基因片段相比,线粒体细胞色素C氧化酶亚基Ⅰ (cytochrome C oxidase subunitⅠ,COⅠ)基因有许多优点,相对保守又有足够的变异,并且序列长度适中。DNA条形码技术 (DNA Barcoding)是通过对一个标准目的基因的DNA序列进行分析而进行物种鉴定的技术[14-15]。目前,线粒体COⅠ基因已成为研究动物DNA条形码最重要的标准基因。Hebert等[14-15]对动物界 11 门 2238 个物种的COⅠ基因序列进行分析,发现其序列间的差异能够很好地区分所有研究物种,并认为在动物界中COⅠ基因是合适的DNA条形码标准基因。本研究中,以大口黑鲈为材料,用PCR扩增COⅠ基因的部分区段并测序,比较分析亚种内、亚种间的遗传结构差异,并构建系统树,旨在为两个亚种的鉴别以及遗传多样性的分析提供参考。
1 材料与方法
1.1 材料
试验用大口黑鲈北方亚种21尾,于2011年9月21日取自天津市天祥水产有限责任公司,体长为 (15.18±3.67)cm;大口黑鲈佛罗里达亚种19尾,于2012年1月11日由中国水产科学研究院珠江水产研究所提供,体长为 (16.34±2.26)cm。
1.2 方法
1.2.1 基因组DNA的提取 在鲜活状态下,从每尾试验鱼体上剪取大小约1.0 cm×0.5 cm的鳍条,用苯酚-氯仿抽提法提取总基因组DNA,4℃下保存备用[16]。
1.2.2 mtDNA COⅠ基因区段的扩增及凝胶电泳依据GenBank登录的大口黑鲈mtDNA全序列(DQ536425),参考文献[17-22]中的 COⅠ序列通用引物和变异集中位置,通过Blast软件比对确定相对保守的序列,利用Primer Premier 5.0软件[23]设计引物。上游引物命名为 MSA5524:5'TCTATTTAGTATTTGGTGCTTGAG 3';下游引物命名为 MSA6972:5'GAGGAGGGCAGCCGTGAA 3',两个引物的结合位点均位于COⅠ基因区段内。以提取的总基因组DNA为模板,使用所设计的引物对每尾试验鱼的COⅠ区段进行扩增,将PCR产物委托华大基因公司进行测序,依据峰图确认,获得碱基排列顺序清晰的区段。
1.3 数据处理
以GenBank中的大口黑鲈mtDNA全序列(DQ536425)为参考,用 CLUSTAL 1.83软件[24]对测序结果进行同源序列比对分析,确定PCR扩增产物位于COⅠ区段内。用DNAsp 4.0软件[25]统计两个亚种的单倍型数以及单倍型多样性指数、核苷酸多样性指数、平均核苷酸差异性。用Mega 4.0软件[26]统计每个个体所得区段的碱基组成和碱基差异,基于Kimura 2-parameter计算两个亚种间的遗传距离,并构建NJ系统树。
2 结果与分析
2.1 大口黑鲈COⅠ区段的PCR扩增及测序
大口黑鲈两个亚种mtDNA COⅠ区段的PCR扩增产物经10 g/L琼脂糖凝胶电泳后,每个个体均得到约1400 bp的扩增片段 (图1)。PCR产物经华大基因公司电泳、片段回收并测序。
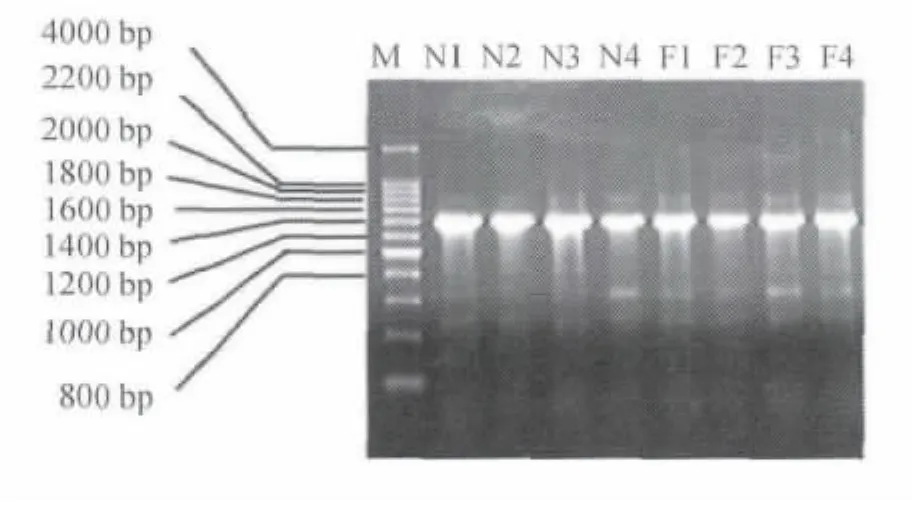
图1 大口黑鲈两个亚种COⅠ区段PCR产物的电泳图Fig.1 The electrophoresis of PCR product of COⅠregion in two subspecies of largemouth bass
2.2 测序结果分析
2.2.1 序列长度、单倍型和遗传多样性 用Contig Express Project软件对双向测序结果进行拼接,删除测序起始部分峰图杂乱的序列,获得碱基排列顺序清晰的1337 bp片段。与GenBank上的序列(DQ536425)比对后发现,获得的片段是mtDNA COⅠ基因的部分序列,位于COⅠ基因的第110个~第1446个碱基处。北方亚种21尾个体间未见差异,只有1种单倍型,命名为N1,其单倍型多样性指数 (H)、核苷酸多样性指数 (Pi)和平均核苷酸差异性 (k)均为0;佛罗里达亚种19尾个体中有5种单倍型,分别命名为F1、F2、F3、F4和F5,5种单倍型中分别包含6、6、2、1、4个个体,佛罗里达亚种的 H、Pi和 k分别为0.784、0.003 42、4.573。将两个亚种共6种单倍型的序列提交到GenBank,获得的序列号为KF176376~KF176381。
2.2.2 两个亚种的序列比较
(1)碱基组成与差异。用Mega 4.0软件计算大口黑鲈两个亚种测序得到COⅠ区段的碱基频率(表1)。两个亚种中A+T含量都高于G+C含量。碱基组成与鱼类、两栖类、爬行类、鸟类和哺乳类等动物的mtDNA蛋白质编码基因的特征相同[27-28]。
两个亚种鱼序列结构的差异性表现在亚种内和亚种间两方面,亚种间的差异性远大于亚种内(表2)。其中,北方亚种个体间未见差异,与GenBank中大口黑鲈的序列 (DQ536425)进行比对,有5个碱基出现置换;佛罗里达亚种个体间碱基变异较多,两两单倍型间出现1至8个碱基的差异,5个单倍型间共有11个碱基的差异。两个亚种不同个体间存在52个碱基的差异,其中两个亚种间完全不同的碱基有41个,可作为DNA-COⅠ条形码来鉴定两个亚种。

表1 大口黑鲈两个亚种COⅠ区段序列的碱基频率Tab.1 Nucleotide frequencies of COⅠregion in the two subspecies of largemouth bass %
(2)两个亚种的遗传距离与系统发育树。基于Kimura 2-parameter(K2-P)计算两亚种内和亚种间COⅠ区段序列的遗传距离 (表3)。结果表明,两个亚种间的遗传距离>佛罗里达亚种内遗传距离>北方亚种内遗传距离。
运用Mega 4.0软件,基于Kimura 2-parameter模型,采用1000次抽样重复检测,将两个亚种6种单倍型序列与GenBank中大口黑鲈序列(DQ536425)构建NJ系统树。从图2可以看出,GenBank中大口黑鲈序列与北方亚种 (N1)聚为一支,佛罗里达亚种 (F1~F5)聚为另一支。

表2 大口黑鲈两个亚种COⅠ区段序列变异位点Tab.2 The variation sites of COⅠregion in the two subspecies of largemouth bass

表3 大口黑鲈两个亚种COⅠ区段种内和种间的平均K2-P遗传距离Tab.3 The genetic distance of mtDNA COⅠregion in the two subspecies of largemouth bass based on Kimura 2-parameter model
3 讨论
近年来,鱼类mtDNA的研究逐步向序列分析的方向发展,序列遗传多样性分析及DNA条形码已成为 COⅠ区的研究重点[18-22,27-28]。

图2 基于Kimura 2-parameter模型构建的大口黑鲈两个亚种COⅠ序列的NJ树Fig.2 NJ tree of COⅠregion in the two subspecies of largemouth bass based on the Kimura 2-parameter model
Lutz-Carrillo等[29]运用微卫星DNA对大口黑鲈两个亚种进行了检测分析,认为佛罗里达亚种的遗传多样性高于北方亚种。李胜杰等[30]对线粒体DNA D-loop区段的序列分析结果表明,佛罗里达亚种的核苷酸多样性指数高于北方亚种。本研究中应用COⅠ序列分析,结果显示,21尾北方亚种个体间没有变异,19尾佛罗里达亚种不同个体间有变异存在,其H、Pi和k指数均大于北方亚种,表明佛罗里达亚种遗传多样性高于北方亚种。至于北方亚种的个体间没有变异,遗传多样性指标均为0的可能原因:一是大口黑鲈引进时基础群体数量较小,养殖过程中发生了近交和遗传漂变等现象[30];二是北方亚种物种本身的特性,即北方亚种是一个遗传多样性较低的类别,这与其他学者使用其他遗传标记检测的结果相似[29-31]。
大口黑鲈两个亚种在形态上十分相似,较难区分,虽然二者的侧线鳞数有所不同,北方亚种侧线鳞数为59~68,佛罗里达亚种的侧线鳞数为69~73,但由于测量时存在误差,很难有效地鉴别两个亚种[30,32]。Nedbal等[31]运用 RFLP 技术研究了两个亚种的线粒体DNA,8种酶切结果表明,两个亚种具有不同的单倍型。Williams等[33]运用 RAPD技术鉴定了两个亚种,筛选出3个引物,扩增出15个特异性标记,可有效地鉴别两个亚种。张大莉等[34]对两个亚种做了同工酶检测,发现两个亚种在IDH-1*和MDH-2*两个基因座位上的基因完全不同,可依此将两个亚种完全分开。Li等[35]分析了采自中国3个养殖场共14尾北方亚种 (每个养殖场取4~5尾)和采自美国的5尾野生北方亚种与5尾佛罗里达亚种,研究了COⅠ区807 bp序列的单核苷酸多态性,认为两个亚种的COⅠ有明显区别。Hebert等[14-15]对包括脊索动物在内的11个门类动物的2238个物种间的遗传差异进行比较后,得到种内遗传距离大多数在1%以下,很少超过2%。由此认为,3%的遗传距离可以作为种类鉴别的标准。本研究中,通过对两个亚种COⅠ区段1337 bp的检测分析,得到两亚种间的Kimura 2-parameter遗传距离为0.035 13,作者认为,依据COⅠ序列可有效地将大口黑鲈北方亚种与佛罗里达亚种分开。对大口黑鲈两个亚种COⅠ序列的检测分析,可为种类鉴别、遗传多样性分析和遗传育种等研究提供参考依据。
[1] 白俊杰.加州鲈鱼[C]//王清印,余来宁,杨宁生.中国水产生物种质资源与利用.北京:海洋出版社,2005:303-310.
[2] Bailey R M,Hubbs C L.The black basses(Micropterus)of Florida,with description of a new species[J].Occasional Papers of the Museum of Zoology(University of Michigan),1949,516:1 -40.
[3] Philipp D P.Stocking Florida largemouth bass outside its native range[J].Transactions of the American Fisheries Society,1992,121:686-691.
[4] 楼允东.我国鱼类引种研究的现状与对策[J].水产学报,2000,24(2):185 -192.
[5] 翟中和,王喜忠,丁明孝.细胞生物学[M].北京:高等教育出版社,2000:207.
[6] 肖武汉,张亚平.鱼类线粒体DNA的遗传与进化[J].水生生物学报,2000,24(4):384 -391.
[7] 杨明生,熊邦喜.动物mtDNA的研究及在鱼类生态学中的应用[J].孝感学院学报,2005,25(3):23 -27.
[8] 汪泰初,刘朝良,肖林珍.线粒体基因组(mtDNA)的研究进展[J].安徽农业科学,2006,34(10):2068 -2071.
[9] 潘宝平,卜文俊.线粒体基因组的遗传与进化研究进展[J].生物学通报,2005,40(8):1 -3.
[10] 邹习俊,韩雪,韩虎峰.鱼类mtDNA及其非编码区的研究概况[J].贵州畜牧兽医,2009,33(3):23 -25.
[11] Liu J X,Gao T X,Yokogawa K,et al.Differential population structuring and demographic history of two closely related fish species,Japanese sea bass(Lateolabrax japonicus)and spotted sea bass(Lateolabrax maculatus)in Northwestern Pacific[J].Molecular Phylogenetics and Evolution,2006,39:799 -811.
[12] 郝君,杨蔷,鲍迪,等.6种mtDNA D-loop及其邻近区段的序列比较分析[J].大连海洋大学学报,2013,28(2):160 -165.
[13] 徐钢春,魏广莲,李建林,等.基于线粒体DNA D-loop序列分析养殖刀鲚与湖鲚的遗传多样性[J].大连海洋大学学报,2012,27(5):448 -452.
[14] Hebert P D N,Ratnasingham S,deWaard J R.Barcoding animal life:cytochrome c oxidase subunitⅠdivergences among closely related species[J].Proceedings of the Royal Society B,2003,270:S96-S99.
[15] Hebert P D N,Cywinska A,Ball S L,et al.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society B,2003,270:313 -321.
[16] Perez- Enriquze R,Taniguchi N.Genetic structure of red sea bream(Pagrus major)population of Japan and the Southwest Pacific,using Microsatellite DNA Markers[J].Fisheries Science,1999,65(1):23 -30.
[17] Palumbi S R,Kessing B D.Population biology of the transarctic exchange:mtDNA sequence similarity between Pacific and Atlantic sea urchins[J].Evolution,1991,45:1790 -1805.
[18] Ward R D,Zemlak T S,Innes B H,et al.DNA barcoding Australia's fish species[J].Philosophical Transactions of the Royal Society B,2005,360:1847 -1857.
[19] 张龙岗,郭金峰,孟庆磊,等.澳洲宝石鲈线粒体DNA COⅠ基因的克隆与序列分析[J].生物技术通报,2011(4):116-119.
[20] 张凤英,马凌波,施兆鸿,等.3种鲳属鱼类线粒体COⅠ基因序列变异及系统进化[J].中国水产科学,2008,15(3):392-399.
[21] 柳淑芳,陈亮亮,戴芳群,等.基于线粒体COⅠ基因的DNA条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用[J].海洋与沼泽,2010,41(3):223 -232.
[22] 张凤英,马凌波,施兆鸿,等.两种鲷属鱼类线粒体COⅠ基因片段序列的比较[J].上海水产大学学报,2006,15(4):403-408.
[23] 翟中会,陈希南,王娟.利用Primer Premier 5.0进行引物设计[J].西北医学教育,2008,16(4):695 -698.
[24] Thompson J D,Gibson T J,Plewniak F,et al.The CLUSTAL_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25(24):4876 -4882.
[25] Rozas J,Sánchez - DelBarrio J C,Messeguer X,et al.DnaSP,DNA polymorphism analyses by the coalescent and other methods[J].Bioinformatics,2003,19(18):2496 -2497.
[26] Tamura K,Dudley J,Nei M,et al.MEGA4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Molecular Biology and Evolution,2007,24(8):1596 -1599.
[27] 彭居俐,王绪祯,王丁,等.基于线粒体COⅠ基因序列的DNA条形码在鲤科鲌属鱼类物种鉴定中的应用[J].水生生物学报,2009,33(2):271 -276.
[28] Broughton R E,Roe B A.The complete sequence of the zebrafish(Danio rerio)mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA[J].Genome Research,2001,11:1958-1967.
[29] Lutz- Carrillo D J,Nice C C,Bonner T H,et al.Admixture analysis of Florida largemouth bass and Northern largemouth bass using microsatellite loci[J].Transactions of the American Fisheries Society,2006,135:779 -791.
[30] 李胜杰,白俊杰,叶星,等.基于线粒体D-loop区探讨我国养殖大口黑鲈的分类地位和遗传变异[J].海洋渔业,2008,30(4):291-296.
[31] Nedbal M A,Philipp D P.Differentiation of mitochondrial DNA in largemouth bass[J].Transaction of the American Fisheries Society,1994,123:460 -468.
[32] 前田政善.オオクチバス[C]//川那部浩哉,水野信彦.日本の淡水鱼.东京:山と泮谷社,1989:494-502.
[33] Williams D J,Kazianis S,Walter R B.Use of random amplified polymorphic DNA(RAPD)for identification of largemouth bass subspecies and their intergrades[J].Transactions of the American Fisheries Society,1998,127:825 -832.
[34] 张大莉,杨蔷,郝君,等.大口黑鲈北方亚种和佛罗里达亚种的同工酶分析[J].天津农学院学报,2012,19(3):8 -12.
[35] Li S J,Bai J J,Cai L,et al.The complete mitochondrial genomes of largemouth bass of the northern subspecies(Micropterus salmoides salmoides)and Florida subspecies(Micropterus salmoides floridanus)and their applications[J].Mitochondrial DNA,2012,23(2):92-99.
张大莉1,董仕1,白俊杰2,苗建发3,胡莹1,李胜杰2,苗建新3,张兴华1,马冬梅2
(1.天津师范大学生命科学学院天津市动植物抗性重点实验室,天津300387;2.中国水产科学研究院珠江水产研究所,广州510380;3.天津市天祥水产有限责任公司,天津301500)
Sequence analysis of mtDNA COⅠregion in northern and Florida subspecies of largemouth bass Micropterus salmoides
ZHANG Da-li1,DONG Shi1,BAI Jun-jie2,MIAO Jian-fa3,HU Ying1,LI Sheng-jie2,
MIAO Jian-xin3,ZHANG Xing-hua1,MA Dong-mei2
(1.Tianjin Key Laboratory of Animal and Plant Resistance,College of Life Science,Tianjin Normal University,Tianjin 300387,China;2.Pearl River Fisheries Research Institute,Chinese Academy of Fishery Sciences,Guangzhou 510380,China;3.Tianjin Tianxiang Fishery Company Limited,Tianjin 301500,China)
The PCR technology was used to amplify the mtDNA COⅠregion of 21 individuals of northern subspecies and 19 individuals of Florida subspecies of largemouth bassMicropterus salmoides,and about 1400 bp DNA fragments were obtained,in which the sequence of 1337 bp fragment were obtained by sequencing.Comparing with COⅠ region of mtDNA complete sequence of largemouth bass(GenBank:DQ536425),there were five base substitutions and the haplotype detected was only one within northern subspecies.There were five haplotypes within Florida subspecies,and the 41 bases substitution was detected in all individuals of Florida subspecies compare with northern subspecies.The six haplotypes has GenBank serial numbers from KF176376 to KF176381 and the base composition and base differences were calculated by Mega 4.0 software,with haplotype diversity index(H)of 0,nucleotide diversity index(Pi)of 0 and average nucleotide diversity(k)of 0 in northern subspecies.Florida subspecies has H of 0.784,Pi of 0.003 42 and k of 4.573.The two subspecies have average Kimura 2 - parameter genetic distance of 0.035 1.There was higher genetic diversity in mtDNA COⅠ region in Florida subspecies than in northern subspecies,indicating that the sequence of mtDNA COⅠ region can be used as a DNA barcode for identification of the two subspecies of largemouth bass.
Micropterus salmoides;COⅠregion;genetic diversity;genetic distance;NJ phylogenetic tree
S965.199;Q953
A
10.3969/J.ISSN.2095 -1388.2014.03.002
2095 -1388(2014)03 -0212 -05
2013-11-28
天津市农业科技成果转化与推广项目 (201104120);科技部农业科技成果转化基金资助项目 (2012GB23260562)
张大莉 (1988—),女,硕士研究生。E-mail:913801465@qq.com
胡莹 (1963—),女,副教授。E-mail:huying265@126.com
