高GC含量DNA模板的PCR扩增
2013-10-29邢玉华戴素琴刘体颜王微谭俊杰曲国龙刘刚陈惠鹏
邢玉华 ,戴素琴 ,刘体颜 ,王微 ,谭俊杰 ,曲国龙 ,刘刚 ,陈惠鹏
1.军事医学科学院 生物工程研究所,北京 100071;2.吉林大学 生命科学学院,吉林 长春 130012;3.安徽大学 生命科学学院,安徽 合肥 230039
链霉菌属于放线菌,是革兰阳性菌,其基因组中GC含量很高(>70%)。链霉菌能够产生多种活性物质,尤其是能够产生丰富的抗生素,目前75%的天然抗生素都来自链霉菌[1]。达托霉素(daptomycin)是一种新型环脂肽类抗生素,2003年9月经美国食品和药物管理局(FDA)批准在美国上市。达托霉素具有在体外抗绝大多数临床革兰阳性菌的作用,主要用于耐药菌的感染治疗[2]。达托霉素的生物合成经非核糖体肽合成酶(NRPS)途径,其合成基因成簇排列在染色体上,共同调控达托霉素的合成[3]。因此,对达托霉素合成基因的克隆与分析,对了解达托霉素的调控机制,提高达托霉素的产量有重要意义。
达托霉素的生物合成基因簇总长128 kb,其平均GC含量为71%,我们曾尝试用常规PCR方法分段扩增,但很难将其扩增出来。由于许多非编码区的序列是高度保守的,密码子优化的方法也受到限制。因此,我们选择10个1 kb的片段进行扩增,尝试通过对PCR反应条件进行优化,包括加入不同的PCR添加剂[4-8]及调整PCR循环程序[9-10],从而实现对高GC含量的达托霉素生物合成基因的扩增,同时也实现了对长达6 kb的高GC含量DNA的扩增。
1 材料与方法
1.1 材料
玫瑰孢链霉菌(Streptomyces roseosporus)31568菌株购自美国ATCC;大肠杆菌DH5α、DH10B化学感受态购自北京博迈德公司;pMD18-T载体和LA Taq聚合酶购自TaKaRa公司;Q5高保真DNA聚合酶、7-deaza-dGTP、dNTP、各种限制性内切酶均购自NEB公司;质粒小提试剂盒、DNA凝胶回收试剂盒均购自Axygen公司;DyNAzymeⅡDNA聚合酶购自Thermo公司;链霉菌基因组提取试剂盒购自GENMED公司;DMSO及其他化学试剂均购自Sigma公司。
1.2 引物设计
我们将达托霉素生物合成基因簇分成128个平均长度为1080 bp的片段,分别进行扩增,序列之间带有重叠序列,方便后续拼接。用Primer Premier 5.0软件设计引物,两端带有XbaⅠ和HindⅢ酶切位点,由北京奥科鼎盛公司合成,序列见表1。
1.3 模板的制备
用GENMED公司的链霉菌属基因组DNA提取试剂盒提取玫瑰孢链霉菌基因组DNA,DNA的浓度和质量经NanoDrop 2000分光光度计测定。
1.4 PCR扩增体系
LA Taq聚合酶反应体系(共50 μL)包括25 μL 2×GC缓冲液Ⅰ、400 μmol/L dNTP、引物各0.5 μmol/L、100 ng基因组DNA、2.5 U LA Taq聚合酶。
Q5高保真聚合酶反应体系(共50 μL)包括10 μL 5×Q5反应缓冲液、10 μL 5×Q5 High GC En⁃hancer、200 μmol/L dNTP、引 物 各 0.5 μmol/L、50 ng基因组DNA、1 U Q5聚合酶。反应体系中分别加入不同浓度的DMSO(1%~4%)、7-Deaza-dGTP∶dGTP(1∶1或 3∶1)。
1.5 PCR扩增程序
LA Taq聚合酶的扩增程序:94℃预变性5 min;94℃变性30 s,固定温度(55~68℃)退火30 s,72℃延伸1 min,30个循环;72℃延伸5 min。
Q5聚合酶的扩增程序:98℃预变性30 s;98℃变性10 s,固定温度(55~68℃)退火30 s,72℃延伸30 s,30个循环;72℃延伸2 min。
Touch down PCR循环程序:98℃预变性30 s;98℃变性 10 s,71℃退火 30 s,72℃延伸 30 s,退火温度每个循环降低0.2℃,30个循环;之后98℃变性10 s,65℃退火 30 s,72℃延伸 30 s,15个循环;72℃延伸2 min。
1.6 PCR产物分析及测序
取 20 μL PCR 产物,加入 4 μL 6×上样缓冲液,行1%琼脂糖凝胶电泳,EB染色,用凝胶成像系统分析拍照,目的条带用凝胶回收试剂盒回收。Q5高保真聚合酶扩增的DNA需用DyNAzymeⅡDNA聚合酶加A处理,LA Taq聚合酶扩增的片段则直接与pMD18T载体连接,转化大肠杆菌DH5α感受态细胞,次日挑克隆,酶切鉴定挑选阳性克隆测序(测序由北京奥科鼎盛公司进行),比对测序结果,进行保真度分析。

表1 引物及序列
2 结果
2.1 达托霉素生物合成基因簇DNA片段的LA PCR扩增
以玫瑰孢链霉菌基因组DNA为模板,用LA Taq DNA聚合酶使用GC缓冲液进行扩增,能扩增出2个片段(401和678),其他均没有目的条带。对401和678进行测序,发现有大量突变和错配。
2.2 达托霉素生物合成基因簇DNA片段PCR扩增条件的优化
鉴于后续的克隆表达,我们选用了高保真的Q5 DNA聚合酶进行扩增。首先摸索了不同退火温度对PCR扩增的影响,发现较高的温度能极大地提高扩增量和产物特异性(图1)。但单独提高退火温度对于某些DNA(如067和1245)的扩增是无效的,因此,针对这些片段还须进行其他条件优化。
有文献报道,在反应体系中加入增强剂可以提高反应的特异性和产量。常用的增强剂有甘油、DMSO、甲酰胺、7-deaza-dGTP和镁离子等,对减少高GC区域的二级结构等都有一定的功效。我们主要探讨了不同浓度的DMSO和7-deaza-dGTP对扩增效率的影响。由于我们使用的商品化聚合酶试剂盒中的GC增强剂成分不明,按照说明书尝试加入1%~4%的DMSO,结果表明DMSO能显著地提高1245的扩增量(图2),但多次结果也表明DMSO对扩增的特异性没有太大帮助,甚至有时会降低产物的特异性。同时,我们探讨了7-deaza-dGTP对扩增的影响。7-deaza-dGTP[11-12]是dGTP的类似物,能够减少二级结构形成。我们在反应体系中分别加入100 μmol/L 7-deaza-dGTP和100 μmol/L dGTP,及150 μmol/L 7-deaza-dGTP和50 μmol/L dGTP。对于片段067,在加入7-deaza-dGTP后,在较高的退火温度下能够看到有目的条带扩增出来,但仍然很弱(图3A)。因此,我们选用touch down PCR进行扩增,结果表明能将067扩增出来。用touch down PCR并同时添加7-deaza-dGTP进行扩增,发现目的条带的扩增量没有增加反而降低了,但产物特异性有很大提高(图3B)。测序表明加入7-deaza-dGTP后扩增的突变及错配碱基数减少,而且原本测序经常信号中断的克隆也大大得到改善(图4),可见7-deaza-dGTP能够提高产物的特异性及确保测序反应的顺利进行。
2.3 达托霉素生物合成基因簇DNA片段的PCR扩增
我们曾尝试将DMSO与7-deaza-dGTP组合添加到PCR反应体系中,结果发现扩增效果没有改变,说明DMSO与7-deaza-dGTP在这种体系中并不存在协同作用,或者可能会相互抑制,其原因目前还不清楚。因此,考虑到后续拼接、克隆及表达,我们选用touch down PCR及添加7-deaza-dGTP作为我们克隆达托霉素生物合成基因簇的常规方法。应用这种方法,我们将选择的10个DNA片段全部扩增出来,继而扩增了所有的达托霉素合成基因。电泳图中虽然还存在一些非特异性条带,但每个片段的扩增量均能达到克隆的要求,无须重新设计引物扩增。图5为PCR产物纯化后的电泳结果。
2.4 PCR扩增达托霉素生物合成基因簇长片段DNA
利用touch down PCR及添加7-deaza-dGTP的方法,我们还进一步对不同长度的达托霉素生物合成基因簇DNA片段进行了扩增,1~6 kb均有目的条带出现(图6)。为了验证长片段高GC含量DNA的PCR产物的保真度,连接到pMD18-T载体上进行测序,结果表明直到6 kb的序列全都正确,说明利用这种方法进行长片段高GC含量DNA的PCR扩增是可行的。利用以上条件我们尝试扩增更长的片段,结果7、8 kb片段的扩增没有成功,但也足以满足拼接所需。
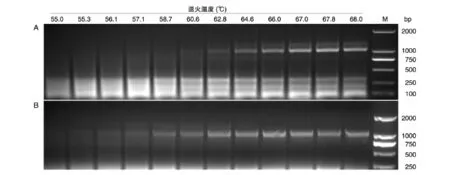
图1 不同退火温度对高GC含量DNA PCR的影响
3 讨论
达托霉素的生物合成基因簇DNA平均GC含量很高,经常出现多个连续的G或C,而且还含有重复序列。笔者在前期用常规PCR方法很难(甚至不可能)扩增出基因。高GC含量DNA的PCR扩增的难点在于模板DNA形成的二级结构,使得DNA模板比较难变性,94℃下可能变性不完全;退火时引物与模板不能很好地结合,容易形成错配;延伸过程也不能很好地进行,从而使扩增效率大大降低。PCR添加剂降低熔解温度,有助于引物退火,并辅助DNA聚合酶延伸通过二级结构区。本研究结果表明DMSO和7-deaza-dGTP对于提高PCR扩增效率有很大帮助,而且采用touch down PCR的方法,先高温下退火再缓慢降温,能极大地提高PCR产物扩增的特异性。我们向反应体系中加入1%~4%的DMSO,对大多数的DNA片段均能提高其扩增产量,而且以4%的DMSO效果最佳。但同时我们还发现,虽然扩增量提高,但反应的特异性降低,出现了很多非特异性条带,可能是降低了熔解温度所致。我们继续加大DMSO的量,发现几乎扩增不出任何条带,经分析认为可能是DMSO抑制了聚合酶的活性。文献报道7-deaza-dGTP会增加PCR扩增的产量和特异性[13],但我们的研究表明7-deaza-dGTP的加入不会增加产物的扩增量,反而会降低,原因还有待于考察。然而7-deaza-dGTP确实可以提高产物的特异性和保真度,而且使测序反应更容易进行。
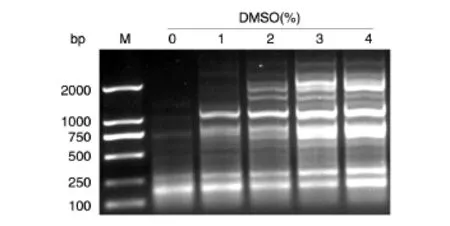
图2 DMSO对PCR扩增高GC含量DNA模板(1245)的影响
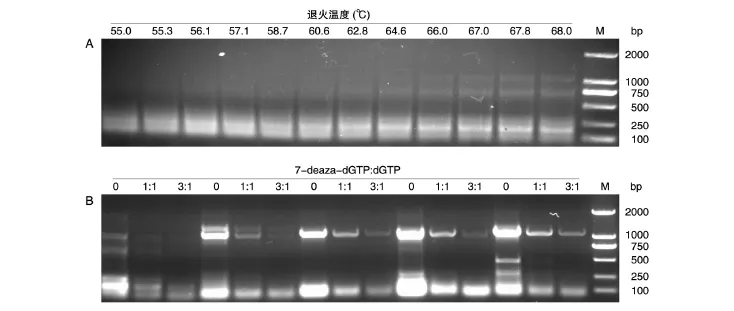
图3 7-deaza-dGTP对PCR扩增高GC含量DNA模板(067)的影响
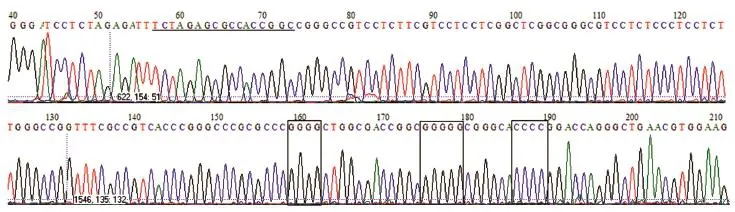
图4 片段1245测序的部分结果
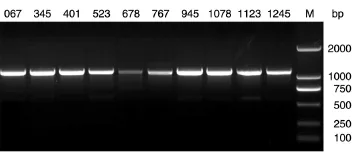
图5 达托霉素生物合成基因簇部分片段的PCR扩增结果
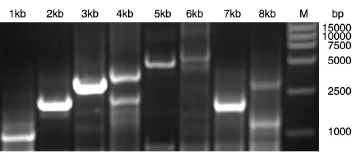
图6 PCR扩增不同长度高GC含量DNA的结果
以上结果表明我们建立了采用touch down PCR并添加7-deaza-dGTP或DMSO进行高GC含量DNA扩增的程序。基于此方法,我们成功地将达托霉素的生物合成基因簇(128 kb)分段扩增出来,经内切酶消化后可用于后续拼接。此外,我们还尝试用该方法进行长片段DNA的扩增,实现了长达6 kb的高GC含量DNA的扩增,但对于更长片段的扩增(>8 kb)没有成功,还须进一步摸索,比如探讨反应过程中添加聚合酶等方法。综上,我们建立了扩增长片段高GC含量DNA的方法,对研究达托霉素的作用机制以及提高其产量将有很大的帮助。
[1]Newman D J.Natural products as leads to potential drugs:an old process or the new hope for drug discovery[J].J Med Chem,2008,51(9):2589-2599.
[2]Barry A L,Fuchs P C,Brown S D.In vitro activities of dap⁃tomycin against 2,789 clinical isolates from 11 North Ameri⁃can medical centers[J].Antimicrob Agents Chemother,2001,45(6):1919-1922.
[3]Miao V,Coëffet-Le Gal M F,Brian P,et al.Cloning,se⁃quence analysis and heterologous expression of the daptomy⁃cin biosynthetic gene cluster of Streptomyces roseosporus[J].Microbiology,2005,151(5):1507-1523.
[4]Mamedov T G,Pienaar E,Whitney S E,et al.A fundamen⁃tal study of the PCR ampli fi cation of GC-rich DNA templates[J].Comput Biol Chem,2008,32(6):452-457.
[5]Sahdev S,Saini S,Tiwari P,et al.Amplifcation of GC-rich genes by following a combination strategy of primer design,enhancersand modifed PCR cycle conditions[J].MolCell Probes,2007,21(4):303-307.
[6]Zhang Z,Yang X,Meng L,et al.Enhanced amplifcation of GC-rich DNA with two organic reagents[J].Biotechniques,2009,47(3):775-779.
[7]Musso M,Bocciardi R,Parodi S,et al.Betaine,dimethyl sulf⁃oxide,and 7-deaza-dGTP,a powerful mixture for amplifca⁃tion of GC-rich DNA sequences[J].J Mol Diagn,2006,8(5):544-550.
[8]Jensen M A,Fukushima M,Davis R W.DMSO and betaine greatly improve amplification of GC-rich constructs in de no⁃vo synthesis[J].PLoS ONE,2010,5(6):e11024.
[9]Frey U H,Bachmann H S,Peters J,et al.PCR-amplifcation of GC-rich regions:slowdown PCR[J].Nat Protoc,2008,3(8):1312-1317.
[10]Wei M,Deng J,Feng K,et al.Universal method facilitating the ampli fi cation of extremely GC-rich DNA fragments from genomic DNA[J].Anal Chem,2010,82(14):6303-6307.
[11]McConlogue L,Brow M A,Innis M A.Structure-independent DNA amplifcation by PCR using 7-deaza-2'-deoxyguanosine[J].Nucleic Acids Res,1988,16(20):9869.
[12]Shore S,Pau N.Robust PCR amplifcation of GC-rich targets with hotstart7-deaza-dGTP[J].BioTechniques,2010,49(5):841-843.
[13]Sahdev S,Saini S,Tiwari P,et al.Amplification of GC-rich genes by following a combination strategy of primer design,enhancersand modified PCR cycle conditions[J].MolCell Probes,2007,21(4):303-307.
