野生型及突变型HIV-1LTR驱动的荧光素酶稳定表达细胞株的建立
2013-04-08杨少宗林师冠王晓辉孔垂瑾曲喜英朱焕章
刘 林,杨少宗,林师冠,王晓辉,孔垂瑾,曲喜英,朱焕章
(1.复旦大学 生命科学学院 遗传工程国家重点实验室,上海 200433;(2.河南 洛阳职业技术学院,洛阳 471001)
获得性免疫缺陷综合征(Acquired Immuno Deficiency Syndrome,AIDS)病人体内HIV-1潜伏感染细胞的存在,是造成病人在高效抗逆转录病毒疗法(Highly Active Antiretroviral Therapy,HAART)治疗中止后外周血中病毒拷贝数重新反弹的主要原因[1,2].在这个病毒储藏库中,HIV-1整合在宿主细胞基因组中但病毒基因并不表达,使得该细胞与正常细胞并无区别从而逃避免疫系统的清除作用[3].当这些潜伏细胞受到细胞因子等刺激而活化后,病毒基因就能得以激活并产生新的病毒,从而使血液中病毒拷贝数反弹[4].
目前一种新型的“激活 清除”治疗策略,就试图通过特定的激活剂来激活潜伏病毒,并辅以HAART 治疗来阻止新产生病毒的传播,最终利用免疫系统的作用来清除潜伏感染细胞[5,6].由于在感染HIV-1的病人体内,每106个静止CD4+T细胞中才有一个整合有复制完全型的HIV-1,这意味着一个病人体内的潜伏感染的细胞数不会超过107[7].因此,建立有效的用于药物筛选的模型,是成功筛选高效、低毒性HIV潜伏激活剂的一个关键.
HIV-1基因表达极其依赖于宿主细胞内的转录因子[8].造成HIV-1潜伏感染的一个重要分子机制就是转录激活相关转录因子的缺乏[9].在HIV-1潜伏感染的建立和再激活中,NF-κB和Tat扮演了至关重要的角色[4,8].静止记忆CD4+T淋巴细胞核中缺乏活化的NF-κB,是这些淋巴细胞中促使HIV-1原病毒潜伏的关键因素[10].Tat蛋白是HIV-1基因正常转录所必需的,缺少了Tat的作用会导致病毒基因不能够正常转录表达而促使病毒在宿主细胞内潜伏[8].在这过程中,NF-κB和Tat通过分别结合到HIV-1 LTR中κB和TAR元件上发挥作用.因此,本文旨在构建野生型及κB或TAR元件突变型HIV-1LTR驱动的荧光素酶表达载体,并将其转染人肾表皮细胞293,通过G418筛选以获得荧光素酶稳定表达细胞克隆.由于TNF-α是一种有效的激活HIV-1表达的激活剂,它能够特异性激活下游NF-κB信号通路[9],可以用来研究这些细胞模型对于TNF-α的反应是否符合预期.我们进一步用激活剂TNF-α对该体外系统进行处理,以初步探讨模型在筛选HIV-1潜伏激活药物及信号通路机制研究方面的作用.
1 材料与方法
1.1 材料
pHIV LTR-Luc、pHIV LTRΔκB-Luc和pHIV LTRΔTAR-Luc由Warner C.Greene实验室提供.
大肠杆菌DH5α、pcDNA3.0质粒均为本实验室保存;各种限制性内切酶,以及T4DNA连接酶均购自TaKaRa公司.PCR引物、DNA序列测定均由上海英骏生物技术有限公司完成.
1.2 方法
1.2.1 质粒构建
以pHIV LTR-Luc报告质粒为模板,用Primer Premier 5软件进行引物设计,PCR扩增HIV LTRLuc片段.引物由上海英骏公司合成,序列如下:LTRNdeI-5'-GGTACATATGTGGAAGGGCTAATT TGGTC-3'和LUCXhoI-5'-AATACTCGAGATTCGTTAAAATAGTACAGTGACCC-3'.PCR反应按以下参数进行:94℃3min;(94℃30s,56℃30s,72℃150s)×32;70℃10min.胶回收PCR产物,用NdeⅠ和XhoⅠ分别酶切PCR扩增产物和pcDNA3.0载体.回收DNA片段,并用T4DNA连接酶16℃连接过夜.连接产物用氯化钙法转化大肠杆菌DH5α,在氨苄青霉素(终浓度50μg/mL)的LB平板中培养过夜后,挑选菌落抽提质粒进行酶切和测序鉴定.
1.2.2 转染及稳定细胞株的筛选
24孔板中每孔接种5×105个293细胞,待培养板中细胞覆盖率在95%以上,用Lipofectamin 2000转染试剂按照2μL∶0.8μg比例转染pcDNA3.0-HIV LTR-Luc及其突变型质粒.细胞在含800μg/mL新霉素(G418)培养液培养14d后,将细胞消化后按梯度稀释种在96孔板中.待能够看到明显克隆后,寻找单个细胞克隆用胰酶消化扩大培养,并提取基因组DNA进行PCR扩增鉴定.
1.2.3 荧光素酶活性检测
稳定细胞克隆在高糖的DMEM(Gibco产品)细胞培养液中(含10%胎牛血清,100U/mL青霉素,100g/mL链霉素),37℃、5%CO2细胞培养箱中培养.当24孔细胞培养板中细胞覆盖率达到95%以上,加入终浓度为10ng/mL TNF-α[11]或10μmol/L PMA(Phorbol-12-myristate-13-acetate,PMA)的培养液培养.培养18h让药物充分作用后,弃去上清,用PBS清洗细胞.然后按照荧光素酶检测试剂盒(Dual Luciferase Assay System,Promega)说明书步骤操作:弃去培养基,用500μL 1×PBS清洗细胞;将PBS完全弃去后,每孔加入100μL的细胞裂解液,在摇床上裂解15min.将上述裂解细胞及上清收集到1.5mL的EP管内,12000r/min离心1min.保留上清进行后续检测.样品放冰上,用冷光仪(Lumat LB 9507)检测细胞内荧光素酶的表达水平.同时设立不加药组、加药组和没有转染质粒的人胚胎肾细胞293细胞作为空白组.仪器参数设置见Lumat LB 9507操作手册.加药组与未加药组荧光素酶检测值的比值可以用来表示药物对于该体外系统中细胞中LTR的激活效果.每个实验重复3次以获得平均值.
2 结 果
2.1 HIV-1LTR驱动的荧光素酶表达质粒的构建和稳定细胞株的筛选
以pHIV LTR-Luc为模板进行PCR,扩增出HIV LTR-Luc片段.然后通过Nde I和XhoI酶切,连接到pcDNA3.0质粒的相应位置,获得pcDNA3.0-HIV LTR-Luc表达质粒.LTR上κB和TAR元件突变的突变型质粒构建方法也与之类似,区别之处在于PCR的模板分别为pHIV LTRΔκB-Luc和pHIV LTRΔTAR-Luc.
将质粒分别转染293细胞,经G418筛选获得稳定细胞株293-HIV LTR-Luc、293-HIV LTRΔκB-Luc和293-HIV LTRΔTAR-Luc.然后,分别提取基因组DNA进行PCR扩增鉴定,扩增结果琼脂糖凝胶电泳进行检测.如图1所示,PCR得到略小于2500bp的特异性条带,与预期2300bp片段大小相符.PCR扩增片段回收送样测序,测序结果用MegaAlign软件进行比对.比对结果显示,293-HIV LTRΔκB-Luc中LTR上350nt位置的2个κB串联序列被缺失突变,而293-HIV LTRΔTAR-Luc则在490nt附近有4bp的缺失(见图1中下划线标注位置).这些突变位点与野生型LTR中顺式元件所在位置相符.
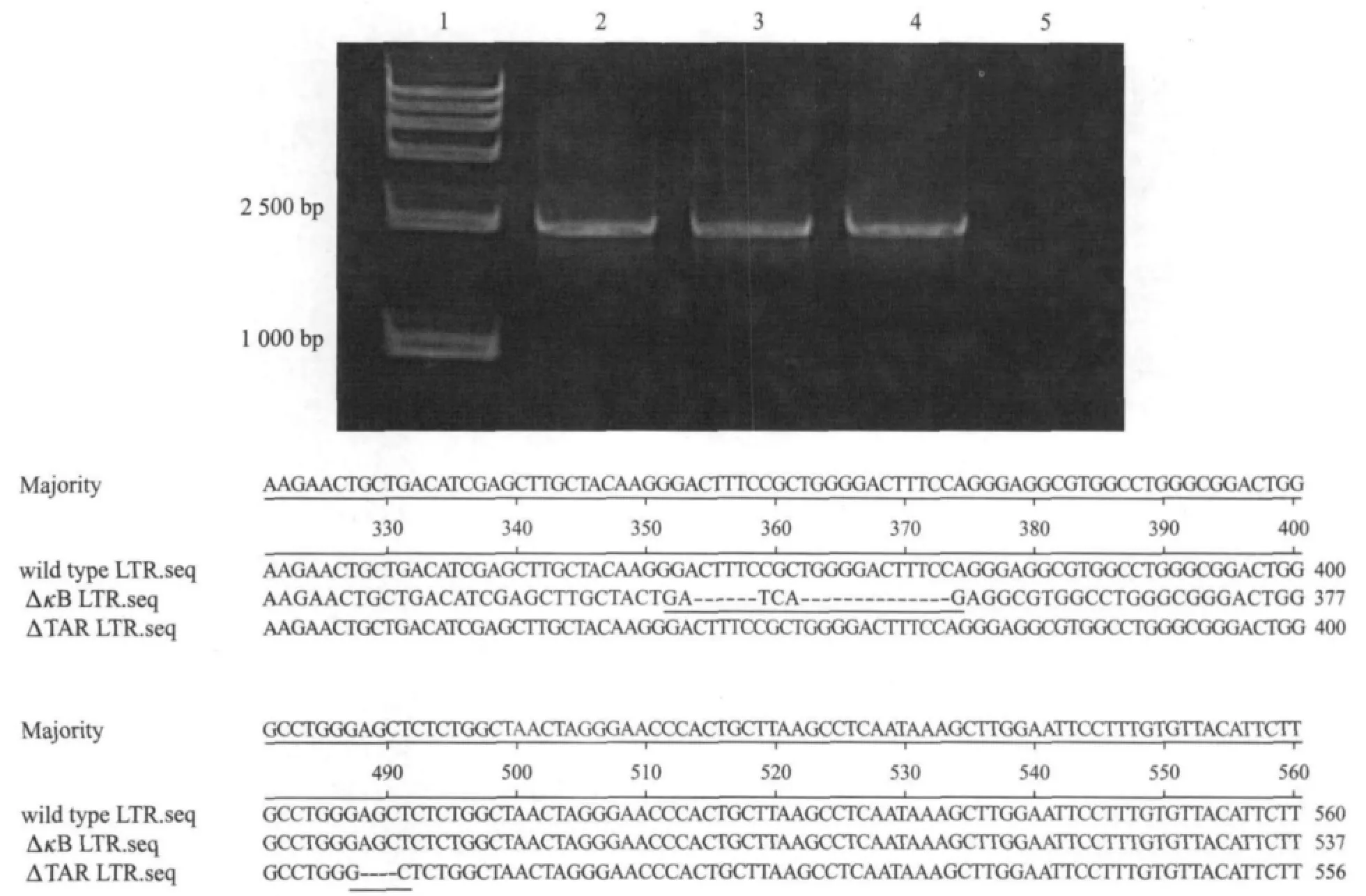
图1 细胞株基因组DNA的PCR检测及序列比对结果Fig.1 The results of DNA PCR and sequence alignment
2.2 TNF-α对HIV-1LTR驱动的荧光素酶稳定表达细胞模型的激活效果
为了验证这些细胞模型能够通过荧光素酶表达反映HIV-1LTR的活性,我们采用10ng/mL的TNF-α[11]对这些细胞模型进行处理冷光仪可以检测荧光素酶表达水平,来反映TNF-α对于不同细胞模型的激活效果我们发现,对于野生型LTR的细胞模型来说,10ng/mL的TNF-α激活18h后荧光素酶表达水平是激活前的2.87±0.09倍(与对照相比,P=0.017),PMA处理为激活前的1.7±0.11倍;而同样条件下,缺失了κB位点的细胞株无明显激活效果(TNF-α为0.96±0.07倍,PMA为1.00±0.13,P>0.05,见图2)对于TAR位点突变的细胞株,TNF-α对荧光素酶表达水平的激活效果与野生型LTR细胞株相比较弱(1.51±0.01倍,P<0.05),PMA处理为1.3±0.12倍.
2.3 TNF-α对于同一细胞模型中不同细胞克隆的激活结果

图2 TNF-α及PMA对野生型及突变型LTR细胞模型的激活效果Fig.2 The effects of TNF-αand PMA in wild-type and mutant LTR cell model
为了研究同一细胞模型中不同细胞克隆之间LTR对于荧光素酶的调控是否有较大区别,我们分别对该体外系统中每个模型选择了3个不同的细胞克隆,同样用10ng/mL TNF-α处理18h.结果显示TNF-α对同一细胞模型不同细胞克隆的荧光素酶表达水平的激活有一定的差异,但是这种差异并没有统计学上的意义(P>0.05,见图3).

图3 TNF-α对同一细胞模型中不同细胞克隆的激活效果Fig.3 The effects of TNF-αin different cell clone of the same model
3 讨 论
HIV-1潜伏感染是造成抗病毒疗法无法根除病人体内病毒的主要原因.因此,如何清除HIV-1潜伏感染细胞是目前艾滋病治疗的一个热门方向[5].而由于病人体内潜伏感染细胞极其稀少,每个病人体内最多也只有107个潜伏感染细胞[7],这迫切需要我们建立一个有效的药物筛选模型来辅助药物筛选和进行机制研究.
HIV-1LTR作为病毒基因转录重要的调控区,上面包含许多与转录调控因子结合的顺式元件[12].这些顺式元件与相应的转录因子信号相互作用形成了一个复杂的调控网络.在HIV-1潜伏感染的建立和维持过程中,NF-κB和Tat参与了至关重要的角色[4,8].NF-κB一般情况下位于细胞质中,与其抑制因子I-κB结合;在细胞信号传导进来时,NF-κB进入细胞核结合到HIV-1LTR上的两个串联的κB增强子序列[13].继而通过直接募集组蛋白乙酰转移酶(HATs)到HIV-1LTR上,触发原病毒的激活[14].Tat是由病毒基因组编码的一种转录调控蛋白.研究表明,HIV-1转录的时候,TAR先转录形成一小段有着茎环结构的RNA.Tat可以结合到由TAR转录形成的RNA,募集转录复合体的一些重要组分,从而保证HIV-1转录延伸的顺利进行[15].在缺失Tat的条件下,转录起始正常但是只产生一小段无效的转录本.Tat表达水平作为转录活化和潜伏的开关[16],当Tat在其他启动子驱动下反式表达时,HIV-1原病毒持续性活化并且不能进入潜伏[16].
突变型HIV-1LTR可以帮助我们有效地进行药物筛选和信号通路机制研究.Yang等就用缺失κB位点的NL4-3LTR驱动的LUC报告质粒建立瞬时转染细胞模型,筛选到细胞内转录因子Ets的一个突变型ΔVII-Ets-1能够有效地激活静止CD4+T细胞中的HIV-1表达[17].Williams等也通过ΔκB LTR的LUC报告质粒,来研究Prostratin激活潜伏HIV-1表达的信号通路[10].与这些瞬时转染系统相比,我们建立的稳定表达系统具备许多优点.由于瞬时转染细胞中质粒DNA没有整合进基因组DNA中,会在细胞分裂的过程中被逐渐稀释.这使得瞬时模型不能传代,每次实验前都需重新转染;而所用转染试剂的不同,所转染的质粒DNA的量,以及细胞状态都会影响后续实验结果.而在稳定表达细胞株中,质粒DNA整合进基因组,随着基因组进行复制,因此可以传代使用,也避免了不同实验者以及不同批次转染实验中所产生的误差,结果重复性高.为了验证模型的有效性,我们用TNF-α对细胞模型进行激活,对比不同细胞模型和同一细胞模型中不同细胞克隆中荧光素酶报告基因的表达水平,能够很好地说明我们建立的这套体外系统能够很好地反映不同药物对于HIV-1LTR的激活效果.
由于我们构建的细胞模型与用野生型病毒[18,19]或者慢病毒[20]感染T淋巴细胞后筛选出来的潜伏感染细胞有所不同,细胞中整合的病毒基因组并非处于潜伏感染状态.因此,进一步证明所筛选出来的药物能够激活潜伏感染病毒,还需在专门的潜伏感染细胞中进行验证.此外,我们构建的模型中HIV-1LTR上与潜伏感染相关的转录调控顺式元件得到了突变,能够进一步用来筛选通过特定信号通路来激活HIV-1表达的药物.我们期望这套体外系统能够为HIV-1激活剂的筛选工作提供便利.
[1]Davey R T,Bhat N,Yoder C,et al.HIV-1and T cell dynamics after interruption of highly active antiretroviral therapy(HAART)in patients with a history of sustained viral suppression[J].Proc Natl Acad Sci USA,1999,96(26):15109-15114.
[2]Persaud D,Zhou Y,Siliciano J M,et al.Latency in human immunodeficiency virus type 1infection:No easy answers[J].Journal of Virology,2003,77(3):1659-1665.
[3]Han Y F,Wind-Rotolo M,Yang H C,et al.Experimental approaches to the study of HIV-1latency[J].Nature Reviews Microbiology,2007,5:95-106.
[4]Williams S A,Greene W C.Regulation of HIV-1latency by T-cell activation[J].Cytokine,2007,39(1):63-74.
[5]Richman D D,Margolis D M,Delaney M,et al.The challenge of finding a cure for HIV infection[J].Science,2009,323(5919):1304-1307.
[6]Colin L,Van Lint C.Molecular control of HIV-1postintegration latency:implications for the development of new therapeutic strategies[J].Retrovirology,2009,6:111.
[7]Chun T W,Carruth L,Finzi D,et al.Quantification of latent tissue reservoirs and total body viral load in HIV-1Infection[J].Nature,1997,387(6629):183-188.
[8]Coiras M,Lopez-Huertas M R,Perez-Olmeda M,et al.Understanding HIV-1latency provides clues for the eradication of long-term reservoirs[J].Nature Reviews Microbiology,2009,7(11):798-812.
[9]Quivy V,De Walque S,Van Lint C.Chromatin-associated regulation of HIV-1transcription:implications for the development of therapeutic strategies[J].Subcell Biochem,2007,41:371-96.
[10]Williams S A,Chen L F,Kwon H,et al.Prostratin antagonizes HIV latency by activating NF-kappaB[J].J Biol Chem,2004,279(40):42008-17.
[11]Quivy V,Adam E,Collette Y,et al.Synergistic activation of human immunodeficiency virus type 1 promoter activity by NF-kappa B and inhibitors of deacetylases:potential perspectives for the development of therapeutic strategies[J].Journal of Virology,2002,76(21):11091-11103.
[12]Pereira L A,Bentley K,Peeters A,et al.A compilation of cellular transcription factor interactions with the HIV-1LTR promoter[J].Nucleic Acids Res,2000,28(3):663-8.
[13]Nabel G,Baltimore D.An inducible transcription factor activates expression of human immunodeficiency virus in T cells[J].Nature,1987,326(6114):711-3.
[14]Lusic M,Marcello A,Cereseto A,et al.Regulation of HIV-1gene expression by histone acetylation and factor recruitment at the LTR promoter[J].EMBO J,2003,22(24):6550-61.
[15]Karn J.The molecular biology of HIV latency:breaking and restoring the Tat-dependent transcriptional circuit[J].Curr Opin HIV AIDS,2011,6(1):4-11.
[16]Pearson R,Kim Y K,Hokello J,et al.Epigenetic silencing of human immunodeficiency virus(HIV)transcription by formation of restrictive chromatin structures at the viral long terminal repeat drives the progressive entry of HIV into latency[J].J Virol,2008,82(24):12291-303.
[17]Yang H C,Shen L,Siliciano R F,et al.Isolation of a cellular factor that can reactivate latent HIV-1 without T cell activation[J].Proc Natl Acad Sci USA,2009,106(15):6321-6326.
[18]Folks T M,Clouse K A,Justement J,et al.Tumor necrosis factor-alpha induces expression of human immunodeficiency virus in a chronically infected T-cell clone[J].Proc Natl Acad Sci USA,1989,86(7):2365-2368.
[19]Folks T M,Justement J,Kinter A,et al.Cytokine-induced expression of HIV-1in a chronically infected promonocyte cell-line[J].Science,1987,238(4828):800-802.
[20]Jordan A,Bisgrove D,Verdin E.HIV reproducibly establishes a latent infection after acute infection of T cells in vitro[J].EMBO Journal,2003,22(8):1868-1877.
