正交设计优化草地早熟禾SRAP-PCR反应体系及引物筛选
2012-09-18任小巍王瑜袁庆华
任小巍,王瑜,袁庆华
(1.兰州大学草地农业科技学院,甘肃兰州730020;2.中国农业科学院北京畜牧兽医研究所,北京100193)
正交设计优化草地早熟禾SRAP-PCR反应体系及引物筛选
任小巍1,2,王瑜2,袁庆华2
(1.兰州大学草地农业科技学院,甘肃兰州730020;2.中国农业科学院北京畜牧兽医研究所,北京100193)
采用L9(34)正交试验设计方法,对草地早熟禾(Poapratensis)基因组DNA SRAP-PCR反应体系中的Taq DNA聚合酶、Mg2+、引物及dNTP四因素的用量进行优化,并比较不同模板DNA用量对扩增的影响,建立草地早熟禾SRAP-PCR最佳反应体系,同时,利用该体系对SRAP引物进行筛选。结果表明,草地早熟禾SRAP-PCR最佳反应体系为Taq DNA聚合酶1.0U、Mg2+1.75mmol·L-1、引物0.25μmol·L-1、dNTP 220μmol·L-1、40ng模板DNA、2μL 10×PCR buffer,总体积20μL。运用该体系从100对SRAP引物中筛选出43对引物能够产生清晰稳定的扩增条带且多态性丰富。优化体系的建立及引物的筛选可为今后利用SRAP标记技术对草地早熟禾进行遗传多样性分析、图谱构建、种质资源鉴定奠定技术基础。
草地早熟禾;SRAP标记;正交试验设计;引物筛选
相关序列扩增多态性(Sequence-related Amplified Polymorphism,简称SRAP),是2001年由Li和Quiros[1]在芸薹属(Brassica)中首次应用的一种基于PCR的分子标记技术,该标记针对基因启动子与内含子中胸腺嘧啶、腺嘌呤含量丰富而外显子中胞嘧啶、鸟嘌呤含量丰富的特点,设计特定引物进行扩增反应,因不同个体启动子、内含子与间隔区长度不同而使扩增条带产生多态性。SRAP标记具有简便、稳定、产率中等以及在基因组中分布均匀等特点[2-3],已被广泛应用于种质资源遗传多样性分析[4-5]、遗传图谱构建[6-7]、比较基因组学[8]、重要性状基因标记[9-10]等方面。
草地早熟禾(Poapratensis)为冷季型草坪草,具有绿期长、色泽翠绿、质地均一柔软等优点,在全世界温带地区广泛种植[11-13]。目前,ISSR[14]、RAPD[15-16]分子标记技术在草地早熟禾研究中均有应用,而SRAP标记方面的研究目前尚未见报道。本研究首次利用正交设计,对草地早熟禾SRAPPCR反应体系进行优化,并利用所得最佳反应体系对SRAP引物进行筛选,以获得草地早熟禾最佳SRAPPCR反应体系和多态性丰富的SRAP引物组合,以期为草地早熟禾SRAP分子标记研究奠定基础。
1 材料与方法
1.1 供试材料 试验所用的4份草地早熟禾材料(表1)由中国农业科学院北京畜牧兽医研究所牧草种质资源研究室提供,SRAP-PCR体系优化的DNA模板来自巴林。SRAP引物由上海生工生物工程公司合成,dNTP和Taq DAN聚合酶为TaKaRa产品。
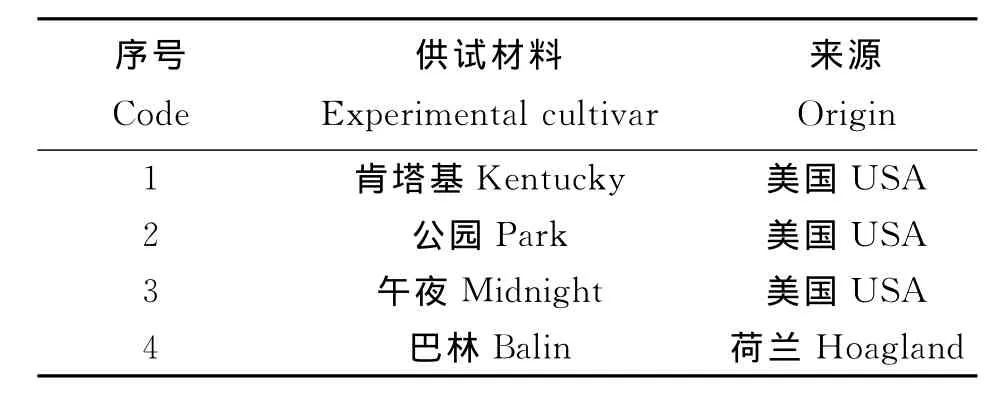
表1 供试材料的名称与来源Table 1 Names and sources of experimental plants
1.2 试验方法
1.2.1 基因组DNA的提取与检测 以苗期草地早熟禾叶片为材料,采用改进的CTAB法[17]提取基因组DNA,通过0.8%琼脂糖凝胶电泳检测提取DNA的质量,并以一定浓度的λDNA作为定量参照标准,稀释样品DNA浓度至50ng·μL-1,-20℃保存备用。
1.2.2 PCR反应体系正交试验设计 采用L9(34)正交试验设计,对Taq DNA聚合酶、Mg2+、引物及dNTP进行4因素3水平筛选(表2、表3),所用引物为Me1和Em3组合(表4)。在20μL PCR反应体系中,含有2μL 10×PCR buffer和50ng模板DNA,其余各成分含量按表3进行制备。
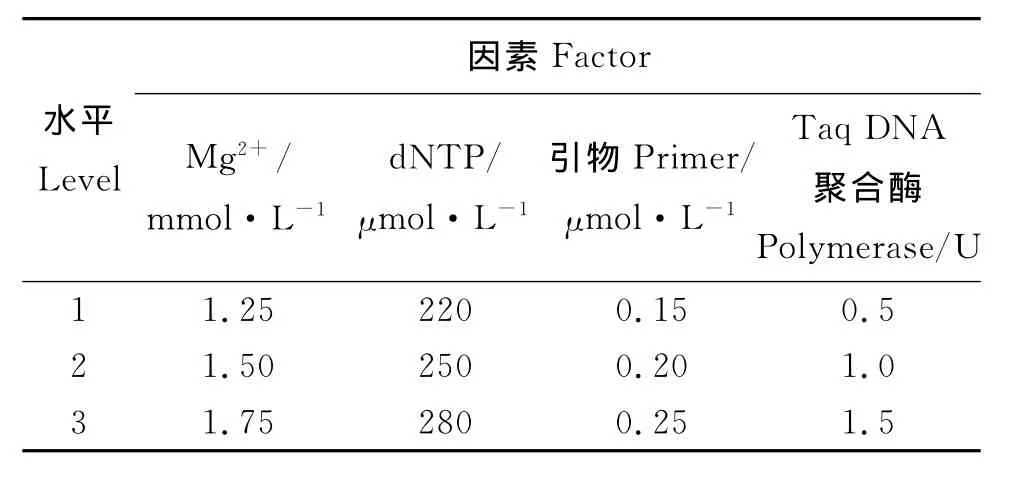
表2 SRAP-PCR体系的因素和水平Table 2 Factors and levels of SRAP-PCR system
PCR扩增程序:94℃预变性4min;94℃变性1 min,37℃退火1min,72℃延伸1min,5个循环;94℃变性1min,50℃退火1min,72℃延伸1min,35个循环;循环结束后72℃延伸7min,4℃保存。扩增结束后,取7.5μL PCR产物与1.5μL 6×Loading buffer混匀,点入含0.5μg·mL-1EB的2%琼脂糖凝胶中,在100V电压下电泳60min,电泳结束后,将凝胶置于紫外凝胶成像仪中观察照相。
1.2.3 模板DNA用量优化 应用正交试验筛选出的最佳组合,对模板DNA用量进行优化。引物选用Me1、Em3组合,模板DNA用量设置12个梯度,依次为10、15、20、25、30、35、40、45、50、60、70、80ng。
1.2.4 引物筛选及优化体系的应用 依据上述试验结果确定的SRAP-PCR最佳反应体系,以巴林基因组DNA为模板,对供试的100对引物组合(表4)进行筛选;从筛选得来的引物中随机选取6对引物组合,对4份草地早熟禾种质材料进行多态性验证。

表3 SRAP-PCR[L9(34)]正交试验设计Table 3 Orthogonal design of SRAP-PCR[L9(34)]
2 结果与分析

表4 SRAP引物序列Table 4 Primer sequences of SRAP
2.1 SRAP-PCR正交试验扩增结果分析 依据谱带的数量及强弱,将SRAP-PCR反应体系9个组合的扩增结果由好至差依次排列为7、3、4、8、2、5、1、9、6(图1)。1、2、5、6和9五个组合条带数量过少,不宜采用;3、4、7、8四个组合条带均较多,经仔细比较后发现组合7的条带数量最多且清晰度高。因此,选择该组合为草地早熟禾SRAP-PCR的最佳反应体系,即4因素用量分别为Taq DNA聚合酶1.0U、Mg2+1.75mmol·L-1、引物0.25μmol·L-1、dNTP 220μmol·L-1,总体积20μL。
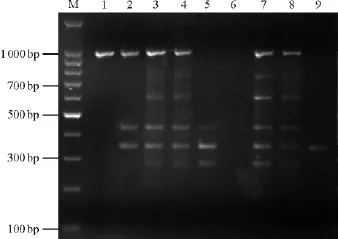
图1 正交设计SRAP-PCR反应体系扩增结果Fig.1 Amplification results of SRAP-PCR system under orthogonal design
参照张丽等[18]的统计方法,对9个组合正交试验结果进行分析(表5),其中K代表该因子在某水平下参与反应所扩增出的总条带数;k代表该因子在某水平下的平均扩增条带数,即K的平均值,其大小反映了该因子的不同水平对反应体系的影响状况,k值越大表明该水平越好;R代表不同水平下该因子得到的条带最大平均值与最小平均值之差,即k的最大值与最小值之差,R值越大,表明该因子对扩增结果影响越显著。从k值的统计结果来看,Taq DNA聚合酶以水平3好,Mg2+以水平3好,引物以水平3好,dNTP以水平1好,即Taq DNA聚合酶1.5U、Mg2+1.75mmol·L-1、引物0.25 μmol·L-1、dNTP 220μmol·L-1,该体系与组合7最接近,仅在Taq DNA聚合酶浓度上略有差异,而直观分析得出的最佳体系组合也为组合7,从而双重确认组合7为最佳体系组合。由R的统计结果表明在被试3个水平下,Taq DNA聚合酶、Mg2+、引物及dNTP四因素对扩增结果的影响由强到弱依次为引物>Taq DNA聚合酶=dNTP>Mg2+。

表5 正交试验结果的统计分析Table 5 Statistic result of orthogonal design
2.2 模板DNA用量的确定 由于模板DNA的用量不同,草地早熟禾SRAP-PCR扩增结果存在较大差异(图2):当模板DNA用量过低(≤25ng)或过高(≥60ng)时,条带数量很少或几乎无条带;当模板用量为30~50ng时条带数量多,且清晰稳定,各处理间均匀一致,说明SRAP扩增模板DNA浓度适应范围较宽。此5个处理基因组DNA用量平均为40ng,而在40ng条件下扩增条带清晰无杂带。因此,最终确定20μL体系中模板DNA的含量为40ng。
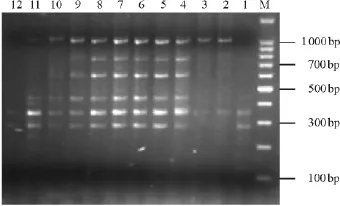
图2 不同模板DNA浓度扩增结果Fig.2 Amplification results under different template DNA concentration
2.3 SRAP引物筛选及验证 利用优化好的SRAP-PCR反应体系,对供试的100对SRAP引物进行筛选,有43对引物能够产生清晰稳定且多态性丰富的条带(表6)。从中随机选出6对(Me1和Em10、Me2和Em1、Me3和Em1、Me5和Em2、Me6和Em9、Me10和Em1)对4份草地早熟禾进行SRAP-PCR扩增,结果表明(图3),6对引物共扩增出34条谱带,平均每对引物扩增出5.7条;其中,多态性谱带17条,占总数的50%,每对引物均扩增出了多态性条带,平均每对引物扩增出的多态性条带数为2.8条(表7)。由此说明,筛选出的43对SRAP引物能够产生丰富的多态性,可应用于草地早熟禾遗传多样性分析、遗传图谱构建、重要性状基因标记等方面的研究。
3 讨论与结论

表6 筛选出的SRAP引物Table 6 Screened SRAP primer

图3 优化的SRAP-PCR反应体系在部分引物组合中的扩增结果Fig.3 Amplification results of primers in optimized SRAP-PCR system
在PCR反应体系中各个因素间都会发生相互作用,如Mg2+受dNTP的拮抗作用,而Taq DNA聚合酶是Mg2+依赖性酶,Mg2+浓度过高或过低都会直接影响Taq DNA聚合酶的活性,而Taq DNA聚合酶又是引物与模板DNA结合后进行延伸的必要因素。因此,反应体系中任何一种因素的改变都会导致扩增结果发生变化[19-22]。与以往的单因素PCR优化设计相比,正交试验设计优化PCR反应体系可以综合考察各因素及其交互作用,减少工作量,降低试验成本,快速获得满意的试验结果[23-25]。本研究采用正交试验设计优化草地早熟禾SRAPPCR反应体系,并参照张丽等[18]的方法对各因素不同水平组合的正交结果进行了统计分析,得出在本研究中各因素对反应体系的影响由大到小依次为引物>Taq DNA聚合酶=dNTP>Mg2+,而运用该方法得出的最佳体系组合与直观分析得出的最佳体系组合最为接近,肯定了该体系的可行性。

表7 6个多态性引物组合的扩增结果Table 7 Amplification results of six polymorphism primer combinations
利用SRAP、AFLP、ISSR三种分子标记方法对毛竹(Phyllostachysheterocycla)种质的鉴别表明,SRAP的单一分子标记一致性高于AFLP和ISSR两种标记[26]。与RAPD标记相比,SRAP聚类分析的石斛属植物遗传关系结果更接近于基于形态特征的分类结果,能更准确地揭示品种间的地域特性及亲缘关系,而且SRAP标记技术具有更高的标记效率[27]。本研究也表明,SRAP标记应用于草地早熟禾中产生的多态性丰富,同样可以对草地早熟禾优良性状标记、遗传图谱构建、遗传多样性分析等方面进行更深入的研究。另外,在本研究中,对PCR产物的检测选用了琼脂糖凝胶电泳,若选用灵敏度更高的聚丙烯酰胺凝胶电泳,则可以大大提高检测的分辨率,多态性条带数量也会相应增多,更利于进行深入研究。
本研究应用正交试验设计对草地早熟禾SRAP-PCR反应体系进行优化,最终得到的最佳反应体系:Taq DNA聚合酶1.0U、Mg2+1.75 mmol·L-1、引物0.25μmol·L-1、dNTP 220 μmol·L-1、40ng模板DNA、2μL 10×PCR buffer,总体积20μL;利用最佳反应体系对SRAP引物进行筛选,共得到43对引物能够产生清晰稳定、多态性丰富的条带;从中选取6对引物对4份草地早熟禾材料进行扩增,结果表明,该反应体系稳定可靠,且各引物能够产生丰富的多态性,可用于草地早熟禾SRAP分子标记研究。
[1]Li G,Ouiros C F.Sequence-related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:It’s application to mapping and gene tagging inBrassica[J].Theoretical and Applied Genetics,2001,103:455-461.
[2]李润芳,惠荣奎,邓瑞宁,等.三叶草遗传多样性的SRAP分析[J].草业科学,2010,27(12):53-57.
[3]罗燕,白史且,彭燕,等.菊苣种质资源遗传多样性的SRAP研究[J].草业学报,2010,19(5):139-147.
[4]文雁成,王汉中,沈鑫雄,等.用SRAP标记分析中国甘蓝型油菜品种的遗传多样性和遗传基础[J].中国农业科学,2006,39(2):246-256.
[5]易杨杰,张新全,黄琳凯,等.野生狗牙根种质遗传多样性的SRAP研究[J].遗传,2008,30(1):94-100.
[6]Lin Z,He D,Zhang X,etal.Linkage const ruction and mapping QTL for cotton fibre quality using SRAP,SSR and RAPD[J].Plant Breeding,2005,124(2):180-187.
[7]林忠旭,张献龙,聂以春,等.棉花SRAP遗传连锁图构建[J].科学通报,2003,48(15):1676-1679.
[8]Li G,Gao M,Yang B,etal.Gene for gene alignment between theBrassicaandArabidopsisgenomes by direct transcriptome mapping[J].Theoretical and Applied Genetics,2003,107:168-180.
[9]赵光伟,徐永阳,徐志红,等.甜瓜抗白粉病基因SRAP分子标记筛选[J].西北植物学报,2010,30(6):1105-1110.
[10]邸青,张淑江,章时蕃,等.大白菜缘枯病抗性基因的SRAP标记筛选[J].中国蔬菜,2010(16):21-25.
[11]张晓波,赵艳.草地早熟禾根际胶质芽孢杆菌的分离及鉴定[J].草业科学,2010,27(6):138-142.
[12]杜建雄,侯向阳,刘金荣.草地早熟禾对干旱及旱后复水的生理响应研究[J].草业学报,2010,19(2):31-38.
[13]陈雅君,祖元刚,刘慧民,等.干旱对草地早熟禾膜质过氧化酶和保护酶活性的影响[J].中国草地学报,2008,30(5):32-36.
[14]童阿玛.基于ISSR标记的草地早熟禾遗传多样性研究[D].西宁:青海大学,2009.
[15]宁婷婷,张再君,金诚赞,等.早熟禾品种间遗传多样性分析[J].遗传,2005,27(4):605-610.
[16]田志宏,邱永福,严寒,等.用RAPD标记分析草地早熟禾遗传多样性[J].草地学报,2006,14(2):120-123.
[17]侯思名,段继强,梁雪妮,等.苎麻总DNA提取的CTAB法优化方案[J].西北植物学报,2005,25(11):2193-2197.
[18]张丽,周兰英,肖千文,等.正交试验设计在建立杜鹃花RAPD-PCR反应体系中的应用[J].北方园艺,2007(5):124-126.
[19]郑轶琦,王志勇,郭海林,等.正交设计优化假俭草SRAP-PCR反应体系及引物筛选[J].草业学报,2008,17(4):110-117.
[20]王志勇,袁学军,刘建秀,等.狗牙根SRAP-PCR反应体系优化及引物筛选[J].草地学报,2008,17(3):79-85.
[21]李丽,郑晓鹰,柳李旺.用SRAP标记分析黄瓜品种遗传多样性及鉴定品种[J].分子植物育种,2006,4(5):702-708.
[22]陈万胜,王元英,罗成刚,等.利用正交设计优化烟草SRAP反应体系[J].分子植物育种,2008,6(1):177-182.
[23]张平湖,刘冠明.橄榄SRAP-PCR体系的建立和优化[J].中国农学通报,2010,26(15):86-88.
[24]邹小云,邹晓芬,陈伦林,等.花生SRAP-PCR反应体系的正交设计优化[J].分子植物育种,2010,8(4):822-826.
[25]Ren Y,Wang D Y,Zhang Y D,etal.Optimization of SRAP-PCR in hot pepper[J].Molecular Plant Breeding,2004,2(5):689-693.
[26]郭起荣,任立宁,牟少华,等.毛竹种质分子鉴别SRAP、AFLP、ISSR联合分析[J].江西农业大学学报,2010,32(5):0982-0986.
[27]樊洪泓,李廷春,邱婧,等.石斛属几种植物遗传关系的SRAP和RAPD比较分析[J].中草药,2010,41(4):627-732.
Optimization of SRAP-PCR system on Poa pratensis using orthogonal design and selection of primers
REN Xiao-wei1,2,WANG Yu2,YUAN Qing-hua2
(1.College of Pastoral Agricultural Science and Technology,Lanzhou University,Lanzhou 730020,China;2.Institute of Animal Science,Chinese Academy of Agricultural Science,Beijing 100193,China)
An orthogonal design was used to optimize a SRAP-PCR system forPoapratensiswith 4factors(Mg2+,dNTP,primer and Taq polymerase)at 3levels plus the concentration of template DNA.The optimized SRAP-PCR system was:2μL 10×PCR buffer,40ng template DNA,Mg2+1.75mmol·L-1,dNTP 220μmol·L-1,primer 0.25μmol·L-1,Taq DNA polymerase 1.0Uin a total of 20μL reaction mixtures.With the optimized system,43primer combinations were selected among 100primer combinations,which produced abundant polymorphism bands.This study optimized SRAP-PCR system and selected the proper primers inP.pratensis,which would play an important role in genetic diversity analyses,map construction,germplasm identification inP.pratensiswith SRAP markers.
Poapratensis;SRAP markers;orthogonal design;selection of primers
YUAN Qing-hua E-mail:yuanqinghua@hotmail.com
S688.4;Q945.79
A
1001-0629(2012)03-0411-06
2011-05-06 接受日期:2011-05-25
国家“十二五”支撑项目(2011BAD17B01)
任小巍(1984-),男,河北张家口人,硕士,主要从事牧草种质资源研究。E-mail:xiao521gui@126.com
袁庆华 E-mail:yuanqinghua@hotmail.com
