实验室养殖条件下麦穗鱼Pseudorasbora parvaeDNA浓度与生物量相关性曲线的拟合与比较分析
2023-07-17郭娟曾燏郑羽晨赵雨双钟清
郭娟 曾燏 郑羽晨 赵雨双 钟清
摘 要:为探究实验室养殖条件下麦穗鱼(Pseudorasbora parva)eDNA浓度与其生物量间关系,根据麦穗鱼线粒体COⅠ基因序列设计特异性引物,通过实时荧光定量PCR(qPCR),得到qPCR标准曲线,采用线性、指数和对数3种模型对麦穗鱼生物量与eDNA浓度的相关性曲线进行拟合和比较分析。结果表明:基于CO I基因设计的引物对麦穗鱼DNA具有特异性扩增,扩增效率为90.94%;qPCR的阈值循环数(Cycle threshold,Ct值)与已知浓度或拷贝数的样品(标准品)间呈现良好的线性关系(R2=0.995 2),说明标准品的拷贝数与Ct值之间相关的可信度较好。通过3种曲线进一步拟合比较分析显示:线性模型为麦穗鱼生物量与eDNA浓度间的最佳拟合模型,其R2高达0.933 8,y=72.087x+81.978。这表明在本试验条件下,eDNA浓度与麦穗鱼生物量呈正相关性。
关键词:eDNA浓度;生物量;麦穗鱼
中图分类号:S932 文献标识码:A DOI 编码:10.3969/j.issn.1006-6500.2023.06.008
Abstract:In order to investigate the relationship between the environmental DNA (eDNA) concentration and biomass of Pseudorasbora parva reared in the laboratory, specific primers were designed based on the mitochondrial COI gene sequence of Pseudorasbora parva. The qPCR standard curve was obtained through quantitative real-time PCR (qPCR). The correlation curves between Pseudorasbora parva biomass and eDNA concentration were fitted and compared using linear, exponential, and logarithmic models. The results showed that the primers designed based on the COI gene specifically amplified Pseudorasbora parva DNA, with an amplification efficiency of 90.94%. The cycle threshold (Ct) value of qPCR showed a good linear relationship with known concentration or copy number of samples (standards) (R2=0.995 2), indicating a good credibility of the correlation between the copy number of standards and the Ct value. Further fitting and comparison of the three curves revealed that the linear model was the best fit for the relationship between Pseudorasbora parva biomass and eDNA concentration, with a high R2 of 0.933 8 and y=72.087x+81.978. This indicates a positive correlation between eDNA concentration and Pseudorasbora parva biomass under the experimental conditions.
Key words: eDNA concentration; biomass; Pseudorasbora parva
监测鱼类生物量是入侵物种种群管理工作的重要组成部分,然而,直接使用传统捕捞调查方法会破坏水生生态环境,也难以准确评估个体小和群众数量少的物种生物量[1]。近年来,随着分子生物学的发展,eDNA技术作为一种新的监测方法,凭借便捷、环境友好、经济高效等优点在珍稀濒危物种和入侵物种监测领域发挥了重要作用[2-3]。环境DNA(Environmental DNA,eDNA)是指从環境样本中(水、土壤、空气等)提取到的总DNA[4]。目前,eDNA主要运用于4个方面。一是检测目标物种的有无,通过检测水体中是否有目标物种释放的DNA来判断水体中是否有该物种的存在[5];二是生物多样性评估,高通量测序的出现使得从一个eDNA样品中同时检测多种生物的DNA成为可能,现阶段已被用于水生生态系统生物多样性的检测[6-7];三是鱼类分布,利用eDNA方法可以识别出一个特定物种在一个大区域(如海湾和大湖泊)上的分布,明确目标物种分布将有助于管理和监测海洋和大型湖泊中的鱼类[8];四是生物量估算,对目标生物的生物量进行调查是水生生物资源监测的重要内容[9-10]。
近年来我国开展了大量与eDNA技术相关的研究,但主要运用的是检测目标物种、生物多样性评估和鱼类分布3个方面,关于eDNA技术评估生物量的研究较少[1]。鱼类资源的评估不仅要定性地评估鱼类多样性,也要定量地评估生物量,实现这些将有助于eDNA技术成为管理鱼类资源的一般工具[11]。生物量是指某一特定时刻单位空间的个体数、质量,如何科学地开展生物量评估是鱼类种群生态的一个重难点问题,准确的生物量评估有助于鱼类资源的合理开发利用和外来入侵物种的有效管理,建立eDNA浓度与生物量间关系是基于eDNA技术探究野生鱼类生物量的重要依据[12]。
麦穗鱼(Pseudorasbora parva)隶属于鲤形目(Cypriniformes)、鲤科(Cyprinidae)、鮈亚科(Gobioninae)、麦穗鱼属(Pseudorasbora)。麦穗鱼主要以浮游动物为食,分布广,世代周期短,繁殖能力和适应能力强,已成功入侵全球超过30个国家[13-14]。当前通过传统种群生态学方法难以准确的评估麦穗鱼种群的生物量,eDNA方法的大量应用使得这一难题的顺利解决成为了可能。目前关于麦穗鱼的研究主要包括麦穗鱼养殖、生活史和疾病等方面,而基于eDNA评估麦穗鱼生物量未见报道[15-17]。
因此,本试验以实验室人工养殖的麦穗鱼为目标物种,通过设计麦穗鱼的特异性引物,实时荧光定量PCR建立标准曲线和熔解曲线,采用线性、指数和对数3种模型对麦穗鱼生物量与eDNA浓度的相关性曲线进行了拟合和分析。研究结果有助于建立实验室条件下eDNA浓度与其生物量的最佳相关性曲线,为eDNA浓度评估野外麦穗鱼生物量提供理论支撑。
1 材料与方法
1.1 特异性引物设计
针对麦穗鱼的线粒体CO I基因设计特异性引物。在GenBank上检索麦穗鱼的基因序列,根据已发表的麦穗鱼序列(基因序列号MT805712.1),利用相关软件Primer-Blast设计引物,使用引物分别对麦穗鱼DNA和12种鮈亚科鱼类DNA进行扩增,PCR试验程序为:95 ℃预变性2 min;95 ℃变性20 s,55 ℃退火20 s,72 ℃延伸10 s,40个循环;72 ℃延伸7 min。扩增后对PCR产物凝胶电泳检测,DNA的胶回收、连接和测序,最后比对测序结果是否是目的基因。
1.2 eDNA采集和提取
在5个鱼缸(50 cm×45 cm×40 cm)中加入50 L曝气水,温度为5 ℃。分别在5个缸中放置0、2、4、8、16尾麦穗鱼,平均每尾质量4.5 g。试验期为7 d,7 d内不喂食,不换水。在试验第7天采集样本,使用消毒杀菌后的EP管取养殖缸表层水50 mL,每个缸做3次重复,设置1个蒸馏水阴性对照,共计18个样本。采样后立即用直径为50 mm,孔径为0.45 μm的混合纤维素滤膜进行无菌真空抽滤装置抽滤,每次抽滤之前用无菌水冲洗过滤漏斗,防止交叉污染。抽滤完成后,用无菌镊子将每张滤膜单独卷起来,放入5 mL 的离心管中并做好标记,按照DNeasy PowerWater kit(Qiagen,Germany)试剂盒的操作方法提取eDNA,將收集到的DNA放置-20 ℃保存。
1.3 实时荧光定量PCR
使用TOYOBO的SYBR Green Real-time PCR Master Mix 试剂盒和 Applied Biosystems StepOneTM qPCR仪。将10 μL的2× SYBR real-time PCR premixture和0.4 μL的正向引物与0.4 μL反向引物(10 μmol·L-1)混合为mixture A体系,上机体系为:8 μL的mixture A和8 μL模板稀释样。试验程序为:95 ℃预变性2 min;95 ℃变性20 s,55 ℃退火20 s,72 ℃延伸10 s,40个循环;72 ℃延伸7 min,在每次运行退火之后采集荧光信号。首先,将目标片段用Taq酶扩增,连接T载体,装入感受态细菌,挑单克隆,摇菌,经测序确认片段正确后,在洁净工作台进行质粒小抽和定量。将标准品依次稀释,形成 101~107 copies·μL-1的7个梯度,制作荧光定量PCR标准曲线。每次试验均设置阴性对照品。为了确定PCR反应效率和定量未知样本,计算并绘制熔解曲线和标准曲线。
1.4 拟合曲线模型的选择
选用线性、指数和对数3种曲线模型进行拟合,利用R软件绘制曲线图,通过比较不同模型曲线拟合度R2的值大小来确定最佳拟合曲线。
2 结果与分析
2.1 提取DNA分析
从水体样本提取的 eDNA和鱼类肌肉中提取的 DNA 浓度分别为20~89 ng·μL-1和376~719 ng·μL-1。提取 eDNA 的OD值(A260/A280)为1.60~2.04,肌肉 DNA 的OD值为 1.79~2.13。尽管有部分eDNA浓度较低,但总体DNA的OD值能满足后续试验需求。阴性对照组和空白组均无扩增。
2.2 麦穗鱼引物和标准曲线
得到麦穗鱼引物F:CCGATTCTCATTGGGGGCTT,
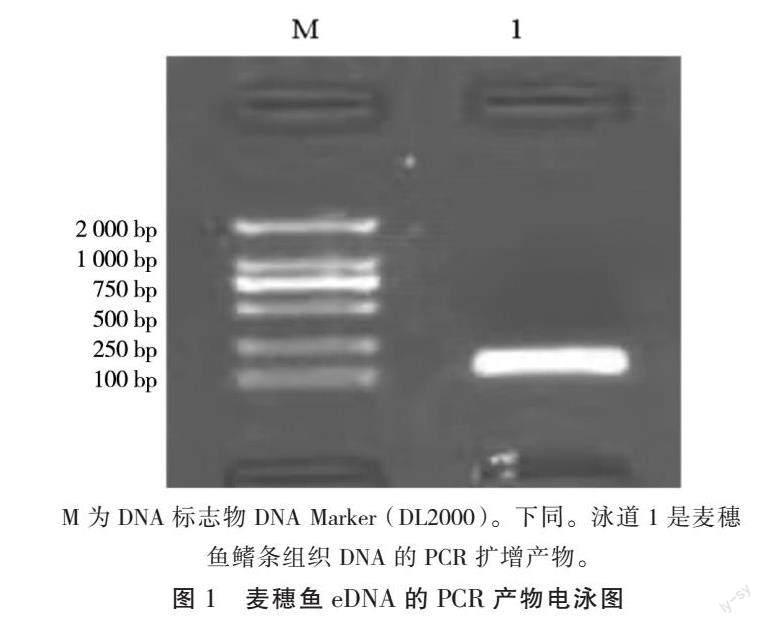
R:CCTGCGAGAGGGGGATAAAC,片段长度188 bp。将麦穗鱼eDNA扩增得到的PCR原液进行凝胶电泳并测序,目的条带单一且明亮清晰,其测序结果在NCBI 网站上经过 Blast 比对分析,其结果显示与原始序列片段同源性达 100%。如图1所示,目的条带清晰明亮且无非特异性扩增。
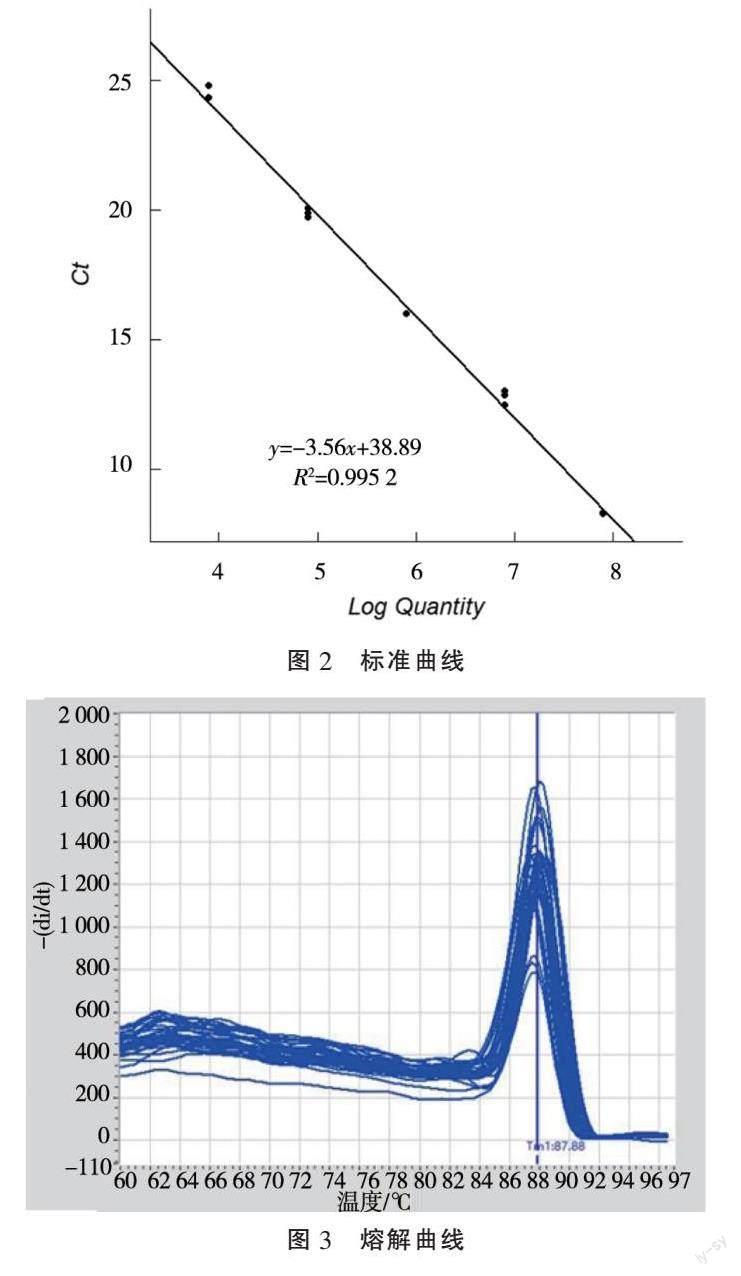
通过实时荧光定量PCR扩增,阈值循环数(Cycle threshold,Ct值)平均值分别为4.83、12.75、14.53、20.09、24.56(图2)。Ct值是指每个反应管内的荧光信号到达设定阈值时所经历的循环数。Ct值与模板的起始拷贝数的对数存在线性关系,起始模板浓度越高,Ct值越小;起始模板量浓度越低,Ct值越大。系统根据标准品浓度与扩增Ct值生成麦穗鱼引物扩增的标准曲线。标准曲线用于确定实验中未知样本的目标DNA的相对或绝对量。标准曲线为:Ct=-3.56 x+38.89,(x为样本中麦穗鱼 eDNA 稀释浓度的对数值),曲线的相关系数 R2=0.995 2,扩增效率为90.94%,说明标准品的拷贝数与Ct值之间相关的可信度较好。反应完成后,得到的熔解曲线如图2所示。熔解曲线是指随温度升高 DNA 的双螺旋结构降解程度的曲线。本研究得到的溶解曲线出现单峰,说明反应具有特异性。
2.3 麦穗鱼eDNA浓度与生物量相关性曲线
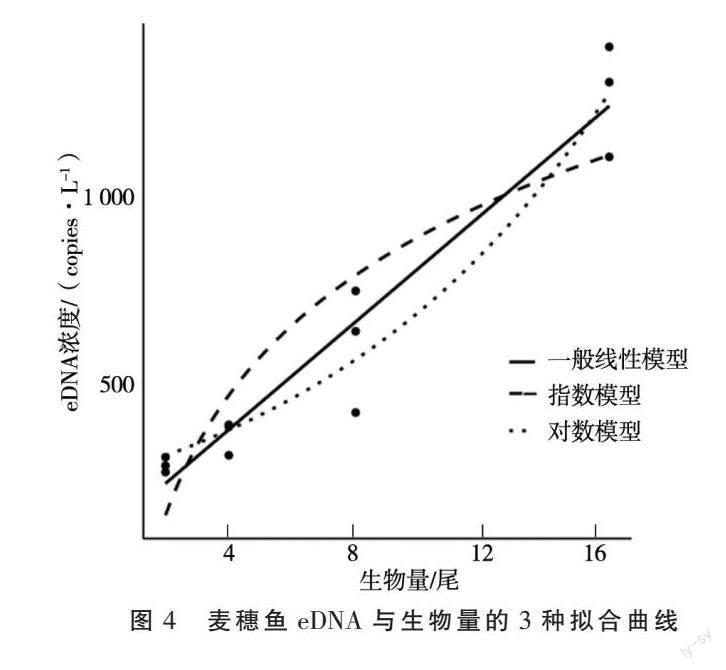
根据公式Ct=-KlogX0+b计算可得(X0表示拷贝数,k 表示标准曲线斜率,b 表示标准曲线截距),0、2、4、8、16尾麦穗鱼养殖缸中得到的平均拷贝数分别为0、275.56、354.03、596.67、1 264.28个·L-1。采用线性、对数和对数3种模型对麦穗鱼生物量与eDNA浓度的相关性曲线进行了拟合。结果如图3所示,3种曲线的拟合分析结果显示:线性模型为麦穗鱼生物量与eDNA浓度的最佳拟合模型,y = 72.087x+81.978,其R2高达0.933 8。eDNA浓度与麦穗鱼生物量呈正相关性,即麦穗鱼生物量越大,eDNA浓度也会越高。
3 讨论与结论
3.1 引物和标准曲线
麦穗鱼eDNA扩增后进行凝胶电泳并测序,目的条带单一且明亮清晰,测序结果为麦穗鱼。这说明基于COI基因设计的引物对麦穗鱼DNA具有特异性扩增,且其扩增效率为90.94%,引物特异性好。
本研究得到的标准曲线为Ct=- 3.56 x + 38.89,曲线的相关系数 R2=0.995 2,说明Ct值与标准品间呈现良好的线性关系,标准品的拷贝数与Ct值之间相关的可信度较好。结果表明,本研究在此麦穗鱼eDNA浓度范围内具有良好的线性关系,所建立的标准曲线能够正确反映出麦穗鱼目的基因的扩增。
实时荧光定量PCR的SYBR Green Ⅰ荧光染料法其优势在于检测方法简便,检测成本低。不足之处是染料与DNA双链分子的结合是非特异性的,它可以和反应体系中的所有DNA分子结合,易受到非特异性扩增和引物二聚体的影响,使定量结果不可靠[18]。解决这一问题可以借助熔解曲线分析法来区分非特异性产物和引物二聚体而排除假阳性[19]。熔解曲线如出现多峰,证明反应非特异或存在二聚体。本研究得到的熔解曲线中出现单峰(图2),说明定量结果是特异可靠的。
3.2 eDNA浓度与生物量相关性曲线
Takahara[9]等发现eDNA 浓度在第6天至第9天之间变化不大,得出eDNA浓度在第6天趋于稳定的结论。因此,本研究在试验第7天进行采样时,eDNA浓度差异不明显,得到的eDNA浓度与生物量的关系更为可靠。
通过3种曲线进一步拟合比较分析显示:线性模型为麦穗鱼生物量与eDNA浓度间的最佳拟合模型,其R2高达0.933 8,y=72.087x+81.978,表明在本试验条件下,eDNA浓度与麦穗鱼生物量呈正相关性。虽然在小型水族箱和人工池塘中eDNA浓度与生物量存在线性关系[20-22],但在湖泊河流中eDNA与生物量的关系会更加的复杂,可能是线性或非线性。闫卉果[9]等在养殖桶中得到岩原鲤eDNA浓度与其生物量为线性关系;Coulter[23]等在河流中发现鲢鱼eDNA浓度与其生物量存在非线性关系。本研究的拟合分析结果显示:线性模型为麦穗鱼生物量与eDNA浓度的最佳拟合模型,y=72.087x+81.978,其R2高达0.933 8,研究结果与养殖桶条件下目标鱼eDNA浓度与其生物量间关系一致。
3.3 eDNA浓度的影响因素
eDNA技术作为一种新的监测方法,具有便捷、环境友好、经济高效等优点。eDNA产生后能在水体中保留一段时间,直至被降解不能检测[24]。因此,它被认为提供了近似的实时数据。eDNA的降解与产生会导致eDNA浓度发生变化,但eDNA浓度的变化受多种因素的影响,进而影响eDNA浓度与其生物量的关系,主要包括生物因素和非生物因素2个方面。
生物因素方面体现在鱼类的生物量会影响eDNA浓度。Michael等[25]發现,当大马哈鱼的密度越高,eDNA浓度也越高;Lacoursière等[26]研究表明,在同一温度条件下,生物量越大,eDNA浓度越高。本研究结果发现,随着养殖缸中麦穗鱼的增加(0、2、4、8、16尾),eDNA浓度也呈正相关的线性关系。除生物因素外,其他非生物因素也会影响eDNA浓度,主要包括温度、紫外线、pH等外界环境因素[27-30]。因此,在实际野外工作中需要综合考虑各种因素,建立麦穗鱼生物量和eDNA浓度关系模型才能准确评估麦穗鱼的种群生物量。
本研究通过麦穗鱼线粒体COI基因设计了麦穗鱼特异性引物,F:CCGATTCTCATTGGGGGCTT,R: CCTGCGAGAGGGGGATAAAC,片段长度188 bp。确定了实验室养殖麦穗鱼eDNA浓度与其生物量的最佳拟合模型为线性模型,y=72.087x+81.978,R2=0.933 8。eDNA浓度与麦穗鱼生物量呈正相关性。
参考文献:
[1] 刁曹鋆, 王闻, 线薇薇, 等. 环境DNA技术在渔业资源生物量评估中的研究进展:现状与展望[J]. 海洋科学, 2022, 46(2): 135-144.
[2] 赵明, 赵梦迪, 马春艳, 等. 环境DNA在水域生态中的研究进展[J]. 中国水产科学, 2018, 25(4): 714-720.
[3] 赵彦伟, 陈家琪, 董丽, 等. 环境DNA技术在水生态领域应用研究进展[J]. 农业环境科学学报, 2021, 40(10): 2057-2065.
[4] THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA[J]. Molecular Ecology, 2012, 21(11): 2565-2573.
[5] FICETOLA G F, PANSU J, BONIN A, et al. Replication levels, false presences and the estimation of the presence/absence from eDNA metabarcoding data[J]. Molecular Ecology Resources, 2015, 15(3): 543-556.
[6] JERDE C L, WILSON E A, DRESSLER T L. Measuring global fish species richness with eDNA metabarcoding[J]. Molecular Ecology Resources, 2019, 19(1): 19-22.
[7] AHN H, KUME M, TERASHIMA Y, et al. Evaluation of fish biodiversity in estuaries using environmental DNA metabarcoding[J]. PLoS One, 2020, 15(10): e0231127.
[8] DEJEAN T, VALENTINI A, DUPARC A, et al. Persistence of environmental DNA in freshwater ecosystems[J]. PLoS One, 2011, 6(8): e23398.
[9] TAKAHARA T, MINAMOTO T, YAMANAKA H, et al. Estimation of fish biomass using environmental DNA[J]. PLoS One, 2012, 7(4): e35868.
[10] KELLY R P, PORT J A, YAMAHARA K M, et al. Harnessing DNA to improve environmental management[J]. Science, 2014, 344(6191): 1455-1456.
[11] 秦傳新, 左涛, 于刚, 等. 环境DNA在水生生态系统生物量评估中的研究进展[J]. 南方水产科学, 2020, 16(5): 123-128.
[12] DOI H, INUI R, AKAMATSU Y, et al. Environmental DNA analysis for estimating the abundance and biomass of stream fish[J]. Freshwater Biology, 2017, 62(1): 30-39.
[13] 杨丽萍, 胡俊仪, 秦超彬, 等. 基于线粒体Cytb分析入侵云南四大水系的麦穗鱼群体遗传结构[J]. 水产学报, 2020, 44(3): 339-350.
[14] 丁瑞华. 四川鱼类志[M]. 成都: 四川科学技术出版社, 1994.
[15] 张凤玉, 刘柳, 张静, 等. 齐齐哈尔地区麦穗鱼吸虫囊蚴感染情况及其种类鉴定[J]. 中国寄生虫学与寄生虫病杂志, 2023, 41(1): 112-116.
[16] 孙欢欢, 朱仁, 冯秀, 等. 西藏湿地外来麦穗鱼生活史特征的适应性研究[J]. 水生生物学报, 2022, 46(12): 1780-1787.
[17] 刘东姣, 杜新科, 王世兴, 等. 水蕹菜和罗莎生菜对麦穗鱼养殖系统水质的影响[J]. 水产学杂志, 2023, 36(1): 89-94.
[18] 袁亚男, 刘文忠. 实时荧光定量PCR技术的类型、特点与应用[J]. 中国畜牧兽医, 2008, 35(3): 27-30.
[19] 陈旭, 齐凤坤, 康立功, 等. 实时荧光定量PCR技术研究进展及其应用[J]. 东北农业大学学报, 2010, 41(8): 148-155.
[20] LACOURSI?魬RE-ROUSSEL A, C?魺T?魪G, LECLERC V, et al. Quantifying relative fish abundance with eDNA: a promising tool for fisheries management[J]. Journal of Applied Ecology, 2016, 53(4): 1148-1157.
[21] SALTER I, JOENSEN M, KRISTIANSEN R, et al. Environmental DNA concentrations are correlated with regional biomass of Atlantic cod in oceanic waters[J]. Communications Biology, 2019, 2(1): 461.
[22] 闫卉果, 董智玲, 马婷婷, 等. 基于环境DNA的岩原鲤检测及生物量评估[J]. 水产学报, 2022, 46(6): 1018-1026.
[23] COULTER D P, WANG P, COULTER A A, et al. Nonlinear relationship between silver carp density and their eDNA concentration in a large river[J]. PLoS One, 2019, 14(6): e0218823.
[24] PIAGGIO A J, ENGEMAN R M, HOPKEN M W, et al. Detecting an elusive invasive species: a diagnostic PCR to detect Burmese python in Florida waters and an assessment of persistence of environmental DNA[J]. Molecular Ecology Resources, 2014, 14(2): 374-380.
[25] TILLOTSON M D, KELLY R P, DUDA J J, et al. Concentrations of environmental DNA (eDNA) reflect spawning salmon abundance at fine spatial and temporal scales[J]. Biological Conservation, 2018, 220: 1-11.
[26] LACOURSI?魬RE-ROUSSEL A, ROSABAL M, BERNATCHEZ L. Estimating fish abundance and biomass from eDNA concentrations: variability among capture methods and environmental conditions[J]. Molecular Ecology Resources, 2016, 16(6): 1401-1414.
[27] JO T, MURAKAMI H, YAMAMOTO S, et al. Effect of water temperature and fish biomass on environmental DNA shedding, degradation, and size distribution[J]. Ecology and Evolution, 2019, 9(3): 1135-1146.
[28] EICHMILLER J J, BEST S E, SORENSEN P W. Effects of temperature and trophic state on degradation of environmental DNA in lake water[J]. Environmental Science & Technology, 2016, 50(4): 1859-1867.
[29] STRICKLER K M, FREMIER A K, GOLDBERG G S. Quantifying effects of UV-B, temperature, and pH on eDNA degradation in aquatic microcosms[J]. Biological Conservation, 2015, 183: 85-92.
[30] LANCE R F, KLYMUS K E, RICHTER C A, et al. Experimental observations on the decay of environmental DNA from bighead and silver carps[J]. Management of Biological Invasions, 2017, 8(3): 343-359.
