桃Ⅱ型NADH脱氢酶家族基因鉴定和表达分析
2022-09-07张春华沈志军马瑞娟张圆圆郭绍雷蔡志翔俞明亮
张春华, 沈志军, 马瑞娟, 张圆圆, 郭绍雷, 蔡志翔, 俞明亮
(江苏省农业科学院果树研究所/江苏省高效园艺作物遗传改良重点实验室,江苏 南京 210014)
Ⅱ型NADH脱氢酶(Type ⅡNADHdehydrogenase,EC 1.6.99.3)广泛存在于真细菌、古细菌、酵母、真菌和植物线粒体中[1-3],基因组测序的100%植物种类中均有Ⅱ型NADH脱氢酶[4]。Ⅱ型NADH脱氢酶家族蛋白质的典型特点是存在一个保守的C端结构域,该结构域在介导蛋白质的二聚化、膜锚定以及底物的识别中发挥着重要作用[5]。Ⅱ型NADH脱氢酶不传递质子,位于线粒体内膜表面(内侧或者外侧),也有位于质体和叶绿体的报道。Ⅱ型NADH脱氢酶相对分子质量低,一般由单个多肽链组成,驱动电子从NADH转移到泛醌[6]。与多聚体复合物I相反,Ⅱ型NADH脱氢酶对鱼藤酮不敏感,通常含有1个FAD非共价结合分子作为氧化还原修复基团[2,7]。
植物能通过Ⅱ型NADH脱氢酶氧化外源NADH和NADPH[8-9]。Ⅱ型NADH脱氢酶介导的NADH氧化可以驱动更快的新陈代谢的产能过程,以及加快碳元素流入生物合成路径,因而可以更高效地合成ATP[5]。每种植物至少含有4种对鱼藤酮不敏感的Ⅱ型NADH脱氢酶[9]。多种植物基因组测序的完成为研究Ⅱ型NADH脱氢酶家族基因提供了便利。根据报道,来自部分植物的Ⅱ型NADH脱氢酶基因已经被鉴定,拟南芥(Arabidopsisthaliana,At)7个[9]、水稻(Oryzasativa,Os)6个、黄豆(Soybean)2个[10]、土豆(Solanumtuberosum,St)2个[11]、玉米(Zeamays,Zm)2个[9,12]、甜菜(Betavulgaris,Bv)2个[12-14]等。另外,来自莱氏衣藻(Chlamydomonasreinhardtii,Cr)7个Ⅱ型NADH脱氢酶已被初步鉴定。
不断增加的证据显示Ⅱ型NADH脱氢酶家族基因在氧化、光呼吸、生物和非生物逆境及其他代谢途径中起重要作用[10,15]。土豆叶片的Ⅱ型NADH脱氢酶蛋白质NDA1依赖光,且对低温敏感,5 ℃冷害处理6 d后,蛋白质表达量下降为原来的10%;在早上蛋白质表达量出现峰值,并可能与昼夜节律和光合作用有关[16-17];输入的蛋白质Ⅱ型NADH脱氢酶在洋地黄素渗透时与膜相关。拟南芥叶片的Ⅱ型NADH脱氢酶基因AtNDA1受光控制,AtNDA2不依赖光,AtNDA2在检测的拟南芥所有组织中均有表达,AtNDA1特异性地在拟南芥地上所有组织中表达,在根中几乎无表达;AtNDB1和AtNDB2在拟南芥所有组织中均有表达,AtNDB3和AtNDB4在拟南芥叶片中不表达,AtNDB4在茎中也不表达;AtNDC1在拟南芥所有检测组织中均有高表达,表明可能与异养代谢、呼吸等活动相关。在莱氏衣藻中,NDA1在复合物I缺乏的情况下,起到氧化基质的作用[18]。Ⅱ型NADH脱氢酶是一类冗余蛋白质,但是拥有良好的可塑性,可以根据环境的不同起到不同的作用,赋予了生物体更好的适应环境的能力[5]。
桃(Prunuspersica)是蔷薇科(Rosaceae)李属(Prunus)植物,果实柔软多汁、营养丰富,深受消费者喜爱,是世界广泛栽培的果树。中国是世界桃生产大国,据联合国粮农组织统计数据库(FAOSTAT)最新统计,2019年中国桃种植面积(84 0919 hm2)和总产量(15 841 928 t),均稳居世界第一,分别占世界桃种植总面积(1 527 052 hm2)的55.07%和总产量(25 737 841 t)的61.55% ;中国桃种植面积排在李、黑刺李、苹果、梨和柿子之后,超过葡萄;总产量仅次于苹果、梨,也已超过葡萄(http://www.fao.org/about/en)。桃是二倍体(2n=2x=16),具有相对小的基因组(约265 Mb),特别是桃Lovell、Chinese Cling全基因组测序获得26 335~27 852个注释基因[19-20],为研究桃每个家族基因的特点和功能提供了坚实的基础。
目前,尚未有桃Ⅱ型NADH脱氢酶家族成员的系统鉴定和分析报道。鉴于此,本研究鉴定了桃Ⅱ型NADH脱氢酶家族成员,并分析了进化特征、基因结构、蛋白质结构、组织表达等特性。研究结果将为进一步验证桃Ⅱ型NADH脱氢酶家族基因在桃生长发育中的功能奠定基础。
1 材料与方法
1.1 桃Ⅱ型NADH脱氢酶家族成员的鉴定
根据拟南芥7个Ⅱ型NADH脱氢酶基因及其编码的蛋白质氨基酸序列,利用BLAST工具检索蔷薇科基因组数据库(http://www.rosaceae.org/node/355)桃参考基因组蛋白质氨基酸序列,将这些候选序列逐一提交到NCBI的在线工具 CD Search(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行保守结构域(NADHdehydrogenase-like)搜索,预测这些蛋白质有无Ⅱ型NADH脱氢酶结构域。存在NADHdehydrogenase-like保守区域的蛋白质就被认为是Ⅱ型NADH脱氢酶蛋白质。
1.2 桃Ⅱ型NADH脱氢酶家族基因克隆
使用植物总RNA提取试剂盒(北京天根生化科技有限公司产品)分别提取桃野鸡红叶芽、老叶、幼叶、一年生枝条的枝表皮、根、幼果肉、熟果肉、熟果皮组织的总RNA,使用1.5%的琼脂糖凝胶电泳和微量紫外可见分光光度计检测总RNA的纯度和浓度,分别以1 μg各组织的总RNA为模板,使用PrimeScriptTMⅡ 1st Strand反转录试剂盒[宝生物工程(大连)有限公司产品]合成cDNA第一条链,并稀释10倍。
根据桃每个Ⅱ型NADH脱氢酶基因的参考基因组CDS序列设计引物(表1),以桃叶片cDNA 为模板,PCR扩增每个桃Ⅱ型NADH脱氢酶基因的开放阅读框(Open reading frame,ORF)全长序列。PCR反应体系50.0 μl:21.0 μl ddH2O,25.0 μl DNA聚合酶混合物,上、下游引物(10 μmol/L)各1.0 μl,cDNA 2.0 μl。PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸60 s,共35个循环;72 ℃延伸10 min。PCR产物经2%琼脂糖凝胶电泳和胶回收后,回收片段与pEASY-T3载体(北京全式金生物技术有限公司产品)连接并转化DH5α感受态细胞,选取菌液PCR鉴定为阳性的克隆,由通用生物系统有限公司测序。

表1 桃Ⅱ型NADH脱氢酶基因克隆所用引物
1.3 桃与其他物种Ⅱ型NADH脱氢酶蛋白质系统进化树的构建
利用软件MEGA 4.1内置的Clustal W程序对6个桃Ⅱ型NADH脱氢酶与拟南芥7个、水稻8个、玉米1个、土豆2个、莱氏衣藻6个Ⅱ型NADH脱氢酶蛋白质氨基酸序列进行比对分析。7个拟南芥Ⅱ型NADH脱氢酶蛋白质氨基酸序列AT1G07180.1、AT2G29990、AT4G28220、AT4G05020、AT4G21490、AT2G20800、AT5G08740下载自http://www.arabidopsis.org/;8个水稻Ⅱ型NADH脱氢酶蛋白质氨基酸序列Os07t0564500-01、Os01t0830100-02、Os01t0830100-01、Os05t0331200-01、Os06t0684000-02、Os06t0684000-01、Os08t0141400-01、Os06t0214900-01下载自https://rapdb.dna.affrc.go.jp;1个玉米Ⅱ型NADH脱氢酶蛋白质氨基酸序列Zm00001eb151430下载自https://plants.ensembl.org/index.html;2个土豆Ⅱ型NADH脱氢酶蛋白质氨基酸序列PGSC0003DMG400021670、PGSC0003DMG400004168下载自https://phytozome-next.jgi.doe.gov;6个莱氏衣藻Ⅱ型NADH脱氢酶蛋白质氨基酸序列XP_001698901.1、XP_001702271.1、XP_001703055.1、XP_001703643.1、XP_001691969.1、XP_001703056.1下载自https://www.ncbi.nlm.nih.gov/。
根据多序列联配的结果,使用MEGA 4.1软件以相邻连接法构建系统进化树,并对构建的系统进化树进行自举评估,随机抽样重复次数设为1 000,其他参数使用系统默认值。并根据拟南芥和水稻Ⅱ型NADH脱氢酶家族的亚家族分类情况进行了桃Ⅱ型NADH脱氢酶家族的亚家族分类。
1.4 桃Ⅱ型NADH脱氢酶基因结构、启动子及保守结构域分析
每个基因序列及所在染色体位置下载自蔷薇科基因组数据库(http://www.rosaceae.org/node/355)。运用在线网站GSDS(http://gsds.cbi.pku.edu.cn/)分析Ⅱ型NADH脱氢酶基因结构,包括外显子、内含子、3′UTR和5′UTR。在GDR数据库下载每个基因起始密码子前的1~2 000 bp,将序列输入Promoter 2.0服务器在线预测Ⅱ型NADH脱氢酶基因启动子。
使用在线工具Multiple EM for Motif Elicitati(MEME, version 4.8.1, http//meme-suite.org/tools/meme)分析每个成员编码蛋白质的保守结构域,并下载LOGO图。参数设置为:保守区域(Motif)数量最多5个,保守区域宽度6~100。
1.5 桃Ⅱ型NADH脱氢酶蛋白质的一级、二级结构预测
利用ExPaSy的Protparam软件(http://expasy.org/tools/protparam.html)预测Ⅱ型NADH脱氢酶蛋白质一级结构及特性,包括氨基酸数目、相对分子质量大小、分子式、总原子数量、稳定性指数、理论等电点、蛋白质亲疏水性等。利用在线工具TMHMM (http://www.cbs.dtu.dk/services/TMHMM/)预测每个蛋白质的跨膜结构域。信号肽预测网站采用https://novopro.cn/tools/signalp.html 和http://www.cbs.dtu.dk/services/SignalP-4.0/。
利用在线工具SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测桃Ⅱ型NADH脱氢酶蛋白质二级结构中α-螺旋、β-折叠和无规则卷曲等在整体结构中所占的比例,窗口宽度设置为17,相似性阈值设置为8。
1.6 桃Ⅱ型NADH脱氢酶基因的表达分析
以各组织cDNA为模板,采用Primer Premier 5.0软件(Premier Biosoft),为每个基因设计实时荧光定量PCR引物(表2),使扩增产物出现在6个基因间的非保守序列,采用SYBR© Premix Ex TaqTM(Tli RNaseH Plus)试剂盒[宝生物工程(大连)有限公司产品]进行荧光定量PCR反应,反应体系和反应程序等参考Zhang等[21]的方法。 使用SPSS20.0 对数据进行差异显著性分析,所有数据以邓肯氏新复极差法进行检验。

表2 桃Ⅱ型NADH脱氢酶基因荧光定量PCR所用引物
2 结果与分析
2.1 桃Ⅱ型NADH脱氢酶家族成员鉴定
根据拟南芥Ⅱ型NADH脱氢酶基因编码的蛋白质检索出6个桃候选Ⅱ型NADH脱氢酶(表3),用Conserved Domain Search软件对其保守结构域进行预测,结果显示,桃有6种Ⅱ型NADH脱氢酶蛋白质。其中,Prupe.6G055600.1、Prupe.4G039000.1、Prupe.3G231400.1只含有1个NADHdehydrogenase-like结构域;Prupe.3G231300.1和Prupe.4G037100.1除了含有NADHdehydrogenase-like结构域外,还含有EF-hand保守结构域,Prupe.3G231300.1氨基酸序列的426~499 aa含有2个EF-hand结构域,Prupe.4G037100.1只含有1个EF-hand结构域,位于390~464 aa;Prupe.5G076700.1氨基酸序列含有1个小的NADH结合结构域,这个小的NADH结合位点被包含在一个更大的、被称作氧化还原辅基的FAD结合结构域里。
2.2 桃Ⅱ型NADH脱氢酶家族基因克隆
从野鸡红叶片中分离出5个基因,根据测序结果和参考基因组序列比对,发现5个Ⅱ型NADH脱氢酶基因与桃参考基因组Lovell品种中5个相应基因的ORF分别完全一致,Prupe.6G055600.1、Prupe.4G039000.1、Prupe.3G231400.1、Prupe.4G037100.1、Prupe.5G076700.1ORF全长分别是1 506 bp、1 653 bp、1 746 bp、1 764 bp、1 620 bp,分别编码501个、550个、581个、587个、539个氨基酸;这些基因分别位于3~6号染色体上(表3)。在多个组织的cDNA文库中,多次设置不同温度梯度和模板浓度,Prupe.3G231300.1都没有被扩增出来(图1)。

a:Prupe.6G055600.1;b:Prupe.3G231400.1;c:Prupe.3G231300.1;d:Prupe.3G231400.1;e:Marker;f:Prupe.4G037100.1;g:Prupe.3G231300.1;h:Prupe.5G076700.1;i:Prupe.4G039000.1;j:RPII。图1 桃6个Ⅱ型NADH脱氢酶家族基因克隆Fig.1 Cloning of type Ⅱ NADH dehydrogenase family genes in peach

表3 桃Ⅱ型NADH脱氢酶家族基因信息
2.3 桃与其他物种Ⅱ型NADH脱氢酶蛋白质系统进化树的构建
为评估桃和其他物种Ⅱ型NADH脱氢酶蛋白质的进化关系,对桃和其他5个物种Ⅱ型NADH脱氢酶家族蛋白质氨基酸序列进行系统进化树构建(图2)。根据不同物种间Ⅱ型NADH脱氢酶家族蛋白质氨基酸序列的相似性和进化关系的远近,特别是根据拟南芥Ⅱ型NADH脱氢酶亚家族蛋白质分类,将桃Ⅱ型NADH脱氢酶蛋白质分成3个亚家族:NDA、NDB、NDC,分别包含2个、3个、1个成员。

图2 桃与其他物种Ⅱ型NADH脱氢酶家族成员系统进化树Fig.2 Phylogenetic tree of type Ⅱ NADH dehydrogenase family members in peach and other species
NDA亚家族中的Prupe.6G055600.1与At1g07180(AtNDA1)和At2g29990(AtNDA2)亲缘关系均较近,同时Prupe.6G055600.1与土豆StPGSC0003DMG400004168(StNDA1,NCBI序列号:CAB52796.1)进化关系也很近,因此,将Prupe.6G055600.1 命名为PpNDA1。Prupe.4G039000.1 命名为PpNDA2。
桃Prupe.3G231400.1与拟南芥At4g28220(AtNDB1)和土豆StPGSC0003DMG400021670(StNDB1)紧密聚在一起,亲缘关系最近,因此,将Prupe.3G231400.1命名为PpNDB1。桃Prupe.3G231300.1与拟南芥At2g20800(AtNDB4)聚在一起,将Prupe.3G231300.1命名为PpNDB4; Prupe.4G037100.1与拟南芥At4g21490(AtNDB3)亲缘关系更近,将Prupe.4G037100.1命名为PpNDB3 (表3)。
桃NDC亚家族中仅含有Prupe.5G076700.1一个成员,其与拟南芥At5g08740(AtNDC1)亲缘关系最近,在同一分支内,因此,Prupe.5G076700.1被命名为PpNDC1(表3)。
桃Ⅱ型NADH脱氢酶家族成员比拟南芥Ⅱ型NADH脱氢酶家族成员少了1个,桃没有与拟南芥AtNDB2相对应的同源基因。
2.4 桃Ⅱ型NADH脱氢酶基因结构、启动子及保守结构域分析
桃Ⅱ型NADH脱氢酶基因结构(图3)显示,NDA亚家族的2个基因均含有8个外显子和7个内含子,而且2个基因之间外显子长度和外显子排列顺序几乎完全相同,但是内含子长度、3′UTR长度和5′UTR长度有很大不同。NDB亚家族的Prupe.3G231400.1(PpNDB1)和Prupe.4G037100.1(PpNDB3)基因均分别含有10个外显子和9个内含子,Prupe.3G231300.1(PpNDB4)含有11个外显子10个内含子,且无上下游3′UTR和5′UTR结构。基因间相比,同一编号的外显子长度几乎相等。Prupe.5G076700.1(PpNDC1)的基因结构与其他亚家族基因不同,没有内含子,由3′UTR、5′UTR和1个长外显子构成。

图3 桃Ⅱ型NADH脱氢酶家族基因结构Fig.3 Gene structure of type Ⅱ NADH dehydrogenase family in peach
桃Ⅱ型NADH脱氢酶家族6个基因的启动子分别位于基因起始密码子前2 000 bp范围的496~546 bp、1 330~1 380 bp、1 771~1 821 bp、1 153~1 203 bp、1 612~1 662 bp、1 678~1 728 bp 处(表4)。

表4 桃Ⅱ型NADH脱氢酶基因启动子分析
保守区域分析结果(图4和图5)显示,每个PpNDA和PpNDB亚家族成员均含有4个相同的保守结构域: motif 1、motif 2、motif 3、motif 5,这4个保守结构域在每个成员的氨基酸序列中出现的先后顺序也相同。PpNDB亚家族的3个成员除了含有上述4个保守结构域外,还含有1个PpNDB亚家族成员特有的保守结构域motif 4。PpNDC亚家族成员只含有保守结构域motif 2和motif 3。
理学作为现代科学技术的基础学科在社会生产生活中有着重要的地位和作用,可“物理是高中最难学的一门课”也已经成为多年来师生们的共识,相当一部分学生因为学不好物理而放弃物理,进而放弃理科选择文科。但对几届学生的调查得知,这些反映高中物理不好学的同学当中有相当一部分在初中其实很喜欢物理,并且成绩也不错。

图4 桃Ⅱ型NADH脱氢酶家族蛋白保守结构域标志Fig.4 Conserved domain markers of type Ⅱ NADH dehydrogenase family proteins in peach

蓝色:motif 1;红色:motif 2 ;桔黄:motif 3;紫色:motif 4;绿色:motif 5 图5 桃Ⅱ型NADH脱氢酶家族蛋白质保守结构域Fig.5 Conserved domain of type Ⅱ NADH dehydrogenase family proteins in peach
2.5 桃Ⅱ型NADH脱氢酶蛋白质一级、二级结构预测
桃Ⅱ型NADH脱氢酶蛋白质一级结构预测结果(表5)表明,桃Ⅱ型NADH脱氢酶蛋白质氨基酸数目介于501~621 aa,相对分子质量介于55 340~70 070;而且发现 NDC亚家族和PpNDA亚家族成员的氨基酸数目和相对分子质量小于PpNDB亚家族成员。
Prupe.3G231400.1(PpNDB1)和Prupe.5G076700.1 (PpNDC1)理论等电点值均小于7,说明是酸性蛋白质;其余4个蛋白质的理论等电点均大于7,是碱性蛋白质。6个蛋白质的不稳定指数介于31.58~39.18,均属于稳定蛋白质。6个桃Ⅱ型NADH脱氢酶蛋白质亲水性指数平均值介于-0.135~-0.308,均是亲水性蛋白质(表5)。
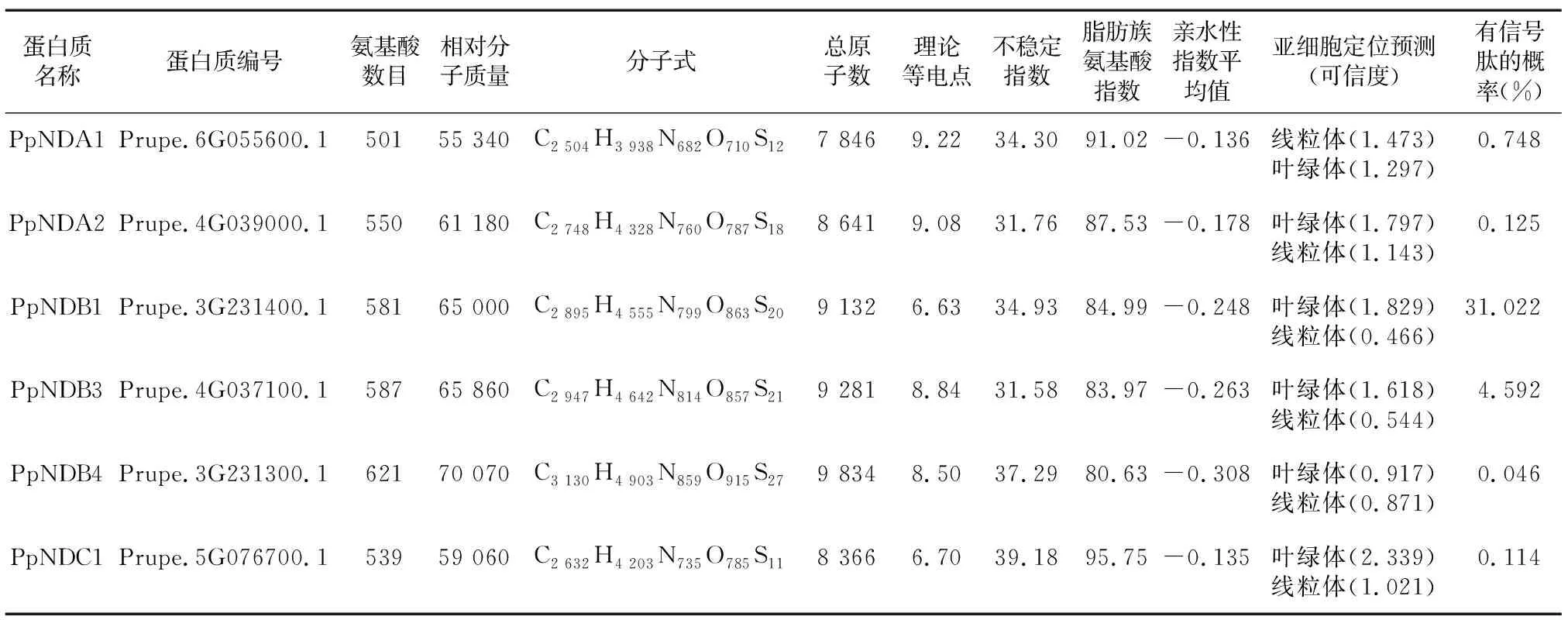
表5 桃Ⅱ型NADH脱氢酶家族蛋白质一级结构分析
亚细胞定位预测结果(表5)显示,6个桃Ⅱ型NADH脱氢酶蛋白质大概率位于线粒体或叶绿体;跨膜结构域预测结果表明,6个蛋白质均无跨膜结构域;6个蛋白质Prupe.6G055600.1、Prupe.4G039000.1、Prupe.3G231400.1、Prupe.4G037100.1、Prupe.3G231300.1、Prupe.5G076700.1有信号肽的概率分别是 0.748%、0.125%、31.022%、4.592%、0.046%、0.114%。
利用SOPMA程序对6个桃Ⅱ型NADH脱氢酶家族蛋白质的二级结构进行预测,结果(表6、图6)显示,PpNDB亚族成员蛋白质的二级结构全部以α-螺旋为主要构成元件,其次是无规则卷曲,β-转角所占比例最低。而PpNDA和PpNDC亚族成员蛋白质的二级结构中占比最高的是无规则卷曲,其次是α-螺旋,β-转角所占比例最低。

表6 桃Ⅱ型NADH脱氢酶家族蛋白质二级结构分析

蓝色:α-螺旋;红色:β-折叠;绿色:β-转角;紫色:无规则卷曲。图6 桃Ⅱ型NADH脱氢酶家族蛋白质二级结构Fig.6 Secondary structure of type Ⅱ NADH dehydrogenase family proteins in peach
2.6 桃Ⅱ型NADH脱氢酶基因表达水平分析
以RPII为看家基因,以叶芽表达量为1,其他7个组织相对于叶芽组织表达量的相对水平进行计算,桃Ⅱ型NADH脱氢酶家族基因在8个组织中的相对表达水平分析结果(图7)表明,Prupe.6G055600.1(PpNDA1)在熟果肉中表达量显著高于老叶、枝表皮和其他组织;Prupe.4G039000.1(PpNDA2) 在根中表达量最高,其次是老叶,再次是叶芽,在果肉中表达量最低;Prupe.3G231400.1(PpNDB1)在老叶和幼果肉中表达量最高,其次是根,再次是叶芽和幼叶,在熟果肉中几乎无表达;Prupe.4G037100.1(PpNDB3)在老叶和根中表达量显著高于其他组织,在熟果肉中几乎不表达,在枝表皮、幼果肉、熟果皮表达量相近。Prupe.3G231300.1(PpNDB4)在枝表皮和熟果肉中检测不到表达,在幼叶和根中表达量显著高于其他组织,在熟果皮、幼果肉、叶芽中表达量相近,没有显著差异;Prupe.5G076700.1(PpNDC1)在老叶中表达量最高,其次是叶芽、幼果肉、枝表皮,再次是幼叶和根,在熟果肉中几乎检测不到表达。

图7 桃Ⅱ型NADH脱氢酶家族基因相对表达水平Fig.7 Relative expression level of type type Ⅱ NADH dehydrogenase family genes in peach
3 讨 论
3.1 桃Ⅱ型NADH脱氢酶家族成员鉴定
本研究鉴定出桃有6个桃Ⅱ型NADH脱氢酶成员,比拟南芥少了一个AtNDB2成员,这表明拟南芥与桃Ⅱ型NADH脱氢酶家族成员数目方面稍有差异,也有很高的保守性;这种物种间的保守性和差异性在其他植物中也被发现,水稻有8个Ⅱ型NADH脱氢酶成员。这在一定程度上验证了以往报道:每种植物线粒体一定程度上至少含有4个不同的Ⅱ型NADH脱氢酶[9]。综合这些研究结果,也说明植物Ⅱ型NADH脱氢酶家族成员数目较少,是较小的家族。
桃Ⅱ型NADH脱氢酶家族被分成3个亚家族,其他植物Ⅱ型NADH脱氢酶家族也是被分为3个亚家族[22],这体现了同一家族基因在不同物种间的保守性。特别是PpNDC亚家族,有且只有1个成员PpNDC1,在拟南芥、水稻、白腐菌及本研究中都得到了证实[22]。
3.2 桃Ⅱ型NADH脱氢酶家族基因克隆
从野鸡红的8种组织cDNA池中均无法扩增出PpNDB4(Prupe.3G231300.1)条带,经过在NCBI中查找该基因的基因组序列,发现与GDR中下载的基因组序列不同,有待进一步研究;但是根据参考基因组Lovell品种中Prupe.3G231300.1的CDS序列设计的qRT-PCR引物能扩增出127 bp目的片段,其表达水平在一些组织中也能被检测到。这与以往拟南芥中发现的类似:AtNDB3无法克隆,并被认为是一个假基因[23]。在萌发过程中的转录组分析结果表明,AtNDB3在萌发早期表达[24]。假基因是基因家族在进化过程中形成的,往往存在于真核生物的多基因家族中。例如,大多数光合作用的基因已经丢失或消失,只保留为假基因[25]。由于假基因在选择压力降低的条件下进化,可以推测出它们的种间变异性增加,因此,这些假基因可能是分类学研究中有用的分子工具[25]。这是首次报道在桃中发现假基因,Prupe.3G231300.1的利用有待进一步深入探索。
3.3 桃Ⅱ型NADH脱氢酶家族基因结构及蛋白质特性
基因结构方面,桃PpNDA亚家族2个基因的外显子数量相同,各8个,这与拟南芥AtNDA亚家族2个基因的外显子数量相等[9]。说明2个物种间NDA亚家族基因在外显子数量上是保守的。比较桃NDA亚家族的2个基因,发现外显子长度和外显子排列顺序、内含子排列顺序几乎完全相同,但内含子长度、上下游长度有很大不同。比较拟南芥NDA亚家族的2个基因,所有内含子的位置、大多数内含子长度都是保守的[9],这说明桃和拟南芥2个物种的NDA亚家族成员基因结构高度保守,除了2个物种间内含子长度略有不同。桃NDB亚家族3个基因的外显子位置、长度、数目、顺序高度保守,内含子长度有差异;比较拟南芥NDB亚家族4个基因,大多数内含子位置保守,和NDB1及NDB2相比,NDB4丢失了几个内含子,这些体现了2个物种间虽然略有差异,但是具有高度保守性。这些基因结构上的保守性也为进化分析结果提供了支持。NDC亚家族唯一成员拟南芥NDC1有9个内含子,而桃NDC亚家族唯一成员NDC1(Prupe.5G076700.1)没有内含子,这可能是自然选择的结果,这种选择是由最小化转录成本所产生的优势驱动的,因为内含子较短,可以减少转录和剪接等过程的能量消耗,从而提高转录效率[26]。
PpNDC1(Prupe.5G076700.1)与拟南芥 AtNDC1(AT5G08740)均是 NDC家族唯一蛋白质,两者的氨基酸序列均含有1个小的NADH 结合结构域,这个小的NADH结合位点被包含在一个更大的、被称作氧化还原辅基的FAD结合结构域里。说明物种间同源基因结构上具有高度保守性,可能起源于同一个祖先。
EF-hand是1个由α-螺旋“E”(一个可能结合钙的环)和第二个α-螺旋“F”组成的基序[27]。含有EF-hand保守区域的蛋白质主要有2类:钙离子传感器,可传递钙离子信号;钙离子信号调节剂,可调节钙离子信号的形状和/或持续时间,或参与钙离子稳态[28]。PpNDB亚家族3个成员中有2个成员含有EF-hand保守结构域,而且PpNDB4(Prupe.3G231300.1)含有2个紧紧相邻的EF-hand结构域,PpNDB3(Prupe.4G037100.1)在390~464 aa含有1个EF-hand结构域且是钙离子结合位点,这与以往报道相同,植物NDB蛋白含有1个EF-hand保守区域,大约70个氨基酸残基长度,它能结合钙从而保证活性,在烟草中已得到证实,NDB活性依赖于钙[4,29]。这间接说明含有EF-hand结构域的氨基酸序列有共同的祖先,一个家族的成员间是同源基因。PpNDB4(Prupe.3G231300.1)蛋白质含有2个紧邻的EF-hand结构域,这与以往报道的EF-hand保守结构域经常成对出现的结论一致[27]。含有EF-hand保守结构域的蛋白质具有一系列多种功能[28],有待在桃中进一步探索和挖掘。
桃NDC1(Prupe.5G076700.1)有1个包含在大的FAD结构域里面的1个小NADH结合结构域,相对分子质量是59 056。这与以往报道相同:含有1个FAD结构域的典型的Ⅱ型NADH脱氢酶,相对分子质量大小是50 000~60 000[2]。拟南芥中也有1个具有FAD结构域的典型的Ⅱ型NADH脱氢酶蛋白质NDC1(AT5G08740.1),相对分子质量是57 018,也介于50 000~60 000。大肠杆菌(Escherichiacoli)NDH和酿酒酵母(Saccharomycescerevisiae)NDI1也分别是包含FAD的单个多肽Ⅱ型NADH脱氢酶蛋白质,相对分子质量是45 000~50 000[9]。这些说明NDC亚家族中的NDC1相对分子质量在植物、细菌、酵母等生物中是不同的,在植物中含有1个FAD结构域的典型的Ⅱ型NADH脱氢酶蛋白质NDC1,相对分子质量符合50 000~60 000的规律[2],在细菌和酵母等生物中NDC相对分子质量范围需要进一步确定和总结。这可能与物种的基因组大小相关,植物基因组比细菌、酵母等基因组大。
3.4 桃Ⅱ型NADH脱氢酶家族蛋白质亚细胞定位预测
最初的体内GFP融合方法确定拟南芥NDA1、NDA2、NDB1、NDB2、NDC1蛋白质都位于线粒体中[9]。Elhafez等[23]通过对分离线粒体进行体外导入分析后确定NDB1、NDB2和NDB4定位于线粒体内膜的外部,NDA1、NDA2和NDC1定位在线粒体内膜的内部。而NDB3因无法克隆,亚细胞定位未知。然而,随后的研究发现,拟南芥Ⅱ型NADH脱氢酶蛋白质事实上是双重定位的,NDA1、NDA2和NDB1被发现定位在线粒体和过氧化物酶体,NDC1被确定定位在线粒体和质体[30]。这些研究得出的结论不同,是因为最初的研究仅使用目标蛋白质的C端GFP标签加上N端前50~100个氨基酸构建载体[22]。
为了得到确定结论,Xu等[22]2013年通过AtNDA1、AtNDA2基因ORF全长C端融合表达,确定AtNDA1和AtNDA2表达的蛋白质定位在线粒体中。如果只把编码AtNDA1、AtNDA2蛋白质最后10个氨基酸的基因融合表达,则发现AtNDA1、AtNDA2表达的蛋白质定位在过氧化物酶体。OsNDA1基因ORF全长C端融合表达,确定OsNDA1表达的蛋白质定位在线粒体中,如果只把编码OsNDA1蛋白质的最后10个氨基酸的基因融合表达,则发现OsNDA1表达的蛋白质不定位在任何位置,OsNDA2表达的蛋白质定位在线粒体和过氧化物酶体;AtNDB1表达的蛋白质定位在线粒体和过氧化物酶体,AtNDB2和AtNDB4表达的蛋白质均只定位在线粒体;OsNDB1和OsNDB2基因ORF全长C端融合表达,确定OsNDB1和OsNDB2表达的蛋白质定位在线粒体中, 如果只把编码OsNDB1和OsNDB2蛋白质最后10个氨基酸的基因融合表达,则OsNDB1和OsNDB2表达的蛋白质定位在过氧化物酶体;AtNDC1和OsNDC1表达的蛋白质均定位在线粒体和质体。在植物中,线粒体和质体的功能也对各种发育和环境作出反应,但有关信号的细节仍然未知;质体功能可以影响线粒体的生物发生和活性[31]。光合作用缺陷细胞可改变线粒体基因在叶中表达和基因组拷贝数,但不影响在根中的表达和基因组拷贝数,这在大麦中较为明显[32]。
本研究中6个Ⅱ型NADH脱氢酶蛋白质被预测定位在线粒体或叶绿体的概率较高,这与其他物种(拟南芥和水稻)C端融合表达确定的亚细胞定位结果[22]既有相同之处,又有差异。PpNDA1被预测的最高可信度的亚细胞定位在线粒体,这与已验证的拟南芥AtNDA1[9]、莱氏衣藻CrNDA1[18]、土豆StNDA1[11]的亚细胞定位结果相同。莱氏衣藻CrNDA2和CrNDA3定位于叶绿体,CrNDA1、CrNDA5、CrNDA6、CrNDA7亚细胞定位结果目前还未知[33];AtNDB2和StNDB1位于线粒体。下一步将对桃Ⅱ型NADH脱氢酶蛋白质亚细胞定位进行试验确定。
3.5 桃Ⅱ型NADH脱氢酶家族基因表达水平
基因表达水平分析结果显示,桃NDB亚家族的3个基因均在老叶、幼叶、根中表达量更高,在熟果肉、熟果皮和枝表皮中表达量相对低或检测不到,说明同一亚家族成员之间不仅在基因结构、蛋白质保守区域、蛋白质理化性质方面具有保守性,在行使的功能方面也可能具有保守性,且在成熟果实中行使的功能少或只在特定条件下的成熟果实中才具有功能。除了Prupe.3G231300.1(PpNDB4) 外,其余5个桃Ⅱ型NADH脱氢酶基因在老叶中的表达水平均分别显著高于其在幼叶中的表达水平,据此推测5个桃Ⅱ型NADH脱氢酶基因在成熟的、深绿的老叶中行使的功能可能强于其在浅绿的嫩叶中行使的功能。Prupe.4G039000.1(PpNDA2)、Prupe.4G037100.1(PpNDB3)、Prupe.3G231300.1(PpNDB4)在白色幼根中表达量较高,且显著高于其在叶芽、枝表皮、幼果肉、熟果肉、熟果皮中的表达水平,据此推测这3个Ⅱ型NADH脱氢酶基因可能在根生长、发育等方面具有重要作用。有研究结果表明,通过线粒体呼吸链中的非磷酸化途径氧化NADH在陆地植物和湿地植物中都很常见;在湿地植物的根部,这些特性尤为显著,所以它们可以在长期缺氧和缺氧胁迫下生存[34-35]。将来可以进一步从Ⅱ型NADH脱氢酶基因入手,解析其在桃树抗涝等方面的分子作用机理。
6个桃Ⅱ型NADH脱氢酶成员的系统性分析为未来基因功能研究提供了重要方向和依据。下一步将通过转基因、亚细胞定位、蛋白质互作等试验验证基因功能,以期明确桃Ⅱ型NADH脱氢酶基因在调控光呼吸、适应生物和非生物逆境(如冷害、涝害等)及其他代谢途径等过程的具体作用机理。
