新一代诊断技术:CRISPR 系统及其在分子诊断中的应用
2022-08-17孙秀兰付旭冉孙嘉笛
孙秀兰, 付旭冉, 鲍 琦, 孙嘉笛
(江南大学 食品学院,江苏 无锡 214122)
分子诊断是通过核酸识别和基因编辑对遗传物质的变化做出诊断的技术[1],在生物医药、环境监测、食品安全防控等领域起着至关重要的作用[2]。 目前,最常用的技术主要有两大类,第一类是以聚合酶链式反应(polymerase chain reaction,PCR)为代表的核酸检测技术,其常作为分子诊断的“金标准”,具有高灵敏度和可靠性,但对设备条件要求高且具有环境依赖性, 基于PCR 方法建立的恒温扩增、基因测序、生物芯片等技术也得到了广泛应用,但这些方法依旧存在技术复杂、成本高、特异性差等问题。 酶 联 免 疫 吸 附 测 定 (enzyme -linked immunosorbent assay,ELISA) 因其快速和高特异性的优势,已经成为了蛋白质和小分子广泛应用的诊断工具,然而,样品前处理的复杂性以及较低的灵敏度限制了其在现场快速精准检测中的应用。
世 界 卫 生 组 织 (World Health Organization,WHO)发布了理想诊断产品的标准ASSURED[3],即为可负担(affordable)、灵敏(sentitive)、特异(specific)、用 户 友 好(user-friendly)、快 速 而 可 靠(rapid and reliable)、免设备(equipment-free)且可交付给最终用户(deliverable to end users)[4]。CRISPR/Cas 系统由于优异的生物传感性能逐渐成为下一代诊断技术的焦点,有望成为开发理想诊断产品的方法。 基于CRISPR/Cas 系统的特异性结合活性和高效反式切割能力,将其与电化学、荧光传感技术相结合,已经成功实现了各种靶标分子的高特异、 高灵敏检测,检测对象也已经从最初的核酸扩展到金属离子、蛋白质、细菌、小分子等,说明了CRISPR/Cas 系统在检测领域具有通用性。 本文中作者重点介绍了两类CRISPR/Cas 系统的特征和机制, 并总结了基于CRISPR/Cas 系统的各种新型生物传感器,列举了针对核酸靶标及蛋白质、生物小分子等非核酸靶标构建的CRISPR/Cas 新型生物传感器, 最后对该领域的发展提出了一些未来展望,以期为进一步开发新型分子诊断技术提供参考。
1 基因编辑及CRISPR/Cas 系统概述
基因编辑是分子诊断技术的基础,目前基因编辑主要有三大利器:锌指核酸酶(zinc-finger nuclease,ZFN)、 转 录 激 活 样 效 应 因 子 核 酸 酶(transcription activator-like effector nuclease,TALEN)和成簇规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)与其蛋白质(CRISPR associated,Cas)共同组成的CRISPR/Cas 系统[5]。 ZFN 是人工修饰的核酸酶,通常由两部分组成,一部分是能够实现特异性识别锌指(zinc-finger,ZF)的DNA 结合域,一般包含3 个ZF 重复结构, 每个ZF 能识别3 个碱基;另一部分是包含非特异性限制性核酸内切酶的DNA切割区域,对DNA 进行特异性切割,形成双链断裂区,以非同源末端连接(non-homologous end joining,NHEJ)的方式使目的基因失活,或借助同源重组(homologous recombination,HR)方式黏合断裂的DNA,从而修复DNA。 其主要应用于科研和医疗领域, 如治疗杜氏肌营养不良症和21 三体综合征等遗传疾病[6]。
TALEN 最初来源于黄单胞菌, 完整的TALEN由包含定位信号的N 结构域、 可识别特定靶DNA的TALE 重复序列以及FoKI 核酸内切酶组成。天然TALEN 元件识别的序列长度为17~18 bp[7],主要是通过将TALEN 靶向并结合DNA 位点, 在FoKI 核酸内切酶的作用下实现对特定点位的剪切。 TALEN技术目前已在全球范围内广泛用于靶向基因编辑,在医疗和动植物育种领域有广泛应用前景。
上述两种酶的作用机制都是通过对双链DNA(double stranded DNA,dsDNA)进行识别和切割, 激活细胞的非同源末端修复或者同源重组修复,依赖于DNA 序列与蛋白质的特异性结合,而这一步骤非常的费时且烦琐, 具有很强的设备依赖性。 CRISPR/Cas 系统通过特异性向导RNA(guide RNA,gRNA)指导核酸内切酶结合靶点,从而实现更简单高效的基因编辑。 不同种类的酶对应的靶点类型不同,包括dsDNA、单链DNA(single stranded DNA,ssDNA)和RNA 等。与其他两种基因编辑系统相比,CRISPR/Cas 系统具有显著优势[8]:1)载体构建简单且靶向效率高,只需要构建一个具有几十个碱基的CRISPR sgRNA 即可与DNA 序列进行匹配,从而介导Cas 效应子对DNA 序列进行切割;2)CRISPR/Cas 系统可编辑的位点分布频率较高,易选择合适的位点进行基因编辑;3)CRISPR/Cas 系统可同时对基因组进行多位点编辑。
简而言之,TALEN 和ZFN 虽然精确度高和可控性强, 但CRISPR/Cas 系统因操作简单和功能强大,在分子诊断及生物传感领域,尤其是现场即时检测(point-of-care testing,POCT)领域有着更为巨 大的潜力,三者的对比见表1[9]。
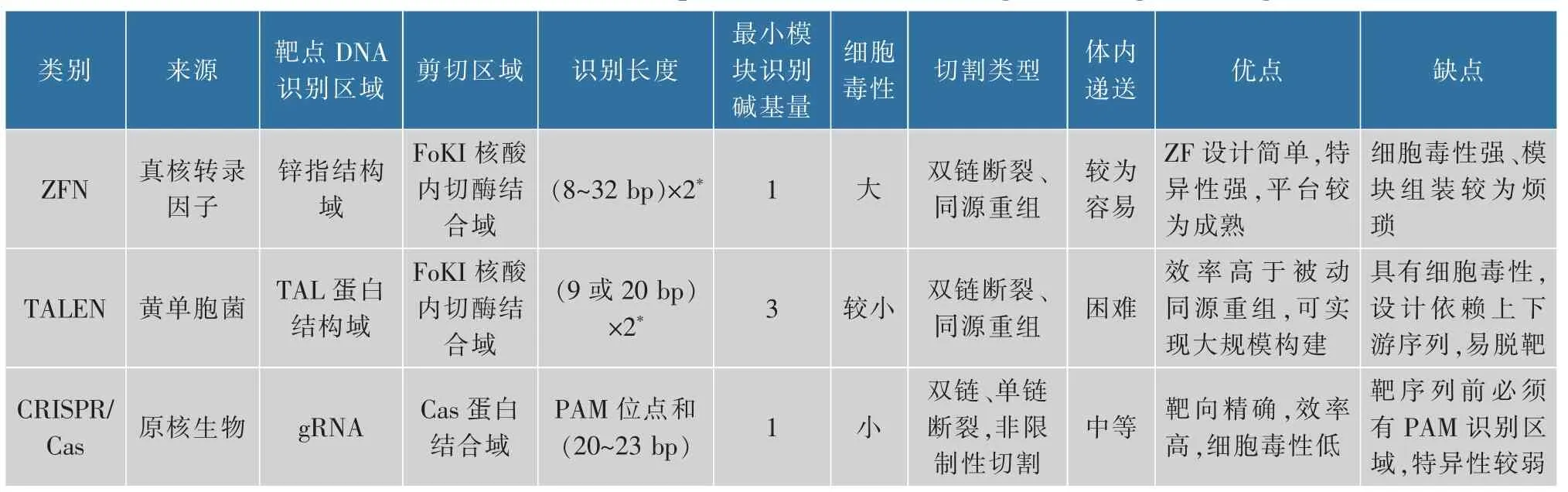
表1 3种常见基因编辑技术的特点及比较Table 1 Characteristics and comparison of three common gene editing technologies
2 CRISPR 系统的分类及特点
CRISPR/Cas 系统依据Cas 效应子的不同可以分为6 大类和32 个亚型。 1 类CRISPR 系统主要通过多个蛋白质协同作用来激活切割活性, 包括Ⅰ型、Ⅲ型和Ⅳ型,效率较低且操作复杂,研究和应用较少;2 类CRISPR 系统的Cas 效应蛋白通常只有一个蛋白质,包括Ⅱ型、Ⅴ型和Ⅵ型[10]。 2 类CRISPR系统作用机制主要包括3 个阶段:第一个阶段是识别可变间隔区,当识别到外来物质时,前间区序列邻近基序(protospacer adjacent motif,PAM)识别外源DNA 片段并整合到宿主CRISPR 基因的两个重复序列之间,作为间隔序列[11];第二阶段是Cas 酶结合gRNA 形成二元复合物, 从而将gRNA 中的CRISPR 引导至DNA 分子上的前间隔序列;第三阶段是gRNA 与前间隔序列配对形成Cas-gRNAPAM 三元复合物[12],特异性识别外源核酸片段,Cas酶被激活, 顺式切割外源核酸片段。 由于2 类CRISPR 系统只需要一个效应蛋白就可以实现对靶核酸位点的切割,操作简单、靶向精确,逐渐成为分子诊断领域的热点, 已发现的2 类CRISPR 系统效应蛋白Cas9、Cas12、Cas13 和Cas14 都有了广泛应用,且具有不同特点,其主要区别见表2。

表2 2 类CRISPR 系统中主要Cas 效应蛋白比较Table 2 Comparison of major Cas effector proteins in class 2 CRISPR system
2.1 Cas9
Cas9 是一种双gRNA 的II 型Cas 效应子,其gRNA 包含一个crRNA 和一个tracrRNA。 crRNA 的3′端与tracrRNA 配对,crRNA 的5′端是游离的靶标序列[13]。 当外源dsDNA 片段进入时,Cas9 结合tracrRNA 形成复合体,然后将crRNA 引导至其与靶标序列的前间隔序列结合,而这个结合取决于前间隔序列和PAM。 与其他II 型CRISPR 系统不同。Cas9 中有两部分起识别作用的结构域, 分别是HNH 结构域和RuvC 结构域[14]。以含有PAM 序列的dsDNA 作为催化底物,在gRNA 的指导下,以PAM位点介导, 使得Cas9 的HNH 结构域和RuvC 结构域分别切割靶标链和非靶标链 (与靶标链互补的链),从而对目的基因进行编辑[1,15]。
2.2 Cas12
Cas12 和Cas9 结 构 和 功 能 相 似,Cas9 和Cas12b 的gRNA 包 括crRNA 和tracrRNA[16],而Cas12a 的gRNA 只包括crRNA, 且只包含RuvC 单个核酸内切酶结构域。Cas12a 在crRNA 介导下特异性识别靶DNA 序列, 结合后触发Cas12a 的构象变化, 导致RuvC 的活性位点暴露, 特异性识别靶标dsDNA, 激活其特异性顺式切割活性和非特异性反式切割活性[17],形成黏性末端并非特异性反式切割 任意ssDNA[18]。 Cas12 中完整的crRNA 长度一般为42~44 nt[16],一般包含19 nt 的重复序列和23~25 nt 的间隔区,其中第19 个U 碱基是严格保守的[15]。
2.3 Cas13a
与2 类中的其他Cas 效应子相比,Cas13a 对单链RNA(single stranded RNA,ssRNA)具有顺式和反式切割活性[19]。 Cas13a 含有两个HEPN 结构域,在crRNA 的引导下,特异性切割带有互补间隔序列的ssRNA。 当Cas13a 结合到ssRNA 靶标后,Cas13a 蛋白被激活并转化为非特异性内切酶,对附近的非靶标ssRNA 具有反式切割活性。Cas13a 的前间隔序列一般为20 nt[20],一般没有特定的切割位点,但显示出对U 碱基的切割偏好。
2.4 Cas14
相较于其他典型的2 类蛋白,Cas14 效应子相对分子质量较小, 只有大约400~700 个氨基酸,包含一个与Cas12a 类似的RuvC 区域[21],通过与crRNA 和tracrRNA 结合切割靶标DNA。 在体外检测中,Cas14 只能识别和切割ssDNA,但其核酸内切酶活性的激发不需要依赖于PAM 序列[22],此外,Cas14在与靶序列结合后也可非特异切割任意ssDNA。
Cas 效应子可以特异性结合靶核酸, 以20~30 nt 的gRNA 为“向导者”。而PAM 序列识别是Cas 效应子检测dsDNA 目标的先决条件[19,23-24],与gRNA互补的ssDNA 也可以被Cas12a 切割, 并且以不依赖于PAM 序列的方式激活Cas12a 的反式切割活性[12]。对于ssRNA 检测,则需要一个3′PFS(非G,相当于PAM 序 列)来 激 活Cas13a 的 切 割 活 性[13,25]。 对 于Cas9、dCas9 和Cas12 来说,gRNA 和靶标核酸的复合物对PAM 序列附近的10 个碱基对非常敏感[19,24,26-27],而Cas13a 则对中心碱基对敏感[13,28]。 当碱基对错配发生在中心碱基(即为敏感区域)时,Cas 效应子的切割效率会显著降低,从而可以用于进行单碱基错配检测。
3 CRISPR 传感技术在分子诊断中的应用
在分子诊断方面,Cas 效应子的切割活性(包括顺式切割和反式切割) 引起了广泛关注与研究,尤其是Cas 效应子的反式切割活性[12,29-31]。报告分子被Cas 效应子切割后, 检测体系将发生显著的信号变化(如电化学或者光学信号)。 基于此,CRISPR/Cas系统成功实现了对包括核酸靶标和非核酸靶标在内的多种生物分子的检测。 各种基于CRISPR/Cas生物传感系统的总结如图1 所示。

图1 基于CRISPR/Cas 的生物传感系统示意图Fig. 1 Schematic representation of CRISPR/Cas-based systems for biosensing
3.1 脱氧核糖核酸检测
单核苷酸多态性 (single nucleotide polymorphism,SNP) 与全基因组关联鉴定的复杂性状和疾病相关,是人类基因组中最常见的变异之一[32]。 Li 等建立了基于LbCas12a 的低成本高效检测系统,被称为一种单小时低成本多用途高效体系(an onehour low-cost multipurpose highly efficient system,HOLMES)用于检测人类SNP。 该体系通过PCR 扩增靶DNA,并通过设计的引物引入PAM 序列,得到的dsDNA 产物会与Cas12a-crRNA 组装形成三元复合物, 切割标记有荧光基团和猝灭剂的ssDNA 探针,从而导致荧光基团与猝灭剂分离,并产生显著增加的荧光信号。 HOLMES 可以快速精准地检测人类SNP,并高特异性区分纯合和杂合基因型[18]。 Li等为了简化操作程序,还构建了基于AsCas12b 的HOLMESv2 系统, 实现了对SNP 和DNA 甲基化的一步核酸检测[20]。
人乳头瘤病毒 (human papillomavirus,HPV)是一种dsDNA 病毒, 与多种恶性肿瘤和癌症有关[33]。70%的宫颈癌由HPV16 和HPV 18 引起[34]。 目前市售DNA 试剂盒不能有效地识别所有致癌的人乳头瘤病毒类型。 Chen 等建立了基于Cas12a 的DNA 内切酶靶向CRISPR 反式切割报告方法(DNA endonuclease -targeted CRISPR transreporter,DETECTR),该方法以LbCas12a 为效应蛋白,采用RPA 等温扩增构建了通用型检测平台。RPA 的优势在于可在37 ℃的条件下快速进行指数扩增, 减少了对设备的依赖性。 该检测平台可以准确地区分HPV16 和HPV18,灵敏度可以达到amol/L 级别。 此外该平台还可以用于检测环境DNA,可成功区分出Salmo salar和Salmo trutta线粒体基因重组质粒,基因组检测的灵敏度为1×10-5ng/μL[12]。 Harrington 等基于Cas14 相关特性,构建了Cas14-DETECTR。 因Cas14 靶向ssDNA,为扩增产生ssDNA,在扩增时,将一条引物进行硫代磷酸酯修饰, 扩增完成后,未被硫代磷酸酯修饰的链会被T7 核酸酶降解, 只保留ssDNA 靶序列[30]。 Cas14 具有更高的特异性和更低的脱靶率, 且可以识别没有PAM 序列的外源DNA,被广泛应用于SNP 检测,例如,目前Cas14 已被成功用于眼球颜色相关基因HERC2的SNP 检测[35]。
非洲猪瘟病毒(African swine fever virus,ASFV)是一种大型有包膜的dsDNA 病毒[36],会导致非洲猪瘟病(African swine fever,ASF)。 ASF 是一种严重的出血性急性传染病, 在家猪和野猪中死亡率接近100%。 Wang 等开发了一种CRISPR/Cas9 介导的侧向流动核酸测定法 (CRISPR/Cas9-mediated lateral flow nucleic acid assay,CASLFA)检测ASFV。 通过重新设计sgRNA 并引入额外的茎环,以实现金纳米粒子(gold nanoparticles,AuNPs)-DNA 探针的结合。靶DNA 在PCR 或RPA 扩增过程中首先被生物素化并与Cas9-sgRNA 二元复合物预孵育。 当检测液添加到横向流动试纸条后, 所得产物与AuNPs-DNA 探针杂交,在结合垫上形成生物素化的扩增子Cas9-sgRNA-AuNPs-DNA 复合物,该复合物被预先包被的链霉亲和素捕获。当有ASFV 存在时,金纳米粒子便会在检测线上聚集,该方法在40 min 内可以完成检测, 检出限为4 copies/mL, 与PCR 检测ASFV 的准确度相当, 说明CRISPR/Cas9 在现场即时检测中具有极强的潜力[37]。 为了进一步提高灵敏度,Wang 等研究了一种CRISPR/Cas12a 横向流动检测方案。扩增的ASFV 激活Cas12a 以切割标有地高辛和生物素的ssDNA 报告基因。 地高辛标记的ssDNA 可以与样品垫上的AuNPs-地高辛抗体复合物结合, 总检测时间为60 min, 可检测低至0.4 copies/mL 的靶DNA[38]。Fu 等将基于Cas12a、RPA 和荧光团猝灭剂(FQ)标记的ssDNA 相结合,用于快速可视化检测AFSV。基于Cas12a 设计的5种crRNAs均显示出显著荧光强度的特异性。 在20 min 内,2 copies DNA 的初始浓度下,在实验组和阴性组之间产生显著差异,该方法具有较高灵敏度且反应时间短[39]。
食品安全问题与人们身体健康息息相关,为有效防控食品安全风险,开发特异、灵敏、快速的食源性致病菌、转基因作物和肉类掺假的核酸检测技术势在必行。Liu 等利用Cas12a 的非特异性ssDNA 切割特性,结合RPA 设计了一种食品安全通用检测平台,称为RPA-Cas12a-FS[40]。CaMV35S启动子广泛用于转基因作物,如转基因玉米、转基因大豆[41]。 目前消费者对转基因生物的接受度仍然很低,一些国家已经强制要求商家在转基因成分上标注阈值[42]。Wu 等通过Cas12a 系统和设计的反应容器,建立了一种超快速和方便的可视化检测CaMV35S启动子的方法[43]。 通过LAMP 和PCR 分别与Cas12a 结合,在大豆粉中可检测到低至0.05%(质量分数)的转基因含量,该方法设计的反应容器有可能成为基于仪器的超高灵敏度方法的补充,并为现场快速检测提供新的解决方案[43]。
食源性致病菌引起食源性疾病,对人们的身体健康造成了极大威胁。 Zhang 等基于Cas12a 系统,建立了一种新颖、 特异和可视化的核酸检测方法,在虾样品中对副溶血性弧菌的检出限为1.02×102copies/μL[44]。大肠杆菌可能导致出血性结肠炎、血性腹泻和肾衰竭等[45],也可以感染水、果汁、牛奶、水果和蔬菜[46]。 Sun 等研究了一种Cas9 触发的两步等温扩增方法,用于检测大肠杆菌O157:H7。 该方法使用金属有机框架UiO66 作为荧光猝灭剂, 在目标dsDNA 存在的情况下,Cas9-sgRNA 复合物被激活,在dsDNA 的非靶标链上引入两个DNA 断裂, 触发新链的合成和延伸, 然后通过RCA 反应产生包含靶标的长ssDNA 与DNA 探针杂交, 从而恢复荧光信号,该方法能够以高特异性检测40 CFU/mL 的大肠杆菌O157:H7[47]。 全球抗生素耐药性以及食品和环境污染的发生率持续增加,特别是由病毒和多重耐药细菌引起的感染。 多重耐药金黄色葡萄球菌(multidrug-resistantStaphylococcus aureus,MRSA)是最重要的多重耐药病原体之一,对多种抗生素耐药。 MRSA 感染导致严重发病率和死亡率的风险极高[48]。 Guk 等构建了一种简便、灵敏和快速的方法,称为CRISPR 介导的DNA 荧光原位杂交(FISH),用于检测MRSA。 该方法中将dCas9-sgRNA 复合物用作识别元件,SYBR Green I 用作dsDNA 染色的报告分子,sgRNA 的易编程性使得dCas9-sgRNA 能够选择性地与MRSA 基因结合,该方法能够检测低至10 CFU/mL 的MRSA 裂解物[49]。Curti 等应用Cas12a来识别与碳青霉烯类耐药基因相对应的靶序列,如blaKPC、blaNDM和blaOXA。 通过无标记阻抗分析,检测时间缩短至60 min 以内,且结果与qPCR 的结果相当,并通过便携式试纸条进行了验证[50]。
3.2 核糖核酸检测
Cas13a 在相应的crRNA 的引导下,可特异性识别RNA,因此多通过Cas13 构建生物传感系统以实现核糖核酸的检测。有研究者利用Cas13a 的反式切割活性, 构建了一种基于Cas13a 的生物传感平台,称为特异性高灵敏度酶报告系统 (specific highsensitivity enzymatic reporter unlocking,SHERLOCK)。通过逆转录重组酶聚合酶扩增(reverse transcription recombinase polymerase amplification,RT-RPA)技术扩增RNA 或RPA 扩增dsDNA, 再经过T7 转录生成RNA 产物,用于激活Cas13a 以产生荧光信号[51]。SHERLOCK 能够在患者血清或尿液样本中检测到寨卡病毒和登革热病毒,并区分不同地区的寨卡病毒。 第1 代SHERLOCK 存在一定的局限性,无法达到便携、多重检测和高灵敏度的性能要求[52]。为了解决这些问题, 张锋所在团队再次构建了SHERLOCKversion2 系统(SHERLOCKv2)[35],利用不同的Cas 酶表现出不同程度的“偏爱”切割特性,同时将多种不同特异性荧光探针引入检测系统,进行多种核酸检测,可在1 份样本中同时检测多种病毒[52]。SHERLOCKv2 还利用侧流层析方法实现了快速检测,当Cas13a 切割RNA 荧光探针时,探针两端连接的6-羧基荧光素(6-carboxyfluorescein,FAM)与生物素分别结合到试纸条的不同区域, 在2 h 内即可将结果直观地展现在试纸条上,摆脱了检测体系对荧光读数仪器的依赖,该方法可通过冻干试剂在室温使用,从而实现了现场即时检测。
微小RNA(microRNAs,miRNA)是具有18~24个核苷酸的小的非编码RNA[53],其对于基因表达的转录后调节是必需的[54]。 miRNA 被发现各种疾病的发生和发展与其异常表达水平密切相关,被广泛作为诊断的生物标志物[55]。 对于miRNA 检测,最常见的策略是基于Cas13 的生物传感系统。Shan 等利用CRISPR/LbuCas13a 的高特异性和简单性直接检测miRNA,这种一步法在30 min 内实现了使检测限低至4.5 amol/L,线性范围为10 amol/L~100 fmol/L[56]。更重要的是,单核苷酸的变化,甚至是目标miRNA末端的变化, 都可以通过合理改变crRNA 进行鉴别。此外,这种基于Cas13a-crRNA 的信号放大策略的实际应用能力通过在复杂生物样品中的miRNA定量检测得到了证明,其具有良好的可靠性、敏感性和方便快捷等特点, 有望在与miRNA 相关疾病的早期诊断中发挥巨大的潜力。 Wang 等利用Cas9开发了一种用于miRNA 检测的滚环DNA 扩增(RCA)-CRISPR-split-HRP(RCH)方法。 靶miRNA可解开哑铃探针以进行RCA 反应, 从而产生重复的反义序列和规则的茎环结构, 其融合的split-HRP-dCas9 复合物将通过sgRNA 装配在RCA 产物上。 因此,split-HRP 催化3′3′5′5′-四甲基联苯胺(TMB)进行氧化以产生颜色变化,该方法可以达到fmol/L 水平的检测灵敏度和单碱基水平的特异性[57]。 同Cas9 相似,Cas12a 生物传感系统也需要将miRNA 转化为DNA 底物再进行检测。Wang 等构建了一种新的生物传感系统,称为Cas12a 自供电和滚环转录(rolling circle transcription,RCT)-释放实时crRNA 募集系统(Cas12a-SCR)。 miRNA 作为引物,沿着挂锁探针启动RCT,不断产生具有pre-crRNA重复的ssRNA。 这些pre-crRNA 与Cas12a 形成Cas12a-crRNA 复合物, 能以amol/L 水平的灵敏度检测miRNA-21。Cas12a-SCR 中的pre-crRNA 是实时循环生成的,不再需要Cas12a-crRNA 的预组装,显著提高了miRNA 检测的信噪比[58]。
2019 新型冠状病毒(2019-nCoV)可在人与人之间广泛传播[59-60],导致新型冠状病毒肺炎的疫情难以控制,研究人员针对该病毒的快速检测方法展开了研究[61]。 Ding 等利用Cas12a 结合RT-RPA 扩增来检测2019-nCoV(SARS-CoV-2),可在20 min内得到单拷贝的灵敏度[62]。为了实现一步到位、简单、快速地对2019 新型冠状病毒现场检测,美国食品药品监督管理局 (Food and Drug Administration,FDA)设计并授权了一种称为“一锅法SHERLOCK”(SHERLOCK testing in one pot,STOP) 来 检 测2019-nCoV,该方法简称STOPCovid。 STOPCovid 将LAMP 和 AapCas12b 酶 结 合, 使 得 两 步 法SHERLOCK 转化为一步法, 通过LAMP 扩增的2019-nCoV 的RNA,激活AapCas12b 以切割LAMP生物素标记的ssDNA 报告分子。 STOPCovid 在70 min 和40 min 内分别实现比色读数和荧光读数。STOPCovid 通过鼻咽拭子取样, 具有97%的准确性和100%的特异性[63]。 为了进一步简化RNA 提取,Joung 等使用磁珠进行目标富集, 在15 min 内即可实现目标物的浓缩,对2019-nCoV(SARS-CoV-2)检出限为33 copies/mL[64]。
埃博拉病毒(Ebola virus,EBOV)是一种ssRNA病毒,属于丝状病毒科[65],可引起致死率很高的埃博拉病毒病(Ebola virus disease,EVD)[66],早期诊断和治疗对于控制EBOV 至关重要。 有研究者利用可编程CRISPR 系统, 并应用聚丙烯酰胺-DNA 水凝胶设计微流体纸基分析装置(μPAD),该装置包括具有拓扑排列亲水区域的多层结构,特定dsDNA 的存在可激活Cas12a 以切割第二层中的ssDNA 交联剂,从而抑制水凝胶的形成。 当缓冲液流经整个装置,在染料存在的情况下可以实现可视化检测。 同时,通过比色长度测量,5 min 即可定量检测病毒的浓度,检出限低至11 amol/L[67]。 此外,该体系使用的无线识别模块被整合到μPAD 中进行数据处理,可实现数据的即时反馈, 基于CRISPR/Cas 的μPAD在便携性、灵敏度和低成本方面表现出优异的性能。
3.3 蛋白质检测
CRISPR/Cas 系统也用于检测非核酸目标,如蛋白质、酶、金属离子和小分子物质等。 通常CRISPR/Cas 系统被用作信号放大器, 因为非核酸的靶标分子不能被直接识别,通常需要掺入识别元件,如功能性核酸(脱氧核酶和适体)、抗体和细菌变构转录因子。
Dai 等利用蛋白质适配体的优异识别能力,构建了一种基于CRISPR/Cas12a 的电化学生物传感器(E-CRISPR)。 蛋白质(TGF-β1)适配体复合物的形成可以阻止Cas12a 切割ssDNA 报告基因, 从而导致电流强度迅速降低,获得报告信号,检测限低至0.2 nmol/L[68]。 此外,该测定方法可用于人间充质干细胞软骨分化过程中的TGF-β1 的检测, 无须复杂的样品预处理。Chen 等开发了一种CRISPR/Cas13a的酶联免疫吸附测定 (Cas13a signal amplification linked immunosorbent assay,CELISA)方法,采用双抗体来捕获蛋白质。 输出荧光信号可以通过T7 RNA 聚合酶转录和CRISPR/Cas13a 反式切割活性进行双倍放大,对白细胞介素6(interleukin,IL-6)和血管内皮生长因子 (vascular endothelial growth factor,VEGF) 的检测限分别为2.29 fmol/L 和0.81 fmol/L,检测灵敏度是传统ELISA 试剂盒的100 倍[69]。Du 等设计了一个基于CRISPR/Cas 生物传感系统的哑铃形尿嘧啶-DNA 糖基化酶 (uracil-DNA glycosylase,UDG)作用底物(eSUDG)。 哑铃形尿嘧啶-DNA 糖基化酶在茎部含有一个尿嘧啶碱基,UDG 可以特异性地去除尿嘧啶碱基以产生无嘧啶位点,该位点可以被Endo.IV 核酸酶进一步切割,这会产生带有3′OH 末端的游离ssDNA。随后,在脱氧核苷酸转移酶(terminal transferase,TdT)的介导下聚合以形成长的PolyT 序列。 由于Cas12a 的crRNA本身具有Poly(A)序列,因此Cas12a 效应子可以被激活并对ssDNA 探针进行切割,从而产生荧光信号[70]。所提出的生物传感器实现了UDG 和Dam MTase 两种酶活性的检测, 检测限分别为5×10-6U/mL 和1×10-4U/mL[70-71]。
3.4 小分子物质检测
开发特异性、简单且可推广的小分子检测方法对于生物应用具有很强的吸引力, 包括环境监测、医学诊断和法医甄别[72-74]。 然而,光谱和质谱技术等“金标准”分析过程耗时,且依赖大型仪器[75]。 Liang等报道了一种CRISPR/Cas12a 和细菌变构转录因子(allosteric transcription factor,aTF)介导的小分子检测器,称为CaT-SMelor。 该系统需要与dsDNA 探针和Cas12a-crRNA 介导的荧光报告基因结合的固定化aTF。 当存在靶标分子的情况下,aTF 通过改变其构象以将dsDNA 从aTF 结合结构域解离, 之后dsDNA 与Cas12a 的结合可以激活Cas12a 非特异性切割ssDNA 报告基因,从而产生恢复的荧光[72]。 该系统的检测时间只需要25 min, 且检测限可达到0.25 nmol/L,满足实际样品(166.4~546.7 mmol/L)尿酸分析的要求[76-77]。
对于非核酸物质的检测通常需要利用特殊的适体将非核酸信号转化为核酸信号。Abnous 等基于AuNPs、CRISPR/Cas12a 和RCA 催化活性开发的比色适体传感器, 将RCA、CRISPR/Cas12a 和AuNPs组合使得在黄曲霉毒素M1(AFM1)存在时形成大的DNA 结构, 作为显色剂的4-硝基苯酚无法进入AuNPs 表面;而当AFM1不存在时,4-硝基苯酚可自由到达AuNPs 表面, 此时的AuNPs 表面仅保留非常短的寡核苷酸, 通过4-硝基苯酚对AuNPs 表面的结构和光谱的影响,可灵敏地识别加标牛奶样品中的AFM1[78]。 Mao 等构建了一种上转换纳米粒子(up conversion nanoparticles,UCNP)探针来检测赭曲霉毒素A (OTA)。 基于OTA 适体构建毒素的dsDNA 探针, 当存在OTA 时,OTA 适体与OTA 竞争结合并释放激活链(OTA 适体互补链),激活链被CRISPR/Cas12a 复合物识别并激活Cas12a 反式切割ssDNA 的活性。 结果表明,OTA 浓度与激活链和荧光值成正比并用于检测玉米粉中的OTA[79]。Wu等通过将CRISPR/Cas14a 系统与二维卟啉金属有机框架纳米片 (2D porphyrin metal -organic framework,pMOFs)相结合,研究了一种新型多功能的Cas14-pMOFs 荧光传感器用于检测饮用水和自来水中的微囊藻毒素-LR,在3.5 h 内可达到19 pg/mL的检测灵敏度[80]。
4 展 望
作为基因编辑工具的固有特性,CRISPR 靶向识别活性已经实现了广泛应用,包括生物传感分析和成像分析等,Cas 效应子的反式切割活性为超灵敏和便携式核酸检测提供了有效的信号输出系统。CRISPR 检测方案有几个共同的特征,如快速、恒温和便捷, 几乎所有的CRISPR 系统都可以在短时间内(如几分钟)在温和的条件下完成反应,这是基于CRISPR 的核酸检测工具最突出的优势。 总之,从表3 中可以看出, 基于结合活性和切割活性,CRISPR/Cas 系统已经成为多种目标检测的通用工具, 包括核酸、蛋白质、金属离子、细菌和小分子。 CRISPR/Cas 技术简单,具有特异性,灵敏度高,可结合多种技术实现目标检测,其性能等同于或优于传统检测方法。虽然已经取得了显著的进步,但CRISPR 诊断工具仍然面临一些挑战。 目前存在问题及解决措施如下所示:

表3 基于CRISPR/Cas 的生物传感平台概述Table 3 Overview of CRISPR/Cas-based biosensing platforms
1)脱靶效应 在CRISPR/Cas 系统中,脱靶效应一直受到关注。 在检测过程中,需要仔细考虑脱靶效应可能导致假阴性或假阳性结果。 多种因素影响脱靶效应, 如Cas 效应子本身对错配的容忍度、crRNA 与Cas 效应子的结合没有发生在 “敏感区域”、设计的向导RNA 序列具有兼容性[81]和靶核酸序列与向导RNA 序列之间的碱基错配程度[82]等。对于Cas 效应子, 不同Cas 效应子的结构特征和来源对脱靶效应有显著影响。 靶标和gRNA 序列之间的碱基错配也是产生脱靶效应的主要原因,完全或部分互补的核酸序列导致了脱靶的可能性,特别是当非种子区域中存在错配的碱基对时,致使一些非靶标序列可能激活Cas 效应子,产生背景信号,并导致CRISPR/Cas 生物传感系统的整体灵敏度相对较低[76,83]。 目前也有一些措施用于减少脱靶效应,如通过人工突变构建了高保真Cas9 效应子[84]、使用Cas9镍酶和成对的向导RNA 来最大化降低脱靶效应[85]。此外, 新发现的Cas 效应子也可以提高靶向活性,如Cas12b[86]、Cas13d[87]。机器学习在脱靶预测和优化向导RNA 设计方面等具有较大的潜力[88-91]。 通过对CRISPR 数据库的不断学习和训练, 脱靶效应将得到精确预测,该方法可广泛应用于CRISPR/Cas 系统。
2)检测目标物的有限性 为了准确识别靶序列,大多数Cas 效应子,如Cas9、Cas12a 和Cas13a,需要包含短序列基序(PAM 或PFS)的靶标[25]。 虽然这一要求可以显著增加CRISPR/Cas 系统的特异性,但也限制了靶标区域的选择。 此外,不同类型和来源的Cas 效应子具有各自的短序列基序(如LbCas12a的5′TTTN 和SpCas9 的3′NGG), 这意味着一个特定的靶序列只能被一个Cas 效应子识别,并降低了CRISPR/Cas 系统的灵活性。 因此,当CRISPR/Cas 系统直接检测目标序列时,可选择的目标序列可能很少,尤其是检测短序列或区分单核苷酸多态性。 此外,为了实现非核酸的检测,一些功能核酸通常被用来将非核酸信号转换成核酸信号。 但是,对短序列基序的要求可能需要在一定程度上改变功能核酸的结构,并进一步影响它们的特异性或检测灵敏度。 减少对短序列的依赖性同时保证其特异性,或许是扩大检测范围的重点。 最近有研究者构建了一个名为SpG 的近似无须PAM 的SpCas9 变体,能够靶向具有非G 的PAM[92]。 随着对Cas 效应子结构的不断研究,更多的Cas 效应子将满足对短序列的检测需求,扩大应用范围。
3)多靶点检测 虽然通过CRISPR/Cas 系统已经成功开发了许多用于各种目标物的检测方法,但从单个样品中同时检测多个目标物仍然难以实现[93-94]。 原因主要归因于一旦Cas 效应子和gRNA的复合物被靶标激活,Cas 效应子就表现出非特异性反式切割活性, 所以依靠CRISPR/Cas 系统本身进行多重检测可能不是一个好的选择。 相反,通过物理分离将Cas 效应子、gRNA 或它们的混合物分离到不同的区域来进行检测应该是更好的解决方案[95]。 但是,仍有许多工作需要考虑,包括设计合适的分离结构(如二氧化硅/玻璃微阵列或硝酸纤维素膜), 建立便携式读出技术 (如比色读数或荧光读数)等。
4)便携式检测平台的设计 为了使CRISPR/Cas 系统更有吸引力, 更广泛地应用于不同目标物的检测, 特别是在资源有限的地区, 利用CRISPR/Cas 系统建立一些简单、低成本、方便的检测平台是非常重要的。 在最近的新型冠状病毒肺炎疫情中,基于CRISPR 的2019 新型冠状病毒检测方法已被广泛研究,并因其高灵敏度和特异性而在控制新型冠状病毒肺炎疫情方面发挥了重要作用[64,96-97],但这些检测方法主要限于实验室使用。 对于通常使用的基于荧光的CRISPR 传感系统, 构建具有高效的光信号收集的小型荧光探测器对于便携式信号读取非常有必要。 目前已有研究者设计了一个口袋大小的荧光检测器用于读取荧光信号变化[98]。 在处理大规模检测时,样本之间可能的交叉污染仍然是一个挑战。 在这种情况下,将CRISPR 检测与工程、微电子和小型化相结合可能会提高检测通量和自动化程度。 微流控平台可以在几平方厘米的系统上集成大型测试实验室的所有功能,包括处理样品、转移溶液、混合试剂、加热等。 未来的工作可能需要专注于建立基于CRISPR 的便携式微流控检测平台,携带所有必要的试剂,整个操作过程可以自动或半自动完成。 此外,便携式检测平台中的CRISPR/Cas 系统试剂可以冻干或脱水,便于运输和储存[31],据报道, 脱水的Cas12a 比Cas12a 溶液具有更好的热稳定性[99]。样本处理对于目标检测也非常重要,在未来发展中,将CRISPR 检测与工程、材料和人工智能相结合的多学科联合将使CRISPR/Cas 系统更加强大。
5)可穿戴设备的研究与应用 将CRISPR 诊断和检测系统集成到可穿戴设备中,可以无创监测生理状况、疾病状态,检测可能接触到的病原体或毒素。CRISPR/Cas 系统与水凝胶耦合构建刺激响应材料,可以将生物信息转化为材料性质的变化[67],为智能生物传感器的发展提供了一条途径。 此外,最近有研究者研制了一种带有冻干CRISPR 传感器的面罩[100],是一种可以实现耐储存、基因可编程和高灵敏度的可穿戴生物传感器,将这些生物传感器集成到与无线网络兼容的服装中,并使用定制的智能手机应用程序提供实时动态。
目前,尽管仍有一些挑战需要克服,但随着对每个CRISPR/Cas 系统机制的不断了解和更多Cas效应子的发现,CRISPR/Cas 系统将以其独特的优势在未来的各种目标检测中变得更加完善。
