菠菜SoNAC转录因子家族的全基因组鉴定与分析
2022-03-09谭献詹葛晨辉戴雨柔孔猛徐晨曦王全华
谭献詹 葛晨辉 戴雨柔 孔猛 徐晨曦 王全华



摘 要: 采用生物信息學分析方法,从菠菜基因组中筛选鉴定了57个菠菜NAC转录因子,并对其基因结构、编码蛋白和系统进化进行了分析;通过荧光定量聚合酶链式反应qRT-PCR分析,研究了高温和盐处理后菠菜叶片中基因的表达模式.研究结果显示:菠菜NAC转录因子可以被归入2组17个亚组,Group Ⅰ包含10个亚组,Group Ⅱ包含7个亚组;染色体定位分析表明,共有36个基因随机分布在菠菜的6个染色体上,其中21个分布在scaffold上,多数基因位于端粒,部分基因在染色体上成簇分布并具有相似的结构;基序分析表明,SoNACs中存在5个高度保守的基序,其中没有一个基序是所有SoNACs都共有,有些亚族(SND)的保守基序被其他基序替换,进一步对保守基序的预测表明,Motif 1,2和3是NAM结构域中的核心基序.为揭示NAC家族在菠菜中的抗逆作用,通过qRT-PCR分析了不同亚族基因在高温和盐胁迫下的基因表达,许多基因被盐分或高温胁迫上调或下调,不同胁迫下存在显著差异表达模式.盐胁迫下,表达模式聚为2类:一类为表达上调、在转录中后期发挥主要作用的成员(,,,);另一类为表达下调,但有2个成员(,)在转录起始阶段发挥主要作用的;高温胁迫下,表达模式聚为3类:第一类为表达上调、在转录中后期发挥主要作用的成员();第二类为表达下调的成员;第三类为表达上调不明显的成员.与高温胁迫相比,盐胁迫下更多基因倾向于高表达,是响应两种胁迫均被上调的基因.因此,进一步对该基因及其亚组成员进一步分析可能有助于建立菠菜的胁迫耐受性.
关键词: 菠菜(); SoNAC; 高温胁迫; 盐胁迫
中图分类号: S 636.1 文献标志码: A 文章编号: 1000-5137(2022)01-0020-12
In this study, bioinformatics analysis was used to screen and identify 57 spinach NAC transcription factors from the spinach genome, and to analyze their gene structures, encoded proteins and phylogenetic evolution. Meanwhile, by using quantitative fluorescence PCR (qRT-PCR), expression patterns of NAC genes were examined in spinach leaves treated by high temperature and salt stress. The results of this study showed that spinach NAC transcription factors were classified into 2 groups and 17 subgroups. Groups I and II consisted of 10 and 7 subgroups, respectively. Chromosome location analysis showed that 36 genes were randomly distributed on six spinach chromosomes, and among them, 21 genes were distributed on scaffolds, most of genes were located in telomeres, and some genes were clustered on chromosomes with similar structures. In addition, motif analysis showed that, there are 5 highly conserved motifs in SoNACs, none of which is common to all SoNACs, and some subfamilies (SNDs) of conserved motifs are replaced by other motifs. Further prediction of conserved motifs shows that Motifs 1, 2 and 3 are core motif in the NAM domain. In order to reveal the stress resistance of NAC family in spinach, the gene expression of different SoNAC subfamilies under conditions of high temperature and salt stress was analyzed by qRT-PCR. The results showed that many genes were up-regulated or down-regulated by salt or high temperature stress, indicating there were significant different expression patterns under different stresses. Under salt stress, the expression patterns of are clustered into two categories, one is the up-regulated members (, , , ) that function mainly in the middle and late stages, and the other is down-regulated members (, ) that work mainly in the initiation stage. By comparison, under high temperature stress, the expression patterns of are clustered into three categories, the first category is the up-regulated member () that functions mainly in the middle and later stages, the second category is down-regulated member, and the third group is the member whose expression is not significantly up-regulated. Compared with high temperature stress, more genes tend to be highly expressed under salt stress. However, is a gene that is up-regulated in response to both stresses. As a consequence, further analysis of this gene and its subgroup members may help to establish stress tolerance in spinach.
spinach (); SoNACs; high temperature stress; salt stress
0 引 言
转录因子是一类在植物生长发育中起关键调控作用的蛋白质,它们通过与启动子区域中的顺式调控元件结合来调节靶基因的时空表达.植物转录因子家族,主要包括AP2,bHLH,ARF,MYC,WRKY和NAC等,是许多生物过程中必不可少的调节剂,其中NAC是植物特有的一类转录因子,在植物干旱、盐和高温胁迫下发挥重要作用.NAC是由NAM,ATAF1/2和CUC2共3个亚族构成的转录因子,是植物最大的转录因子家族之一.典型的NAC蛋白N端包含一个保守的NAM结构域,包含约150个氨基酸,在C端包含一个高度可变的转录调控区.自1996年首次报道NAC蛋白以来,NAC蛋白已在拟南芥、水稻、小麦等许多物种中被鉴定.过表达的拟南芥对干旱和盐胁迫的耐受性增强,水稻通过ABA途径在水稻耐旱和耐盐中发挥正调控作用.通过增强下游胁迫相关基因的表达提高了小麦对干旱胁迫的耐受性.此外,一些拟南芥NAC同源基因,如(一种韧皮部特异表达的NAC),细胞壁调节因子,同源的杨树基因(如,)都能够通过减少细胞大小和数量来降低植物的生长速度.一些NAC通过调节植物的激素水平影响其生长发育,已被确定为番茄果实成熟的正调节因子,调节植物生长、侧根形成、叶片衰老以及果实成熟和软化.对拟南芥、水稻和花生的研究已经证明NAC参与干旱和盐胁迫反应.然而,菠菜作为重要的绿叶菜之一,关于其NAC转录因子家族的研究尚未见报道.
本研究以上海师范大学公布的菠菜基因组数据为资源平台,利用生物信息学相关手段,在菠菜基因组范围内筛选、鉴定菠菜NAC转录因子,分析其基因结构、启动子Motifs分布特征、编码序列特征和分类信息等;通过实时荧光定量-聚合酶链式反应(qRT-PCR)分析了所有SoNACs亚组的代表性基因在高温和盐胁迫下的表达水平,以期为菠菜NAC转录因子的克隆、表达和功能验证等研究奠定基础.
1 材料与方法
菠菜转录因子家族的检索、鉴定
菠菜数据库(http://www.spinachbase.org/)用于鉴定NAC转录因子家族成员.采取两个步骤进行鉴定.首先,使用Pfam 32.0(http://pfam.xfam.org/)下载NAM结构域的隐马尔可夫模型(PF号:PF02365)和训练文件集(PF02365_seed.txt),在HMMER3.3.1(http://www.hmmer.org/)程序下搜索菠菜NAC蛋白,提取E_VALUE大于10的高质量序列以PF02365_seed.txt为训练模型NAC.hmm文件,以此模型执行搜索,将结果去除冗余后进行汇总.通过NCBI-CDD(https://blast.ncbi.nlm.nih.gov/)鉴定候选序列,那些不含NAM結构域或明显具有属于其他蛋白质家族的序列被排除在外.最后剩余的蛋白质序列被视为Sp75的NAC转录因子,被用作后续进一步的分析.
菠菜蛋白的理化性质和亚细胞定位
ExPASy(https://web.expasy.org/protparam/)用于预测 SoNACs蛋白的等电点(pI)和分子质量(MW). WOLF PSORT(https://www.genscript.com/tools/wolf-psort) 被用来预测的亚细胞定位情况.
的保守结构域分析
使用Clustal X 2.1对亚族代表SoNACs进行多序列比对,根据保守序列覆盖范围截取比对结果,DNAMAN 6.0软件被用作保守序列特殊颜色的标记.
基因结构、保守基序和染色体定位分析
SoNACs的基因长度、结构信息、保守基序和染色体定位用TBtools软件分析.被用作展示的motif信息则在TBtools上获得(搜索范围为19~50).
启动子的顺式作用元件预测
截取NAC基因启动子上游2 000 bp序列,用来预测顺式作用元件,在PLANTCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)上进行顺式作用元件搜索,并利用Excel中的VBA程序对所有与应激相关的元件进行统计分析.
菠菜蛋白的系统发育分析
为了研究菠菜NAC 蛋白与双子叶植物拟南芥以及近邻植物甜菜NAC之间的系统发育关系,从植物转录因子数据库PlantTFDB 5.0(http://planttfdb.gao-lab.org/)下载拟南芥和甜菜NAC序列,通过MUSCLE进行多序列比对,并经过trimAL程序剪枝后由MEGA 11.0软件使用邻接方法构建无根的系统发育树.iTOL(https://itol.embl.de/)被用于系统发育树的美化.
植物材料和处理
温度敏感型菠菜材料Sp75由本实验室保存并在此种植与处理.在受控温室培养箱条件下进行实验.将来自Sp75的种子4 ℃浸泡后播种在装有等体积的土壤和蛭石的多孔穴盘中,待长出2片真叶后移栽到陶土盆中,缓苗1周后将植物在36 ℃/36 ℃(白天/黑夜)下处理,分别收集处理前的叶片,及处理后2,6,12,24 h的叶片,立即在液氮中冷冻并在-80 ℃下储存.将2对真叶的幼苗移栽到只含珍珠岩的穴盘中,用NaCl物质的量浓度为300 mmol∙L的盐溶液处理,分别在处理0,2,6,12,24 h收集叶片.
基因表达模式分析
使用Trizol试剂从菠菜叶中提取总RNA,PrimeScript RT-PCR Kit(Takara)用于合成第一链cDNA作为后续分析的模板.反应混合物由1 μL cDNA、正向和反向引物各0.5μL、0.04μL ROX、10 μL TB Green™ Premix Ex Taq™(Tli RNaseH Plus)组成,并加入无菌水使总体积达到20 μL.在ABI 7500 Fast Real-Time System上进行如下扩增:95 ℃,2 min;95 ℃,10 s;60 ℃,34 s;72 ℃,31 s,共40个循环.通过熔解曲线分析验证反应的特异性.每个基因进行3个生物学重复,并以18SrRNA作为内参,使用ΔΔCt方法计算每个实验中每个样品的基因转录水平.用于qRT-PCR的基因是每个亚族的代表.
2 结果与分析
菠菜基因的鉴定和分类
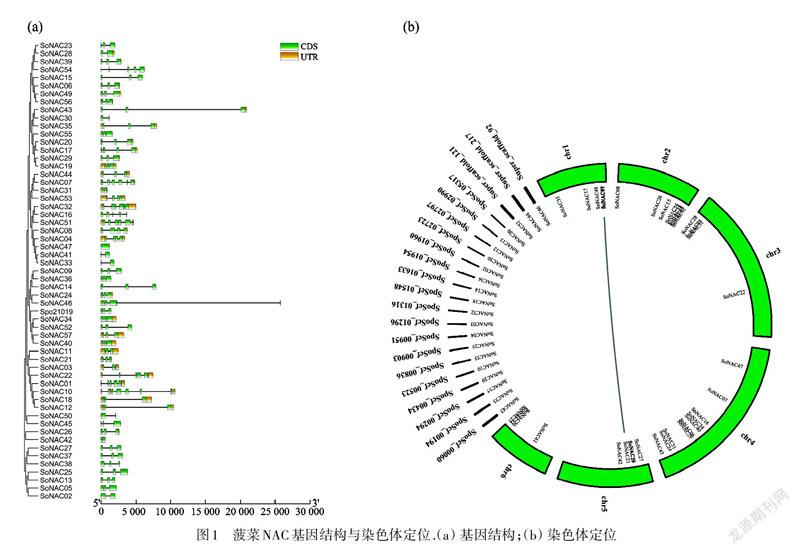
使用配置文件(PF02365)和训练集文件(PF02365_seed.txt)分别鉴定菠菜基因组中的NAC家族成员,2种方式分别鉴定到58个和71个候选成员;对所有结果汇总、去除冗余后进一步在NCBI-CDD上进行确认,得到57个NAC转录因子作为后续的研究对象,本研究中根据NAC成员的基因注释将其编号为SoNAC01-SoNAC57(表1).它们中只有1个()不含内含子结构,绝大多数由2个或2个以上外显子构成,超过一半(56.14%)含有3个外显子,含外显子最多(8个),如图1(a)所示.在编码NAM保守序列的基因里,有11个外显子是连续的,其他的都具跨内含子编码的特性,有2个SoNACs除具有NAM结构域,还含其他家族的保守结构域(含PLN02772超家族,SoNAC47含Herpes_BLLF1超家族).染色體定位分析表明:36个分别被定位于菠菜的6条染色体上,分布在4号染色体上的数目最多(10个),分布在3,5和6号染色体上的数目最少(4个);其他21个SoNACs分布在不同长度的scaffold上,如图1(b)所示.
菠菜转录因子蛋白基本信息分析
通过在线分析软件得到的菠菜NAC转录因子蛋白基本信息分析见表1.由表1可知,菠菜NAC转录因子编码蛋白的大小和氨基酸数目均差异很大,编码氨基酸长度为200~500 aa的NAC占多数,其中最小的SoNAC30由102个氨基酸组成,最大的SoNAC51由663个氨基酸组成,它们对应的蛋白相对分子质量为102~663 ku.等电点范围从4.54(SoNAC32)至10.32(SoNAC30),平均值为6.65.亚细胞定位预测结果显示,45个SoNAC定位于细胞核,占比79%;7个SoNAC定位于细胞质;SoNAC15和SoNAC35定位于氧化物酶体,预测定位于叶绿体的有SoNAC16,SoNAC32和SoNAC47.
菠菜蛋白保守结构域与保守基序分析
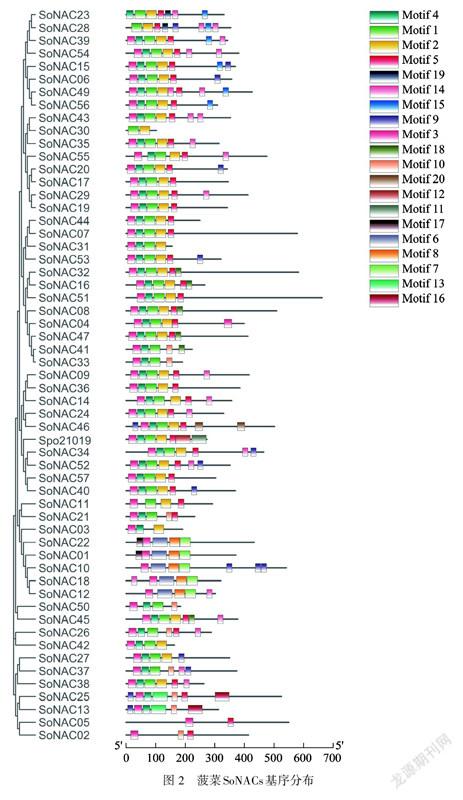
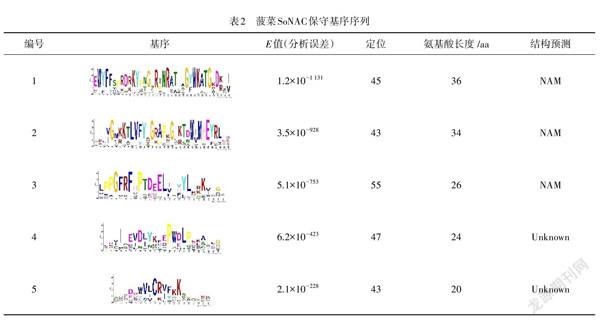
为了揭示SoNAC蛋白结构的多样化,通过MEME搜索发现了20个Motifs(图1).这些Motifs的长度为19~50个氨基酸不等.每个SoNAC含2~8个Motifs,每个SoNAC最多只含7种类型的Motifs. SMART分析表明Motif1~5均为NAM结构,是最保守的部分,其他大多数基序未得到其他功能结构信息(表2).具有Motif 1~5的SoNAC转录因子有36个,占比63.16%,在基因中,缺失Motif 3的有3个(SoNAC23,SoNAC28,SoNAC30),缺失Motif 4的有7个(14.28%),缺失Motif 5的有14个(24.56%),有3个(SoNAC02,SoNAC05,SoNAC30)只含有5个保守基序中的2个(Motif 3和Motif 5、Motif 1和Motif 2),说明Motif 1~3为菠菜NAC转录因子的核心保守基序.此外,有的SoNACs具有2个相同的基序,如SoNAC28,SoNAC54,SoNAC49,SoNAC43含2个Motif 14,SoNAC10含2个Motif 19,但Motif 14和Motif 19的结构域功能尚未注释.
基因结构及家族的系统进化分析
为了揭示菠菜NAC转录因子的系统进化关系,根据OOKA等对拟南芥NAC转录因子的分类,采用NJ(Neighbor-Joining)法构建拟南芥、甜菜和菠菜NAC蛋白的进化树(图3).由图3可知:Group I和 Group II均包含10个亚群,S1~S8为拟南芥中未注释的NAC成员,3个亚组ANAC001,ANAC003和ANAC091中均没有SoNAC转录因子成员,S2~S5,SENU5和TIP都只含一个SoNAC转录因子成员,其他51个SoNACs被分别归入11个亚组.根据拟南芥NAC的基因注释信息对其进行功能预测,其中NAM与发育和形态发生有关;ATAF,NAP参与ABA信号途径,介导拟南芥对盐和ABA的耐受,NAP与SENU5是拟南芥衰老相关的重要因子;NTL是一类跨膜转录因子,主要将外部信号(如盐、冷和渗透胁迫)与植物生长发育联系起来;TIP与拟南芥的病害防御有关;SND和NST与细胞分化相关
红色的实心圆代表菠菜的NAC;绿色的空心三角形代表拟南芥的NAC;蓝色的空心五角星代表甜菜的NAC,拉丁字母代表不同的组,对应的注释标签代表亚族
胁迫相关的顺式作用元件分析
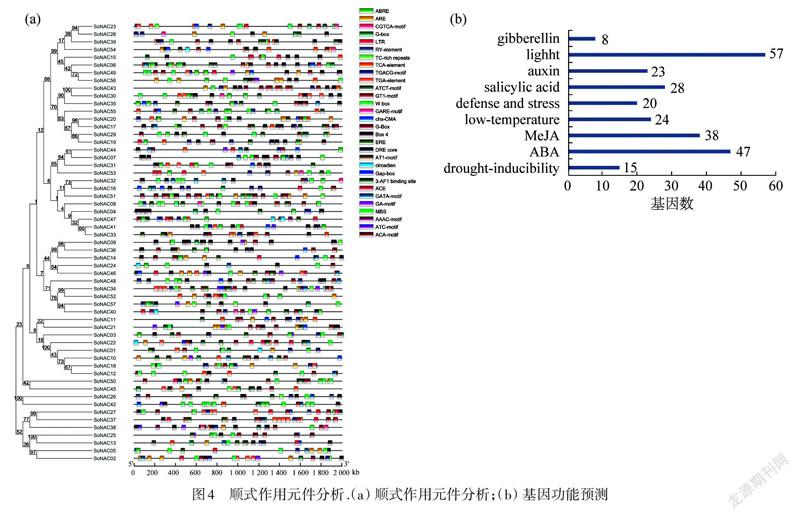
NAC基因在众多应激反应中起着关键作用.如图4(a)所示,通过分析起始密码子(ATG)上游2.0 kb序列中与生物或非生物胁迫响应有关的顺式作用元件,鉴定了30个已知的应激相关顺式作用元件.这些基因的启动子区域内的顺式作用元件的数量为0~10个不等,中数量最多,共有8种不同类型的顺式作用因子,而种类最少的只含1种().所有SoNACs基因包含与光反应相关的顺式作用元件,多数SoNACs参与激素信号响应,较少基因参与低温、干旱等胁迫,如图4(b)所示.47个NAC基因含有1~12个ABRE(GACACGTGGC)拷贝,这是响应参与干旱胁迫耐受性的ABRE依赖性ABA信号传导途径所必须的.20个基因含有1~2个富含TC重复拷贝,其参与防御和应激反应.39个具有1~5个ARE拷贝,这是厌氧诱导所必需的.CGTCA基序参与茉莉酸甲酯(MeJA)介导的应激反应,存在于38个基因中.24个基因具有1~5个LTR元件,其与高温、干旱和盐胁迫有关.在这些启动子区域中还发现了与非生物和生物胁迫响应相关的其他几个元件,例如TGA,W1元件(图4(a)).这些结果表明 NAC基因在响应生物和非生物胁迫时受到转录调控.
高温和盐胁迫下的表达模式分析
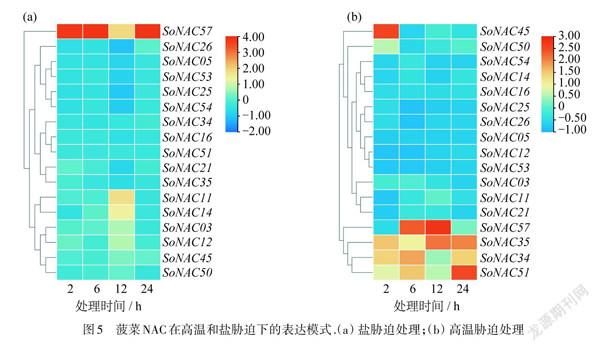
为了揭示菠菜NAC在高温和盐分胁迫下的表达模式,来自菠菜NAC家族的亚族代表被用来进行qRT-PCR分析. 由图5可知:菠菜在高温和盐胁迫下具有不同的表达模式.盐处理下表达模式聚为2类:,,,響应盐处理而表达显著上调,其中和在盐处理12 h后表达丰度最高;另一类为响应盐胁迫呈下调表达的基因,但在盐处理2 h后有显著的上调表达.在高温处理条件下,候选分析的菠菜NAC转录因子表达模式聚为3类,其中受高温诱导,在处理的4个时间点均显著上调表达,和在高温处理12 h后表达水平小幅上调,其他各基因受高温诱导表达水平变化不显著.由此可以推测:可能在菠菜响应高温和盐胁迫时,尤其在响应高温胁迫过程中发挥积极的调控作用,,和可能在菠菜响应盐胁迫时发挥调节作用.
3 讨 论
本研究利用生物信息学方法筛选鉴定出菠菜的57个NAC转录因子,相比已鉴定的拟南芥、番茄和水稻等模式植物,其数目少且相差较大,与甜菜中的NAC数目相近.这种差异可能是其他物种从最早的祖先分化后,发生了更多的重复事件,例如,拟南芥基因组中发生了4次大型重复事件,而菠菜没有经历过基因组复制事件,所以目前发现的NAC转录因子家族数目较少.
NAC转录因子家族的命名源自矮牵牛NAM,拟南芥ATF1,ATF2和CUC2.本研究参照拟南芥NAC转录因子的分组方式,将57个菠菜NAC转录因子分成2组17个亚族,这与YUAN等在花生中的结果(18个亚族)基本一致.菠菜NAC都包含一个保守的NAC转录因子特有的NAM结构域 (PF02365.15),结构域中包含3个保守的NAM基序,主要分布在N端.
NAC參与植物抵抗非生物胁迫、植物形态建成和叶片衰老等多个生命活动.菠菜NAC启动子分析鉴定到与胁迫相关的顺式作用元件ABRE,CGTCA和LRT等,表明其可能受激素依赖或受激素信号途径调控,暗示菠菜NAC存在复杂的调控或被调控现象.根据系统发育分析拟南芥直系同源物的功能预测,SoNAC21与ANAC105单独聚为一个亚族S2,SoNAC45与ANAC32单独聚为一个亚族S4,意味着这2个SoNAC与对应的拟南芥同源基因具有非常相似的功能;TIP亚族的SoNAC16与BvNAC51、SENU5上SoNAC11与BvNAC40互为同源基因并与其他拟南芥NAC聚为一起,暗示着菠菜与甜菜这两个基因在进化上具有共同特征.一些干旱和盐胁迫响应SoNACs显示与先前报道的胁迫响应NAC基因同源,亚族S7的SoNAC14与茄属植物耐旱性的重要调节因子JUB1同源;SoNAC19与拟南芥调节衰老、种子萌发和盐胁迫响应的ANAC092同源;拟南芥中参与建立植物的胁迫耐受性调控的ANAC019,ANAC056和 ANAC072在菠菜中的同源SoNACs可能参与菠菜非生物胁迫耐受性的调控.本研究通过qRT-PCR对17个菠菜NAC进行高温或盐胁迫下的表达模式分析,以确定一些NAC亚族.本研究结果表明:,,在高温胁迫下上调表达,4个基因(,,和)在高温处理12 h后表达水平显著上调,其他处理时间下调表达,猜测不同的SoNAC可能在菠菜响应高温的不同阶段发挥重要作用.不同于热胁迫下的表达模式,盐胁迫下超过一半的候选基因上调表达,其中的表达水平上调15倍.在两种胁迫下和均有显著的表达水平变化,意味着它们在参与高温和盐胁迫耐受性中可能发挥重要作用.
4 结 论
本研究以公布的菠菜基因组数据为基础信息,利用生物信息学方法筛选鉴定出57个NAC转录因子,分析得到了57个菠菜NAC转录因子的保守结构域、Motif、基因结构、启动子顺式作用元件等信息,通过构建进化树获取了菠菜NAC转录因子的分类情况;通过高温和盐胁迫下部分SoNACs的qRT-PCR分析发现,部分SoNACs基因可能参与了高温和盐胁迫耐受性的调控.本研究结果为进一步挖掘菠菜非生物胁迫响应基因,揭示菠菜非生物胁迫抗性调控机制提供了数据支持.
参考文献:
[1] JIA D, JIANG Z, FU H, et al. Genome-wide identification and comprehensive analysis of Nac family genes involved in fruit development in Kiwifruit () [J]. BMC Plant Biology,2021,21(1):44.
[2] OOKA H, SATOH K, DOI K, et al. Comprehensive analysis of NAC family genes in and [J]. DNA Research,2003,10(6):239-247.
[3] NURUZZAMAN M, MANIMEKALAI R, SHARONI A M, et al. Genome-wide analysis of NAC transcription factor family in rice [J]. Gene,2010,465(1/2):30-44.
[4] XUE G, WAY H M, RICHARDSON T, et al. Overexpression of TaNAC69 leads to enhanced transcript levels of stress up-regulated genes and dehydration tolerance in bread wheat [J]. Molecular Plant,2011,4(4):697-712.
[5] WU Y R, DENG Z Y, LAI J B, et al. Dual function of Arabidopsis ATAF1 in abiotic and biotic stress responses [J]. Cell Research,2009,19:1279-1290.
[6] SHEN J, LYU B, LUO L, et al. Corrigendum: the NAC-type transcription factor OSNAC2 regulates aba-dependent genes and abiotic stress tolerance in rice [J]. Scientific Reports,2017,7(1):46890.
[7] GRANT E H, FUJINO T, BEERS E P, et al. Characterization of NAC domain transcription factors implicated in control of vascular cell differentiation in Arabidopsis and [J]. Planta,2010,232(2):52-337.
[8] ZHU M, CHEN G, ZHOU S, et al. A new tomato NAC(NAM/ATAF1/2/CUC2) transcription factor, SlNAC4, functions as a positive regulator of fruit ripening and carotenoid accumulation [J]. Plant and Cell Physiology,2013,55(1):119-135.
[9] YUAN C, LI C, LU X, et al. Comprehensive genomic characterization of NAC transcription factor family and their response to salt and drought stress in peanut [J]. BMC Plant Biology,2020,20(1):454.
[10] XU C, JIAO C, SUN H, et al. Draft genome of Spinach and transcriptome diversity of 120 Spinacia accessions [J]. Nature Communications,2017,8(1):15275.
[11] CHEN C, CHEN H, ZHANG Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data [J]. Molecular Plant,2020,13(8):1194-1202.
[12] LIU Y C, SUN J, WU Y R. Arabidopsis ATAF1 enhances the tolerance to salt stress and ABA in transgenic rice [J]. Journal of Plant Research,2016,129(5):955-962.
[13] LIANG M W, LI H J, ZHOU F, et al. Subcellular distribution of NTL transcription factors in [J]. Traffic,2015,16(10):1062-1074.
[14] REN T, QU F, MORRIS T J. HRT gene function requires interaction between a NAC protein and viral capsid protein to confer resistance to turnip crinkle virus [J]. Plant Cell,2000,12:1917-1926.
[15] MITSUDA N, IWASE A, YAMAMOTO H, et al. NAC transcription factors, NST1 and NST3, are key regulators of the formation of secondary walls in woody tissues of Arabidopsis [J]. Plant Cell,2007,19(1):270-280.
[16] ZHONG R, LEE C, ZHOU J, et al. A battery of transcription factors involved in the regulation of secondary cell wall biosynthesis in Arabidopsis [J]. Plant Cell,2008,20(10):2763-2782.
[17] HARRINGTON S A, OVEREND L E, COBO N, et al. Conserved residues in the wheat () NAM-A1 NAC domain are required for protein binding and when mutated lead to delayed peduncle and flag leaf senescence [J]. BMC plant Biology,2019,19(1):1-2.
[18] YOSHIDA T, FUJITA Y, SAYAMA H, et al. AREB1, AREB2, and ABF3 are master transcription factors that cooperatively regulate ABRE-dependent ABA signaling involved in drought stress tolerance and require ABA for full activation [J]. The Plant Journal: for Cell and Molecular Biology,2009,61(4):672-685.
[19] GERMAIN H, LACHANCE D, PELLETIER G, et al. The expression pattern of the picea glauca defensin 1 promoter is maintained in , indicating the conservation of signaling pathways between angiosperms and gymnosperms [J]. Journal of Experimental Botany,2011,63(2):785-795.
[20] WANG C, RU J, LIU Y, et al. Maize WRKY transcription factor ZmWRKY106 confers drought and heat tolerance in transgenic plants [J]. International Journal of Molecular Sciences,2018,19(10):3046.
[21] JIANG X, NIU Y, XU X, et al. Phylogenetic evolutionand expression analysis of NAC gene family in tomato () [J]. Molecular Plant Breeding,2016,14(8):1948-1964.
[22] LEHNER R, BLAZEK L, MINOCHE A E, et al. Assembly and characterization of the genome of chard ( Ssp. Vulgaris Var. Cicla) [J]. Journal of Biotechnology,2021,333:67-76.
[23] ALZOHAIRY A M, SABIR J S M, GYULAI G B, et al. Environmental stress activation of plant long-terminal repeat retrotransposons [J]. Functional Plant Biology,2014,41(6):557-567.
[24] AIDA M, ISHIDA T, FUKAKI H, et al. Genes involved in organ separation in Arabidopsis: an analysis of the cup-shaped cotyledon mutant [J]. Plant Cell,1997,9(6):841-857.
[25] THIRUMALAIKUMAR V P, DEVKAR V, MEHTEROV N, et al. NAC transcription factor jungbrunnen1 enhances drought tolerance in tomato [J]. Plant Biotechnology Journal,2017,16(2):354-366.
[26] BALAZADEH S, SIDDIQUI H, ALLU A D, et al. A gene regulatory network controlled by the NAC transcription factor ANAC092/AtNAC2/ORE1 during salt-promoted senescence [J]. The Plant Journal: for Cell and Molecular Biology,2010,62(2):250-264.
(責任编辑:顾浩然,冯珍珍)
收稿日期: 2021-11-28
基金项目: 上海市部分地方院校能力建设专项(19070502600)
作者简介: 谭献詹(1997—),男,硕士研究生,主要从事蔬菜遗传育种及分子生物学方面的研究. E-mail: txzmmda@163.com
* 通信作者: 王全华(1963—),女,研究员,主要从事蔬菜遗传育种及分子生物学等方面的研究. E-mail: wangquanhua@shnu.edu.cn
引用格式: 谭献詹, 葛晨辉, 戴雨柔, 等. 菠菜SoNAC转录因子家族的全基因组鉴定与分析 [J]. 上海师范大学学报(自然科学版),2022,51(1):20‒31.
TAN X Z, GE C H, DAI Y R, et al. Genome-wide identification and analysis of spinach SoNAC transcription factor family [J]. Journal of Shanghai Normal University(Natural Sciences),2022,51(1):20‒31.
