利用永久F2群体定位小麦穗部性状相关的QTL
2021-09-23刘阳娜刘丽华张风廷张立平苑少华张明明李宏博庞斌双赵昌平
刘阳娜,刘丽华,张风廷,张立平,苑少华,张明明,李宏博,庞斌双,赵昌平
(北京市农林科学院杂交小麦工程技术研究中心/杂交小麦分子遗传北京市重点实验室,北京 100097)
小麦是我国乃至世界上最主要的粮食作物之一,在粮食生产中占有举足轻重的地位。目前,小麦种植面积日益减少,所以,提高小麦产量尤为重要[1]。穗部性状是小麦重要的产量性状,在小麦产量构成中占据重要地位和作用[2-3]。因此,开展小麦穗粒数、千粒重等主要穗部性状的QTL定位,明确其在染色体上的位置和效应,对利用分子标记辅助育种选择和提高产量具有重要意义。
迄今为止,国内外学者利用不同的分子标记对不同的作图群体(F2、RIL和BC群体)在小麦21条染色体上定位出许多控制小麦穗部性状的QTL[4-8]。近几年随着分子标记和高通量基因分型技术的发展,高密度SNP标记开始应用于小麦遗传连锁图谱的构建和基因定位领域。吴秋红等[9]以269个RIL为材料,利用SNP和SSR在2个环境中共检测到29个穗部性状加性效应QTL。刘 凯等[10]以173个RIL群体为材料,利用90K小麦SNP和DArT芯片技术,在5个环境中进行穗部相关性状的QTL定位,分别检测到7、8、3、5和4个控制千粒重、穗长、穗粒数、可育小穗数和总小穗数的加性QTL,表型解释率均达到8%以上。武炳瑾等[11]利用RIL群体为材料,基于90K SNP芯片,在3个环境下检测到37个控制穗长、小穗数、不育小穗数、穗粒数、千粒重的主效QTL(PVE>10%)。Cui等[12]利用RIL群体,基于660K SNP芯片在10个环境中检测到1个控制穗粒数的主效QTL位点(qKnps-4A),可解释8.0%~21.2%的表型变异。Kuang等[13]利用多小穗雌性10-A与单小穗雄性BE89杂交的F2和F2:3群体为材料,基于SNP和SSR标记在4个不同的环境中共检测到11个与穗部性状相关的QTL,分别在2D、4A、6A、7A和7B染色体上,可解释8.2%~37.8%的表型变异。
前人在利用SNP标记对有关穗部性状进行QTL定位研究时,材料均为基因型纯合的永久性群体(DH、RIL)或F2分离群体,而利用兼有两类群体优势的永久F2群体进行小麦穗部性状QTL定位的研究未见报道。因此,本研究以白玉149×BS366组合含有73个株系的DH群体为材料,构建“永久F2”群体,结合90K SNP芯片基因分型结果,对穗部相关性状进行QTL定位和遗传分析,以期为研究小麦穗部QTL及其遗传效应提供参考,为小麦分子标记辅助育种提供依据。
1 材料与方法
1.1 “永久F2”群体的构建及田间鉴定
以BS366与白玉149杂交组合培育的73个DH群体为材料,根据每个DH群体的开花期调整相应播期,保证不同群体的花期能够相遇。待每个群体发芽后置于4 ℃进行春化处理,之后移栽至云南元谋基地,通过群体间两两随机配制杂交组合,构建232个“永久F2”群体。
232个“永久F2群体”及其亲本于2017―2018年度分别种植于安徽阜阳、北京海淀两地,分别用E1、E2表示。每个“永久F2”群体前后相邻种植其双亲品种,便于获取相关农艺性状,每个环境设两次重复。田间每份材料种植2行,行长1.5 m,行距25 cm,每行30株,采用常规大田田间管理模式。
1.2 表型性状测定及数据处理
于小麦成熟后,每个F2群体随机挑选10个单株,参照李立会等[14]的方法调查穗长、穗粒数、小穗数和千粒重,取平均值用于表型及遗传分析。采用Microsoft Excel 2003软件统计分析数据。
1.3 分子标记筛选及遗传图谱构建
利用Illumina(美国) 公司的Wheat 90K SNP芯片进行全基因组扫描,使用Genome Studio v1.0软件分型。去掉样品分型质量差、无信号、数据缺失率高、双亲间无多态性、最小等位变异率低于0.05的SNP位点,保留具有高质量和双亲间多态性的 SNP 标记用于遗传分析。基于已知染色体组信息,采用QTL IciMapping 4.2软件的BIN功能删除冗余的标记,构建遗传连锁 图谱。
1.4 产量性状QTL定位
利用QTL IciMapping 4.2软件中的完备复合区间作图法ICIM-ADD模型进行QTL定位, 逐步回归的概率为P<0.001,LOD阈值为 3.0[15]。QTL命名规则为:Q+性状+染色体。采用物理定位的方法把本研究鉴定到的主效QTL与前人研究结果进行比较,具体方法为:在Grain Gene网站上查找标记序列,然后在Ensemble Plants数据库(http://plants.ensembl.org/index.html)中利用Blastn功能获取其物理位置。
2 结果与分析
2.1 “永久F2”群体的表型分析
从表1可以看出,基因型和环境对千粒重、穗粒数、穗长和小穗数的影响均达到显著或极显著水平。千粒重、穗粒数、穗长和小穗数各性状在E1、E2两个环境下均存在差异,且4个性状在阜阳的表型稍好于北京。4个穗部性状中,穗粒数和小穗数的遗传力分别为0.55和0.59,说明两个性状受环境和基因型的共同影响;千粒重和穗长的遗传力分别为0.67和0.81,主要受基因型控制,受环境的影响相对较小。
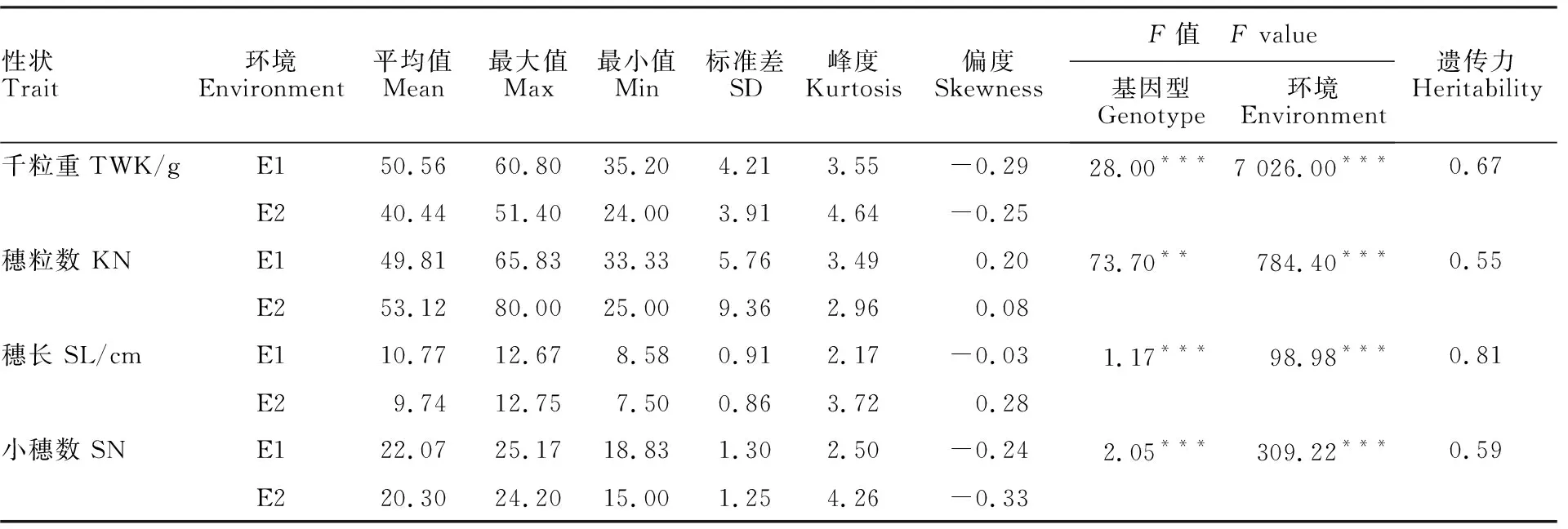
表1 “永久F2”群体穗部性状表型分析 Table 1 Phenotypic analysis of panicle traits of “immortalized F2”population
2.2 遗传图谱构建结果
本试验使用的Wheat 90K SNP芯片共有 81 588个探针,覆盖小麦全基因组。删除不具备重复性、缺失率较高(大于等于10%)和MAF值小于0.05的位点,筛选出材料间有多态性的SNP标记共计8 726个,用于构建遗传连锁图谱。遗传图谱总长19 533 cM,连锁图谱覆盖小麦 21 条染色体,平均SNP标记距离2.24 cM。8 726个SNP标记按染色体合并为3 078个BIN标记,其中,大多数BIN标记定位在A染色体组(1 283个,41.7%)和B染色体组(1 188个, 38.6%)上,仅有19.7%(607个)的BIN定位在D染色体组上。A、B、D基因组染色体图谱总长分别为 7 868.23、6 129.90和5 534.87 cM,分别占总图谱长度的40.3%、31.4%和28.3%。染色体长度为591.4(4B)~1 248.3 cM(7A),每条染色体上定位的BIN标记为45~244个。
2.3 穗部性状QTL的定位结果
利用2个环境及其平均值的表型数据,共定位到96个与穗部性状相关的QTL,分布在19条染色体上,其中23个QTL的表型变异解释率大于5%,为主效 QTL(表 2~5)。
2.3.1 穗长QTL
定位到控制穗长的QTL有20个(表2),分布在2A、2D、3A、4A、4B、5B、5D、6A、6D、7A和7B染色体上,单个QTL可解释1.83%~7.77%的表型变异,其中有10个为主效 QTL。10个QTL加性效应为正值,表明其增效效应来源于母本。其余10个QTL加性效应为负值,表明其增效效应来源于父本。10个QTL显性效应为正值,表明显性等位基因起增效作用。7个QTL的显性效应大于加性效应(显性效应与加性效应比值的绝对值,下同),表现为超显性效应。在10个主效 QTL中,3个QTL表现为超显性效应,分别是QSL.1-5D、QSL.1-6A和QSL.2-6A。
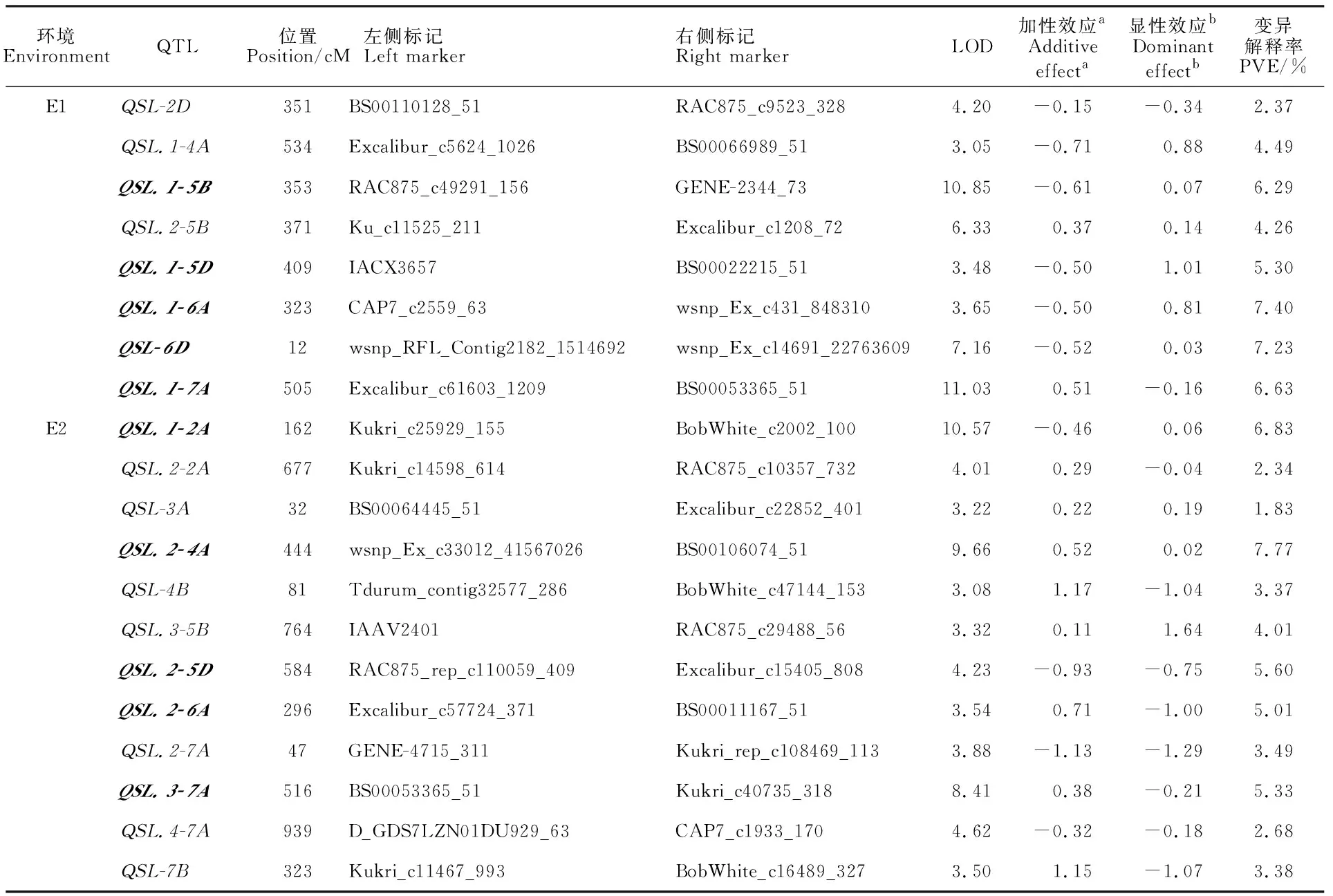
表2 本研究定位的穗长QTLTable 2 QTLs for spike length mapped in this study
2.3.2 小穗数QTL
控制小穗数的QTL有59个(表3),位于除3B和6B以外的19条染色体上,单个QTL可解释0.22%~12.34%的表型变异,其中 4 个是主效QTL。31个QTL加性效应为正值,表明其增效效应来源于母本,28个QTL加性效应为负值,表明其增效效应来源于父本。6个QTL显性效应为正值,表明显性等位基因起增效作用。43个QTL的显性效应大于加性效应,表现为超显性效应。在4个主效QTL中,2个QTL表现为超显性效应,分别是QSN.1-2A和QSN.1-7A。
2.3.3 穗粒数QTL
控制穗粒数的QTL有6个(表4),位于1A、2A、4A和4D染色体上,均为主效QTL,单个QTL可解释6.80%~9.36%的表型变异。4个QTL加性效应为正值,表明其增效效应来源于母本,2个QTL加性效应为负值,表明其增效效应来源于父本。2个QTL显性效应为正值,表明显性等位基因起增效作用。2个QTL的显性效应大于加性效应,表现为超显性效应,分别为QKN.1-2A和QKN.2-2A。
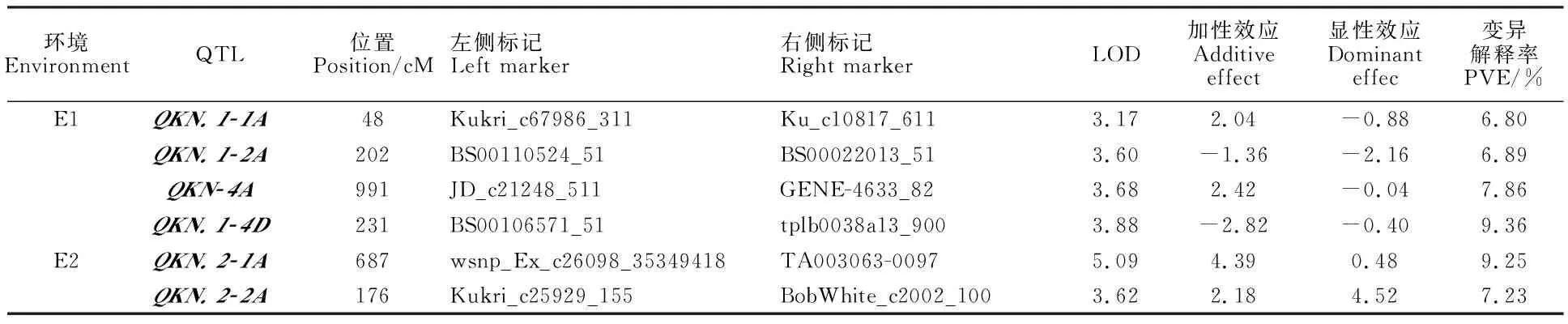
表4 本研究定位的穗粒数QTLTable 4 QTLs for kernels number per spikemapped in this study
2.3.4 千粒重QTL
控制千粒重的QTL有11个(表5),分布于2D、3A、3D、7A和7B染色体上,单个QTL可解释2.57%~6.55%的表型变异,其中3个是主效QTL。6个QTL加性效应为正值,表明其增效效应来源于母本,5个QTL加性效应为负值,表明其增效效应来源于父本。10个QTL显性效应为正值,表明显性等位基因起增效作用。7个QTL的显性效应大于加性效应,表现为超显性效应。在3个主效 QTL中,1个QTL表现为超显性效应,为QTWK.3-7A。
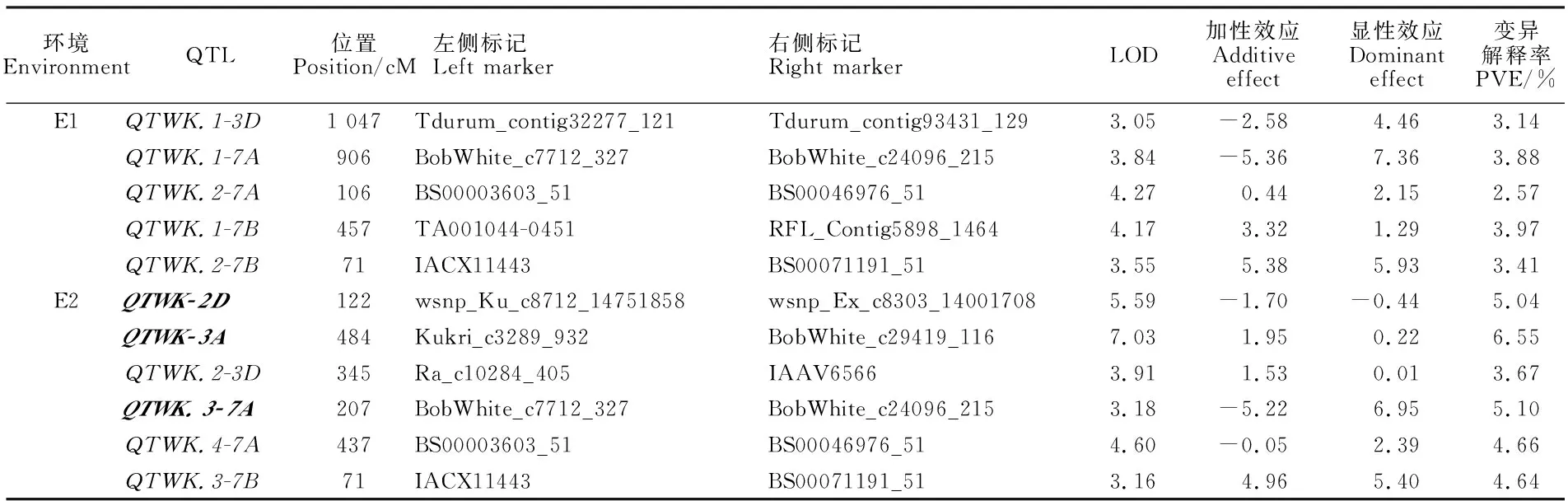
表5 本研究定位的千粒重QTLTable 5 QTLs for thousand-grain weight in this study
3 讨 论
3.1 染色体组等位变异丰富度不均衡
普通小麦是由 A、B、D 染色体组组成的异源六倍体物种,长期进化和人工选育使小麦资源的遗传多样性偏低且遗传基础狭窄,尤其是D染色体组。前人研究认为,3个基因组等位变异丰富度为B>A>D[16-18]。本研究结果认为,A基因组的遗传多样性最高,B基因组次之,D基因组最低,A和B基因组研究结果与前人研究结果不一致,原因可能是本研究所用的SNP芯片和群体与前人不一致。
3.2 QTL定位结果分析
本研究共定位到96个与穗部性状相关的QTL,其中主效QTL有23个,均只在一个环境中检测到,但在阜阳环境下检测到的控制穗长的QSL.1-6A(5.3~615.8 Mb)与在北京环境下检测到的控制穗长的QSL.2-6A(31.0~614.0 Mb)位置有重叠区域;在阜阳环境下检测到的控制穗长的QSL.1-7A(683.5~692.9 Mb)与在北京环境下检测到的控制穗长的QSL.3-7A(692.9~700.7 Mb)位置较近,可能为同一个QTL。
到目前为止,前人定位到许多与小麦穗部性状相关的QTL。对穗长而言,本研究定位到的QSL.1-6A(5.3~615.8 Mb)和QSL.2-6A(31.0~614.0 Mb)与武炳瑾等[11]在6A染色体上检测到的QTL位置(531.5~551.0 Mb)有重叠区域,可能为同一个QTL;本研究定位到的QSL.1-7A和QSL.3-7A的物理位置分别为683.5~692.9 Mb和692.9~700.7 Mb,而武炳瑾等[11]和Fan等[19]也在7A染色体上检测到控制穗长的QTL,但位置不一样。对小穗数而言,本研究定位到的QSN.1-5A(567.9~615.3 Mb)与刘 凯等[10]在5A染色体上检测到的QTL位置(539.5 Mb左右)接近,表型变异解释率为12.34%;本研究定位到的QSN.1-7A物理位置为717.1~717.4 Mb,武炳瑾等[11]也在7A染色体上检测到控制小穗数的QTL,但位置不一样。对穗粒数而言,本研究定位到的QKN.2-2A的物理位置为535.8~710.2 Mb,Shi等[20]在2A染色体上检测到的QTL(物理位置约为612.8 Mb)也在此区间内,可能为同一个QTL;本研究定位到的QKN-4A的物理位置为6.8~645.1 Mb,与Cui等[12]在4A染色体上检测到的QTL位置(物理位置约为683.5 Mb)不一致。对千粒重而言,本研究定位到的QTWK.2-2D物理位置约为481.6 Mb,与宋利强等[21]检测到的控制千粒重的主效QTL (qTkw-2D)位置(561.16~ 574.60 Mb)相差较远,可能是一个新的QTL;本研究定位到的QTWK.3-7A物理位置约为481.6 Mb,与孙宇慧等[22]在4A染色体上检测到的QTL位置(物理位置为5.6 Mb)不一致。
本研究检测到的主效QTL仅在一个环境下能够检测到,可能是穗部性状受基因型和环境的共同影响,穗部性状基因之间也存在相互作用,导致QTL在多环境检测中表现不稳定或环境特异表达,后期将在多环境中种植并设置重复,以消除环境或基因型与环境互作的影响,进一步对这些主效QTL精细定位,为开展与穗部性状相关QTL的克隆和分子标记辅助育种提供基础。
