厌氧砷还原细菌(Clostridium sp. ZY1)分离鉴定及基因组分析
2021-07-01黄华枝廖俊杰朱永闯李湘伟黄伟忠房保柱李文均李金天
张 堃, 周 媛, 黄华枝, 廖俊杰, 朱永闯, 李湘伟, 李 林, 黄伟忠, 房保柱, 李文均, 李金天, 廖 斌*
(1. 广东轻工职业技术学院, 广东 广州 510300;2. 中山大学 生命科学学院, 广东 广州 510275; 3. 华南师范大学 生命科学学院生态科学研究所, 广东 广州 510631)
砷(As)作为毒性较强的致癌物质,是一种常见的天然元素,并且广泛分布于水、土壤、植物等自然环境以及食物中。砷元素除了自然源的释放外,众多人为活动如含砷矿山的开采及冶炼、煤碳的燃烧以及在玻璃制造、饲料加工和染料等生产过程的添加剂等也加速砷元素的释放[1-2]。然而,水环境污染被视为砷产生毒害作用的主要途径之一,当砷元素进入水体后,大部分被富集在沉积物中[3],并且这些砷元素长期处于固液面交换的动态变化中,随着环境条件改变或沉积物在微生物的作用下,砷元素会重返表层,经由食物、饮用水等途径对人类健康构成威胁,并引起公共健康安全事件[4-7]。2014年生态环境部和自然资源部对我国土壤污染状况展开调查,结果显示,我国土壤重点监控的五大重金属(Cd、Hg、As、Pt和Cr)点位超标率均在1%~7%,其中砷元素(As)超标率为 3%[8],我国正面临着越来越严重的砷污染问题。砷元素(As)主要以不同价态砷(As(Ⅴ/Ⅲ))存在于自然环境中,其主要存在形态为无机态砷化物,氧化状态主要为砷酸盐(As(Ⅴ)),而还原态则为亚砷酸盐(As(Ⅲ))[9-11]。不同价态砷元素的环境迁移性及生物毒性也不尽相同。自然环境中,微生物对砷元素的价态和形态转化具有不可忽视的作用[12]。研究表明,微生物是砷元素生物地球化学循环的主要贡献者,并在砷元素的不同转化过程(如氧化、还原、甲基化及去甲基化等)中发挥着举足轻重的作用[13-15]。在厌氧条件下,微生物的活动可以直接或间接影响沉积物中的砷元素及其他的铁氧化物、硫酸盐的形态,进而影响砷在环境中的元素地球化学行为。众多研究表明,呼吸性砷还原菌(Dissimilatory Arsenate-Respiring Prokaryotes,DARPs)是砷元素从沉积层溶解和释放到地表或地下水的主要驱动力[16]。在厌氧条件下,呼吸性砷还原菌利用各类有机物(乳酸盐、甲酸盐等)或无机物(氢气或硫化物)作为电子供体,将低毒性砷(Ⅴ)转化为高毒性砷(Ⅲ)[17]。这些微生物的主要类群为后壁菌门(Firmicutes)和变形菌门(Proteobacteria),如乙酸氧化脱硫单胞菌(Desulfuromonassp. WB3)、杆状菌(Bacillussp. M17、Bacillussp. JMM-4)、Sulfurihydrogenibiumsp.HGMK-1、腐败希瓦菌(Shewanellasp.ANA-3)、硫磺单胞菌(Sulfurospirillumdeleyianum)等[18]。因此,能够直接还原吸附砷酸盐或矿物质,并显著增强不溶性砷的溶解与释放的微生物种类较为多样。本研究对湖南石门砷矿区底泥进行富集培养,利用纯培养手段分离获得1株能够进行厌氧砷还原的菌株ZY1,并发现其属于梭菌属(Clostridium),随后对该菌株进行基因组测序,对基因组进行初步分析,为后续砷还原的微生物机理研究提供菌株资源。
1 材料与方法
1.1 材料
1.1.1 试验材料 用于富集分离的底泥样品(采样深度约10 cm)采自湖南省石门县白云乡雄黄矿(N25°2′57.5″,E113°39′34.1″)。表层覆盖的用于修复的新层土壤导致砷的浓度较低,因天然淋滤、矿化和微生物的作用,底泥以及积水呈酸性,水样pH值4.2。采集的底泥样品置于无菌的50 mL离心管,低温运输,4 ℃保存备用。
1.1.2 仪器与试剂 PCR仪(T100,美国伯乐公司);凝胶成像仪(GEL Logic200 Imaging System,美国伯乐公司);电泳仪(Mini-Sub Cell GT Cell 170-4486,美国伯乐公司);厌氧工作站(ELECTROTEK AW 400SG,英国依莱泰科公司);透射电镜(LKB 2088-020);PCR引物(上海捷瑞公司);DNA聚合酶(RR02AG,宝生物(Takara bio)科技公司);OMEGA E.Z.N.A.®细菌DNA提取试剂盒(D5625,美国);MP细菌基因组提取试剂盒(FastDNA SPIN Kit for Soil,美国),制霉菌素等。
1.1.3 培养基 ① 1号培养基(富集培养基):(NH4)2SO40.1 g, NaCl 1.0 g, MgSO4·7H2O 0.05 g, Na2SO4·2H2O 100 μL (3 mg/L);葡萄糖1.8 g,复合维生素400 μL,微量元素溶液 100 μL,pH 3, 蒸馏水1 000 mL。② 2号培养基(分离培养基):富集培养基配方中加入1.56 g Na2AsO4·7H2O。③ 3号培养基(梭菌增菌培养基)(g/L):蛋白胨10,牛肉浸出粉10,酵母提取物3,可溶性淀粉1,葡萄糖5,氯化钠5,醋酸钠3,L-半胱氨酸盐酸盐0.5,琼脂0.5。复合维生素溶液(mg/L):对氨基苯甲酸5,生物素 5,维生素B25,盐酸吡哆醇1, 维生素B15,烟酸5,维生素B55,硫辛酸5,维生素B120.1;微量元素溶液:FeCl2·4H2O 1.5 g,CoCl2·6H2O 190 mg,MnCl2·4H2O 100 mg,ZnCl270 mg,H3BO262 mg,Na2MoO4·2H2O 36 mg,NiCl2·6H2O 24 mg,CuCl2·2H2O 17 mg, 蒸馏水定容至1 000 mL,pH 3。在上述培养基中加入琼脂糖制成相应的固体培养基。
1.2 方法
1.2.1 厌氧砷还原细菌的厌氧富集与分离 ①样品厌氧富集:50 g底泥样品置于装有300 mL 1号液体培养基的500 mL厌氧瓶中,并向瓶内鼓入混合气(N2/CO2/H2)。为使底泥样品与培养基充分混合,每日摇匀2次,培养40 d。厌氧培养的实验条件:温度30 ℃、湿度40%。②砷还原菌的分离:厌氧工作站中,将1 mL富集培养物与冷却至40 ℃的2号固体培养基混匀倒入平板,凝固后倒置培养,待培养皿中长出菌落,挑取单菌落进行三区划线法培养,获得纯培养菌株。并接种在3号固体培养基上快速培养,同时进行菌株鉴定。2号与3号培养基均加入采样点酸性废水过滤原液(滤膜孔径0.22 μm)100 mL。
1.2.2 菌株ZY1的初步鉴定 ①菌体形态观察:利用透射电镜对菌株的形态进行观察。选取生长1 d的菌落制成悬浮液,2%磷钨酸钠水溶液染色0~1 min,取适量悬液置于载物铜网上,自然干燥后电镜观察。②生理生化性质测定:a.菌株生长曲线:将1 mL种子液接入装有200 mL 3号液体培养基的250 mL三角瓶中,厌氧30 ℃,湿度40%,测定菌株ZY1的生长曲线。b.最适pH:以1%(体积分数)的接种量,将菌株接种于pH值分别为2.96、4.00、5.02、5.72、6.42、7.09、7.84、8.90、9.22的1号液体培养基中,湿度40%,30 ℃条件下观察其生长状况。c.NaCl耐受:将菌株划线接种于含有0%~4.5%盐浓度pH 8的3号固体培养基上,30 ℃,湿度40%,培养观察。d.菌株对不同砷元素(V/Ⅲ)耐受:将菌株分别接种于As(V)浓度为0~200 mmol/L(10 mmol/L梯度递增)和三价砷浓度分别为0.66、1.32、1.98、2.64、3.3、3.96 mmol/L pH 8的3号液体培养基中,30 ℃,湿度40%条件下培养,观察菌株对不同价态砷元素的耐受性。
1.2.3 菌株ZY1的分子生物学鉴定 ①核酸提取:菌株ZY1的DNA使用OMEGA E.Z.N.A.®细菌DNA提取试剂盒提取[19],操作过程做少许调整,加溶菌酶37 ℃预热10 min后,选择加入少许(10 mg)玻璃珠,破碎无需太过剧烈。②16S rRNA基因的PCR扩增:通过细菌16S rRNA通用引物(27f/1492r[20])16S rRNA基因进行扩增,50 μL反应体系与PCR 扩增反应条件参照文献[21],送上海生工生物工程有限公司进行PCR产物测序。③基因序列比对分析:登录EzBioCloud (http://eztaxon-e.ezbiocloud.net/)网站,将上海生工生物工程有限公司测序返回的16S rRNA基因序列进行比对分析其相似性[22]。
1.2.4 菌株ZY1全基因组分析 ①基因组 DNA 的提取及测序:将菌株 ZY1于3号固体培养基上 30 ℃、湿度40%培养24 h,收集菌体用无菌水清洗后,使用QIAGEN细菌基因提取试剂盒提取细菌总基因组DNA,并送至广州基迪奥生物科技有限公司,利用Illumina HiSeq 4 000进行基因组测序。②全基因组de novo拼接与结果评估:原始数据(rawdata)经过FastQC(v0.11.8)[23]和 cutadapt(v1.18)[24]过滤,获得Clean data,随后利用SOAPdenovo进行组装[25]。对初步组装得到的结果利用GapCloser进行修补[26],减少结果中N的数量;利用SSpace软件对Scaffolds进行延伸,最终获得组装的菌株ZY1基因组[27];将拼接得到的基因组与其近缘基因组通过MUMmer[28]进行全局比对,将比对结果通过MUMmerplot绘制点阵图;利用BUSCO对基因组组装质量进行分析[29];采用CG View Server进行基因组圈图绘制。③菌株ZY1功能基因注释与分析:利用GeneMark对基因组进行ORF预测[30],利用diamond(v0.7.9)进行基因注释[31]。主要通过COG和KEGG两个数据库,将菌株ZY1基因组利用COG和KEGG数据库进行注释。④菌株ZY1与砷相关的基因鉴定:根据关键词“arsen”,从全部的COG、KEGG、NCBI-nr数据库中筛选全部潜在的氨基酸序列,通过手动筛选,将序列分成4个不同的基因家族,得到砷相关基因数据库。将步骤④预测得到的菌株ZY1基因组核酸序列与砷相关基因蛋白数据库进行tblastx程序比对,将比对结果进行筛选(e-value ≤1e-5,覆盖度>40%,准确度>40%)。最后通过预测得到的ORF在基因组Scaffold上的坐标,将菌株ZY1基因组中的As相关基因定位到菌株ZY1基因组中。
2 结果与分析
2.1 菌株ZY1的生理生化特征
菌株ZY1在2号固体培养基上菌落白色或透明,圆形凸起,黏稠,产气泡,革兰染色阳性。菌株ZY1长度为5 μm左右,长杆状,有芽胞,具侧生鞭毛(图1)。经过培养优化,菌株ZY1在不同培养基上的生长周期不同,其中1号培养基至少1周长出菌落,2号培养基至少25 d,3号培养基20 h长出明显菌落。

图1 菌株ZY1的透射电镜图Fig.1 The transmission electron microscopy (TEM) of ZY1
菌株ZY1经过3号培养基培养,在600 nm波长下,检测菌液OD值发现,菌株ZY1静止期约为第0至10 小时,对数生长期约为第10至20小时,平台期约为第20小时后(图2)。

图2 菌株ZY1的生长曲线图Fig.2 The growth curve of ZY1
pH耐受试验结果发现,pH为6.4和7.1的培养基在第4天时可以观察到菌株ZY1的生长,pH值为5.0、7.8和8.9的培养基约两周后菌株ZY1生长,其他约4周后生长,从而确定菌株ZY1最适pH生长范围为6.4~7.1。菌株ZY1的NaCl耐受范围为0%~3%,但不同盐浓度对菌株ZY1的生长时间有所影响,随着盐度的增加菌株ZY1的生长周期延长。在砷元素(As(V/Ⅲ)耐受实验中,菌株ZY1能够耐受100 mmol/L 五价砷(As(V)),而三价砷(As(Ⅲ))的耐受浓度仅为1.98 mmol/L。
此外,菌株ZY1的16S rRNA基因序列与菌株北极梭菌(ClostridiumarbustiSL206T)的相似性为99.0%,通过MEGA软件采用邻接法(Neighbour-joining)构建系统发育树。菌株ZY1与ClostridiumpasteurianumDSM 525T聚类在一起,且Maximum-Likelihood(ML)和Maximum Parsimony(MP)方法构建的系统发育树也支持这一聚类关系,并形成稳定的分支(图3)。因此,菌株ZY1应归属于梭菌属(Clostridium)。

图3 菌株ZY1的系统发育树Fig.3 Phylogenetic tree showing the relationship of ZY1 with some other clostridium based on 16S rRNA genes
2.2 菌株ZY1基因组信息
为了进一步了解分离菌株ZY1的代谢潜能和其逆境条件下的生存机制,对其进行基因组测序并进行相关分析。测序获得原始数据(rawdata)1.87 Gb,质控获得干净数据(clean data)1.36 Gb。经过拼接和修补,获得菌株ZY1基因组大小为4.4 M,基因组包括31个Scaffolds,最长的Scaffold为570 737 bp,N50为318 327 bp,GC含量30.6%,基因预测得到4 210个基因,平均长度861 bp,将序列中耐砷和与砷转化的相关基因分成了4个不同的基因家族(表1)。

表1 细菌中耐砷和砷转化的相关基因
将上述预测得到的菌株ZY1基因组核酸序列与砷代谢相关基因蛋白数据库进行tblastx程序比对(表2)。梭状芽胞杆菌(Clostridiumsp.)ZY1的基因组信息中注释到3个arsA和3个arsR,推测菌株ZY1具有五价砷的能力,通过多个arsR基因不断调节还原,多个arsA编码合成ATP酶的ArsA蛋白,为亚砷酸盐转运蛋白的转运过程提供大量的能量,从而极大地增强了解毒作用。

表2 菌株ZY1砷代谢相关基因与相关数据库比对结果
随后对菌株ZY1基因组与近缘巴氏芽胞梭菌(ClostridiumpasteurianumCP1、ClostridiumpasteurianumATCC 6013)的基因组进行全局比对[28],结果表明,大部分序列均能与参考基因组较好地匹配(图4),并且几乎覆盖参考基因组的所有区间,菌株ZY1基因组完整、可靠。

图4 菌株ZY1基因组与参考基因组全基因组对比分析Fig.4 The alignments between draft genomes of strain ZY1 and the reference strains 红点代表正向匹配参考基因组,蓝点代表反向匹配参考基因组ZY1 genome sequences against that of reference genome are plotted as red dots and reverse are plotted as blue ones
利用BLASTx将KEGG、NCBI-nr功能数据库与得到的开放阅读框(ORF)进行序列比对实现功能注释。结果表明,KEGG和NCBI-nr数据库均呈现较高的功能注释率,分别达到94.1%和92.7%。利用CGView Server做出菌株ZY1的基因组圈图(图5),其中最外两层分别表示前导链和后随链中预测得到的基因,第三层表示全基因组的GC含量,第四层表示GC skew(绿色代表正值,洋红色代表负值)。
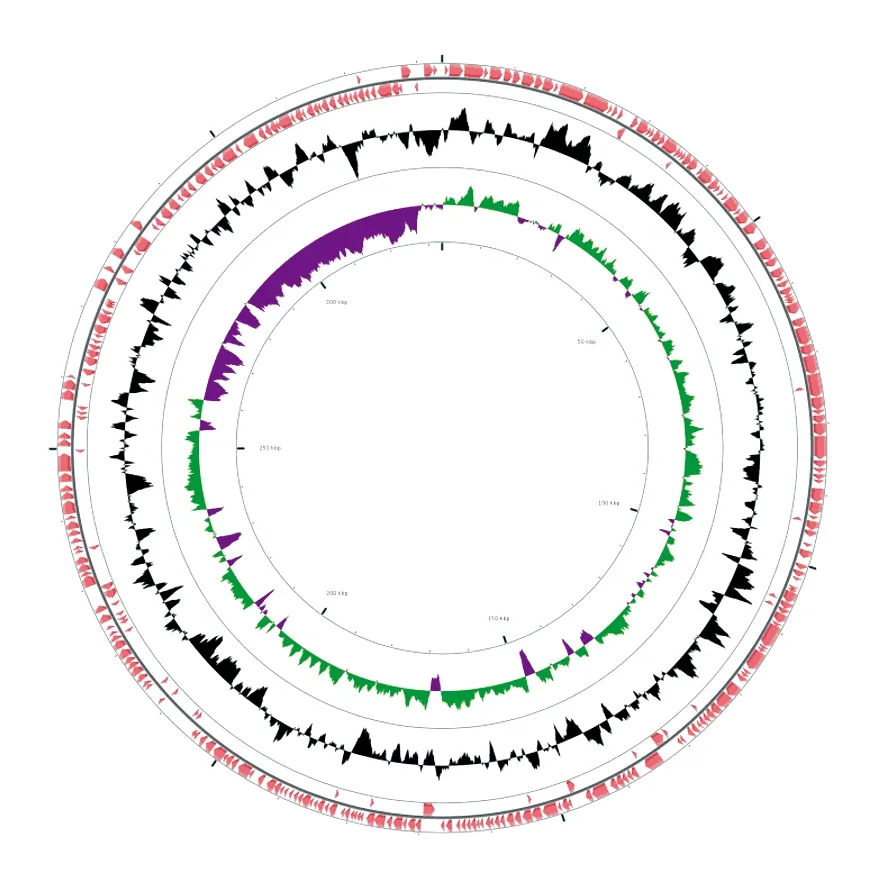
图5 全基因组圈图Fig.5 Graphical map of the ZY1 chromosome
2.3 菌株ZY1全基因组功能注释
在基因预测的基础上,与蛋白质数据库中的蛋白序列进行比对,得到相对应的基因功能信息,从而了解基因功能和其代谢通路的详细信息。在已预测的4 210个基因中,有3 902个基因注释到KEGG数据库中,占预测基因数的92.7%。结果表明,菌株ZY1的代谢通路相关基因的显著聚集从高到低依次为碳水化合物代谢(Carbohydrate metabolism)、氨基酸代谢(Amino acid metabolism)、辅酶因子和维生素代谢(Metabolism of cofactors and vitamins)、能量代谢(Energy metabolism)、膜转运(Membrane transport)和核酸代谢(Nucleotide metabolism),膜转运与辅酶因子和维生素代谢等表明对环境胁迫微生物表现出较强的适应能力。此外,信号转导(Signal transduction)、细胞能动性(Cell motility)、脂肪代谢(Lipid metabolism)和氨基酸代谢(Metabolism of other amino acids)也占据较高的丰度(图6)。

图6 菌株ZY1各功能基因KEGG Categories注释结果Fig.6 KEGG pathway categories classification of strain ZY1
根据KEGG代谢通路统计前20的功能分类丰度结果(图7),从各功能基因大类中细分可知,代谢通路中,嘌呤代谢(Purine metabolism)、嘧啶代谢(Pyrimidine metabolism )、氨基糖和核甘糖代谢(Amino sugar and nucleotide sugar metabolism)、卟啉和叶绿素代谢(Porphyrin and chlorophyll metabolism)、果糖和甘露糖代谢(Fructose and mannose metabolism)、糖酵解/糖质新生(Glycolysis/Gluconeogenesis)(产生少量能量)等代谢在代谢通路中占据主要的丰度,表明菌株ZY1在环境中为了抵抗寡营养环境通过自身不断迅速合成代谢和繁殖提高其生存能力,通过各类代谢获得能量作为生存储备。

图7 KEGG代谢通路统计排名前20的各功能分类丰度Fig.7 Number of genes assigned to KEGG functional pathway for strain ZY1
2.4 菌株ZY1代谢通路预测分析
为了进一步了解菌株ZY1的生长原因,基于基因组学信息分析,从能量代谢和胁迫响应两方面探究其代谢途径、产物,描述其代谢过程。
2.4.1 碳代谢 通过分析碳代谢(Carbon metabolism)所有基因,发现菌株ZY1可能利用葡萄糖、磷酸果糖、磷酸烯醇丙酮酸、丙酮酸。①可以利用丙酮酸羧化酶(Pyruvate carboxylase)将丙酮酸降解为丁酮二酸,但不能再降解为柠檬酸;②利用苹果酸脱氢酶(Malate dehydrogenase)和延胡索酸水合酶(Fumarate hydratase)将丙酮酸降解为延胡索酸;③由丙酮酸脱氢酶(Pyruvate dehydrogenase)或丙酮酸铁氧还蛋白氧化还原酶(Pyruvate-ferredoxin oxidoreductase)催化脱羧生成乙酰辅酶A,再由乙酰辅酶A羧酶(Acetyl-CoA carboxylase)催化生成丙二酰辅酶A;④生成乙酰辅酶A后,利用乙酰辅梅A酰基转移酶生成乙酰基辅酶A。大部分微生物能够通过TCA循环(Tricarboxylic acid cycle)将糖类、脂肪、蛋白质彻底降解成CO2,但是由于缺失柠檬酸合成酶(Citrate synthase),导致草酰乙酸(Oxaloacetate)不能被降解生成柠檬酸,使TCA循环中断,不能彻底氧化有机底物。菌株ZY1属于梭菌属,该属无固碳能力,基因通路显示在Wood-Ljungdahl通路中,由于缺失甲酸盐加氢酶(Formate dehydrogenase)而无法固定CO2。

2.4.3 硫代谢 菌株ZY1含有两种潜在的硫代谢方式:①通过硫酸盐转运系统将胞外的硫酸盐转移至胞内(cys),再通过硫酸盐腺苷酰转移酶(sulfate adenylyl transferase)将硫酸盐(Sulfate)转化为APS(腺苷酰硫酸,Adenylyl sulfate),通过合成酶合成3′-磷酸腺苷-5′-磷酸硫酸酯(3′-phosphoadenosine 5′-phosphosulfate,PAPS);②将硫酸盐转化为腺苷酰硫酸,再通过腺苷酰硫酸还原酶(Adenylylsulfate reductase)还原腺苷酰硫酸为亚硫酸盐,通过sir基因合成亚硫酸盐还原酶还原亚硫酸盐得到硫化物,硫化物再由半胱氨酸合成酶(Cysteine synthase)(cys、MET、ATCYSC)合成L-半胱氨酸,或者硫化物由met基因编码的胱硫醚γ-合成酶(Cystathionine gamma-synthase)生成L-高半胱氨酸(L-Homocysteine)。
2.4.4 胁迫响应 在低pH、低磷且氧化胁迫严重的条件下,微生物会通过自身的抵抗机制克服不利环境,维持自身代谢。如宏基因组分析表明,为抵抗低pH胁迫,微生物可以通过吸收阳离子提高细胞内部的渗透压,以抵抗外部大量H+。将菌株ZY1全基因组基因预测得到的开放阅读框(ORF)与收集得到的砷相关功能基因数据库进行比对,发现了11个砷相关功能基因,其中包括成簇的ars基因家族。
2.5 菌株ZY1砷还原相关基因
根据菌株ZY1砷相关基因信息,初步判断菌株ZY1的砷还原调控机制属于只有ars基因簇调控的砷解毒机制,由于测序原因,arsC、2个arsA和2个arsR的具体位置无法确定,依据全基因组信息可确定其他ars基因的位置,绘制基因调控机制图(图8)。其中为主要参与砷还原的基因簇,arsR负责调控其他的ars基因,砷酸盐由磷酸盐通道进入细胞内,由arsC编码的五价砷还原酶将砷酸盐还原成亚砷酸盐,arsB/acr3编码三价砷转运蛋白,同时arsA编码ATP还原酶,为转运过程提供能量,arsD增强转运蛋白与亚砷酸盐的结合,最终将亚砷酸盐排出胞外,完成解毒过程。实验表明,菌株ZY1具有较好的五价砷耐受能力。

图8 菌株ZY1的砷还原功能基因簇Fig.8 ars-regulated mechanism of ZY1
3 讨 论
砷元素作为一种普遍存在的有毒且致癌的化学元素,广泛分布于各类环境,如地壳、土壤、海水、河水及大气中。除地质变化因素外,更重要的是人为因素,例如金属矿的开采和冶炼、砷产品的加工与使用、煤的燃烧等,导致砷元素污染不断加重。因此,砷污染已经成为较为严重的重金属污染之一,逐渐转变为全球性的问题。微生物作为自然环境中重要的一员,对各种元素及营养物质具有重要的转换作用,具有对砷元素循环的潜质。某些特殊微生物类群具有极强的砷元素适应性,甚至还以砷元素作为必要的生长能源,甚者直接将胞内积累的砷及其化合物,通过自身的氧化还原或甲基化作用对砷元素的不同形态进行转化。利用富集筛选出的抗耐砷微生物,通过多种途径转化砷及其化合物,进而降低砷元素的环境毒性,减轻不同环境砷元素污染问题,实现砷元素污染的微生物修复。
本研究针对湖南石门砷矿区底泥开展砷化物转化微生物资源的富集培养研究,利用纯培养分离获得1株能够进行厌氧砷还原的菌株ZY1,基于16S rRNA基因以及部分生理生化结果初步判定菌株属于梭菌属(Clostridium)。基因组分析结果表明,菌株ZY1具有较好的初级代谢能力,同时,菌株ZY1可以通过两种途径响应pH、重金属和氧化胁迫:一种是ABC转运蛋白(ABC transporters)、双组分系统(Two-component system)、鞭毛装配(Flagellar assembly)、细菌趋药性(Bacterial chemotaxis),通过感受比较不同环境中的化学物质浓度改变自身的运动方向和持续时间,可以通过适当排除体内有毒物质适应艰难的环境;第二种是通过细胞重生,嘌呤、嘧啶代谢和核糖体中的氨酰基-转移核糖核酸(Aminoacyl-tRNA biosynthesis)、同源重组(Homologous recombination)基因的大量存在,表明菌株ZY1的重生能力较强。梭菌属(Clostridium)作为厌氧微生物类群,在特殊生境中进化出适应特殊环境的功能。
梭状芽胞杆菌(Clostridiumsp. ZY1)的基因组信息中注释到3个arsA和3个arsR,可增强其对砷的解毒作用。另外,arsM为甲基砷相关基因,虽然在培养菌株ZY1的溶液中并未检测到甲基砷的存在,但说明了菌株ZY1具有将三价砷转化为甲基砷的潜能,有研究表明一些细菌起始并无甲基化现象,如大肠埃希菌(E.coli)、假单胞菌(Pseudomonassp.)、黄杆菌(Flavobacteriumsp.)、变形杆菌(Proteussp.)经高浓度的砷元素6个月诱导下,可以将无机砷转为甲基态砷化物[32]。因此,推测菌株ZY1的arsM基因可能是来源于自身的基因演化。
菌株ZY1所属的梭菌属(Clostridium)的ars基因簇耐砷原理并未有文献进行深刻分析。2000年,Jones等[33]在研究微生物调节砷还原和砷的溶解性时,从富集培养物中得到1株有五价砷还原作用的梭状芽胞杆菌(Clostridiumsp. CN-8),随后Langner等[34]在探究厌氧条件下微生物还原水溶性五价砷时,对水铁矿吸附五价砷的研究中再次利用梭状芽胞杆菌(Clostridiumsp. CN-8)进行研究。2007年,Stolz等[35]研究畜禽养殖过程中使用硝羟苯胂酸增加砷污染的过程中,发现梭状芽胞杆菌Clostridiumsp. OhILAs能将硝羟苯胂酸转化为低毒的五价砷化物。2016年,Li等[36]在研究砷与硝酸盐污染的地下水环境时,发现梭状芽胞杆菌Clostridiumsp. pxl2能够氧化二价铁离子并降低硝酸盐浓度,同时降低环境中的三价砷化合物的浓度。但是对于梭菌属(Clostridium)的砷元素的转化机理研究并不深入。本研究分离获得的梭状芽胞杆菌(Clostridiumsp. ZY1)表现出五价砷还原能力和代谢潜能。随着现代测序成本的不断降低,梭菌属有很多菌株的基因型已经被测出,但对于该类群的ars基因调控原理是否与其他菌属的有所不同,需要进一步探究。
