单细胞基因组测序技术新进展及其在生物医学中的应用
2021-02-26王卓申笑涵施奇惠
王卓,申笑涵,施奇惠
综 述
单细胞基因组测序技术新进展及其在生物医学中的应用
王卓,申笑涵,施奇惠
复旦大学生物医学研究院, 上海 201100
随着单细胞基因组测序技术的建立与发展,对细胞基因组特征的分析进入了单细胞水平。单细胞的基因组分辨率不但使研究人员能够在单细胞尺度上分析肿瘤细胞的异质性,也使得传统上难以检测的稀有细胞的基因组研究成为可能。这些稀有细胞往往具有重要的生物学意义或临床价值,如癌症患者血液中循环肿瘤细胞(circulating tumor cell, CTC)的基因组检测或三代试管婴儿植入前胚胎细胞的遗传缺陷诊断与筛查(preimplantation genetic diagnosis/screening, PGD/PGS)。本文总结了近年来发展的各种单细胞基因组扩增技术及其优缺点,并介绍了单细胞基因组测序技术在肿瘤生物学和临床检测中的应用,以期为单细胞基因组测序技术在临床检测中应用开发提供参考。
单细胞全基因组;高通量测序;异质性;循环肿瘤细胞
细胞是构成生命体的基本单元,正如物理在原子层面进行研究,化学在分子层面进行研究,生物学基于细胞进行研究。细胞在统一的基因组蓝图和时空特异性调控下,由一个受精卵分化出各种形态、位置、功能不同的细胞,从而构成一个完整的生命体。肿瘤作为一种由基因组异常导致的恶性病变,其细胞之间存在着基因组层面上的异质性,这就使肿瘤成为了一组具有不同基因组特征构成的细胞的集合体。这种异质性的解析对于理解肿瘤的演化规律、耐药机制以及发展有效的治疗方法有着重要的意义。但是,早期的测序技术只能用于包含大量细胞的样本,然后通过计算机重构模拟细胞之间的差异。单细胞测序技术的建立和发展使研究人员能够真正从单细胞水平上研究细胞间的异质性,包括单细胞基因组单碱基突变差异(single-nucleotide variants, SNV)、短序列插入/缺失(insertions and deletions, Indel)和拷贝数变异(copy number variants, CNV)等,以及单细胞基因表达差异和单细胞蛋白修饰差异等[1,2]。除了肿瘤异质性研究以外,单细胞测序所具有的分辨率也使得对具有重要生物学意义或临床价值的稀有细胞的基因组研究成为可能,如循环肿瘤细胞(circulating tumor cell, CTC)或三代试管婴儿植入前的遗传缺陷诊断与筛查(preimplantation genetic diagnosis/screening, PGD/PGS)。单细胞基因组测序主要包括3个部分:单细胞获取、单细胞全基因组扩增和扩增产物测序及分析。本文总结了近年来单细胞基因组扩增技术的发展及优缺点,并介绍了单细胞基因组测序技术在肿瘤生物学和临床检测中的应用。
1 单细胞的获取
单细胞测序首先需要获取感兴趣的单细胞样本。研究中通常采用机械剪切结合酶解方法将组织消化成单细胞悬液,单细胞消化过程为了避免消化不充分从而导致细胞类型不均一,通常采用多种水解酶混合使用,单细胞样本的回收根据实验需要选择合适的技术平台(表1)。使用口吸管[3]或显微操作仪(micromanipulation)[4]回收单细胞,可以借助显微镜直观的观察目标单细胞,可视化、准确、回收成功率高,但对操作人员技术要求较高,单细胞回收通量比较低。口吸管或显微操作技术适用于目标细胞较少且珍贵样本,如循环肿瘤细胞或辅助生殖移植前胚胎细胞。流式细胞分选技术(fluorescence activated cell sorting, FACS)[5]的应用非常广泛,FACS可以高通量、高效的将目标单细胞样本回收到96孔板或384孔板中,与高通量、标准化实验操作兼容。如果借助荧光标记的单克隆抗体可以标记细胞亚群,FACS还可以有选择的回收某一特定类型单细胞。缺点是FACS对细胞有一定损伤,对起始细胞数量有一定要求,细胞数量较少的细胞亚群或珍贵样本不适合使用FACS进行单细胞回收。激光显微切割捕获技术(laser-capture microdissection, LCM)[6]通常被用于分离回收固定染色切片上的目标细胞样本,LCM技术的优点是可以确定单细胞在组织样本中的空间位置,但设备操作者需要对组织样本非常熟悉,因为是从组织原位回收目的细胞,需要操作者分辨出目标细胞和非目标细胞[7],对操作者技术要求也比较高,LCM是唯一能够获取目标细胞空间位置的技术。微流控芯片平台[8,9]用于单细胞回收的优点是通量高,效率高,自动化程度高,能够与下游分子生物学反应集成化,全部在微流控芯片上完成,降低污染,降低人为操作导致的实验偏差,反应体积小,反应效率高,节约试剂使用量,但微流控平台对技术要求比较高,如果自己搭建微流控芯片平台很多普通生物学实验室没有相关技术支持,使用全自动商业化仪器,设备成本较高。
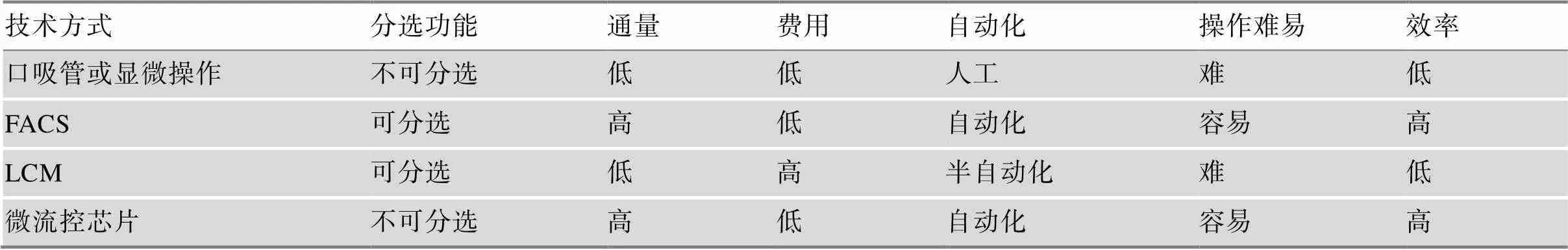
表1 单细胞分离技术优缺点比较
2 单细胞全基因组扩增技术的发展
哺乳动物由受精卵发育成完整个体,受精卵基因组遗传自父亲和母亲,分析基因组特定基因碱基序列可以分析家系遗传追踪。肿瘤细胞积累新的基因突变可以形成新的肿瘤细胞克隆亚群。应用基因测序可以分析肿瘤细胞基因组单碱基突变、插入和缺失、拷贝数变异和染色体结构变异。单细胞基因组DNA大约5.6 pg,满足不了测序要求,即使最新的三代测序技术对样本量要求降低很多[10],也需要对单细胞基因组进行预扩增。单细胞培养可以实现核酸在胞内的扩增,但细胞培养只适用于少量细胞类型,如部分干细胞、肿瘤细胞系等,临床检测所分离的稀有细胞原代培养成功率还不高,暂时不具备普遍应用的价值,肿瘤细胞在人为培养下,可能会积累新的基因突变,因此临床上单细胞基因组预扩增主要使用体外试剂扩增技术。单细胞基因组预扩增技术主要有基于聚合酶链式反应(polymerase chain reaction, PCR)扩增、多重置换扩增(multiple displacement amplification, MDA)和体外转录(transcription technology, IVT)等技术(图1)。
2.1 基于PCR的单细胞基因组扩增技术
随机寡核苷酸引物PCR技术(primer-extension preamplification PCR, PEP-PCR)[11,12]或简并引物PCR技术(degenerate oligonucleotide-primed PCR, DOP-PCR)[13,14]首先应用引物随机结合在单细胞基因组上,通过PCR反应随机扩增全基因组序列。而接头介导的PCR技术(ligation-mediated PCR, LM-PCR)[15]则是使用限制性内切酶Mse I在基因组5ʹ-TTAA-3ʹ位置随机切断单细胞基因组DNA,然后在DNA片段末端加上扩增引物,也是通过PCR反应扩增基因组DNA片段。PEP-PCR、DOP-PCR和LM-PCR技术作为单细胞基因组DNA扩增技术的雏形,最先实现了单细胞基因组DNA的扩增,满足了某些研究的需要。但PCR扩增反应具有碱基偏好性导致基因组扩增存在偏向性,全基因组扩增产物覆盖度不足10%[16],PCR扩增反应碱基错配率偏高,此类单细胞基因组扩增技术不适用于检测基因点突变,假阳性率较高。
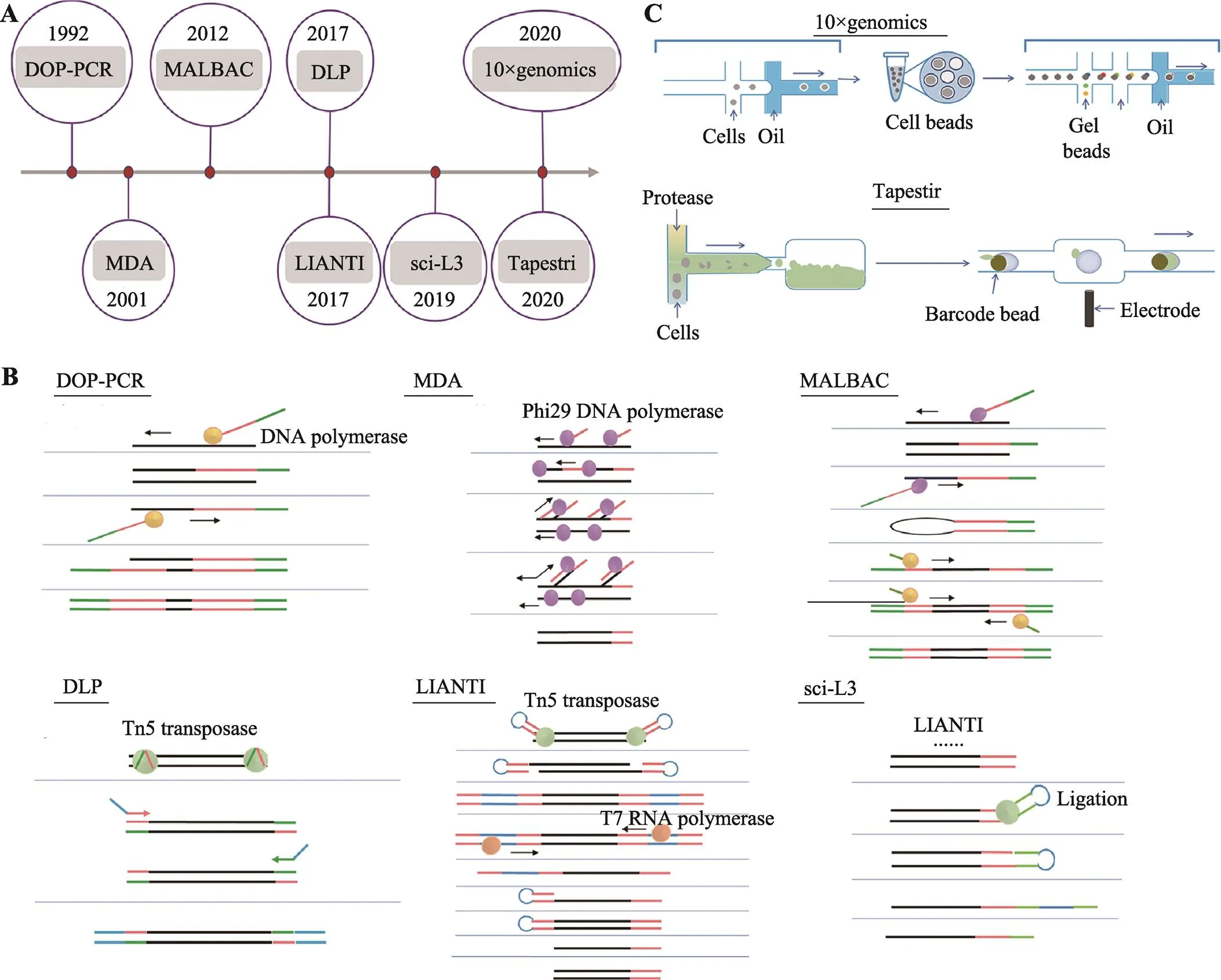
图1 单细胞全基因组扩增技术的发展
A:单细胞全基因组扩增技术发展时间轴;B:人工单细胞全基因组扩增技术;C:自动化单细胞基因组扩增技术。
2.2 多重置换扩增技术
Dean等[17,18]创造性的开发了新的单细胞基因组扩增方法—多重置换扩增技术,phi29 DNA聚合酶在恒温条件下扩增单细胞基因组,phi29 DNA聚合酶具有很强DNA合成活性和链置换活性,扩增产物可大于10 kb,以新合成的DNA子链为模板,继续合成新的DNA子链,所以MDA反应后扩增产量高,而且phi29 DNA聚合酶保真性好,与PCR反应使用的DNA聚合酶相比,扩增产物保真度提高1000倍,基因组扩增覆盖度更高,MDA技术比较适合检测单细胞基因组SNV,但MDA指数扩增会导致有些基因组区域偏向性[19],因此不适用于基因组CNV检测,另外,高效的phi29 DNA聚合酶会导致基因融合和等位基因丢失的现象[20]。为了解决MDA指数扩增所导致的偏向性,Fu等[21]将单细胞基因组随机片段化,借助微流控芯片生成的乳滴将DNA片段和预扩增试剂包裹起来,形成小的空间,借助乳滴中扩增反应最小限制因子来终止扩增反应,避免了传统MDA技术基因组扩增时,基因组某些区域无限扩增的偏向性,同时借助乳滴物理隔离DNA片段,降低了单细胞扩增时人为导致的基因融合,但phi29 DNA聚合酶对不完整DNA扩增效率较低,可能会降低扩增产物基因组覆盖度。MDA技术比较适合新鲜样本的单细胞基因组扩增,不适合用于固定后的单细胞样本。
2.3 多次退火环状循环扩增技术
多次退火环状循环扩增技术(multiple annealing and looping-based amplification cycles, MALBAC)[22]将PCR和MDA技术联合,同时兼顾了单细胞基因组扩增的保真度和均一性。MALBAC单细胞基因组扩增分为两个过程:前5个循环采用Bst DNA聚合酶完成多重置换扩增,完整的扩增产物两端引入互补序列可以形成loop结构,封闭新扩增的DNA子链,避免新合成的DNA子链被当作扩增反应DNA模板;PCR扩增利用高温变性将完整的扩增子loop结构打开,经过PCR反应完成单细胞基因组扩增。MALBAC技术利用loop结构抑制了指数扩增,尽可能的从单细胞原始基因组模板合成子链DNA,第二步扩增使用PCR方法尽可能的保证扩增产物的均一性。MALBAC技术第一步应用多重置换反应将扩增产物全基因组覆盖度提高到90%以上,在一定程度保证了扩增产物的保真性,适用于SNV的检测,同时loop结构巧妙的应用实现了单细胞基因组准线性扩增,抑制了第一步反应指数扩增,尽可能的从单细胞原始基因组模板合成子链DNA,保证了扩增产物的均一性,可用于基因组拷贝数变异的检测。MALBAC技术可以同时用于新鲜样本和固定后的单细胞样本的单细胞基因组扩增,MALBAC技术的应用促进了临床辅助生殖技术的发展[23]。
2.4 单细胞基因组线性扩增技术
Tn5转座酶系统因其可以在基因组DNA上随机打断,在打断位置插入一段已知序列,并且稳定性好的特点,已经成为分子遗传研究和基因诊断应用的常用工具酶[24]。通过转座子介导的单细胞基因组线性扩增技术(linear amplification via transposon insertion, LIANTI)[25]利用Tn5转座酶打断基因组DNA时引入T7启动子,通过体外转录以基因组DNA片段为模板,大量合成mRNA,再通过mRNA合成大量cDNA双链,每次转录合成mRNA都是以最原始的DNA片段为模板,实现了单细胞基因组线性扩增。LIANTI技术使得单细胞基因组测序覆盖提高到97%,同时兼顾了扩增的均一性,而且降低了扩增错误率,等位基因丢失率仅17%。LIANTI技术可同时适用于检测单细胞SNV、Indel和CNV。实验中扩增后的cDNA双链仍然使用传统的高通量测序文库构建方法,仍然是每个单细胞扩增后的DNA片段单独构建文库,所以LIANTI测序通量有限。
2.5 高通量单细胞基因组扩增技术
肿瘤细胞基因组进化重建研究往往需要一次性研究大量肿瘤单细胞基因组突变特征,低通量的单细胞测序技术成本高,高人力成本。2017年,加拿大英属哥伦比亚大学的Hansen课题组和美国俄勒冈健康与科学大学的Adey课题组同时发表文章,报道利用Tn5转座酶开发了大规模、低成本的单细胞基因组CNV测序技术[26,27]。直接建库技术(direct library preparation, DLP)[26]借助微流控平台实现单细胞样本的获取,利用Tn5转座酶打断DNA引入接头时并没有添加index,通过11个PCR循环反应引入index和测序接头,将多个单细胞文库混合后进行基因测序。单细胞组合标签测序技术(single-cell combinatorial index sequencing, SCI-seq)[27]使用FACS将单细胞回收到96孔板中,在Tn5转座酶打断DNA引入接头时添加index1,然后将添加index1的DNA片段混匀后重新分组,通过PCR循环反应引入index2,再将文库混匀后进行基因测序。DLP和SCI-seq技术适用于高通量的、低成本单细胞CNV分析,由于Tn5转座酶系统酶切后的DNA片段存在一些大片段,超出了二代测序读长范围,导致基因组覆盖度降低,所以不适用于单细胞SNV和Indel检测,而且DLP和SCI-seq技术在PCR扩增过程中仍然会引入一定偏向性。为了降低PCR扩增DNA片段带来的偏向性,Yin等[28]将sci技术和LIANTI技术结合并进行了优化,开发了一种高通量单细胞基因组线性扩增技术(sci-L3-WGS)。借助sci组合标签技术,sci-L3-WGS大大提高了LIANTI技术的通量。第一轮加标签,Tn5转座酶随机打断单细胞基因组并加上标签1,第二轮加标签2和T7转录启动子序列连接到DNA片段末端,T7转录启动子的引入,可以实现体外转录反应,以DNA片段为模板,线性扩增的方式合成大量mRNA,再将mRNA逆转录合成cDNA,合成cDNA第二条链的时候再引入标签3,最后在DNA片段末端加上测序接头,完成的文库DNA片段含有3种标签组合,因此sci-L3- WGS技术可以实现高通量单细胞基因组线性扩增。sci-L3-WGS技术细胞回收率高达90%,以更少的测序数据达到更高覆盖度,单细胞基因组可覆盖约97,000片段,测序数据高达86%为有效reads,比LIANTI技术61%的有效reads提高了25%。
以上高通量技术虽然一定程度上解决了单细胞基因组测序细胞通量问题,自动化平台则可以进一步减少人为操作,大大提高实验效率。Tapestri技术平台最先使用微流控平台实现单细胞多基因突变检测[29],采用油包水的方式高通量的分离单细胞,使用多重PCR特异性扩增多个目标区域靶序列,引入barcode,通过高通量测序实现单细胞靶基因SNV检测,同时利用扩增的目标片段可以低分辨率的分析单细胞CNV,只是扩增区域有限,覆盖度不是很高。单细胞捕获效率有限,需要一次性上机大量单细胞样本,不适合少量单细胞样本的检测。10× genomics是另一款基于微流控自动化、高通量的检测单细胞基因组拷贝数变异的平台[30],10×genomics进行单细胞基因组测序时需要进行两次捕获,第一次将磁球与单细胞包裹在一起,通过磁球上引物随机扩增单细胞基因组片段,第二次将凝胶球与磁球包裹起来,将barcode标记到单细胞DNA片段上,一次操作可以检测数千个肿瘤单细胞基因组拷贝数变异,进而基于肿瘤单细胞CNV特征对肿瘤细胞进行分群,因为需要进行两次捕获,所以捕获率只有15%左右。
3 单细胞基因组测序在生物医学中的应用
3.1 单细胞基因组测序用于肿瘤异质性研究
肿瘤组织由肿瘤祖细胞发展而来,在肿瘤形成过程中,肿瘤细胞不断积累新的基因突变,形成肿瘤内异质性。肿瘤异质性是肿瘤复发、转移和耐药的主要因素之一。研究肿瘤异质性与肿瘤临床诊断和治疗息息相关。肿瘤基因测序通常是提取肿瘤组织中大量细胞基因组DNA,检测肿瘤细胞基因突变,但这样检测的基因突变结果代表的是多种类型细胞平均化的结果。单细胞基因组扩增技术的建立,可以从单细胞水平研究肿瘤细胞基因突变,进而比较肿瘤单细胞之间的基因突变异质性。肿瘤异质性研究早期,单细胞基因组扩增主要基于PCR技术完成,虽然扩增后的基因组覆盖度有限,仍然可以满足基因组拷贝数变异的分析。
Klein等[31]从乳腺癌患者骨髓中分离到稀有肿瘤细胞,扩增肿瘤细胞单细胞基因组,利用比较基因组杂交技术探索肿瘤但细胞CNV突变分析。Martelotto 等[32]基于PCR技术建立了肿瘤组织石蜡切片(formalin-fixed paraffin-embedded, FFPE)样本单细胞CNV 分析方法。应用NGS建立了一种重现性较好、准确度较高的FFPE样本单细胞CNV 分析方法,有助于利用临床肿瘤FFPE样本,研究肿瘤进化机制。美国德州大学MD安德森癌症中心Navin课题组应用单细胞基因组测序技术分析乳腺癌肿瘤单细胞CNV特征,加深了对乳腺癌肿瘤细胞进化机制的理解;应用单细胞基因组测序技术比较了原位肿瘤组织和转移肿瘤组织中肿瘤单细胞CNV变异差异,结果发现转移灶肿瘤细胞与原位灶肿瘤细胞具有类似的CNV变异,通过肿瘤单细胞CNV分析明确了乳腺癌转移确实是由原位肿瘤进展转移到其他器官形成转移病灶的,而不是由完全不同的独立肿瘤细胞克隆形成的[16]。通过乳腺癌肿瘤单细胞CNV分析,确定了乳腺癌肿瘤细胞“爆发式”进化模式,即肿瘤细胞CNV突变在乳腺癌原位肿瘤病灶早期就已经出现,并形成单克隆或多克隆,在一段时间内保持一定的稳定[33]。应用空间单细胞基因组测序技术研究乳腺原位导管癌和侵袭性导管癌肿瘤细胞克隆组成,揭示了乳腺癌多克隆共转移模式。肿瘤原位灶肿瘤细胞多个克隆可以同时转移到新的位置形成转移病灶[34]。
MDA技术的建立大大提高了单细胞SNV和Indel的检测,Xu等[35]应用MDA技术详细的描绘了一位肾癌患者肿瘤单细胞SNV突变谱,从单细胞水平证明了肾癌相关突变基因VHL和PBRM1可能与肿瘤发生没有直接关系,而且此肾癌患者肿瘤组织中并没有鉴定出明显的主克隆亚群,通过单细胞基因突变检测预示着肿瘤异质性要比之前的认识复杂的多。Hou等[36]和Wang等[37]应用单细胞测序分析了骨髓瘤和乳腺癌单细胞SNV突变谱,揭示了肿瘤单细胞之间碱基突变异质性。应用Tapestri高通量技术平台,在急性髓细胞性白血病(acute myeloid leukemia, AML)患者治疗后肿瘤单细胞中检测到携带NRAS、KRAS和FLT3突变的,提供了新的用药靶点[38]。虽然肿瘤细胞系具有均匀的染色体拷贝数变异,但Velazquez-Villarreal等[30]借助10×genomics高通量单细胞DNA拷贝数检测平台,从单细胞水平揭示了肿瘤细胞系单细胞之间拷贝数变异差异,发现了肿瘤细胞系中多个亚克隆,提示肿瘤细胞系较我们之前认识的更复杂。
3.2 单细胞基因组测序用于CTC检测
单细胞测序技术的发展促进了稀有细胞的检测与应用。血液CTC是由肿瘤组织脱落进入血液循环系统的肿瘤细胞,CTC计数可以用于患者预后疗效评估。Jonas等[39]应用单细胞测序分析了非转移性乳腺癌患者骨髓中播散肿瘤细胞(disseminated tumor cell, DTC)和原位肿瘤基因突变特征,确定了DTC的来源。Carlotta等[40]应用单细胞测序分析了脑转移乳腺癌患者CTC基因突变特征,揭示了乳腺癌脑转移相关信号通路基因突变潜在靶点。应用单细胞测序比较肺癌肺静脉血和外周血中CTC和原位肿瘤细胞及转移病灶肿瘤细胞基因突变特征,结果发现CTC携带了一部分原位肿瘤细胞基因突变特征,而与转移病灶肿瘤细胞基因突变特征更相近,明确了CTC是肺癌转移的中间途径[41,42]。Carter 等[43]和Su等[44]在各自研究中,应用单细胞测序检测了小细胞肺癌患者外周血中CTC单细胞CNV 变异特征,基于CTC单细胞CNV突变特征提出了患者接受化疗反应预测模型,用于患者化疗预后评估。吴保军等[45]应用单细胞基因组CNV变异分析,从肺癌患者恶性胸腔积液中鉴定出携带不同CNV变异的肿瘤细胞亚群,从单细胞水平揭示了肺癌患者恶性肿瘤细胞异质性。
3.3 单细胞基因组测序用于胚胎移植前基因诊断和筛查
胚胎移植前PGD主要用于具有家族式遗传病家庭进行试管婴儿移植前胚胎基因诊断,可以确保新生儿不携带相关基因异常而进行的胚胎移植前基因检测;胚胎移植前PGS主要是针对体外受精胚胎进行的染色体倒位等异常的筛查。通常用来做PGD 或PGS的样本为极体或囊胚期单细胞,能够用于检测的DNA含量很少,并且能够检测的靶点比较少。单细胞基因组高通量测序技术的发展大大提高了PGD或PGS检测成功率:如CGH芯片、SNP杂交芯片以及NGS测序。Wells等[46]借助DOP-PCR单细胞扩增技术第一次完成了第一极体单细胞全基因组扩增,并采用CGH芯片成功检测到胚胎染色体异常情况。Daina等[47]使用MDA扩增胚胎活检单细胞样本,第一次完成了Lynch综合征家庭开展单细胞双因子PGD,可以从体外培养的胚胎中挑选既健康又具有整数倍性的胚胎用于胚胎移植。Tobler等[48]回顾性分析了498个胚胎的PGD/PGS结果,比较了SNP 芯片和CGH 杂交芯片用于MDA扩增胚胎单细胞染色体异常检测情况,提示SNP芯片和CGH杂交芯片具有类似的异常检出率。Huang等[49]利用3位孕妇捐献23枚胚胎样本,应用MALBAC技术,比较了MALBAC-scWGS技术、SNP芯片及CGH芯片用于PGD/PGS的准确性,MALBAC-scWGS检测结果与SNP芯片或者CGH芯片结果重复性达到78.26%,其中有8个胚胎使用三种检测技术的检测结果完全一致,验证了MALBAC-scWGS在PGD/ PGS检测中的应用价值。Wang等[50]将此技术应用拓展到线粒体基因异常性疾病的PGD/PGS检测中。单细胞基因组测序技术的发展极大的提高了临床产前诊断技术的应用和准确性。
4 结语与展望
单细胞基因组扩增技术和高通量测序技术相结合极大的促进了单细胞基因组测序技术的发展,使得研究细胞之间基因差异达到单细胞水平。应用单细胞基因组测序技术,研究者对肿瘤多样性、肿瘤异质性以及肿瘤克隆进化机制不断加深理解,同时单细胞基因组测序技术促进了CTC检测和PGD/ PGS临床的应用。多种单细胞基因组测序技术各有优缺点,在科研或临床应用时应该根据实际需求选择合适的单细胞基因组测序技术。单细胞基因组测序技术的应用促进了肿瘤异质性研究,但仍然需要发展能够同时兼顾基因组扩增保真性和均一性的技术,提高肿瘤单细胞SNV、CNV、SV等基因突变的检测。随着肿瘤单细胞研究不断深入,人们对单细胞多组学的需求会越来越高。Macaulay等[51]最早将单细胞基因组和转录组测序联合使用,同时检测同一个单细胞样本的基因组和转录组,通过单细胞测序发现HCC38-BL细胞系10%细胞存在11号染色体三倍体,并通过FISH技术做了验证,Tang等[52]应用单细胞基因组和转录组测序技术分析了黑色素瘤患者不同部位肿瘤单细胞的突变负荷和基因表达特征。肿瘤细胞基因突变负荷检测揭示了被遮蔽的部位黑色素细胞突变负荷少于暴露于阳光的部位,间歇性暴露于阳关的背部和四肢的突变负荷多于脸部、脖子等暴露在阳光下的部位。Zhou等[53]联合单细胞基因组和转录组测序技术对结直肠癌肿瘤组织、淋巴结转移组织、癌旁组织、外周血样品以及健康人外周血中非上皮细胞进行了单细胞测序。研究发现人体正常组织中免疫细胞、成纤维细胞以及血管内皮细胞中广泛存在少量正常携带拷贝数变异。转录组测序分析在肿瘤组织成纤维细胞中BGN、RCN3、TAGLN、MYL9和TPM2表达量明显上调,鉴定出肿瘤微环境中成纤维细胞特异性表达的肿瘤标志基因。Hou等[54]又开发了单细胞基因组、甲基化组和转录组测序技术,Zachariadis等[55]应用Tn5转座子系统进一步提高了单细胞基因组和转录组测序的细胞通量。未来单细胞组学技术开发不仅仅考虑提高细胞通量,多组学技术联合开发,同时还需要考虑增加组织空间位置信息。随着技术的发展,单细胞测序技术会展现出更强的应用价值,供研究者选择在科学研究中选择。
[1] Zhou Y, Wang CJ, Zhu CC, Chen JR, Cheng M, Deng YL, Guo Y. Single-cell gene variation analysis method for single gland., 2017, 39(8): 753–762.周彦, 王超杰, 朱纯超, 陈江荣, 程酩, 邓宇亮, 郭妍. 组织单腺体内单细胞的基因变异分析方法. 遗传, 2017, 39(8): 753–762.
[2] Zhao LN, Wang N, Yang GL, Su XB, Han ZG. A method for reliable detection of genomic point mutations based on single-cell target-sequencing., 2020, 42(7): 703–712.赵利楠, 王娜, 杨国良, 苏现斌, 韩泽广. 基于单细胞靶向测序探究基因碱基突变的方法. 遗传, 2020, 42(7): 703–712
[3] Nagano T, Lubling Y, Stevens TJ, Schoenfelder S, Yaffe E, Dean W, Laue ED, Tanay A, Fraser P. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure., 2013, 502(7469): 59–64.
[4] Babbe H, Roers A, Waisman A, Lassmann H, Goebels N, Hohlfeld R, Friese M, Schröder R, Deckert M, Schmidt S, Ravid R, Rajewsky K. Clonal expansions of CD8(+) T cells dominate the T cell infiltrate in active multiple sclerosis lesions as shown by micromanipulation and single cell polymerase chain reaction., 2000, 192(3): 393–404.
[5] Dalerba P, Kalisky T, Sahoo D, Rajendran PS, Rothenberg ME, Leyrat AA, Sim S, Okamoto J, Johnston DM, Qian DL, Zabala M, Bueno J, Neff NF, Wang JB, Shelton AA, Visser B, Hisamori S, Shimono Y, van de Wetering M, Clevers H, Clarke MF, Quake SR. Single-cell dissection of transcriptional heterogeneity in human colon tumors., 2011, 29(12): 1120–1127.
[6] Frumkin D, Wasserstrom A, Itzkovitz S, Harmelin A, Rechavi G, Shapiro E. Amplification of multiple genomic loci from single cells isolated by laser micro-dissection of tissues., 2008, 8: 17.
[7] Bhattacherjee V, Mukhopadhyay P, Singh S, Roberts EA, Hackmiller RC, Greene RM, Pisano MM. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells., 2004, 39(1): 58–64.
[8] Marcus JS, Anderson WF, Quake SR. Microfluidic single-cell mRNA isolation and analysis., 2006, 78(9): 3084–3089.
[9] White AK, VanInsberghe M, Petriv OI, Hamidi M, Sikorski D, Marra MA, Piret J, Aparicio S, Hansen CL. High-throughput microfluidic single-cell RT-qPCR., 2011, 108(34): 13999–14004.
[10] Pushkarev D, Neff NF, Quake SR. Single-molecule sequencing of an individual human genome., 2009, 27(9): 847–850.
[11] Snabes MC, Chong SS, Subramanian SB, Kristjansson K, DiSepio D, Hughes MR. Preimplantation single-cell analysis of multiple genetic loci by whole-genome amplification., 1994, 91(13): 6181–6185.
[12] Zhang L, Cui XF, Schmitt K, Hubert R, Navidi W, Arnheim N. Whole genome amplification from a single cell: implications for genetic analysis., 1992, 89(13): 5847–5851.
[13] Telenius H, Carter NP, Bebb CE, Nordenskjöld M, Ponder BA, Tunnacliffe A. Degenerate oligonucleotide-primed PCR: general amplification of target DNA by a single degenerate primer., 1992, 13(2): 718–725.
[14] Cheung VG, Nelson SF. Whole genome amplification using a degenerate oligonucleotide primer allows hundreds of genotypes to be performed on less than one nanogram of genomic DNA., 1996, 93(25): 14676–14679.
[15] Klein CA, Schmidt-Kittler O, Schardt JA, Pantel K, Speicher MR, Riethmüller G. Comparative genomic hybridization, loss of heterozygosity, and DNA sequence analysis of single cells., 1999, 96(8): 4494–4499.
[16] Navin N, Kendall J, Troge J, Andrews P, Rodgers L, McIndoo J, Cook K, Stepansky A, Levy D, Esposito D, Muthuswamy L, Krasnitz A, McCombie WR, Hicks J, Wigler M. Tumour evolution inferred by single-cell sequencing., 2011, 472(7341): 90–94.
[17] Dean FB, Nelson JR, Giesler TL, Lasken RS. Rapid amplification of plasmid and phage DNA using Phi 29 DNA polymerase and multiply-primed rolling circle amplification., 2001, 11(6): 1095–1099.
[18] Dean FB, Hosono S, Fang LH, Wu XH, Faruqi AF, Bray-Ward P, Sun ZY, Zong QL, Du YF, Du J, Driscoll M, Song WM, Kingsmore SF, Egholm M, Lasken RS. Comprehensive human genome amplification using multiple displacement amplification., 2002, 99(8): 5261–5266.
[19] de Bourcy CF, De Vlaminck I, Kanbar JN, Wang JB, Gawad C, Quake SR. A quantitative comparison of single-cell whole genome amplification methods., 2014, 9(8): e105585.
[20] Lasken RS, Stockwell TB. Mechanism of chimera formation during the multiple displacement amplification reaction., 2007, 7: 19.
[21] Fu YS, Li CM, Lu SJ, Zhou WX, Tang FC, Xie XS, Huang YY. Uniform and accurate single-cell sequencing based on emulsion whole-genome amplification., 2015, 112(38): 11923–11928.
[22] Zong CH, Lu SJ, Chapman AR, Xie XS. Genome-wide detection of single-nucleotide and copy-number variations of a single human cell., 2012, 338(6114): 1622– 1626.
[23] Yao YX, La YF, Di R, Liu QY, Hu WP, Wang XY, Chu MX. Comparison of different single cell whole genome amplification methods and MALBAC applications in assisted reproduction., 2018, 40(8): 620–631.姚雅馨, 喇永富, 狄冉, 刘秋月, 胡文萍, 王翔宇, 储明星. 不同单细胞全基因组扩增方法的比较及MALBAC在辅助生殖中的应用. 遗传, 2018, 40(8): 620–631.
[24] Picelli S, Björklund AK, Reinius B, Sagasser S, Winberg G, Sandberg R. Tn5 transposase and tagmentation procedures for massively scaled sequencing projects., 2014, 24(12): 2033–2040.
[25] Chen CY, Xing D, Tan LZ, Li H, Zhou GY, Huang L, Xie XS. Single-cell whole-genome analyses by Linear Amplification via Transposon Insertion (LIANTI)., 2017, 356(6334): 189–194.
[26] Zahn H, Steif A, Laks E, Eirew P, VanInsberghe M, Shah SP, Aparicio S, Hansen CL. Scalable whole-genome single-cell library preparation without preamplification., 2017, 14(2): 167–173.
[27] Vitak SA, Torkenczy KA, Rosenkrantz JL, Fields AJ, Christiansen L, Wong MH, Carbone L, Steemers FJ, Adey A. Sequencing thousands of single-cell genomes with combinatorial indexing., 2017, 14(3): 302– 308.
[28] Yin Y, Jiang Y, Lam KWG, Berletch JB, Disteche CM, Noble WS, Steemers FJ, Camerini-Otero RD, Adey AC, Shendure J. High-throughput single-cell sequencing with linear amplification., 2019, 76(4): 676–690.e10.
[29] Pellegrino M, Sciambi A, Treusch S, Durruthy-Durruthy R, Gokhale K, Jacob J, Chen TX, Geis JA, Oldham W, Matthews J, Kantarjian H, Futreal PA, Patel K, Jones KW, Takahashi K, Eastburn DJ. High-throughput single-cell DNA sequencing of acute myeloid leukemia tumors with droplet microfluidics., 2018, 28(9): 1345– 1352.
[30] Velazquez-Villarreal EI, Maheshwari S, Sorenson J, Fiddes IT, Kumar V, Yin YF, Webb MG, Catalanotti C, Grigorova M, Edwards PA, Carpten JD, Craig DW. Single-cell sequencing of genomic DNA resolves sub-clonal heterogeneity in a melanoma cell line., 2020, 3(1): 318.
[31] Klein CA, Schmidt-Kittler O, Schardt JA, Pantel K, Speicher MR, Riethmüller G. Comparative genomic hybridization, loss of heterozygosity, and DNA sequence analysis of single cells., 1999, 96(8): 4494–4499.
[32] Martelotto LG, Baslan T, Kendall J, Geyer FC, Burke KA, Spraggon L, Piscuoglio S, Chadalavada K, Nanjangud G, Ng CKY, Moody P, D'Italia S, Rodgers L, Cox H, da Cruz Paula A, Stepansky A, Schizas M, Wen HY, King TA, Norton L, Weigelt B, Hicks JB, Reis-Filho JS. Whole-genome single-cell copy number profiling from formalin-fixed paraffin-embedded samples., 2017, 23(3): 376–385.
[33] Gao RL, Davis A, McDonald TO, Sei E, Shi XQ, Wang Y, Tsai PC, Casasent A, Waters J, Zhang H, Meric-Bernstam F, Michor F, Navin NE. Punctuated copy number evolution and clonal stasis in triple-negative breast cancer., 2016, 48(10): 1119–1130.
[34] Casasent AK, Schalck A, Gao RL, Sei E, Long A, Pangburn W, Casasent T, Meric-Bernstam F, Edgerton ME, Navin NE. Multiclonal invasion in breast tumors identified by topographic single cell sequencing., 2018, 172(1–2): 205–217.e212.
[35] Xu X, Hou Y, Yin XY, Bao L, Tang AF, Song LT, Li FQ, Tsang S, Wu K, Wu HJ, He WM, Zeng L, Xing MJ, Wu RH, Jiang H, Liu X, Cao DD, Guo GW, Hu XD, Gui YT, Li ZS, Xie WY, Sun XJ, Shi M, Cai ZM, Wang B, Zhong MM, Li JX, Lu ZH, Gu N, Zhang XQ, Goodman L, Bolund L, Wang J, Yang HM, Kristiansen K, Dean M, Li YR, Wang J. Single-cell exome sequencing reveals single- nucleotide mutation characteristics of a kidney tumor., 2012, 148(5): 886–895.
[36] Hou Y, Song LT, Zhu P, Zhang B, Tao Y, Xu X, Li FQ, Wu K, Liang J, Shao D, Wu HJ, Ye XF, Ye C, Wu RH, Jian M, Chen Y, Xie W, Zhang RR, Chen L, Liu X, Yao XT, Zheng HC, Yu C, Li QB, Gong ZL, Mao M, Yang X, Yang L, Li JX, Wang W, Lu ZH, Gu N, Laurie G, Bolund L, Kristiansen K, Wang J, Yang HM, Li Y, Zhang XQ, Wang J. Single-cell exome sequencing and monoclonal evolution of a JAK2-negative myeloproliferative neoplasm., 2012, 148(5): 873–885.
[37] Wang Y, Waters J, Leung ML, Unruh A, Roh W, Shi XQ, Chen K, Scheet P, Vattathil S, Liang H, Multani A, Zhang H, Zhao R, Michor F, Meric-Bernstam F, Navin NE. Clonal evolution in breast cancer revealed by single nucleus genome sequencing., 2014, 512(7513): 155–160.
[38] McMahon CM, Ferng T, Canaani J, Wang ES, Morrissette JJD, Eastburn DJ, Pellegrino M, Durruthy-Durruthy R, Watt CD, Asthana S, Lasater EA, DeFilippis R, Peretz CAC, McGary LHF, Deihimi S, Logan AC, Luger SM, Shah NP, Carroll M, Smith CC, Perl AE. Clonal selection with Ras pathway activation mediates secondary clinical resistance to selective FLT3 inhibition in acute myeloid leukemia., 2019, 9(8): 1050–1063.
[39] Demeulemeester J, Kumar P, Møller EK, Nord S, Wedge DC, Peterson A, Mathiesen RR, Fjelldal R, Zamani Esteki M, Theunis K, Fernandez Gallardo E, Grundstad AJ, Borgen E, Baumbusch LO, Børresen-Dale AL, White KP, Kristensen VN, Van Loo P, Voet T, Naume B. Tracing the origin of disseminated tumor cells in breast cancer using single-cell sequencing., 2016, 17(1): 250.
[40] Riebensahm C, Joosse SA, Mohme M, Hanssen A, Matschke J, Goy Y, Witzel I, Lamszus K, Kropidlowski J, Petersen C, Kolb-Kokocinski A, Sauer S, Borgmann K, Glatzel M, Müller V, Westphal M, Riethdorf S, Pantel K, Wikman H. Clonality of circulating tumor cells in breast cancer brain metastasis patients., 2019, 21(1): 101.
[41] Chemi F, Rothwell DG, McGranahan N, Gulati S, Abbosh C, Pearce SP, Zhou C, Wilson GA, Jamal-Hanjani M, Birkbak N, Pierce J, Kim CS, Ferdous S, Burt DJ, Slane-Tan D, Gomes F, Moore D, Shah R, Al Bakir MA, Hiley C, Veeriah S, Summers Y, Crosbie P, Ward S, Mesquita B, Dynowski M, Biswas D, Tugwood J, Blackhall F, Miller C, Hackshaw A, Brady G, Swanton C, Dive C, Consortium T. Pulmonary venous circulating tumor cell dissemination before tumor resection and disease relapse., 2019, 25(10): 1534–1539.
[42] Ni XH, Zhuo ML, Su Z, Duan JC, Gao Y, Wang ZJ, Zong CH, Bai H, Chapman AR, Zhao J, Xu LY, An TT, Ma Q, Wang YY, Wu MN, Sun Y, Wang SH, Li ZX, Yang XD, Yong J, Su XD, Lu YY, Bai F, Xie XS, Wang J. Reproducible copy number variation patterns among single circulating tumor cells of lung cancer patients., 2013, 110(52): 21083–21088.
[43] Carter L, Rothwell DG, Mesquita B, Smowton C, Leong HS, Fernandez-Gutierrez F, Li YY, Burt DJ, Antonello J, Morrow CJ, Hodgkinson CL, Morris K, Priest L, Carter M, Miller C, Hughes A, Blackhall F, Dive C, Brady G. Molecular analysis of circulating tumor cells identifies distinct copy-number profiles in patients with chemosensitive and chemorefractory small-cell lung cancer., 2017, 23(1): 114–119.
[44] Su Z, Wang ZJ, Ni XH, Duan JC, Gao Y, Zhuo ML, Li RY, Zhao J, Ma Q, Bai H, Chen HY, Wang SH, Chen XX, An TT, Wang YY, Tian YH, Yu JY, Wang D, Xie XLS, Bai F, Wang J. Inferring the evolution and progression of small-cell lung cancer by single-cell sequencing of circulating tumor cells., 2019, 25(16): 5049–5060.
[45] Wu BJ, Wang Z, Dong Y, Deng YL, Shi QH. Identification and single-cell sequencing analysis of rare tumor cells in malignant pleural effusion of lung cancer patients., 2019, 41 (2): 175–184.吴保军, 王卓, 董宇, 邓宇亮, 施奇惠. 肺癌恶性胸腔积液中稀有肿瘤细胞的鉴定与单细胞测序分析. 遗传, 2019, 41 (2): 175–184.
[46] Wells D, Escudero T, Levy B, Hirschhorn K, Delhanty JDA, Munné S. First clinical application of comparative genomic hybridization and polar body testing for preimplantation genetic diagnosis of aneuploidy., 2002, 78(3): 543–549.
[47] Daina G, Ramos L, Obradors A, Rius M, Martinez- Pasarell O, Polo A, Del Rey J, Obradors J, Benet J, Navarro J. First successful double-factor PGD for lynch syndrome: monogenic analysis and comprehensive aneuploidy screening., 2013, 84(1): 70–73.
[48] Tobler KJ, Brezina PR, Benner AT, Du LK, Xu X, Kearns WG. Two different microarray technologies for preimplantation genetic diagnosis and screening, due to reciprocal translocation imbalances, demonstrate equivalent euploidy and clinical pregnancy rates., 2014, 31(7): 843–850.
[49] Huang J, Yan LY, Fan W, Zhao N, Zhang Y, Tang FC, Xie XS, Qiao J. Validation of multiple annealing and looping- based amplification cycle sequencing for 24-chromosome aneuploidy screening of cleavage-stage embryos., 2014, 102(6): 1685–1691.
[50] Shang W, Zhang YS, Shu MM, Wang WZ, Ren LK, Chen F, Shao L, Lu SJ, Bo SP, Ma SJ, Gao YM. Comprehensive chromosomal and mitochondrial copy number profiling in human IVF embryos., 2018, 36(1): 67–74.
[51] Macaulay IC, Haerty W, Kumar P, Li YI, Hu TXM, Teng MJ, Goolam M, Saurat N, Coupland P, Shirley LM, Smith M, Van der Aa N, Banerjee R, Ellis PD, Quail MA, Swerdlow HP, Zernicka-Goetz M, Livesey FJ, Ponting CP, Voet T. G&T-seq: parallel sequencing of single-cell genomes and transcriptomes., 2015, 12(6): 519–522.
[52] Tang J, Fewings E, Chang D, Zeng HL, Liu SS, Jorapur A, Belote RL, McNeal AS, Tan TM, Yeh I, Arron ST, Judson-Torres RL, Bastian BC, Shain AH. The genomic landscapes of individual melanocytes from human skin., 2020, 586(7830): 600–605.
[53] Zhou Y, Bian SH, Zhou X, Cui YL, Wang WD, Wen L, Guo LM, Fu W, Tang FC. Single-cell multiomics sequencing reveals prevalent genomic alterations in tumor stromal cells of human colorectal cancer., 2020, 38(6): 818–828.e5.
[54] Hou Y, Guo HH, Cao C, Li XL, Hu BQ, Zhu P, Wu XL, Wen L, Tang FC, Huang YY, Peng JR. Single-cell triple omics sequencing reveals genetic, epigenetic, and transcriptomic heterogeneity in hepatocellular carcinomas., 2016, 26(3): 304–319.
[55] Zachariadis V, Cheng HT, Andrews N, Enge M. A highly scalable method for joint whole-genome sequencing and gene-expression profiling of single cells., 2020, 80(3): 541–553.e5.
Advances in single-cell whole genome sequencing technology and its application in biomedicine
Zhuo Wang, Xiaohan Shen, Qihui Shi
The advent and development of single-cell whole-genome sequencing (scWGS) technology has shed lights on the genomic heterogeneities within biosamples at the single-cell resolution. The technology is particularly well-established in the recent decade and witnesses a variety of clinical applications, such as circulating tumor cell (CTC) detection and preimplantation genetic diagnosis/screening (PGD/PGS). In this review, we summarize the latest practical breakthroughs of scWGS in the field of biomedicine, with the hope of providing a guideline to apply single-cell genomic sequencing in clinical researches.
single cell whole genome; high throughput sequencing; heterogeneity; circulating tumor cell
2020-12-01;
2021-01-03
中国博士后科学基金面上项目(编号:2019M651377)和上海市“超级博士后”激励计划项目(2018-2020)资助[Supported by China Postdoctoral Science Foundation Grant (No. 2019M651377), and Shanghai “Super Postdoctoral Fellow” Program (2018-2020)]
王卓,博士,研究方向:循环肿瘤细胞鉴定与单细胞测序。E-mail: wangzhuoibs@fudan.edu.cn
施奇惠,博士,研究员,研究方向:液态活检与单细胞分析。E-mail: qihuishi@fudan.edu.cn
10.16288/j.yczz.20-363
2021/1/22 10:34:26
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210122.0900.002.html
(责任编委: 方向东)
